Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Ingeniería y competitividad
versão impressa ISSN 0123-3033
Ing. compet. vol.15 no.1 Cali jan./jun. 2013
Sintonización de clasificadores difusos por medio de algoritmos genéticos inicializados con FCM
Fuzzy classifiers tuning using genetic algorithms with FCM-based initialization
Carlos E. Celemín-Páez
Laboratorio de Automática e Inteligencia Computacional (LAMIC) Universidad Distrital Francisco José de Caldas, Bogotá D.C., Colombia
E-mail: carloscelemin@gmail.com
Hair A. Martínez-Gómez
Laboratorio de Automática e Inteligencia Computacional (LAMIC) Universidad Distrital Francisco José de Caldas, Bogotá D.C., Colombia
E-mail: hair.martinez@hotmail.com
Miguel Melgarejo
Laboratorio de Automática e Inteligencia Computacional (LAMIC) Universidad Distrital Francisco José de Caldas, Bogotá D.C., Colombia
E-mail: migbet@gmail.com
Eje temático: Ingeniería eléctrica y electrónica / Electric and electronics engineering
Recibido: Junio 29 de 2011
Aceptado: Abril 16 de 2013
Resumen
Este artículo presenta una técnica de inicialización para un Algoritmo Genético Simple el cual sintoniza un Sistema de Inferencia Difusa que actúa como clasificador. La técnica propuesta usa el algoritmo de agrupamiento Fuzzy C-means (FCM) para generar la población inicial del Algoritmo Genético Simple. Se consideran dos problemas de referencia en clasificación con el fin de validar el algoritmo propuesto y compararlo con un Algoritmo Genético Simple. Los resultados muestran que es posible lograr una reducción significativa en el número de generaciones para encontrar un clasificador objetivo usando la técnica propuesta.
Palabras Claves: Algoritmos de agrupamiento, algoritmos genéticos, clasificadores difusos, sistemas difusos.
Abstract
This paper presents an initialization technique for a Simple Genetic Algorithm that tunes a Fuzzy Inference System working as a classifier. The proposed technique uses the Fuzzy C-Means (FCM) clustering algorithm to generate the initial population of a Simple Genetic Algorithm. Two classification problems are considered to validate the proposed algorithm and to compare it against a Simple Genetic Algorithm with random initialization. Results show that it is possible to achieve a reduction in generations necessary for finding a desired classifier by using the proposed technique.
Keywords: Clustering algorithms, genetic algorithms, fuzzy classifiers, fuzzy systems.
1. Introducción
Entre las aplicaciones de los algoritmos genéticos (AG), se encuentra la optimización de Sistemas de inferencia difusa (SID) para solucionar problemas de clasificación. Algunos de los trabajos previos en este tema son: Peña (2004) presenta un ejemplo de diagnóstico de cáncer usando un algoritmo genético simple (AGS), el cual sintoniza un SID para clasificar entre muestras benignas y malignas. Además propone un enfoque coevolutivo para modelar un SID y lo valida sobre el problema de diagnóstico de cáncer de mama de la Universidad de Wisconsin y el problema de clasificación de flores de la especie Iris.
Liu et al. (2006) desarrollan una aproximación de un Algoritmo Rápido Genético Difuso proponiendo un nuevo método de agrupamiento para obtener la población inicial y una modificación del operador genético de cruce. El método propuesto se valida con tres problemas de clasificación, los dos primeros corresponden a las bases de datos Phoneme y Satimage del proyecto ELENA y el tercero corresponde a la base de datos Waveform del repositorio de aprendizaje de maquina de UCI.
Stavrakoudis et al. (2009) proponen el uso de un clasificador genético difuso para la clasificación de cobertura de tierra de imágenes multiespectrales. Bazrafshanet al. (2010) modifican el clasificador difuso de Ishibuchi, Ishibuchi et al. (1995), Ishibuchi et al. (2004), Irizarry (2005), para manejar problemas de alta dimensión y prueba éste con el problema de identificación del golpe de las teclas.
Los AG para generar su población inicial usan, en la mayoría de los casos, inicialización aleatoria. El objetivo de usar este tipo de inicialización es tratar de cubrir en su totalidad el espacio de búsqueda y así reducir la posibilidad de converger en un mínimo local, Holland (1992). Una de las dificultades asociadas a este tipo de inicialización es que se requiere un número elevado de iteraciones, llamadas generaciones, para encontrar un individuo adecuado que solucione un problema específico debido a que la población inicial posee una gran variedad genética, Bramlette (1991).
Una forma para lograr una reducción en el número de generaciones necesarias para encontrar un resultado adecuado es usar conocimiento previo deducido del problema para generar la población inicial, Louis & Johnson (1999). En trabajos anteriores se han propuesto diversas técnicas para inicializar un AG. Bramlette (1991) usa el mejor de n individuos elegidos de forma aleatoria para obtener la población inicial del AG. Gordon (1993) presenta un método para deducir por medio de un reporte de alto nivel las reglas para crear la población inicial de un AG. Rahnamayan et al. (2007) proponen un novedoso enfoque de inicialización que usa el aprendizaje basado en oposición para generar la primera población de los algoritmos evolutivos. Dong et al. (2004) presentan un algoritmo híbrido que usa la fase de inicialización del algoritmo de agrupamiento Fuzzy C-Means con muestreo multiestado aleatorio (MRFCM) para crear una población inicial de centros de clases, luego usan esta población en el algoritmo de agrupamiento genético difuso mejorado (GFGA) para ser optimizada y de esta forma construir un clasificador en un menor número de iteraciones del proceso evolutivo. Cabe aclarar que en esa propuesta no se usa un SID como clasificador, por tanto no se hace uso de un conjunto de reglas para la clasificación ni se optimizan parámetros de conjuntos difusos, en este caso el clasificador obtenido funciona mediante el resultado del agrupamiento difuso (centros y pertenencias).
Este trabajo propone una metodología para configurar un modelo híbrido que combina Fuzzy C-Means, Bezdek (1981), y AG para construir un clasificador difuso. En primer lugar, el algoritmo usa la técnica de aprendizaje no supervisado para extraer las medias y las desviaciones estándar de los datos de entrenamiento para construir los conjuntos difusos del SID. Luego, el algoritmo aprovecha el conocimiento extraído para deducir la población inicial reduciendo el espacio de búsqueda de la evolución del AG.
El algoritmo genético inicializado con Fuzzy C-Means (AGIFCM) se valida con el problema de diagnóstico de cáncer de mama e Iris de Fisher, Frank & Asuncion (2010). Varios SID con diferente número de reglas se obtienen por medio del algoritmo propuesto y un AGS, comparándose la cantidad de generaciones empleadas para obtener un desempeño específico en cada caso. Los resultados muestran que el algoritmo propuesto encuentra los parámetros del sistema difuso usando menos generaciones que el AGS dado un nivel de error preestablecido. Esta propuesta permite sintonizar un SID clasificador en función de todos sus parámetros, a diferencia de las otras propuestas de inicialización consultadas, además el algoritmo de agrupamiento tiene un costo computacional bajo que no influye negativamente en el tiempo de evolución.
2. Inicialización del AG usando FCM para problemas de clasificación
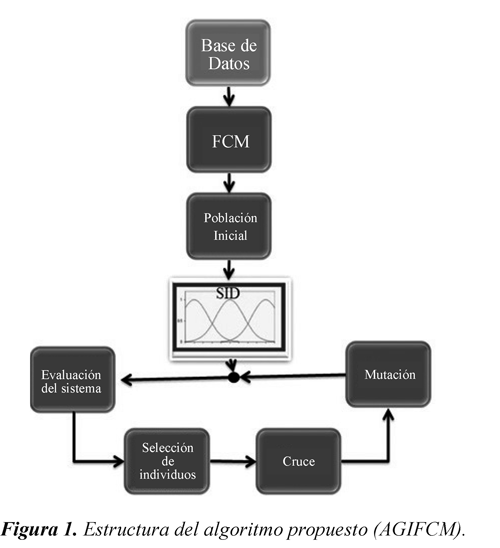
En la Figura 1 se presenta el esquema del AGIFCM. En primer lugar a partir de la base de datos y usando el algoritmo de agrupamiento difuso FCM se extraen los parámetros de la población inicial para construir un SID que posteriormente se sintoniza finamente usando un AG. De esta forma, el genotipo de cada individuo que se quiere generar en el proceso de inicialización es un vector que tiene dentro de sus elementos las medias de cada universo para todas las reglas al igual que las desviaciones estándar (estos parámetros definen los conjuntos del antecedente) y los centros en el consecuente asociados a cada regla. En las siguientes secciones se presentan en primer lugar las características del SID, posteriormente se presenta el proceso de extracción de los parámetros a partir del algoritmo FCM y finalmente se presenta un ejemplo para aclarar la metodología propuesta.
2.1 Descripción del SID
En este trabajo, se considera construir un SID, Mendel (2001), Yager & Zadeh (1995), usando la expansión en funciones de base difusa (EFBD), Wang (1997), con las siguientes características: Fusificador tipo Singleton, funciones de pertenencia Gaussianas, T-norma producto, máquina de inferencia con operación producto y defusificador por promedio de centros, como se observa en la Ec(1).
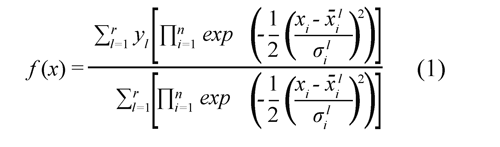
Donde r es el número de reglas, n es el número de entradas, xi es el i-ésimo dato del vector de entrada x, xil es la media del conjunto l de la i-ésima entrada, σil es la desviación estándar del conjunto l en la i-ésima entrada y yl es el centro del conjunto l en el consecuente.
Este SID está definido por una cantidad de reglas múltiplo del número de clases necesarias. Por tanto, en la Ec(2) se da el número de reglas:
r = Φ * nC (2)
Siendo Φ un entero positivo. A cada clase se le asocia un número igual de reglas, esto es una consideración que se hace debido a la ausencia de conocimiento sobre cada problema de clasificación, en caso de requerir una distribución diferente de reglas para cada clase el proceso evolutivo debería reajustar la estructura establecida en la inicialización.
Como se mencionó antes, el algoritmo propuesto obtendrá los parámetros de la población inicial, basado en adquirir conocimiento de algunos datos de entrenamiento y describirlos como reglas. Un individuo es un vector formado por r*n medias y desviaciones y r centros como lo muestra la Figura 2.
2.2 Descripción de la extracción de los parámetros de la población inicial a partir del algoritmo FCM
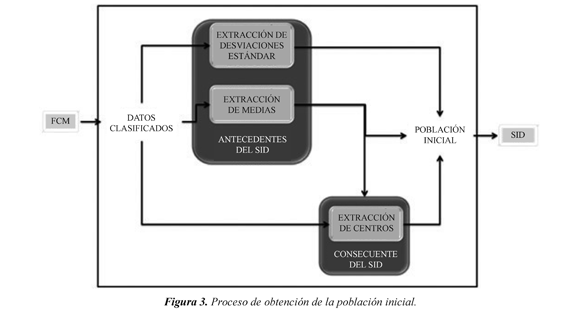
El método de inicialización empieza al generar tantos grupos como número de clases deseadas. Por tanto, se obtiene la matriz de partición difusa U por medio del FCM. La información de esta matriz se usa en el proceso evolutivo como se muestra en la Figura 1. El bloque para obtener la población inicial se especifica en la Figura 3, en esta Figura cada uno de los 2 parámetros del antecedente se puede hallar en paralelo, mientras que el centro del consecuente, depende de las medias escogidas en el antecedente ya que estas tienen un valor de salida asociado (la media es la componente de entrada de un vector de entradasalida escogido aleatoriamente, por tanto el consecuente está relacionado con la componente de salida). Los 3 bloques internos se exponen en las siguientes subsecciones.
El tamaño de la matriz de partición difusa U es nC × N, donde nC es el número de clases definido y N es la cantidad de datos de entrenamiento. Los grados de pertenencia notados como μik (1≤ i ≤ nC , 1 ≤ k ≤N ) de cada dato a cada clase ubicados en la matriz de partición, se obtienen en la Ec(3).
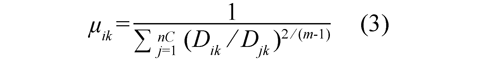
El valor Dik es la distancia entre el dato k y el centro de la clase i, mientras que Djk es la distancia entre el mismo dato k y cada uno de todos los centros decada clase, m es conocido como el parámetro de borrosidad y puede seleccionarse cualquier valor del conjunto (1,â) pero normalmente se usa el valor 2 por costo computacional.
Así pues, se toman algunos vectores de entrada y conociendo la respectiva clase y su pertenencia, de acuerdo con los resultados entregados por el FCM, se asigna cada uno a una regla de acuerdo con la clase a la cual pertenece. Así, cuando Φ=1, cada clase tendrá asociada una regla, cuando Φ=2 a cada clase se le asignan dos reglas, es decir que Φ define el número de reglas asociadas a cada clase.
Cada vector de entrada de los datos de entrenamiento se etiqueta en la clase a la que tiene mayor pertenencia en la matriz de partición difusa, la pertenencia a un grupo dice que tanto se parece un dato a ese grupo, es decir si es más alta se considera mayor similitud y por esto se puede asociar el dato a ese grupo. Ki (1≤ i ≤ nC ) es el vector indexado construido con las direcciones en la base de datos de los vectores que pertenecen a la clase i de acuerdo con el resultado del agrupamiento, y asociando un vector de pertenencias U i con el grado al que pertenecen a la clase i los elementos de Ki . Con la información contenida en estos vectores se puede encontrar los parámetros de la población inicial como se muestra a continuación.
Después de ejecutar el FCM se toman npop * Φ datos de entrenamiento de cada clase, siendo npop el tamaño de la población definido para el proceso evolutivo, y su respectiva pertenencia (es decir se extraen de U i y Ki ). Al remover o repetir elementos de estos vectores según sea necesario en cada clase, se asegura que los nuevos vectores U'i y K'i tengan de tamaño npop * Φ, como lo definen la Ec(4) y Ec(5).
U'i = [U'i j ]; 1 ≤ j ≤ npop * Φ (4)
K'i = [K'i j ]; 1 ≤ j ≤ npop * Φ (5)
A cada individuo le corresponde igual número de muestras de cada clase, así que los conjuntos de muestras U i' y K'i son distribuidos en una cantidad de npop subconjuntos cuya intersección sea nula y su unión obtenga nuevamente a U i' y K'i , cada subconjunto es de tamaño Φ y su obtención se puede lograr como muestra la Ec(6).
K'i 1 = [ K'i b ]; 1 ≤ b ≤Φ
K'i 2 = [ K'i b ]; Φ + 1 ≤ b ≤ 2Φ
K'i = [K'i ]; (npop -1) * Φ +1≤ b ≤ npop * Φ npopb (6)
En general cada subconjunto de cualquier clase le corresponde aun solo individuo y en la Ec(7) se obtiene cada subconjunto.
K'i p = [K'i b ]; ( pop -1) * Φ +1≤ b ≤ p * Φ (7)
Donde 1 ≤ p ≤ npop, siendo p el número del subconjunto K'i p de la clase i que le corresponde al individuo p.
En la Ec(8) se observa como los subconjuntos componen a K'i.
K'i = [K'i K'i ... K'i ] (8)
Igualmente se tiene que distribuir el vector U'i en subconjuntos con las mismas características, de la misma forma en la Ec (9).
U'i p = [μ'i b ]; ( p -1) * Φ +1≤ b ≤ p * Φ (9)
2.2.1 Obtención de medias
Teniendo en mente que el patrón de una clase tiene diversas pertenencias y ocupa diferentes regiones en el espacio de características, se puede obtener un mejor modelado de cada clase tomando diferentes datos de entrenamiento y asignándolos como medias de los conjuntos en el antecedente. Cada una de estas reglas es un caso representativo de posibles vectores de entrada que se van a clasificar. Entonces la activación de cada regla depende de como el vector de entrada se parezca al dato con el que se construye cada regla.
Como cada vector de entrada tiene n dimensiones, significa que hay n universos de discurso en el antecedente. Por tanto, cada vector de entrada asignado a un individuo tiene las medias de los conjuntos de una regla en todos los universos de discurso. De esta manera, todos los individuos reciben nC*Φ muestras de la base de datos de entrenamiento, las cuales serán las medias de los conjuntos en el antecedente.
Teniendo en cuenta que K'i contiene las índices de los datos de la clase i en la base de entrenamiento, el vector de datos de la clase i en la entrada l es la Ec(10).
DBil = [ dbl (K'i)]; 1 ≤ l ≤ n (10)
Por tanto, el valor de las medias corresponde al valor de la base de datos de entrenamiento apuntada por el vector K'i p como se muestra en la Ec(11).
x̅ ilp=[ dbl (K'i p )]; 1 ≤ l ≤ n (11)
Donde i corresponde a la clase en la base de entrenamiento, l corresponde al universo de entrada y p (1 ≤ p ≤ npop) es el número del subconjunto K'i p de la clase i que le corresponde al individuo p.
2.2.2 Cálculo de las desviaciones estándar
Las desviaciones estándar para los conjuntos en el antecedente se obtienen proyectando todos los datos de entrenamiento de cada clase a cada una de las n dimensiones. Allí se calcula la desviación estándar de estos datos, la cual se asociará a ese respectivo universo de discurso. Como cada clase tendrá asociado Φ conjuntos en el antecedente, teniendo en cuenta que Φ es un parámetro definido por el usuario al principio del algoritmo, la desviación calculada se escala dividiendo entre Φ.
Ya que cada desviación estándar calculada es un único valor para cada clase, se adhiere ruido gaussiano a este valor en cada uno de los conjuntos de la misma clase de todos los individuos con el fin de tener diversidad genética en toda la población. La distribución del ruido que se agrega tiene una media en cero para que se exploren en igual probabilidad, valores por encima y por debajo de la desviación de las clases en cada universo, pero con mayor concentración cerca del valor original. La desviación del ruido es un parámetro que puede ser configurable al inicio del algoritmo, pero con el fin de no distorsionar el conocimiento extraído en la etapa de agrupamiento, se establece igual al valor de dispersión calculado para cada grupo resultante del agrupamiento.
La Desviación Estándar (DE) asociada a los elementos de cada una de las clases es una matriz de npop*Φ×n y se calcula con la Ec(12) donde cada una de las filas le corresponde a un individuo diferente.
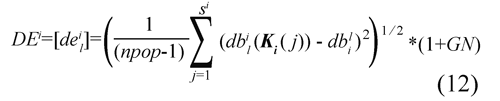
Donde si es el tamaño del vector Ki y GN es ruido gaussiano con media cero y desviación estándar uno.
2.2.3 Inicialización de centros en el Consecuente
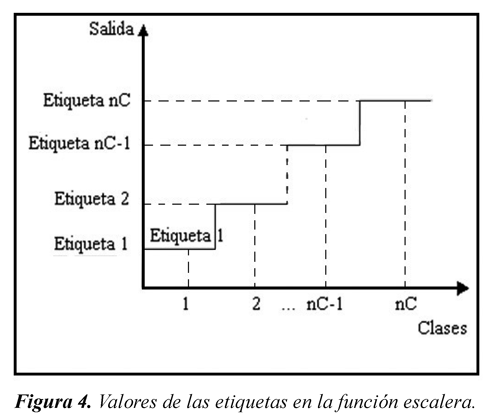
Debido a que el SID estima valores continuos a la salida, cada clase es asociada con un valor etiqueta. Cuando un vector de entrada sea evaluado por este sistema, el resultado deberá ser cercano al valor de su respectiva etiqueta para que la clasificación sea correcta. De esta forma se construye una función escalera como la mostrada en la Figura 4, Pedrycz et al. (2003), Peña (2004), y Zhou et al. (2007).
El centro de los conjuntos en el consecuente será un valor cercano al respectivo valor de la etiqueta de su clase. El valor exacto se relaciona con la pertenencia a la clase del dato en el que se basa la regla, contenido en el vector U'i. De manera que mientras la pertenencia es más cercana a uno, el centro de esta regla debe estar más próximo a su valor de etiqueta establecido.
Para la clase i, el vector de centros está en la Ec(14), mientras que αi que es la mitad de la distancia entre la etiqueta de la clase i y la siguiente se calcula con la Ec(13). Se podría definir αi como el umbral entre esas dos clases.
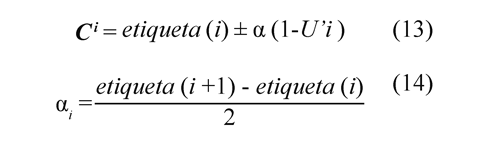
Donde todos los c^i elementos están distribuidos en toda la población y los valores de los umbrales (th) que restringen el rango de cada clase se obtienen con la Ec(15).
th1 = etiqueta (i) ± αi (15)
3. Metodología experimentalEl método propuesto se prueba con dos problemas clásicos de clasificación Wisconsin Diagnostic Breast Cancer e Iris Database, Frank & Asuncion (2010). Se compara contra los resultados obtenidos con el AGS.
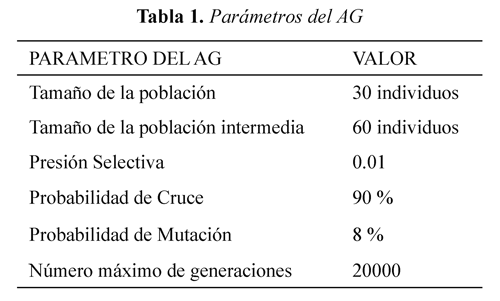
Los parámetros del AG para el algoritmo propuesto y para el AGS están listados en la Tabla 1 y se obtienen luego de una serie de experimentaciones previas para determinar los parámetros con los mejores resultados. En los dos casos estos algoritmos se caracterizan por tener una codificación real para los genotipos de los individuos, se realiza selección por ruleta, el operador de cruce es de tipo aritmético ya que los cromosomas de los nuevos individuos son el promedio de la información genética de sus padres y además, se aplica mutación uniforme al modificar un parámetro al azar por un valor aleatorio con probabilidad uniforme dentro del intervalo delimitado por el universo de discurso respectivo en los individuos a quienes la probabilidad de mutación les obligue a mutar. Como función de aptitud los dos algoritmos utilizan la tasa de error de clasificación (TEC). Los algoritmos se corren y finalizan cuando un individuo alcanza alguno de los tres TEC establecidos (10% y 5%).
Para los parámetros mencionados anteriormente se varía el número de reglas del SID y cada caso se evalúa teniendo en cuenta la cantidad de generaciones empleadas para obtener la solución. El algoritmo se corre para cada caso 20 veces. Se calcula la media y la desviación Estándar del número de generaciones usadas en cada caso para obtener la TEC establecida y de esta forma comparar los dos algoritmos.
En el caso del método propuesto, se varía el parámetro de fusibidad (m) en la etapa de inicialización. Cuando el desempeño de alguno de los individuos de la población no llega al mínimo establecido por la TEC antes de 20000 generaciones, se detiene el proceso evolutivo y se considera que el proceso de optimización no converge.
Los resultados de clasificación alcanzados por los SID entrenados con el AGIFCM no son comparados con los de otros trabajos, ya que para esto se debería acondicionar las bases de datos de la misma forma (utilizar mismas instancias y atributos) para que los resultados sean comparables, aún así con esto las diferencias entre los desempeños podrían deberse al SID utilizado ya que las características de éste como lo son el tipo de Fusificador, de funciones de pertenencia, de T-norma, de máquina de inferencia y de defusificador influyen en el desempeño de forma diferente para cada problema, esa variación paramétrica no está contemplada en los objetivos de este trabajo, concentrando el análisis de los resultados no tanto en el clasificador sino en el desempeño del proceso de sintonización y su costo computacional. Sin embargo se puede decir que para los dos problemas las tasas de desempeño de los mejores clasificadores obtenidos se encuentran en el mismo nivel que las alcanzadas por trabajos como el de Peña (2004) y los trabajos con quienes él compara sus resultados, pero una revisión detallada de la precisión y exactitud de los clasificadores es un poco distante a los objetivos de la propuesta de este trabajo
4. Discusión y resultados
4.1 Diagnóstico de cáncer de mama
Para este primer problema, se usa la base de datos llamada Wisconsin Diagnostic Breast Cancer (WDBC), Frank & Asuncion (2010). Esta base de datos cuenta con 569 casos de los cuales 357 corresponden a muestras benignas y 212 a muestras malignas. Cada registro cuenta con 31 características. Tres de las 31 características (textura, suavidad extrema y área extrema) disponibles son seleccionadas como entradas al sistema, a partir de un trabajo previo, Wolberget al. (1995). La salida toma únicamente dos valores (dos clases), benigno o maligno.
En este caso el valor de la etiqueta para la clase benigno es tag1=0.3 y para la clase maligno es tag2=0.7, de este modo el valor de umbralización se establece en 0.5 y α=0.2.
Para los casos benignos el valor de salida del SID debe ser menor que el umbral (0.5) y para los caos malignos, mayor que éste para una adecuada clasificación.
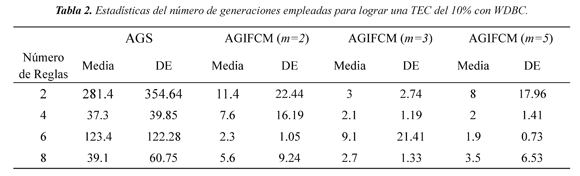
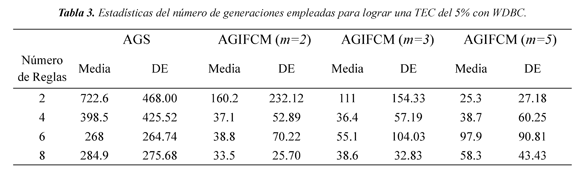
Las estadísticas consignadas en las Tabla 2 muestran que la población inicial de AGIFCM tiene mejor desempeño que la del AGS. Obsérvese que para diferentes valores de fusibidad el algoritmo híbrido alcanza la TEC objetivo en la primera generación para el 95 % de las experimentaciones, es decir que la primera generación tiene un desempeño que satisface la TEC, caso inexistente para el AGS.
En todos los casos la cantidad de generaciones requeridas son reducidas en relación con el AGS. El peor de los casos para comparar es con un clasificador de 6 reglas y m=5, el número de generaciones usadas con el algoritmo propuesto es 2.73 veces más pequeña que con el AGS. El mejor de los casos se presenta con un clasificador de 2 reglas y el mismo valor del parámetro de fusibidad, en el cual el AGS invierte 28.56 veces más generaciones que el AGIFCM.
En las Tablas 2 y 3 se observa que el algoritmo híbrido utiliza menos generaciones que el AGS para llegar a los mismos resultados. Esto se asocia al impacto que tiene el hecho de empezar con una buena población. Los resultados dan a conocer que con el AGIFCM se tiene en general una menor TEC inicial para todos los individuos, ya que hasta el peor error inicial no es tan alto como en el AGS. Lo mismo sucede con la TEC promedio ya que se ve reducida al aplicar la inicialización propuesta.
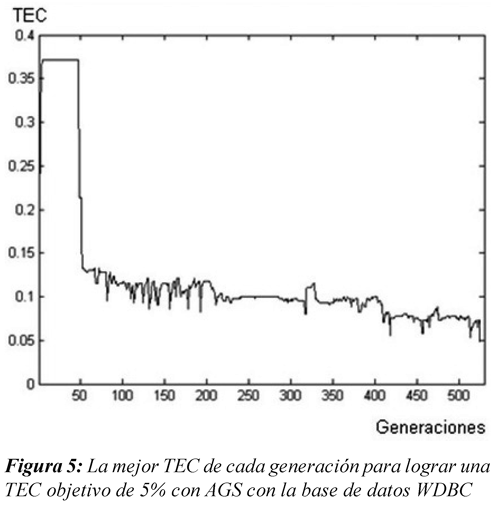
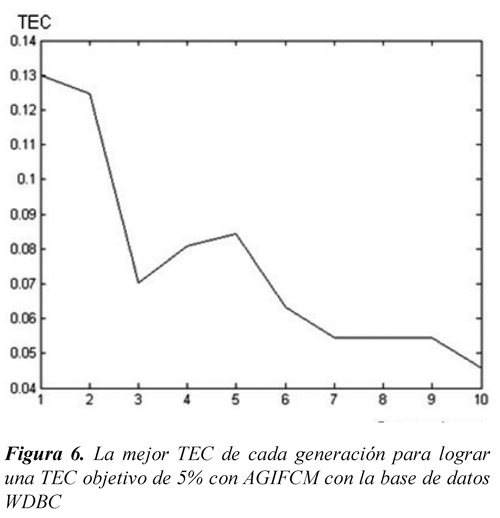
En las Figuras 5 y 6 se presenta la menor TEC de cada generación en el proceso de sintonización de un SID con 4 reglas y con criterio de parada de un error mínimo de clasificación del 5% (En este caso se establece m=2 para AGIFCM). En estas figuras se observa como el desempeño inicial del AGIFCM mostrado en la Figura 6 es aproximadamente la mitad del logrado por el AGS mostrado en la Figura 5, esta característica influiría significativamente en el número de generaciones empleado en el proceso evolutivo.
4.2 Problema de clasificación de flores Iris
La base de datos está organizada en tres clases (Iris Setosa, Iris Versicolour e Iris Virginica), cada caso tiene 50 registros y 4 características, Frank & Asuncion (2010).
Los valores de las etiquetas para las 3 clases son: tag1=0.2;tag2=0.5; tag3=0.8. El valor de umbralización entre la primera y la segunda clase es 0.35, mientras que entre la segunda y la tercera es 0.65, en este caso α=0.15.
Cuando el vector de entrada corresponde a la clase Iris Setosa, el valor de salida del SID debe ser menor a 0.35, si la clase es Iris Versicolor el valor de salida debe estar entre 0.35 y 0.65, por ultimo si la entrada corresponde a Iris Virginica la salida debe ser mayor a 0.65 para una correcta clasificación.
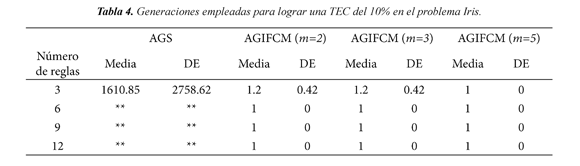
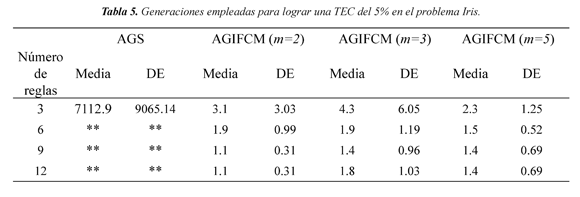
En las Tablas 4 y 5, las casillas sombreadas significa que el 30% de las corridas la evolucion no converge, y los dos asteriscos (**) significa que en el 40% o más de las corridas no hay convergencia.
La función escalera propuesta para obtener con un SID clasificador es un problema practicamente no resuelto con el AGS, unicamente con 3 reglas seobtienen resultados satisfactorios. Sin embargo no se puede asegurar que siempre converga para este caso. Con AGIFMC, los resultados muestran que en todas las variaciones de número de reglas, fusibidad y valores de la TEC, la media de las generaciones empleadas es menor que 5. Los resultados evidencian que para este problema el AGIFCM alcanza desempeños que el AGS no puede lograr ya que converge prematuramente.
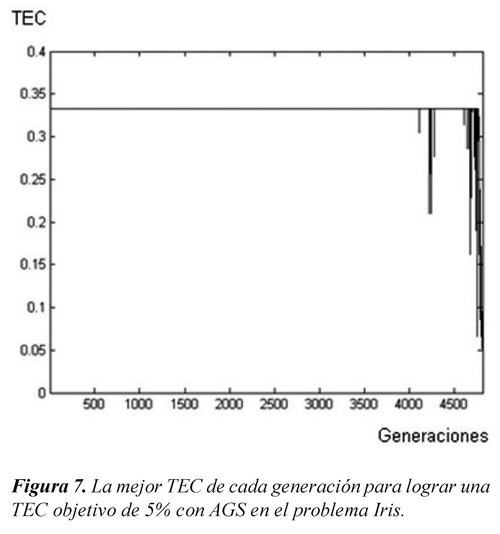
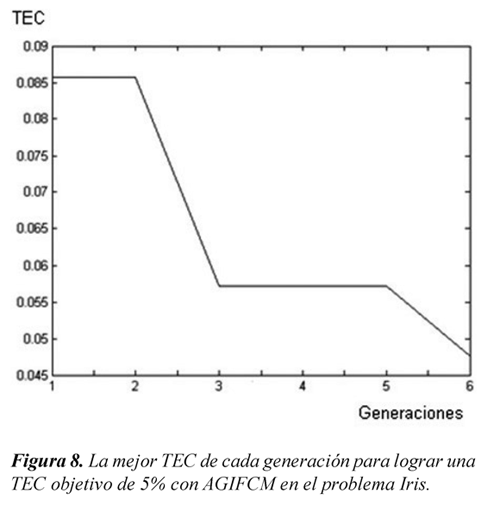
En las Figuras 7 y 8 se presenta la menor TEC de cada generación en el proceso de sintonización de un SID con 4 reglas, y como criterio de parada un error mínimo de clasificación del 5% (En este caso se establece m=2 para el AGIFCM). En estas Figuras se observa también que el error inicial es menor para el AGIFCM. Para este problema particular la inicialización favorece al proceso evolutivo dado que se observa que a través de las generaciones el desempeño de la población va mejorando. En cambio con el AGS, la mínima TEC se mantiene constante la mayor parte del tiempo debido a que hay una clase completamente mal clasificada inicialmente. Este problema tiende a corregirse sino solo en algunos casos después de un número significativo de generaciones.
5. Conclusiones
En este artículo se presentó una propuesta metodológica de inicialización de un AGS orientado a la optimización de clasificadores difusos, la cual extrae información relevante de la base de datos por medio del Algoritmo de Agrupamiento Difuso FCM. Con el conocimiento extraído se obtiene un conjunto de parámetros adecuados para la construcción de una población inicial de clasificadores.
Se realizaron dos experimentos en problemas de clasificación típicos y se evidenció a partir de los resultados que el método propuesto permite obtener un clasificador difuso con un nivel de desempeño particular en un número de generaciones significativamente menor en comparación con un AGS inicializado aleatoriamente. Esta conclusión se soporta en el hecho que la población inicial obtenida a partir del FCM tiene un menor error promedio de clasificación que la generada aleatoriamente marcando un mejor punto inicial para la optimización evolutiva.
6. Referencias bibliográficas
Bazrafshan, F., Javanbakht, A., & Mojallali, H. (2010). Keystroke identification with a genetic fuzzy classifier. Proceedings of II International Conference on Computer Engineering and Technology (ICCET) (pp. 136-140). Chengdu, China: IEEE. [ Links ]
Bezdek, C. (1981). Pattern Recognition with Fuzzy Objective Function Algorithms. New York: Plenum Press. [ Links ]
Bramlette, M. F. (1991). Initialization, Mutation and Selection Methods in Genetic Algorithms for Function Optimization. R.K. Belew & L. B. Booker (Eds.). Proceedings of Fourth International Conference on Genetic Algorithms (pp. 100-107). San Francisco (CA): Morgan Kaufmann Publishers Inc. [ Links ]
Dong, Y.-Y., Zhang, Y.-J., & Chang, C.-L. (2004). Multistage random sampling genetic-algorithmbased fuzzy c-means clustering algorithm. Third International Conference on Machine Laming and Cybernetic, 4, 2069-2073. Shangai, Aug. 26-29. [ Links ]
Frank, A., & Asuncion, A. (2010). UCI Repository of Machine Learning Databases. Irvine (CA): University of California, School of Information and Computer Science [Online]. Recovered 2013/06/15 http://archive.ics.uci.edu/ml [ Links ]
Gordon, D. F. (1993). A Multistrategy Learning Scheme for Assimilating Advice in Embedded Agents. Proceedings of the Second International Workshop on Multistrategy Learning (pp. 218-233). Harper's Ferry (WV): NAVAL RESEARCH LAB WASHINGTON DC. [ Links ]
Holland, J. H. (1992). Adaptation in Natural and Artificial Systems. Cambridge (MA): MIT. [ Links ]
Irizarry, R. (2005). Fuzzy classification with an artificial chemical process. Chemical Engineering Science, 60 (2), 399-412. [ Links ]
Ishibuchi, H., Nakashima, T. & Nii, M. (2004). Classification and Modeling with Linguistic Information Granules: Advanced Approaches to Linguistic Data Mining. London: Springer. [ Links ]
Ishibuchi, H., Nozaki, K., Yamamoto, N., & Tanaka, H. (1995). Selecting Fuzzy If-Then Rules for Classification Problems using Genetic Algorithms. IEEE Transaction on Fuzzy Systems, 3(3), 260- 270. [ Links ]
Liu, M., Yuan, B., Chen, J., & Miao, Z. (2006). A fast Genetic Fuzzy approach and its application in Classifier Fusion. 8th International Conference on Signal Processing, 3, 16-20. [ Links ]
Louis, S. J. & Johnson, J. (1999). Robustness of Case-Initialized Genetic Algorithms. A. N. Kumar & I. Russell (Eds.). Proceedings of the Twelfth International Florida Artificial Intelligence Research Society Conference, (pp. 129-133). Orlando (FL): AAAI Press. [ Links ]
Mendel, J. M. (2001). Uncertain Rule-Based Fuzzy Logic Systems: Introduction and New Directions. Upper Saddle River (NJ): Prentice-Hall. [ Links ]
Pedrycz, W., Reformat, M. (2003). Evolutionary fuzzy modeling. IEEE Transactions on Fuzzy Systems, 11 (5), 652- 665. [ Links ]
Peña-Reyes, C. A. (2004). Coevolutionary Fuzzy Modeling. Lecture Notes in Computer Science. 3204. London: Springer. [ Links ]
Rahnamayan, S., Tizhoosh, H.R., & Salama, M.M.A. (2007). A Novel Population Initialization Method for Accelerating Evolutionary Algorithms. Computers and Mathematics with Applications, 53(10), 1605-1614. [ Links ]
Stavrakoudis, D.G., & Theocharis, J.B. (2009). A genetic fuzzy rule-based classifier for land cover image classification. IEEE International Conference on Fuzzy Systems. Jeju Island, Korea, august 20-24. IEEE. [ Links ]
Wang, L.-X.(1997). A Course In Fuzzy Systems and Control. Upper Saddle River (NJ): Prentice-Hall, Inc. [ Links ]
Wolberg, W.H., Street, W.N., Heisey, D.M., & Mangasarian, O.L. (1995). Computer-derived nuclear features distinguish malignant from benign breast cytology. Human Pathology, 26 (7), 792-796. [ Links ]
Yager, R. R. & Zadeh, L. A. (1994). Fuzzy Sets, Neural Networks, and Soft Computing. New York: Van Nostrand Reinhold. [ Links ]
Zhou, E. & Khotanzad, A. (2007). Fuzzy classifier design using genetic algorithms. Pattern Recognition, 40 (12), 3401-3414. [ Links ]

Revista Ingeniería y Competitividad por Universidad del Valle se encuentra bajo una licencia Creative Commons Reconocimiento - Debe reconocer adecuadamente la autoría, proporcionar un enlace a la licencia e indicar si se han realizado cambios. Puede hacerlo de cualquier manera razonable, pero no de una manera que sugiera que tiene el apoyo del licenciador o lo recibe por el uso que hace.













 -
-