Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Ingeniería y competitividad
versão impressa ISSN 0123-3033
Ing. compet. vol.16 no.2 Cali jul./dez. 2014
Una arquitectura de integración semántica de datos e imágenes médicas
An architecture for semantic integration of data and medical images
Marta Millán
E-mail:martha.millan@correounivalle.edu.co
Maria Trujillo
E-mail: maria.trujillo@correounivalle.edu.co
Daniel Valencia
E-mail: daniel.valencia@correounivalle.edu.co
Escuela de Ingeniería de Sistemas y Computación, Facultad de Ingeniería, Universidad del Valle, Cali, Valle del Cauca
Eje temático: INGENIERÍA DE SISTEMAS/SYSTEMS ENGINEERING
Recibido: 08 de Noviembre de 2011
Aceptado: 11 de Diciembre de 2013
Resumen
En las organizaciones prestadoras de servicios de salud, existen diferentes fuentes de datos (e.g. historia clínica, datos demográficos, archivos DICOM) de naturaleza distinta, que están dispersas en medios de almacenamiento y que provienen de fuentes heterogéneas. Adicionalmente, el formato DICOM brinda la posibilidad de almacenar información del paciente y de las imágenes médicas. Estos archivos son administrados en PACS, sin embargo los PACs no brinda herramientas de apoyo al diagnóstico. En este artículo se presenta una arquitectura de integración de datos orientada a enriquecer imágenes médicas mediante metadatos extraídos de un vocabulario controlado. La arquitectura fue instanciada en un prototipo que ofrece mecanismos de anotación - manual y automática - de imágenes y estrategias de búsqueda y recuperación de datos e imágenes diferentes a los tradicionales usando palabras claves o descriptores MPEG-7. La anotación se basa en un vocabulario controlado que forma parte de una taxonomía de conceptos y términos médicos.
Palabras clave: anotación de imágenes, imágenes médicas, integración y visualización de datos, vocabulario controlado.
Abstract
In organisations providing health services, there exist different data sources (e.g. clinical history, demographics data, DICOM files) of diverse nature, which are scattered storage and come from heterogeneous sources. Additionally, the DICOM format stores patient information and medical images. These files are managed in PACS, however PACs does not provide diagnostic support tools. In this paper, a data integration architecture oriented to enrich medical images using metadata extracted from a controlled vocabulary is presented. The architecture was instantiated in a prototype that provides image annotation mechanisms - manual and automatic - and strategies for searching and retrieving data and images using keywords or descriptors MPEG-7, which are different from traditional ones. The annotation is based on a controlled vocabulary that is part of a taxonomy of concepts and medical terms.
Keywords: annotation of images, controlled vocabulary, data integration and visualization, medical imaging.
1. Introducción
El auge de las tecnologías digitales ha impactado el dominio médico con fuentes de información que apoyan el diagnóstico, siendo el área de imágenes, una de las de mayor desarrollo, generando grandes repositorios con información digital. Las más avanzadas tecnologías, puestas al servicio de los médicos y pacientes, como soporte a un diagnóstico preciso son, entre otras, los rayos X, el ultrasonido, la tomografía y la resonancia magnética.
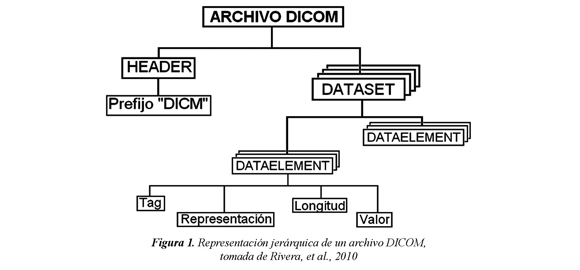
Los equipos de captura de imágenes médicas almacenan los datos usando el formato Digital Imaging and Communication in Medicine (DICOM), un estándar diseñado para garantizar la interoperabilidad entre los sistemas que manipulan, almacenan, imprimen y transmiten imágenes médicas. Este estándar fue creado por la American College of Radiology (ACR) y la National Electrical Manufacturers Association (NEMA). Un archivo DICOM consta de un encabezado que sirve para verificar que el archivo efectivamente es un archivo DICOM y una serie de Data Sets integrados por una secuencia de Data Element, como se ilustra en la Figura 1. Un Data Set representa un elemento del mundo real de DICOM y a su vez un Data Element almacena valores de dicho elemento. Los Data Element se agrupan y organizan en Group Element, donde cada integrante del grupo se identifica por un Data Element Number, (Rivera et al. 2010).
En general, los archivos DICOM guardan dos tipos de datos: datos demográficos del paciente y datos de las imágenes. Esta información es administrada por un Picture Archiving and Communication System (PACS) (Mansoori et al., 2012; Chen et al., 2011). Sin embargo, los PACs no cuentan con herramientas de apoyo al diagnóstico. Un estudio, por ejemplo, de Resonancia Magnética (RM) o de Tomografía Computarizada (TC), puede contener cientos de imágenes, algunas de las cuales podrían no ser de interés o representativas para el diagnóstico. Por otra parte, además de las imágenes médicas existen otras fuentes de datos (e.g. historia clínica, datos demográficos) de naturaleza distinta, que están dispersas en diferentes medios de almacenamiento y que provienen de fuentes heterogéneas. Si al momento de interpretar una imagen un radiólogo pudiera acceder a información adicional de estas diversas fuentes, es posible que ésta facilite y oriente su análisis. Adicionalmente, las imágenes que un radiólogo -o conjunto de radiólogos- ha analizado en el pasado se podrían enriquecer semánticamente mediante anotaciones o metadatos a distintos niveles de granularidad.
La anotación semántica de imágenes ha sido una estrategia ampliamente usada para abordar el problema de la brecha semántica. Las anotaciones semánticas que usan metadatos relacionan dos tipos de entidades: una imagen y un concepto o término de un vocabulario. Esta relación facilita, posteriormente, recuperar imágenes de manera más precisa, a partir de la nueva información asociada con la imagen. Estas anotaciones o metadatos pueden usar vocabularios conocidos y compartidos por la comunidad de médicos, de tal modo que este consenso facilite llevar a cabo tareas intensivas de conocimiento, integrar esta información, enriquecerla semánticamente y ofrecer técnicas y herramientas de recuperación y acceso a los datos de forma eficiente y efectiva.
A pesar de que existen estrategias y métodos para la recuperación de imágenes a partir de sus descriptores de bajo nivel, éstas solo se pueden usar cuando se tiene una imagen que represente lo que se desea recuperar. Esto se debe a la dificultad que existe para asociar los descriptores de bajo nivel - que son representaciones numéricas de características visuales como color, textura y forma - con la descripción semántica del contenido de una imagen.
En este artículo se presenta una arquitectura de integración semántica de datos e imágenes médicas implementada en un prototipo para la validación de la propuesta de integración. La arquitectura propuesta integra información de datos demográficos, imágenes, anotaciones semánticas usando un vocabulario controlado y descriptores MPEG-7. Una estrategia de recuperación híbrida se integra en la arquitectura, con el propósito de brindar a los usuarios médicos una forma de organizar y almacenar casos médicos de interés y así facilitar una eficiente recuperación. El prototipo desarrollado, usando software libre, ofrece mecanismos para indexar las imágenes, seleccionar, manualmente, una región de interés (ROI) de la imagen, anotar imágenes o ROIs a partir de un vocabulario controlado de forma manual y automática y recuperar imágenes del repositorio. El prototipo ofrece varios mecanismos de búsqueda y recuperación de información: a partir de descriptores de bajo nivel de una imagen de ejemplo, con base en descriptores demográficos o en términos de la taxonomía médica o combinando estos mecanismos. En forma novedosa, se integran los datos demográficos del paciente y datos de las imágenes contenidos en los archivos DICOM usando descriptores de MPEG-7 y un vocabulario controlado que soporta una estrategia de recuperación híbrida, con el propósito de brindar a los usuarios médicos una forma de organizar y almacenar casos médicos de interés y así facilitar una eficiente recuperación, así como las anotaciones realizadas usando un vocabulario controlado brindan un herramienta de apoyo al diagnóstico en la medida en que un radiólogo puede consultar imágenes con el mismo diagnósticos.
El resto del artículo está estructurado en 4 secciones. En la sección 2, se presentan y discuten trabajos relacionados. En la sección 3, se presenta la arquitectura y se detalla cada uno de los componentes que la integran. En la sección 4, se describen las experiencias de desarrollo del prototipo como comentarios finales.
2. Trabajos relacionados
Integrar información clínica, diagnósticos e imágenes, generalmente anotadas, ha sido una fuente de desarrollo de sistemas y herramientas computacionales (Mhiri et al., 2009; Jonquet et al., 2008; Teodorescu et al., 2008a; Teodorescu et al., 2008b; Wei & Barnaghi, 2008; Rubin et al., 2008; Caicedo et al.,2007; Lacoste et al., 2007). En (Jonquet et al., 2008) se describe un sistema que permite anotar e indexar datos biomédicos. De acuerdo con los autores, el problema de extraer datos específicos ajustados a las necesidades de los investigadores en biomedicina se basa en crear etiquetas consistentes, para cada elemento de los repositorios públicos. En particular, en la anotación, se utiliza un diccionario que integra todos los nombres y sinónimos del conjunto de ontologías disponibles. Las anotaciones se registran en tablas con información sobre el elemento anotado, el concepto asociado con el elemento y el contexto en el cual se hace la anotación. (Teodorescu et al., 2008a ; Teodorescu et al., 2008b) introducen el método denominado de fusión semántica, a partir del cual y con base en una red semántica, integran información de imágenes de resonancia magnética y de tomografía con metadatos del metathesaurus de la National Library of Medicine.
La anotación semántica de imágenes se ha usado, generalmente, como estrategia de solución al problema de la brecha semántica. Anotar una imagen semánticamente implica asociarle un concepto frecuentemente extraído de vocabularios controlados, taxonomías u ontologías. En Wang et al. (2008), se discuten enfoques de anotación de imágenes: textual (anotadas manualmente por humanos) y automática (indexadas por su contenido visual mediante descriptores de bajo nivel de la imagen). La asociación entre estos dos tipos de anotación es difícil (brecha semántica). En este sentido, proponen una estrategia de recuperación de imágenes basada en contenido. Wang et al. (2006), muestran que la recuperación de imágenes se mejora cuando se combina tanto texto como características de la imagen.
Para hacer explícita la semántica del contenido de imágenes médicas, Rubin et al. (2008), construyen una ontología (OWL-Dl) para facilitar la anotación. Con el uso de ontologías de dominio se integra al análisis de imágenes médicas un conjunto de herramientas que se ofrecen a los médicos para anotar las imágenes. El objetivo es integrar contenido semántico y datos relacionados con su contenido.
En el dominio médico se han desarrollado algunas soluciones que integran semánticamente fuentes de datos heterogéneas como MEDICO (Sonntag & Möller, 2009) y ACGT (Martín et al., 2008) (Advancing Clinico-Genomic Trials on Cancer). MEDICO (Möller M. & Sintek M., 2007; Möller M. et al., 2009; Sonntag D. & Möller M., 2009) hace parte del proyecto THESEUS, cuyo propósito es construir un motor de recuperación de imágenes médicas. Las imágenes se consultan con base en el contenido semántico y en descriptores de bajo nivel. La arquitectura de MEDICO incluye una jerarquía de ontologías, cuyos conceptos se usan para anotar las imágenes, dotándolas así de semántica. Las consultas se hacen a través de imágenes de ejemplo, conceptos (palabras de la ontología), o lenguaje natural. Como resultado, la interfaz construye una consulta en SPARQL, para recuperar imágenes, datos del paciente y metadatos almacenados en formato DICOM o RDF.
Por su parte, ACGT (Long et al., 2009; Martín et al., 2008), busca proveer acceso integrado a bases de datos clínicos, genéticos e imágenes, a través de una infraestructura grid, con el fin de dar soporte a estudios sobre cáncer. Para ello, cuenta con un mediador, que funciona como un servicio, que provee herramientas de descubrimiento de conocimiento y que, a su vez, es un cliente de los wrappers que dan acceso a las bases de datos. Para abordar los problemas de semántica presentes al integrar las fuentes de datos, se usa la ontología Master Ontology.
Teniendo en cuenta que la anotación manual de imágenes es un proceso dispendioso y que contar con un conjunto grande de imágenes anotadas manualmente por especialistas médicos es poco práctico, varias estrategias, usando técnicas de aprendizaje automático, se han propuesto para hacer anotación automática (Dan Burdescu et al. 2012; Zhang et al., 2012; Ke et al., 2012; Byoung et al., 2012; Wang et al., 2008; Mueen et al., 2008; Caicedo et al., 2007; Racoceanu et al., 2006; Lacoste, 2006). Sin embargo, cualquier proceso de anotación automática requiere necesariamente de una base de datos de recursos anotados con base en la cual los algoritmos de aprendizaje se pueden aplicar. DanBurdescu et al. (2013) describen un sistema que permite la anotación de imágenes médicas y su recuperación basada en semántica y en contenido. Una vez segmentadas las imágenes, las regiones de la imagen se describen usando información extraída de MeSH (Medical Subject Headings).
Zhang et al. (2012) analizan y comparan métodos de anotación automática basados en redes neuronales, árboles de decisión y máquinas de soporte vectorial. Se trata de construir automáticamente un modelo de un concepto, con base en muestras de imágenes anotadas con ese concepto. Después, este modelo se usa para anotar imágenes nuevas.
De acuerdo con Ke et al. (2012), las bases de datos anotadas son difíciles de construir y por esa razón existen pequeños conjuntos de imágenes anotadas. Una estrategia de anotación basada en una comunidad web se propone. La estrategia incluye tareas de preprocesamiento de datos y particularmente hace énfasis en la aplicación de algoritmo KNN (K-nearest neighbour) para anotar las imágenes. Usar KNN requiere necesariamente contar con un conjunto de imágenes de entrenamiento que deben estar anotadas.
Byoung et al. (2012) por su parte, introducen un mecanismo de anotación múltiple de imágenes médicas basado en palabras clave. La anotación semántica usa clasificación basada en métodos que combinan local wavelet-based center symmetric-local binary patterns with random forests.
Caicedo et al. (2007), proponen un método de recuperación de imágenes a partir de dos fases: pre-procesamiento y recuperación. En la fase de pre-procesamiento, se toma una imagen original y a partir de histogramas y de meta-características, que se calculan, a su vez, a partir de histogramas, se genera una base de datos de características y se construye un modelo de clasificación. La fase de recuperación se activa mediante una consulta basada en una imagen de ejemplo, retornando las imágenes de la base de datos más similares. Racoceanu et al. (2006) y Lacoste (2006), proponen un marco de referencia para la recuperación de imágenes médicas, basado en máquinas de soporte vectorial y en técnicas de indexamiento basadas en el metathesaurus UMLS, para aportar información semántica asociada al contenido visual de las imágenes.
3. Arquitectura de integración semántica de datos e imágenes médicas
En esta sección se presenta una arquitectura de integración semántica de datos e imágenes médicas, con el propósito de facilitar la recuperación de imágenes integrando, además, diferentes tipos de datos. En particular se considera integrar: datos demográficos como filtros para refinar las búsquedas, etiquetas semánticas disponibles en forma de anotaciones usando un vocabulario controlado y descriptores visuales extraídos de una imagen de ejemplo o de una región de interés.
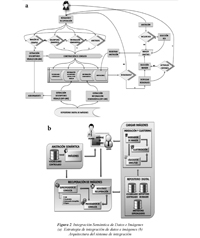
El mecanismo de integración se muestra en la Figura 2(a), la anotación manual que hace un médico, se puede hacer sobre una ROI o sobre una imagen completa. La imagen anotada, sus descriptores visuales de bajo nivel y la información demográfica asociada a la imagen se integran con las anotaciones que contienen términos que pertenecen al vocabulario controlado. De esta manera, se enriquecen semánticamente las imágenes médicas, para después facilitar su recuperación. Una anotación es una relación <Imagen-ROI, Médico, Datos demográficos, Diagnóstico, Términos del vocabulario>.
Cuando un médico selecciona una imagen para anotarla, el sistema le ofrece la opción de ver la información asociada con otras anotaciones, si estas existen. De esta manera, un residente en formación, podría etiquetar una imagen y después, verificar si su anotación coincide o discrepa de otras, hechas por otras especialistas.
Cuatro componentes se integran en la arquitectura propuesta como se ilustra en la Figura 2(b): Repositorio Digital, Recuperación de Imágenes, Anotación Semántica, e Indexación y Clustering.
3.1 el repositorio digital
El repositorio digital almacena, en sistema de archivos, los DICOM de los estudios realizados a los pacientes, los datos demográficos son extraídos como metadatos de las cabeceras DICOM, el tipo de órgano del estudio, los datos de los especialista que hacen anotaciones sobre las imágenes y un conjunto de términos que constituyen un vocabulario controlado para hacer la anotación. Los términos del vocabulario controlado que se utilizan en la anotación como metadatos de la imagen, forman parte de alguna de las ontologías médicas existentes (e.g. RadLex, ICD9). El repositorio digital se pobla con estudios que los médicos almacenan en este. Cada una de las imágenes incluidas en un DICOM, se exporta al formato JPG y la ruta de la imagen resultante se almacena en la base de datos junto con la información asociada. Esto con el propósito de, posteriormente, facilitar su acceso y carga desde la interfaz del sistema, durante el proceso de anotación textual o de recuperación.
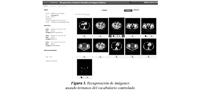
Los médicos especialistas que se registran en el sistema pueden cargar estudios DICOM completos o imágenes individuales, como se muestra en la Figura 3. Algunas de las imágenes de los estudios completos se pueden marcar como relevantes.
3.2. Recuperación de imágenes
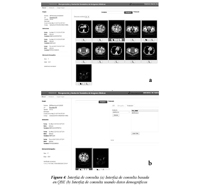
En este componente, se gestiona la búsqueda y la recuperación de las imágenes médicas, a partir del repositorio digital de imágenes. Algunos mecanismos de recuperación se ofrecen al usuario, de manera que pueda parametrizar los criterios con los cuales se recupera la información almacenada. La recuperación se puede hacer basada en Query By Example (QBE) - recuperación basada en una imagen ejemplo -, basada en información demográfica o en descriptores semánticos basados en el vocabulario controlado. La Figura 4(a), muestra la interfaz de consulta basada en QBE.
Particularmente, para el caso de recuperación basado en QBE, el médico carga una imagen como ejemplo para recuperar las top-n más similares a esta. La similitud se calcula con base en los descriptores de bajo nivel (MPEG-7) de la imagen. Este mecanismo de recuperación implica que el usuario ofrezca la imagen de referencia, y que ésta se indexe para extraer sus descriptores de bajo nivel. Para reducir el espacio de búsqueda, una estrategia de indexación se usa para agrupar imágenes similares. Diferentes clusters se generan por cada órgano. El órgano al cual hace referencia un estudio o una imagen, se extrae, como metadato, de la cabecera del DICOM. Por esta razón, y debido a que este metadato se registra manualmente, es posible que se introduzcan términos equivocados.
Los datos demográficos, tales como el grupo etario y el género, se extraen de la cabecera de cada archivo DICOM. Las búsquedas pueden incluir filtros o condiciones a partir de los cuales se restringe los resultados a aquellas que satisfacen las condiciones especificadas por el usuario, como se ilustra en la Figura 4(b).
Con base en el vocabulario controlado se pueden anotar, manualmente, las imágenes. Los términos del vocabulario, a su vez, sirven también como criterios de recuperación de imágenes, como se muestra en la Figura 5. Cuando un médico utiliza términos del vocabulario controlado como parte de los criterios de recuperación, el sistema le retorna las imágenes y/o estudios que coincidan en anotaciones utilizadas en la especificación de la consulta.
Una vez especificada la consulta, el sistema retorna, en forma de grilla, las imágenes resultantes del proceso de recuperación. Haciendo click sobre cada imagen resultante, se puede acceder a la información de las anotaciones (si existen) incluida la fecha, hora, autor (médico) y términos usados en la anotación.
3.3 Anotación semántica
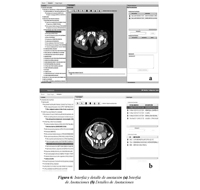
Este componente permite relacionar un conjunto de descriptores semánticos (etiquetas que pertenecen al vocabulario controlado) con una imagen o a una región de interés (ROI) de una imagen. Un usuario médico cuenta con herramientas para seleccionar una ROI sobre una imagen (e.g. ROI libre, polígono o circunferencia). Un médico puede acceder a una anotación en dos momentos. El primero, cuando recupera imágenes resultantes de una consulta, y el segundo, cuando carga una imagen en el repositorio de imágenes. En el primer caso, cada una de las imágenes resultante de la consulta puede contener anotaciones. Para acceder a la anotación se hace click en el icono de anotación, lo que produce que el sistema despliegue los detalles de la anotación como se muestra en la Figura 6(b). En esta interfaz se resaltan, a la izquierda, los términos usados en la anotación de la imagen o de la ROI, que forman parte una taxonomía, con base en la cual, se ordena el vocabulario controlado. Se muestran además, la imagen, en el centro, y todas sus anotaciones, en el lado derecho.
Un usuario puede navegar sobre las anotaciones o sobre los términos de la jerarquía. Cuando esto se hace, el sistema resalta o una ROI a la que pertenece la anotación o la imagen completa. En la parte superior de la interfaz se despliegan las utilidades disponibles para la selección de una ROI.
En el segundo caso, cuando el usuario sube una imagen, no se despliega la taxonomía, puesto que la imagen aún no se ha anotado. Para hacer una anotación, el usuario puede usar el campo de autocompletado ubicado en la parte derecha de la interfaz. Cuando se empieza a digitar un término, el sistema despliega los posibles términos del vocabulario con los cuales coincide. De esta forma, se controla que las anotaciones solo incluyan términos del vocabulario. La anotación puede ser simple, cuando se utiliza un solo término, o compuesta cuando se usan varios términos al momento de anotar.
3.4 Indexación y clustering
A partir de una imagen, en este componente, se extraen sus descriptores de bajo nivel (MPEG-7). El proceso de extracción de los descriptores de bajo nivel se puede hacer de dos modos: off-line u on-line. Es off-line, cuando se realiza de forma independiente al curso normal de eventos en el sistema, por ejemplo sobre un lote de archivos DICOM y es on-line cuando se realiza durante el curso normal de eventos en el sistema, por ejemplo cuando se carga la imagen de referencia en una consulta QBE.
Para hacer más eficiente el proceso de recuperación de imágenes basadas en consultas QBE, un mecanismo de cluster se usa para agrupar las imágenes más similares, en términos de descriptores de bajo nivel y para reducir el espacio de búsqueda, a este proceso se le conoce como indexación.
Las imágenes previamente cargadas en el repositorio son pre-procesadas mediante la extracción de descriptores de bajo nivel. En un proceso off-line, se utiliza K-means [Kanungo et al. (2002)] para generar los clusters. El valor de K y los valores iniciales de los centroides se determinan usando la propuesta de Chen et al. (2008).
4. Conclusiones
Este proyecto encontró en el uso de las tecnologías libres el medio para avanzar en la implementación de las funcionalidades esperadas por parte del equipo de investigación, garantizando su extensión en el futuro cercano debido a la disponibilidad de documentación y ausencia de patentes y/o licencias que comprometan la libre distribución y uso de la tecnología resultante. Luego de un análisis de diferentes tecnologías web, se decidió usar el framework Vaadin (http://www.vaadin.com, 2010), cuya arquitectura orientada a servidor facilitó dirigir las tareas dependiendo de su carga computacional. Es decir, tareas computacionalmente livianas como la selección de regiones de interés y la generación de interfaces gráficas en general, al lado del cliente, en este caso al navegador web, y ocupar al servidor Apache Tomcat con las tareas computacionalmente costosas como la extracción de descriptores visuales de bajo nivel a partir de las imágenes y de información demográfica del paciente de la cabecera de los archivos DICOM, la construcción de consultas y recuperación de información a partir de múltiples criterios de búsqueda que incluyen información demográfica, anatómica (órganos), QBE y palabras dentro de un vocabulario controlado.
El prototipo se puede extender para ofrecer otros mecanismos de selección de ROIs, para integrar múltiples vocabularios que tengan en cuenta, por ejemplo, la especialidad del médico (internista, radiólogo, etc.), las condiciones de captura de la imagen, incrementando el tipo de metadatos utilizados.
5. Referencias
Byoung, C. K., JiHyeon, L., & Jae-Yeal, N. (2012). Automatic medical image annotation and keyword-based image retrieval using relevance feedback. J Digit Imaging, 25, 454-465. [ Links ]
Caicedo, J. C., González, F., & Romero, E. (2007). Content-Based Medical Image Retrieval Using Low-Level Visual Features and Modality Identifications. Advanced in Multilingual and Multimodal Information Retrieval. 8th Workshop of the Cross-Language Evaluation Forum CLEF. (pp. 615-622). [ Links ]
Chen J., Qin Z., & Jia, J. A. (2008). Weighted Mean Subtractive Clustering Algorithm. Information Technolognal, 7, 56-360. [ Links ]
Chen, J., Bradshaw, J., & Nagy, P. (2011). Has the Picture Archiving and Communication System (PACS) Become a Commodity? Journal of Digit Imaging, 24 (1), 6-10. [ Links ]
Dan Burdescu, D., Mihai, C. G., Stanescun, L., & Brezovan, M. (2013). Automatic image annotation and semantic based image retrieval for medical domain. Neurocomputing, 109, 33-48. [ Links ]
Deselaers, T., Deserno, T.M., & Müller, H. (2007). Automatic medical image annotation in ImageCLEF 2007: Overview, results, and discussion. Pattern Recognition Letters, 29, 1988-1995. [ Links ]
Gupta, A., Bug, W., Marenco, L., Qian, X., Condit C., Rangarajan A., et. al. (2008). Federated access to heterogeneous information resources in the Neuroscience Information Framework (NIF). Neuroinformatics, 6 (3), 205-217. [ Links ]
Jonquet, C., Musen, M. A., & Shah, N. (2008, Jan.). A system for ontology-based annotation of biomedical data. In, Data Integration in the Life Sciences (pp. 144-152). Berlin, Germany: Springer Berlin Heidelberg. [ Links ]
Kanungo, T., Mount, D.M., Netanyahu, N.S., Piatko, C.D., Silverman, R., & Wu, A.Y. (2002). An Efficient K-Means Clustering Algortihm: Analysis and Implementation. IEEE Transactions on Pattern Analysis and Machine Intelligence, 24 (7), 881-892. [ Links ]
Ke, X., Li, S., & Chen, G. (2013). Real web community based automatic image annotation. Journal Computers and Electrical Engineering, 39 (3), 945-956. [ Links ]
Lacoste, C., Chevallet, J., Lim, H., Hoang, D.T., Xiong, W., Racoceanu, D. et. al. (2006). Inter-Media Concept-Based Medical Images Indexing and Retrieval with UMLS at IPAL. CLEF. Lecture Notes in Computer Science, 4730, 694-701. [ Links ]
Long, R., Antani, S., Deserno, T., & Thoma, G. (2009). Content-Based Image Retrieval in Medicine: Retrospective Assessment, State of the Art, and Future Directions. Intl. Journal of Healthcare Information Systems and Informatics, 4(1), 1-16. [ Links ]
Marenco, L., Li, Y., Martone, M., Sternberg, P., Shepherd, G., & Miller, P. (2008). Issues in the Design of a Pilot Concept-Based Query Interface for the Neuroinformatics Information Framework. Neuroinformatics, 6 (3), 229-239. [ Links ]
Martín, L., Anguita, A., Maojo, V., Bonsma, E., Bucur, A., Vrijnsen, J. et. al. (2008). Ontology Based Integration of Distributed and Heterogeneous Data Sources in ACGT. In Proceedings of the First International Conference on Health Informatics, HEALTHINF 2008, vol. 1 (pp. 301-306). Madeira, Portugal. [ Links ]
Mansoori, B., Erhard, K., & Sunshine, J. (2012). Picture Archiving and Communication System (PACS) implementation, integration & benefits in an integrated health system. Academic Radiology, 19 (2), 229-235. [ Links ]
Mhiri, S., Depres, S., & Zagrouba, E. (2008). Ontologies for the Semantic-Based Medical Image Indexing: An Overview. In, Proceedings of International Conference on Information and Knowledge Engineering, 311-317. [ Links ]
Möller, M., Regel, S., & Sintek, M. (2009). RadSem: Semantic Annotation and Retrieval for Medical Images. The Semantic Web: Research and Applications, 6th European Semantic Web Conference, ESWC 2009. Lecture Notes in Computer Science, 5554, 21-35. [ Links ]
Möller, M., & Sintek, M. (2007). A Generic Framework for Semantic Medical Image Retrieval. Proceedings of the Knowledge Acquisition from Multimedia Content (KAMC) Workshop. Genoa, Italy. [ Links ]
Möller, M., Vyas, N., Sintek, M., Regel, S., & Mukherjee, S. (2009). Visual Query Construction for Cross-Modal Semantic Retrieval of Medical Information. Proceedings of the Malaysian Joint Conference on Artificial Intelligence MJCA. Kuala Lumpur, Malasya. [ Links ]
Mueen, A. , Zainuddin, R., & Baba, MS. (2008). Automatic multilevel medical image annotation and retrieval. Journal of Digital Imaging, 21(3), 290-5. [ Links ]
Racoceanu, D., Lacoste, C., Todorescu, R., & Vuillemenot, N. (2006). A Semantic Fusion Approach between Medical Images and Reports Using UMLS. Third Asia Information Retrieval Symposium, AIRS . Lecture Notes in Computer Science, 4182, 460-475. [ Links ]
Rubin, D.L., MongKolwat, P., Kleper, V., Supekar, K., & Channin, D.S. (2008). Medical Imaging on the Semantic Web: Annotation and Image Markup. AAAI Spring Symposium Series, Semantic Scientific Knowledge Integration. Technical Report SS-08-05. Stanford, California. [ Links ]
Shah, N.H., & Musen, M.A. (2007). Which Annotation did you mean? Stanford Medical Informatics Technical Report, SMI-2007-1247. [ Links ]
Sonntag, D., & Möller, M. (2009). Unifying Semantic Annotation and Querying in Biomedical Images Repositories. Proceedings of the International Conference on Knowledge Management and Information Sharing (KMIS-09). INSTICC Press, 2009, 89-94. Madeira, Portugal. [ Links ]
Teodorescu, R., Cernazanu-Glavan, C. Cretu, V., & Racoceanu, D. (2008). The use of medical ontology in a knowledge-based semantic fusion system. 4th International Conference on Intelligent Computer Communication and Processing, ICCP 2008, 265-268. [ Links ]
Teodorescu, R., Racoceanu, D., Leow, W., & Cretu, V. (2008). Prospective Study for Semantic Inter-Media Fusion in Content-Based Medical Image Retrieval. Medical Imaging Technology, 26, 1-11. [ Links ]
Rivera, J. H., Trujillo, J. P. & Serna, W. (2010). Descripción del estándar DICOM para un acceso confiable a la información de las imágenes médicas. Scientia et Technica, 1 (45), 289-294. [ Links ]
Tutac, A. E., Racoceanu, D., Putti, T., Xiong, W., Leow, W., & Cretu, V. (2008). Knowledge-Guided Semantic Indexing of Breast Cancer Histopathology Images. International Conference on BioMedical Engineering and Informatics. IEEE Computer Society, 107- 112. [ Links ]
Wei, W., & Barnaghi, P.M. (2007). Semantic Support for Medical Image Search and Retrieval. Proceeding BIEN '07, Proceedings of the fifth IASTED International Conference: biomedical engineering, ACTA Press. (pp. 315-319). Anaheim (CA), USA. [ Links ]
Wang, C., Zhang, L., & Zhang, H-J. (2008). Learning to reduce the semantic gap in web image retrieval and annotation. SIGIR '08: Proceedings of the 31st Annual International ACM SIGIR Conference on Research and Development in Information Retrieval. (pp. 355-362). Singapore, Singapore. [ Links ]
Zhang, D., Islam, M., & Lu, G. (2012). A review on automatic image annotation techniques. Pattern Recognition, 45 (1), 346-362. [ Links ]

Revista Ingeniería y Competitividad por Universidad del Valle se encuentra bajo una licencia Creative Commons Reconocimiento - Debe reconocer adecuadamente la autoría, proporcionar un enlace a la licencia e indicar si se han realizado cambios. Puede hacerlo de cualquier manera razonable, pero no de una manera que sugiera que tiene el apoyo del licenciador o lo recibe por el uso que hace.