1. Introducción
El cultivo de cacao (Theobroma cacao) en Colombia actualmente ocupa alrededor de 164.000 Ha, en el año 2018 se produjeron 56.867 toneladas de cacao, por debajo de los 500 kilogramos por hectárea año lo cual representa un crecimiento del 3.7 % en comparación de la producción del grano en el año 2015, en el cual se registraron 54.798 toneladas (1. Estas plantaciones se han caracterizado por el uso de bajos niveles de tecnología para su manejo, lo que ha generado la reducción gradual de la productividad; que, sumado a la incidencia de enfermedades como la Moniliasis, producida por el hongo Moniliophthora roreri2, viene causando graves pérdidas económicas, siendo este otro factor limitante para la producción nacional. Adicionalmente el manejo tradicional de esta enfermedad se limita únicamente a la remoción de frutos enfermos, siendo poco conocidos y aplicados otros métodos que contribuyan al manejo biocultural.
Actualmente la Federación Nacional de Cacaoteros indican que la afectación por esta enfermedad alcanza el 80% en todo el cultivo de cacao en el departamento Norte de Santander. Una las principales razones de esta alta incidencia es el manejo poco tecnificado que se presenta en los cultivos. Está comprobado que el control biológico es un método efectivo y una alternativa al uso de agentes químicos que contaminan suelos y fuentes hídricas, que se basa en el uso de los principios ecológicos como los organismos benéficos y/o sus productos, aprovechando así los beneficios de la biodiversidad en la agricultura 3. Diversos estudios realizados en Norte de Santander se han centrado en el aislamiento, identificación y conservación de microorganismos en el control de la “moniliasis” en el cultivo de cacao 4-6. Estos estudios tienen como objetivo primordial, mitigar en gran medida la incidencia de la “moniliasis” en el departamento. Sin embargo, la tendencia actual se centra en el análisis molecular de los genomas de estos microrganismos con el fin de conocer sus fortalezas genético-moleculares y así poder generar productos comerciales con alto valor agregado en la lucha contra esta enfermedad en la región.
Este trabajo tuvo como objetivo encontrar genes de interés en los procesos patogénicos llevados a cabo por hongos antagonistas, así como obtener información sobre las capacidades biotecnológicas de este tipo de cepas con el fin de poder implementar marcadores moleculares que se podrían usar en actividades de control de calidad de microorganismos antagonistas que se producen comercialmente, los cuales son regulados mediante pruebas de patogenicidad.
2. Metodología
Se seleccionaron 6 cepas aisladas de plantaciones de cacao de Norte de Santander con actividad antagonista contra M. roreri, pertenecientes al banco de cepas de la facultad de ciencias agrarias y del ambiente. Trichoderma yunnanense (HA001), Beauveria bassiana (HE002-3), Penicillium citrinum (HD011-5), Purpureocillium lilacinus (GIAV 2-3), Penicillium rubens (HE001-5,) y Aspergillus nidulans (HD017-18).
2.1. Extracción de ADN
La extracción se llevó a cabo tomando micelios crecidos en caldo Dextrosa Sabouraud modificado (Dextrosa 40 g/l, extracto de levadura 5 g/l, Bactopeptona 10 g/l a pH 7,5,) estos se coloraron en agitación a 28 °C por 24 - 48 horas en baño seco 7. Una vez transcurridas las 24-48h, se ejecutó el protocolo propuesto por 8, para la extracción de ADN el cual consistió en el método de solventes orgánicos (fenol-cloroformo).
2.2. Estandarización de técnica de amplificación de ADN polimórfico al Azar (RAPD)
La estandarización de la técnica de RAPD se llevó a cabo según el protocolo propuesto por 7 en un termociclador de gradiente “MultigeneTM Optimax” (Labnet). La mezcla de reacción consistió en los iniciadores Operon Technology, serie E (OPE-01 - OPE-10) (Tabla 1). Se utilizó el ADN del fago λ digerido con la enzima de restricción Hind III, como patrón de peso molecular (100pb).
Cada muestra se analizó mediante electroforesis en geles de agarosa 1,0 % (p/v) diluida en buffer TBE 1X (Trisborato, EDTA 0,5M pH 8), a 100 voltios, utilizando como intercalante GelRed®. Los geles se visualizaron en un fotodocumentador ChemiDoc™ Imaging Systems Bio-Rad.
Tabla 1 Iniciadores para RAPD
| Iniciador* | Secuencia | Referencia |
|---|---|---|
| OP E-01 | 5´- CCC AAG GTC C - 3´ | Dávila, et al, 2001 9 |
| OP E-02 | 5´- GGT GCG GGA A- 3´ | |
| OP E-03 | 5´- CCA GAT GCA C- 3´ | |
| OP E-04 | 5´- GTG ACA TGC C- 3´ | |
| OP E-05 | 5´- TCA GGG AGG T- 3´ | |
| OP E-06 | 5´- AAG ACC CCT C- 3´ | |
| OP E-07 | 5´- AGA TGC AGC C- 3´ | |
| OP E-08 | 5´- TCA CCA CGG T- 3´ | |
| OP E-09 | 5´- CTT CAC CCG A- 3´ | |
| OP E-10 | 5´- CAC CAG GTG A- 3´ |
*Los iniciadores fueron producidos por Operon Technologies Inc. (9
El análisis de los datos se realizó mediante el registro de los productos de amplificación, asignando (0) para ausencia y (1) para presencia de bandas en matrices binarias. Así mismo se construyeron matrices de distancia usando el índice de Jaccard y el dendograma mediante análisis de agrupamiento UPGMA. Por otra parte, se determinó el contenido de información polimórfica (PIC). En conjunto los análisis permitieron visualizar las relaciones genéticas entre los aislamientos. Todos los análisis se realizaron usando el programa InfoGen.
2.3. Identificación de genes de proteasas
Para la identificación de genes de proteasa en las cepas en estudio, se siguió la metodología propuesta 10 y 11, usando los iniciadores para el gen pr1A (Tabla 2). Las cepas HB012 y HB015 se usaron como controles positivos.
Tabla 2 Iniciadores para gen de proteasa
| Gen | Iniciador* | Secuencia | Referencia |
|---|---|---|---|
| pr1A | Pr1ALF | 5´- CATCAGTGGCATGGACTACGTT- 3´ | Rosas y Avalos-de-León en 2014 10 |
| Pr1ALR | 5´- GGAAGCAATGGCGCCTTT- 3´ | ||
| pr1B | Pr1BLF | 5´AGCGCCGGCGAAGGAACA3´ | |
| Pr1BLR | 5´CGCCGCCCACCGAGACAAAC3´ | ||
| pr1C | Pr1CLF | 5´CACGCCCGATCTCAGCAAGTAT3´ | |
| Pr1CLR | 5´CTCAGAGCGCCCGCCAGTAATG3´ | ||
| Pr1D | Pr1DLF | 5´CAAATAATCCGCGCCCTCCAATGT3´ | |
| Pr1DLR | 5´ACGCCCGTCCCTCAAACTCC3´ | ||
| pr1E | Pr1ELF | 5´TACCCTCGCGCCTCTTGAACTA3´ | |
| Pr1ELR | 5´TCGGAATATCGGTGTCGGTAAT3´ | ||
| Pr1F | Pr1fLF | 5´TGCTACCGGGGAGGATGACT3´ | |
| Pr1FLR | 5´CTGGGGCAAAAATGGTGACA3´ |
*El iniciador fue sintetizado por Isla S.A.S
La extracción de ADN de cada cepa fúngica se llevó a cabo de acuerdo con el protocolo descrito anteriormente. El ADN (1 µl) se colocó en un tubo de microcentrífuga que contenía 1,25 U de ADN Taq polimerasa (Promega), 1x tampón Taq, 0,2 µM de iniciador, dNTPs 0,2 mM y 14,8 µl de H2O miliQ para un volumen final de 25 µl. La amplificación se realizó con un termociclador de gradiente “MultigeneTM Optimax (Labnet) con un ciclo de desnaturalización inicial a 96 °C durante 2 minutos y un programa de ciclos programado para 30 ciclos (con un ciclo que consiste en desnaturalización a 94 °C durante 45 segundos) recocido a 60 °C, durante 1 min, y extensión a 72 °C durante 1 min), y un paso extra de extensión a 72 °C durante 7 min. Después de la amplificación, 1 µl de muestra de cada mezcla de PCR se mezcló con 4 µl de GelRed® y se sometió a electroforesis en un gel de agarosa al 1.5% en tampón TBE 1X a 100 V durante 1 h. Los geles se visualizaron en un fotodocumentador ChemiDoc™ Imaging Systems Bio-Rad.
3. Resultados y discusión
3.1. Extracción y visualización de ADN genómico de los hongos antagonistas
En este trabajo se realizó la extracción de ADN de 6 aislamientos de hongos antagonistas que se obtuvieron del banco de cepas de la facultad de ciencias agrarias. Un óptimo método de extracción de ADN debe garantizar el mantenimiento de la integridad física y bioquímica de la molécula y por lo tanto incrementar sus rangos de pureza y concentración 12. Se obtuvo ADN de cada extracción según lo propuesto por Suárez 1. Por medio de la corrida de electroforesis se logró visualizar una óptima calidad y cantidad de ADN de las 31 repeticiones que se realizaron de los 6 aislados (Figura 1). A partir de allí se pudo continuar con los análisis moleculares de los hongos antagonistas.
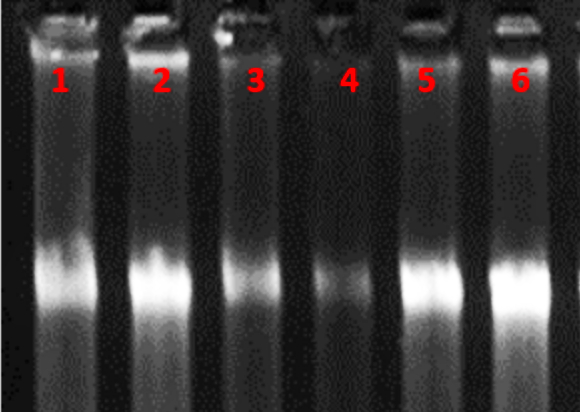
Figura 1 Gel de electroforesis de ADN en agarosa al 1,5 %. Línea 1: Trichoderma yunnanense; Línea 2: Beauveria bassiana; Línea 3: Penicillium citrinum; Línea 4: Purpureocillium lilacinus; Línea 5: Penicillium rubens; Línea 6: Aspergillus nidulans.
Se logró apreciar claramente las bandas de Trichoderma yunnanense (HA001), Beauveria bassiana (HE002-3), Penicillium citrinum (HD011-5), Purpureocillium lilacinus (GIAV 2-3), Penicillium rubens (HE001-5,) y Aspergillus nidulans (HD017-18), demostrando satisfactoriamente la extracción de ADN genómico.
3.2. Estandarización de técnica de amplificación de ADN polimórfico al Azar (RAPD
Los 10 iniciadores RAPD generaron 134 bandas amplificables y reproducibles (Figura 2). El iniciador OPE-09 no generó ninguna banda, lo cual indicó que, bajo las condiciones de amplificación, por lo menos una de las secuencias determinadas no existía en ninguno de los hongos para que estos iniciadores prealinearan, por lo tanto, no se tuvo en cuenta en el análisis. Los iniciadores OPE-06 y OPE-08 generaron el mayor número de bandas polimórficas, 19 y 17, respectivamente, lo cual los sugiere como buenos iniciadores para hacer un “fingerprinting”. El contenido de información polimórfica (PIC) de los iniciadores fue similar con valores entre 0,31 y 0,34 (Tabla 3).
Tabla 3 Tipos y números de alelos detectados por los iniciadores de RAPD de los 6 aislamientos de hongos antagonistas.
| Iniciador | BP | BM | BT | PIC | SD |
|---|---|---|---|---|---|
| OPE_01 | 12 | 0 | 12 | 0.31 | 0.02 |
| OPE_02 | 16 | 1 | 17 | 0.30 | 0.01 |
| OPE_03 | 9 | 2 | 11 | 0.33 | 0.01 |
| OPE_04 | 16 | 0 | 16 | 0.34 | 0.01 |
| OPE_05 | 15 | 0 | 15 | 0.33 | 0.01 |
| OPE_06 | 19 | 0 | 19 | 0.33 | 0.01 |
| OPE_07 | 16 | 0 | 16 | 0.31 | 0.01 |
| OPE_08 | 17 | 0 | 17 | 0.34 | 0.01 |
| OPE_10 | 14 | 0 | 14 | 0.34 | 0.01 |
| Total | 134 | 3 | 134 |
BM= Bandas monomórficas, BT= Bandas totales, BP= Bandas polimórficas, PIC= Contenido de información polimórfica y SD= Desviación estándar
De los nueve iniciadores utilizados se lograron observar tres de las bandas que resultaron ser monomórficas para las muestras empleadas. Esto permitió comprobar la presencia de bandas de longitudes similares en otras especies de hongos antagonistas. Estos hallazgos podrían dar indicios de la posibilidad de utilizar los iniciadores involucrados como marcadores de factores de patogenicidad bajo condiciones específicas
En el análisis de los datos de la técnica se pudo evidenciar variabilidad intra e interespecífica entre los aislamientos examinados, formando 4 grupos (Figura 2). El primer grupo incluye al aislamiento Aspergillus nidulans (HD017-18), este se encontró ligeramente disperso de los demás aislamientos a una distancia de 0,76 lo cual indicó que comparte un 24% del reservorio genético. Además, se encontró un segundo grupo el cual se evidencio la presencia de dos aislados Trichoderma yunnanense (HA001), y Beauveria bassiana (HE002-3). La distancia de este grupo fue de 0,68, compartiendo el 32% de las características con el resto de la población. Un tercer grupo estuvo constituido por los aislados Penicillium citrinum (HD011-5) y Penicillium rubens (HE001-5), este estuvo separado 64% de los demás aislamientos, como era de esperarse al pertenecer al mismo género las especies guardan ciertas similitudes en su genoma. El último grupo estuvo formado por Purpureocillium lilacinus (GIAV 2-3) a una distancia de 70%.

Figura 2 Dendrograma de los coeficientes de distancia (1-S de Jaccard) obtenido mediante algoritmo de UPGMA con datos de RAPDs en 6 cepas de hongos antagonistas.
Se espera que el análisis de una especie altamente heterocigota mediante la técnica RAPD subestime las distancias genéticas, ya que los marcadores RAPD son marcadores dominantes 13. La detección de loci heterocigotos utilizando técnicas de marcadores codominantes daría relaciones genéticas más realistas. Sin embargo, considerando los ahorros de tiempo y costos, los RAPD han demostrado una resolución adecuada de las variedades locales que se utilizarán con fines de reproducción 14. Teniendo en cuenta los resultados obtenidos mediante los marcadores RAPD se encontró una relación entre los grupos de los aislamientos con la naturaleza y característica de cada uno de ellos, esto demostró una alta variabilidad entre los mismos, lo cual indica que los RAPD son una técnica confiable para la caracterización de aislamientos de hongos antagonistas. Los resultados son corroborados por 15 quienes demostraron que los marcadores RAPD revelan una gran variabilidad inter e intraespecífica realizado en cepas de Trichoderma examinadas. Así mismo, 16 resaltan que el uso de RAPD permitió demostrar variabilidad entre los aislamientos de Trichoderma, como se indica en la Figura 3.
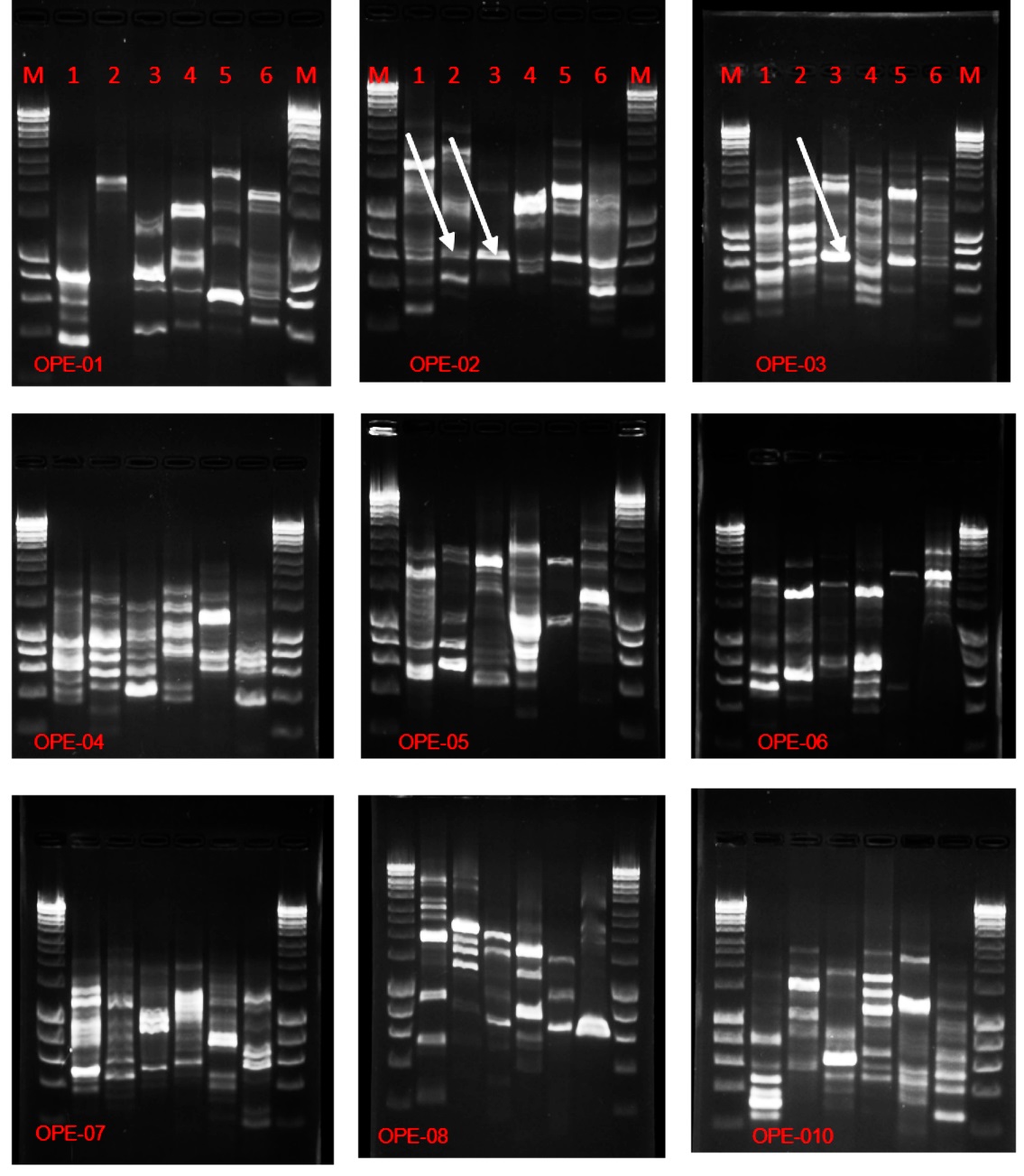
Figura 3 Patrones de bandeo en gel de electroforesis al 1%. Carril 1: Beauveria bassiana; Carril 2: Penicillium Rubens; Carril 3: Penicillium citrinum; Carril 4: Aspergillus nidulans; Carril 5: Purpureocillium lilacinus; Carril 6: Trichoderma yunnanense; Carril M: marcador de 1 kb. Primers OP_E01 hasta OP_E10.
3.3. Identificación de genes de proteasas.
La Figura 4 muestra un gel de agarosa que representa una correcta amplificación del control positivo (Metarhizium anisopliae) con un tamaño aproximado de 1400 pb 11. De las seis cepas en estudio tres de ellas Beauveria bassiana, Trichoderma yunnanense y Purpureocillium lilacinus evidenciaron la presencia de una banda de 300, 400 y 320 pb aproximadamente. Esta amplificación se produjo mediante el uso de los oligos pr1BF-R y pr1CF-R respectivamente. Los resultados indicaron que las tres cepas albergan el gen pr1. Este gen ha sido reportado pertenecer a una familia multigénica de 11 isoformas encontradas en el género Metarhizium17. La presencia de este gen podría representar una ventaja en comparación con otras cepas de antagonistas en la capacidad patogénica y de control de plagas y enfermedades
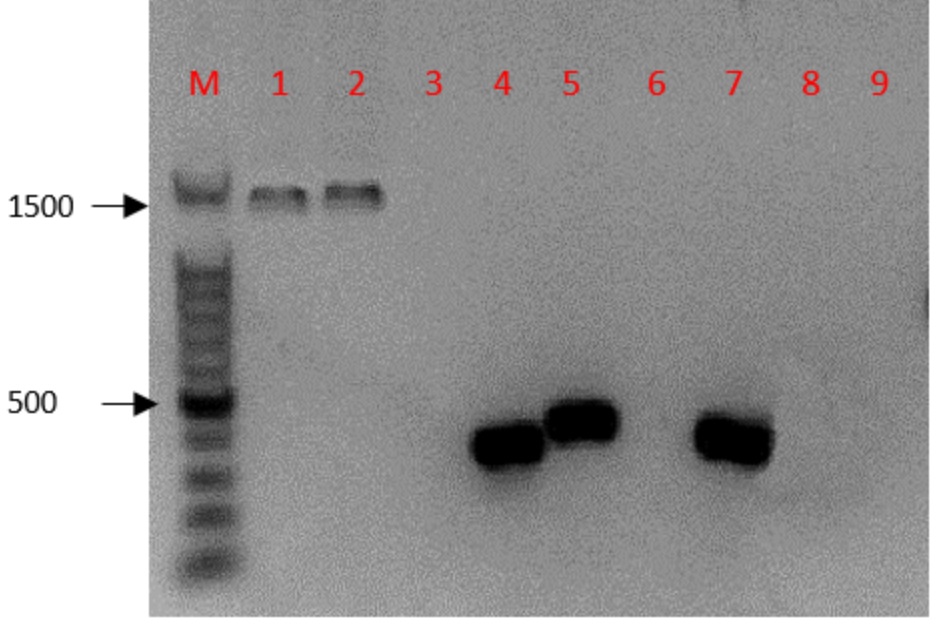
Figura 4 Gel de electroforesis al 1,5%. Carril M: marcador de peso molecular (100 pb); Carriles 1 y 2: control positivo; Carril 3: control negativo (agua); Carril 4: Beauveria bassiana; Carril 5: Trichoderma yunnanense; Carril 6: Aspergillus nidulans; Carril 7: Purpureocillium lilacinus; Carril 8: Penicillium citrinum; Carril 9: Penicillium Rubens.
La interacción entre los patógenos potenciales y sus antagonistas, se da de forma natural en el medio ambiente, la acción directa de los antagonistas repercute de forma directa a mitigar las enfermedades asociadas a los cultivos de interés agrícola 18. Las condiciones medioambientales naturales propician un equilibrio dinámico de los microorganismos en la superficie de las plantas y en el suelo. Diversos mecanismos de acción de los antagonistas como antibiosis, competencia por espacio o por nutrimentos, interacciones directas con el patógeno (micoparasitismo y lisis enzimática) e inducción de resistencia, se han descrito para controlar el desarrollo de patógenos 19.
Entre los antagonistas existe un tipo de interacción directa con los patógenos llamado parasitismo. El parasitismo es una simbiosis antagónica entre organismos en la que uno vive a expensas de otro. En este proceso están involucradas una serie de enzimas extracelulares tales como B1,3-glucanasa quitinasa, proteasa y celulasa, las cuales escinden las estructuras de virus, bacterias y hongos parasitados. Existen varias evidencias de hongos hiperparásitos como lo son Trichoderma sp., Beauveria y Metarhizium, los cuales utilizan el parasitismo como el mecanismo más importante. Uno de los géneros más estudiado con actividad antagónica hacia organismos patógenos ha sido Trichoderma en contra de Rhizoctonia solani, un patógeno de suelos agrícolas, además ya se encuentran en el mercado diversas formulaciones desarrolladas a partir de ellos 20.
Diversos hongos antagonistas y entomopatógenos excretan una gran variedad de enzimas como son proteasas, quitinasas, fosfolipasas, lipasas, esterasas, fosfatasas, etc., que se encargan de causar antibiosis y de hidrolizar la cutícula de los insectos 21. Dentro de las proteasas se encuentran las del tipo de las subtilisina. Muchas de estas proteasas son sintetizadas cuando el hongo es cultivado en medios con cutícula de insecto, y se consideran entre los principales factores de virulencia 17.
La evolución de las subtilisinas (Pr1) ha dado como resultado un número relativamente alto de isoformas en diversos hongos, actualmente se han reportado 11 genes de subtilisinas en los cuales el número y el nivel de expresión varía ampliamente 21. Múltiples isoformas del gen pr1 con diferencias sutiles en la especificidad pueden actuar sinérgicamente en la degradación de la proteína cuticular de los insectos y demás organismos plaga, permitiendo al hongo operar en condiciones ambientales cambiantes 22.
4. Conclusiones
Por medio del uso de la técnica de RAPDs, se logró una clasificación inicial de la población de hongos antagonistas aislados e identificados en el laboratorio de Biotecnología Molecular Así mismo se demostró la diversidad de los aislados. Además, se pudo amplificar tres genes de las cepas Beauveria bassiana, Trichoderma yunnanense y Purpureocillium lilacinus, que resultaron codificar para una familia de proteasas destinadas a ejercer actividad antagonista contra un amplio rango de hospederos. Gracias a la amplificación de estos genes se puede inferir que las cepas en estudio son buenos candidatos iniciales para formulación de biopreparados destinados al control biológico de organismos plaga en cultivos de interés agrícola en la región.















