Introducción
El género Annona presenta una amplia diversidad genética, y aunque varias de sus especies producen frutos comestibles con una pulpa altamente valorizada, factores como la susceptibilidad de la piel y el corto período de duración, afectan a muchas de sus especies y no ocupan un lugar destacado actualmente en el comercio de frutas tropicales.
Para mantener y aumentar su cuota de mercado, las exigencias para la industria son el desarrollo de variedades de alta calidad adaptables a las diversas condiciones abióticas y bióticas del ambiente y lograr producir rendimientos aceptables para el productor (Reina et al., 1996). Por ello, la importancia de conocer la variación genética dentro de una población hará factible el diseño de planes eficientes en el manejo, aprovechamiento y preservación de los recursos genéticos (Guarino y Maxted 1996; Otero-Arnaiz et al., 2005). Esta información sistematizada es indispensable para programas de reproducción adecuados y de selección de variedades resistentes a enfermedades y parásitos (Harlan, 2009; Marshall y Brown 1975).
Muchas de estas especies son originarias de América Central y del norte de Suramérica. La chirimoya (A. cherimola) es una excepción, ya que su origen se ubica en la región subtropical de los Andes (Popenoe, 1975). En diversas especies del género el número de cromosomas es 2n=2x=14 ó 16, a excepción de la especie A. glabra que es tetraploide (Kessler, 1993). Por su parte, A. muricata es originaria de América y África tropical. Es un árbol tropical, reconocido en Colombia por el potencial económico y por las propiedades nutricionales de su pulpa. Su fruto se considera exótico con características sensoriales excelsas que le brindan un potencial para su utilización como producto fresco o transformado en la fabricación de bebidas, mermeladas, néctares y otras conservas. Incluso las semillas reportan un efecto potencial contra el cáncer, así como otras aplicaciones médicas (Márquez-Cardozo, 2009).
Pese a estas características, su cultivo en el país participa con solo 6.481 ha, de un área total de 259.409 dedicadas al cultivo de fruta (Agronet, 2018).
Los microsatélites (SSR, por sus siglas en inglés, simple sequence repeats), se han utilizado ampliamente como herramienta para responder a ciertas preguntas dentro de las poblaciones naturales, con relación por ejemplo al flujo de genes y al mapeo genético (Zaghloul et al., 2006). La naturaleza codominante y polimórfica de los SSR los hace altamente informativos en comparación con otros marcadores. Además de ser selectivamente neutrales y de seguir un patrón de herencia mendeliana, lo que favorece su utilización para la detección de patrones demográficos (Selkoe y Toonen, 2006).
Se ha demostrado que cebadores diseñados para algunas especies pueden utilizarse para detectar polimorfismos en loci homólogos de especies relacionadas, como en el caso de algunas plantas tropicales (Squirrell et al., 2003; Zucchi et al., 2003; Nybom, 2004; Dayanandan et al., 1997; Arnold et al., 2002; Escribano et al., 2004; Mix et al., 2004; Schnell et al., 2005; Escribano et al., 2008). En el caso de la familia Annonaceae, se han amplificado con éxito regiones microsatélites en diferentes especies, a partir de cebadores diseñados para la amplificación de regiones microsatélites en A. cherimola (Escribano et al., 2004; Escribano et al., 2008; Salazar et al., 2010). En el presente estudio se evaluó la diversidad genética de poblaciones de A. muricata procedentes de la región central de Colombia, a partir de regiones microsatélites altamente discriminativas empleadas en otras especies de la misma familia.
Materiales y métodos
Material vegetal
Se analizó un total de 102 cultivares de A. muricata colectados en cuatro fincas del departamento de Caldas: Portugal (Palestina), Zaragoza (Palestina), Carminales (Palestina), El Bosque (Neira) y en una finca del departamento de Risaralda: Halcones (Pereira).
Los materiales provenientes de Palestina han sido cultivares originalmente propagados sobre portainjertos nativos regionales. Y los materiales provenientes de Risaralda, desde sus inicios se establecieron por semilla. Se colectó el tejido foliar joven y sano de cada individuo, se pulverizó mediante nitrógeno líquido y se preservó en ultra congelador a - 80°C.
Obtención del ADN genómico
Se siguió el protocolo de extracción de ADN descrito por Doyle y Doyle (1990), modificado por Marulanda et al. (2007). La calidad del ADN se verificó en geles de agarosa. La verificación de la relación del radio a una absorbancia entre 260/280 nm se realizó mediante un Nanodrop 2000 (Thermo Fisher, USA).
Amplificación de regiones microsatélites (SSR)
Se evaluaron 102 genotipos de A. muricata mediante 16 marcadores moleculares SSR, seleccionados a partir de regiones polimórficas en A. cherimola reportadas por Escribano et al. (2004), Escribano et al. (2008) (Tabla 1). Para las reacciones de amplificación se usó el máster MangoMixTM de Bioline, y se ajustaron las condiciones de acuerdo con Escribano et al. (2008) así: de cada cebador y 25 ng de ADN, para un volumen final de reacción de 12,5 μL. El perfil térmico se ajustó con base en los parámetros de Escribano et al. (2007) y Hilje-Rodríguez (2008) así: 35 ciclos de 30 seg a 94°C, desnaturalización inicial 1 min. a 94°C, seguido de 30 seg. a 94°C, 30 seg a 55°C, y 1 min a 72°C, y una extensión final por 5 min a 72°C.
Tabla 1 SSR usados para la evaluación de la diversidad genética de 102 materiales de A. muricata.
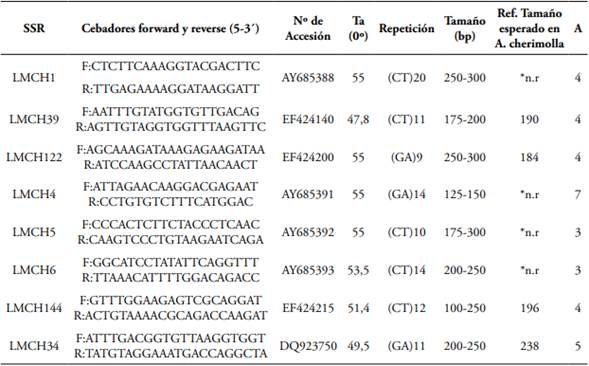
Nota. Identificación del Locus, secuencias de cada cebador (Forward - Reverse), Número de Accesión GenBank, (Ta) temperatura de anillamiento, repetición de motivos, intervalo y tamaño esperado de los fragmentos amplificados de acuerdo a A. chirimolla en Escribano et al. (2004) y Escribano et al. (2008), (*n.r) no registrado, (A) número de alelos.
Los productos de amplificación se llevaron a electroforesis en geles denaturantes de poliacrilamida al 6% y se observaron mediante tinción con nitrato de plata. Como control negativo se utilizó agua de PCR y como marcador de peso molecular el HyperLadderTM 50bp de Bioline.
Análisis de la información
Los resultados obtenidos se registraron en una matriz numérica binaria (1 y 0), representada por la presencia de bandas (1) o ausentes de ellas (0). Para la evaluación y análisis del poder de polimorfismo de los SSR, se calculó el contenido de información polimórfica (PIC), así: valores de PIC superiores a 0,5 indican loci altamente polimórficos, valores de PIC entre 0,25 y 0,5 se consideran medianamente informativos y valores de PIC inferiores a 0,25 se consideran poco informativos (Piñero et al., 2008). La selección de las bandas polimórficas se realizó considerando como locus polimórfico aquél en el cual la frecuencia del alelo más común fue inferior al 95%. Los análisis estadísticos para la estimación de la diversidad genética poblacional se realizaron a través del software GenAlEx6 (Peakall y Smouse, 2006). Se realizó un agrupamiento de los 102 individuos mediante el programa estadístico NTSYS 2.02i (Rohlf, 1997), y empleando el algoritmo de agrupamiento UPGMA (Unweighted Pair Group Method with Arithmetic Mean o ligamiento promedio, utilizando la media aritmética no ponderada) (Sneath y Sokal, 1973).
Resultados
Se realizó el análisis de diversidad genética para los 102 cultivares de A. muricata a partir de 16 microsatélites y considerando todas las localidades (municipios). Ocho SSR dieron como resultado patrones de banda claros y polimórficos entre las accesiones, y sugieren la presencia de más de un loci en la especie. El porcentaje de amplificación fue del 50%, lo que indica que la tasa de transportabilidad de los SSR varía entre géneros, y acorde a las distancias filogenéticas. Se encontró una tasa de migración poblacional (Nm > 1) que revela flujo genético.
Los ocho SSR detectaron 30 alelos, todos polimórficos, con un intervalo de 2 a 7 alelos por locus (promedio de 1,84 alelos por locus). En general, toda la población registró bajo número de heterocigotos. Las heterocigosidades observadas y esperadas fluctuaron entre 0,276 a 0,584 con un promedio de 0,449; y de 0,322 a 0,373 con un promedio de 0,342, respectivamente (Tabla 2). Estos valores de He son contrastantes con lo reportado por Talamantes et al. (2020) en A. muricata (promedio He= 0,164, fluctuaciones entre 0,175 y 0,144). Promedios superiores de heterocigosidades han sido reportados en A. cherimolla (Ho= 0,40 y He= 0,57) (Escribano et al., 2008). En A. cherimolla (Ho= 0,43 y He= 0,49) por Escribano et al. (2004). Y en A. squamosa (He= 0,47) por Salazar et al. (2010).
Tabla 2 Estadísticos descriptivos Na, Ne, Ho, He y F para A. muricata usando SSR.
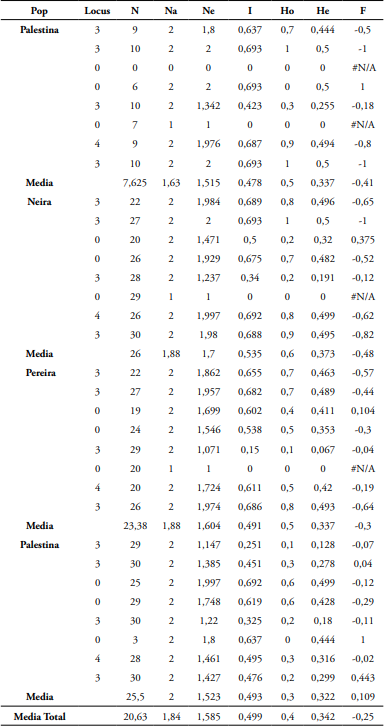
Nota. (N) # de alelos, (Na) # de alelos diferentes, (Ne) alelos efectivos, (I) índice de información, (Ho, He) heterocigosidad observada y esperada, (F) índice de fijación.
LMCH4 fue el SSR que registró el mayor número de alelos (7). Y LMCH5, el menor (2). Pereira fue la población que registró la mayor frecuencia alélica con el microsatélite LMCH4= 0,771. Mientras que Palestina la menor (0,5). El mayor porcentaje de loci polimórficos (P) estuvo representado para Palestina (100%), con una media poblacional de 87,5%. De igual manera esta población fue la única que registró alelos únicos (0,125) (Tabla 3). Este valor de P es mayor a los valores reportados para A. muricata en México por Talamantes et al. (2020) (76,67%).
Tabla 3 Estimativos alélicos entre las poblaciones de A. muricata usando SSR.
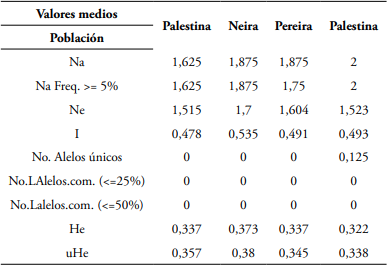
Nota. Valores medios, poblaciones, (Na) # de alelos diferentes, (Na Freq.) frecuencia del # de alelos diferentes, (Ne) alelos efectivos, (I) índice de información, (No. alelos únicos) # de alelos únicos, (No. Loci con alelos comunes) # de alelos comunes, heterocigosidad esperada, (uHe) heterocigosidad esperada imparcial.
El análisis poblacional por pares de Nei reveló una mayor distancia genética y una menor identidad genética entre los cultivares procedentes de Palestina (Nei D= 0,249 y Nei I= 0,780) (Tabla 4).
Tabla 4 Distancia genética e identidad genética poblacional de Nei por pares.
| Pop1 | Pop2 | Nei D | Nei I |
|---|---|---|---|
| Palestina | Neira | 0,081 | 0,922 |
| Palestina | Pereira | 0,115 | 0,891 |
| Neira | Pereira | 0,024 | 0,976 |
| Palestina | Palestina | 0,249 | 0,78 |
| Neira | Palestina | 0,155 | 0,857 |
| Pereira | Palestina | 0,075 | 0,927 |
Nota. (Nei D) distancia genética de Nei, (Nei I) identidad genética de Nei.
El análisis AMOVA registró la mayor variación entre los individuos (74%), en contraste con la variación entre las poblaciones (15%). Una variación baja fue registrada dentro de los individuos (11%). La varianza registrada entre las poblaciones es similar a la registrada por Talamantes et al. (2020) (12%).
El PIC (0,875) se consideró con alto poder informativo (Wright, 1951). El coeficiente Fst poblacional fue de 0,168, sugiriendo una moderada diferenciación genética en la población (Tabla 5). Wright (1951) clasifica la diferenciación genética de acuerdo con el Fst de la siguiente forma: 0-0,05 = poca diferenciación genética, 0,05-0,15 = moderada diferenciación genética, 0,15-0,25 = grande y > 0,25 = muy grande. El Nm para la estimación del flujo genético en la población, fue de 3,534 (Tabla 5).
Tabla 5 Estadísticos poblacionales F y estimativos Nm en A. muricata usando SSR.
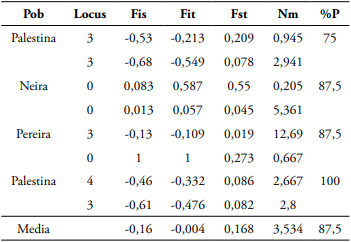
Nota. (Fis) coeficiente de endogamia de un individuo (I) respecto de la subpoblación (S), (Fit) coeficiente de endogamia de un individuo (I) respecto a la población total, (Fst) efecto de las subpoblaciones (S) comparado al total de la población (T), (Nm) tasa efectiva de migración, (%P) porcentaje de Loci polimórficos.
El valor de la He fue similar entre las poblaciones. El valor promedio fue de 0,342. Inferior a lo reportado en A. cherimola (He= 0,50 y 0,57) (Escribano et al., 2007; 2008) respectivamente. Y en A. squamosa (He= 0,47), por Salazar et al. (2010). Otros valores notablemente inferiores a los hallados en la presente investigación, en A. muricata (He= 0,164) son reportados por Talamantes et al. (2020) en México, usando marcadores moleculares SRAP. El Rst calculado para toda la población fue de 0,146, con una fluctuación entre 0,101 y 0,404 (Tabla 6).
Tabla 6 Estadísticos poblacionales RST en A. muricata usando SSR.

Nota. (Pop1) población 1, (Pop2) población 2, (Rst) estadístico R, (LinRst) estadístico R linear. Valores poblacionales Rst y LinRst por pares.
Los agrupamientos e índices de similitud obtenidos a partir de las bandas polimórficas se aprecian en el dendrograma (Figura 2). Se observan tres grupos principales que comparten aproximadamente un 57% de su genoma, según el coeficiente de Dice. Un primer grupo (G1), está constituido por individuos procedentes de Palestina, Neira y Pereira, y presenta una marcada tendencia a generar subgrupos entre las mismas procedencias.
G2 estuvo constituido por individuos procedentes de Palestina y Pereira, con una marcada tendencia de re-agrupamiento entre los materiales procedentes de Palestina.
G3 presentó las menores similitudes entre los materiales. La agrupación de ocho materiales procedentes de Palestina y Pereira reveló un 54% de similitud con las agrupaciones de G2 y G3. El individuo 71, procedente de Pereira, registró un 45% de similitud con las agrupaciones de G2.
Aunque G1 agrupó el mayor número de materiales de las tres localidades (Palestina, Pereira y Neira), es el grupo con menores distancias genéticas.
Sintetizando, Tabla 7 y Figura 1:
Tabla 7 Resumen del análisis AMOVA.
| Source | df | SS | MS | Est. Var. | % |
|---|---|---|---|---|---|
| Among Pops | 3 | 495,030 | 165,010 | 2,778 | 15% |
| Among Indiv | 97 | 2952,515 | 30,438 | 14,162 | 74% |
| Within Indiv | 101 | 213,500 | 2,114 | 2,114 | 11% |
| Total | 3661,045 | 19,054 | 100% |
Nota. La tabla muestra el resumen del análisis de varianza AMOVA.
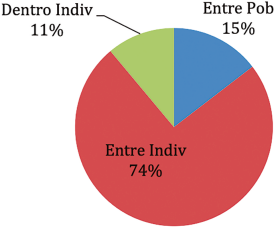
Nota. La figura muestra los porcentajes de diferenciación de la variabilidad genética entre poblaciones e individuos (Peakall y Smouse, 2006).
Figura 1 Porcentajes del análisis de varianza molecular.
Discusión
Ocho marcadores SSR polimórficos presentaron un PIC de 0,875, con ellos fue posible la discriminación efectiva entre los individuos y las agrupaciones generadas a través del coeficiente de Dice (Figura 2). En concordancia con autores como Escribano et al. (2004) y Escribano et al. (2008), quienes emplearon estos mismos SSR en la evaluación de la diversidad genética de otras especies del género Annona.
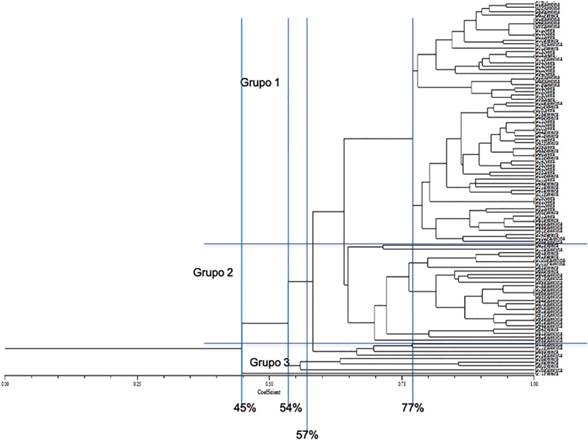
Nota. La figura muestra los agrupamientos e índices de similitud de 102 individuos de A. muricata obtenidos mediante el coeficiente de Dice (Rohlf, 1997).
Figura 2 Dendrograma de 102 individuos de A. muricata, obtenido con el índice de similitud de Dice a partir de ocho marcadores moleculares SSR.
El estadístico Nm para la estimación del flujo genético (3,534) reveló que en la población de A. muricata es posible superar los efectos de la deriva génica, previniendo la diferenciación local, lo que soporta los resultados del Fst. Según Wright (1951), una tasa de migración >1 en cada generación, es suficiente para contrarrestar la diferenciación genética debida a deriva génica.
El valor de P (87,5%) es mayor a los valores reportados para A. muricata en México por Talamantes et al. (2020) (76,67%). Estos datos ratifican un mayor poder de discriminación de los SSR en estos análisis de diversidad genética, en comparación con los marcadores SRAP empleados por los autores. Seguramente, la naturaleza codominante de los SSR prima sobre la detección de secuencias codificantes detectadas por los SRAP, pese a que sean más conservadas entre los cultivares como lo es para A. muricata.
El coeficiente de diferenciación genética (Fst= 0,168) obtenido en el presente estudio, reveló una moderada diferenciación genética en la población, debido probablemente a la cercanía geográfica de las localidades muestreadas. Dicha cercanía podría contribuir a favorecer la polinización cruzada entre los individuos sin limitaciones geográficas, ejerciendo quizás una presión evolutiva sobre las condiciones propias de los factores intrínsecos de las estructuras reproductivas de la especie que han limitado el flujo de genes a través de la polinización cruzada. Esto permite comprender que el flujo genético (Nm > 1) obtenido en este estudio, estaría representado entre los individuos a través de su contribución con la variabilidad genética pese al bajo número de heterocigotos registrado para la población (He= 0,449), atribuido probablemente a la presión de selección a través del tiempo de las semillas empleadas para la obtención de los portainjertos francos, nativos y adaptados a zonas de producción durante largos periodos, como de las copas usadas en la injertación.
Este hecho estaría siendo corroborado con los resultados obtenidos a partir del coeficiente de diferenciación poblacional (Rst= 0,146), y por la diferenciación de la variabilidad genética intra e interespecífica de la población generada por el estadístico AMOVA.
Los datos arrojados por el análisis poblacional por pares de Nei reportan una mayor distancia genética y una menor identidad genética entre los cultivares procedentes de Palestina (Nei D= 0,249 y Nei I= 0,780) (Tabla 4), probablemente ha sido derivado del tipo de material de siembra empleado, semillas, y a la interacción genotipo ambiente de las copas utilizadas para el establecimiento del material en esta región, lo que podría estar contribuyendo a una expresión que favorece las condiciones propias de las estructuras reproductivas que limitan la polinización cruzada.
Con base en los resultados obtenidos a partir de los estadísticos genéticos y a las agrupaciones generadas a través del coeficiente de similaridad de Dice, es posible obtener una primera representación de la variabilidad genética de la guanábana en Colombia, en la que se destaca un importante esfuerzo en la evolución de sus sistemas reproductivos, principalmente en aquellos factores externos que favorecen la segregación y la efectividad en la generación de nuevos y diversos embriones, pese a la continua selección de materiales con caracteres morfo-agronómicos de interés, que ponen al límite la diversidad de la especie y en riesgo su supervivencia ante escenarios de cambio climático, nuevas plagas y enfermedades.
El intercambio de semillas entre productores de las regiones más cercanas, probablemente ha favorecido la generación de nuevas combinaciones alélicas debido a la recombinación genética durante el proceso de formación gamética. Hecho corroborado con 5, 6 y 7 alelos obtenidos en el presente estudio, destacando a Pereira como la población que registró el mayor número de alelos (0,771), y a Palestina con el registro de alelos únicos (0,125).
Así mismo, este sistema tradicional de propagación de la especie, semillas, refleja la variabilidad que existe dentro de cada población y su grado de aislamiento. Por ejemplo, para nuestro caso, el análisis AMOVA registró la mayor variación entre los individuos (74%), y la menor dentro de los individuos (11%). Estas actividades podrían incrementar a largo plazo el acervo genético de las plantaciones y de esta manera disminuir la vulnerabilidad de estas ante las amenazas ambientales actuales.
Si bien es clara la necesaria inclusión de otros cultivares provenientes de otras regiones del país, los resultados preliminares del presente estudio permiten sospechar con esta dinámica de reproducción sexual entre los individuos cercanos y las distancias genéticas que reflejan similitudes de sus genomas hasta de un 77% (Figura 2), que sería poco útil la selección de materiales con fines de introgresión genética. Sin embargo, en un inicio podría fijarse la atención en una selección inicial sobre los materiales más distantes genéticamente que por consiguiente reportaron menores similitudes en sus agrupaciones, y sobre ellos garantizar una estabilidad genética en el tiempo. Estos genotipos son los correspondientes al grupo 3, y el genotipo 71, que fue el material con la mayor distancia genética. En ambos casos son materiales principalmente provenientes de Risaralda, seleccionados por el productor sobre la base de sus características sobresalientes de campo sin considerar la procedencia.
El conocimiento preliminar de la diversidad y estructura genética en guanábana (A. muricata) para Colombia, es esencial para identificar con mayor precisión los riesgos actuales y planificar medidas de protección, selección y aprovechamiento sostenible del cultivar. Los resultados de este estudio muestran niveles bajos de diversidad genética para A. muricata, similar a lo ocurrido en el estudio de Talamantes et al. (2020). Seguramente, debido a que la población analizada correspondió a material para producción y a las características propias del material de origen, discutidas previamente. Estos valores fueron contrastantes con mayores diversidades genéticas reportadas para otras anonas (Escribano et al., 2007; Escribano et al., 2008; Salazar et al., 2010).
En aras de mitigar este panorama en la especie, e impulsar la conservación e incremento de los niveles de diversidad genética, una estrategia inicial podría basarse en la identificación y selección de materiales individuales portadores de riqueza alélica. Un acercamiento preliminar a la selección de materiales promisorios a partir de estos genotipos del grupo 3, y el genotipo 71, sería oportuna como contribución inicial de lo que sería la base de un programa de mejoramiento genético para el cultivo de la especie en Colombia (Agronet, 2022), acompañado de una evaluación de nuevos materiales procedentes de otros lugares del país con características morfo-agronómicas de interés y que respondan mejor a las principales limitantes del cultivo.
Conclusiones
Fue posible tener un primer reporte de la representación de la diversidad genética de A. muricata en Colombia, a través del uso de marcadores SSR que lograron discriminar a los 102 materiales evaluados, y elucidar la situación actual de los cultivares analizados.
Con los valores obtenidos a partir de los estadísticos empleados, se vislumbra un primer acercamiento en la identificación de materiales promisorios basado en la selección individual de materiales portadores de riqueza alélica, y en aquellos materiales que reportaron una menor similitud en las agrupaciones.














