Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Colombiana de Biotecnología
versão impressa ISSN 0123-3475
Rev. colomb. biotecnol v.13 n.1 Bogotá jan./jun. 2011
ARTÍCULO DE INVESTIGACIÓN
Estudio del polimorfismo genético de las células de la médula ósea y del sistema nervioso central de ratas mediante la técnica de RAPD
Study of genetic polymorphism of the bone marrow and the central nervous system of rats cells by RAPD technique
Título corto: RAPD a células de ratas.
Esteban Alberti 1 , Jorge Fraga 2 , Rocío García 1 , Elizabeth Hernández 1 , Karelys de la Cuetara 1 , Lázara Castillo 1 , Teresa Serrano 1.
1 Centro Internacional de Restauración Neurológica, La Habana, Cuba. alberti@neuro.ciren.cu
2 Instituto de Medicina Tropical "Pedro Kourí", La Habana, Cuba.
Recibido: enero 09 de 2010 Aprobado: mayo 23 de 2011
Resumen
Los modelos experimentales en rata han sido de gran utilidad en las evaluaciones terapéuticas o de reemplazo de células en enfermedades neurodegenerativas. Se ha comprobado que las células de la médula ósea (CMO) de ratas pueden diferenciarse en células que no forman parte de sus linajes normales. Hay evidencias de estos procesos de trans-diferenciación, pero aún no se conocen los mecanismos moleculares que activan estos procesos. El propósito de nuestro trabajo fue estudiar el polimorfismo genético del ADN de los tipos celulares que conforman las CMO y las células del sistema nervioso central (SNC), estríatales y de la corteza de ratas mediante la técnica de RAPD. Las CMO, las células mononucleares (CMMO), las células estromales (CEMO) y las del SNC fueron obtenidas de ratas, y su ADN genómico fue purificado y amplificado mediante la técnica de RAPD, utilizando 15 cebadores al azar. Se construyó un dendograma de las bandas de amplificación generadas utilizando el método de UPGMA. Las células estudiadas según el análisis del RAPD quedaron en 2 grupos bien definidos, pudiéndose diferenciar las CEMO del resto de las células estudiadas. Los cebadores OPA-6, 7 y 12, mostraron el polimorfismo genético de los linajes de células estudiadas. Mediante la técnica de RAPD se demostró la variabilidad genética entre las CEMO y las CMMO, células estriadas y de corteza que mostraron una homogeneidad genética, proponiéndose marcadores específicos de RAPD para cada grupo de células. Este es el primer estudio del polimorfismo genético de las CMO y del SNC de ratas.
Palabras clave: células de la médula ósea (CMO), células del sistema nervioso central, polimorfismo genético, RAPD.
Abstract
Experimental models have been of grate usefulness in the therapeutic or replacement cells in neurodegenerative diseases. It has been demonstrated that bone marrow cells (BMC), can be difefferentiated in cells that do not form part of their normal lineage. There is evidence of these trans-differentiation processes in these cells, but nevertheless, molecular mechanisms that activate these differentiation process still not known. The purpose of our work was to study the genetic polymorphism of those cellular types; that conform the rat bone marrow cells (BMC) as well as those of the central nervous system (CNS), striatum cells and cortex ones, trough RAPD technique. BM, mononuclear cells (BMMC), estromal cells (BMSC) and the CNS cells were obtained from rats and genomic ADN was purified and amplified through RAPD technique, using 15 random primers. A dendogram was constructed according to UPGMA method of the amplifying RAPD bands. Studied cells as- according to the RAPD analysis- were grouped into 2 well- defined groups, as CEMO coud be differentiated from the rest of studied cells. OPA-6, 7 and 12 primers showed the genetic polymorphism of the studied lineages cells. Also will be proposed specific RAPD genetic markers. Through RAPD technique permitted the genetic variability was demonstrated betwen BMEC and BMMC of striated cells and of cortex, which demonstratd a genetic homogeneity through RAPD technique so specific genetic markers of RAPD were thus propose for each group of cells. These constitute the first study on genetic polymorphism of BMC and CNS.
Key words: Bone marrow cells, central nervous system, genetic polymorphism, RAPD.
Introducción
Las células de la médula ósea (CMO), células mononucleares (CMMO) y células estromales (CEMO) son de gran interés por su posible uso terapéutico en enfermedades neurodegenerativas. Se ha comprobado que estas células pueden diferenciarse, tanto in vitro como in vivo, en células que no forman parte de sus linajes normales: músculo esquelético y cardíaco, hepatocitos, glias y neuronas, por lo que son consideradas un candidato prometedor para la terapia celular (Feron, 2007; Hernández et al., 2007; Rosser et al., 2007; Hess y Borlongan, 2008).
Las CMO son una fuente importante de células madres que pueden ser utilizadas con un alto grado de confiabilidad para el trasplante celular en aquellas enfermedades donde se ha demostrado que pueden jugar un papel fundamental en la regeneración del tejido dañado (Brazelton et al., 2000; Feron, 2007; Rosser et al., 2007). Además de las células propias del Sistema Nervioso Central (SNC), las CMO constituyen una fuente alternativa para ser empleadas en el trasplante de células en pacientes con enfermedades neurológicas (Feron, 2007; Rosser et al., 2007). El trasplante de CMO ha sido utilizado de manera exitosa para reducir el déficit motor que aparece como secuela del daño neuronal (Zhao et al., 2002) en animales que han sufrido un daño cerebral traumático y en isquemia estriatal (Brazelton et al., 2000; Mezey et al., 2000; Mahmood et al., 2001; Feron, 2007). Más recientemente, se ha utilizado el trasplante de CMO autólogas para revertir el daño provocado por la pérdida de células gabaérgicas, así como en otros tipos de células (Weissman et al., 2001; Alberti et al., 2005).
Se ha demostrado la plasticidad de las poblaciones de células madre de la médula ósea. Esta propiedad se refiere a la capacidad que presenta una célula madre adulta de generar una célula especializada de un tejido diferente al que le dio origen (Brazelton et al., 2000). Así, se ha comprobado que las células hematopoyéticas de la médula ósea son capaces de diferenciarse como microglía y macroglía, una vez trasplantadas en el cerebro de ratones adultos (Mezey et al., 2000; Zhao et al., 2002). Las CMMO, como fuente de células madre adultas-hematopoyéticas y estromales, podrían ser útiles en el tratamiento de enfermedades neurodegenerativas. Estas células son de fácil obtención, y en los seres humanos podrían utilizarse como trasplante autólogo, lo que evitaría los procesos de rechazo inmunológico (Brazelton et al., 2000; Mezey et al., 2000; Mahmood et al., 2001; Zhao et al., 2002).
Para el estudio de los mecanismos relacionados con la aparición de alguna enfermedad neurodegenerativa (Enfermedad de Parkinson, Enfermedad de Huntington, Enfermedad de Alzheimer, entre otras), y el desarrollo de métodos terapéuticos o de reemplazo de células, se ha hecho necesaria la introducción de modelos experimentales en rata que mimeticen estas enfermedades, dado que el cerebro de estos animales, por características estructurales y fisiológicas, es muy parecido al cerebro humano (Alberti et al., 2005; 2009).
En la actualidad existen métodos moleculares que permiten complementar los estudios inmunohistoquímicos e inmunofenotípicos sistemáticos tradicionales para hacer una caracterización genética de estas células (Alberti et al., 2009). Sin embargo, aún no se ha estudiado el polimorfismo genético en el ADN genómico de los tipos celulares que componen las CMO (CMMO y CEMO) y las células embrionarias del cerebro (corteza y estriado) de ratas. Estos tipos de estudio son de gran importancia, ya que al conocer las características moleculares de estas células se aportan datos de interés relacionados con su estado de diferenciación y su potencialidad terapéutica como sustitutas en los trasplantes en enfermedades neurodegenerativas, lo que nos permitiría hacer estudios más profundos en las células humanas (Alberti et al., 2005; 2009; Serrano et al., 2005).
La técnica del ADN polimórfico amplificado al azar (RAPD, del inglés Random Amplified Polymorphic DNA) es una metodología que permite hacer estudios de este tipo en las células. Los fragmentos polimórficos en el ADN proveen un método muy eficaz de obtener marcadores genéticos aplicables a organismos diferentes y capaces de detectar diferencias entre individuos que muestran una relación genética estrecha (Welsh y Mc Clelland, 1990; Williams et al., 1990; Cushwa et al., 1996). Mediante esta técnica se determina el polimorfismo genético debido a cambios de bases en sitios de unión del cebador al molde, inserciones y deleciones del genoma completo, y no de un fragmento de gen, por tanto, el análisis del polimorfismo genético que representa es más completo (Williams et al., 1990; Bardakci, 2001).
Se ha demostrado que la información provista por los polimorfos en la RAPD es consistente con aquella obtenida por medio de otras técnicas moleculares que se usan para detectar el polimorfismo genético como las del PCR-Polimorfismo de las longitudes de los fragmentos de restricción (PCR-RFLP, del inglés Restriction Fragment Lenght Polymorphism), Polimorfismo de los fragmentos de amplificación (AFLP, del inglés Amplified Fragment Lenght Polymorphism), y microsatélites, cada una con sus ventajas y diferencias (Welsh y Mc Clelland, 1990; Williams et al., 1990; Cushwa et al., 1996; Savelkoul et al., 1999; Scott y Straus, 2000; Bardakci, 2001).
Todas estas propiedades hacen de la RAPD una técnica muy atractiva para estudios de variabilidad genética de especies animales y vegetales (Scott y Straus, 2001), de ahí que se proponga para evaluar el polimorfismo genético de células de ratas con diferentes linajes.
El presente trabajo tuvo como objetivo estudiar el polimorfismo genético de los distintos linajes de células (CMO, CMMO y CEMO) de ratas, mediante la técnica de RAPD, proponiéndose marcadores de RAPD específicos para las células estudiadas.
Materiales y métodos
Animales de laboratorio
Se utilizaron 20 ratas de la línea isogénica Sprague Dawley(SD), 10 ratas machos y 10 ratas hembras en estado de gestación (17 días), con un peso corporal entre 250 y 300 g (Cenpalab, La Habana, Cuba). Se distribuyeron 5 animales por jaula, con acceso libre al agua y al alimento, y con ciclos de luz / oscuridad de 12 h. Las ratas fueron sacrificadas en diferentes momentos del estudio, 5 ratas para la obtención de CMMO, 5 para la obtención de CEMO, 5 ratas en estado de gestación para obtener muestras de tejido del estriado y otras 5 para obtener muestras de tejido de la corteza. Se incluyeron todos los animales que no mostraron signos de infección o lesión de algún tipo en cara o extremidades, falta de pelaje u otra alteración que indicara que no era una rata sana. Todos los animales fueron manipulados según las normas del Consejo Canadiense en el Cuidado de Animales para el cuidado y uso de animales de laboratorio (Canadian Council on Animal Care, 1984; Castillo et al., 2003; Alberti et al., 2005).
Obtención de CMO
Las ratas fueron anestesiadas por vía intraperitoneal con hidrato de cloral al 7% (0,6 mg/kg de peso corporal). Se realizó un corte de la piel en las patas traseras, decolando el tejido paralelo al hueso y se les extrajeron ambos fémures. El hueso extraído se colocó en una placa de Petri con solución salina al 0,9% durante 30 min. Las CMO se obtuvieron pasando solución PBS (NaCl 8 g/L; KCl 0,2 g/L; Na2HPO4 1,09 g/L; KH2PO4 0,26 g/L, pH 7,2) estéril y a temperatura ambiente con una jeringuilla a través de las epífisis de ambos fémures (Alberti et al., 2005; 2009). Las CMO obtenidas fueron recogidas en tubos estériles y lavadas por centrifugación a 1500 rpm (Centrífuga Eppendorf 5810R Alemania, rotor F34-6-38) a 20 °C. Se usó tinción con azul tripán para verificar la viabilidad de las células (Canadian Council on Animal Care, 1984; Alberti et al., 2005; 2009).
Separación de las CMMO por gradiente de densidad
Las CMO en suspensión fueron lavadas con PBS a temperatura ambiente y centrifugadas 3 veces. Las centrifugaciones se realizaron durante 10 min a 2000 rpm a 20 °C. Posteriormente, se colocaron 2,5 ml de Ficoll-HypaqueTM-Plus (Amershan-Pharmacia Biosciences, Suecia) en un tubo de cristal graduado y se depositaron sobre el mismo, 5 ml de las células suspendidas en PBS. Se centrifugó durante 45 min a 2800 rpm a 20 °C. Se extrajo la capa de células mononucleadas succionando con una pipeta las que seguidamente fueron lavadas tres veces con PBS a temperatura ambiente y estéril. Posteriormente, el sobrenadante fue desechado y el sedimento celular fue resuspendido en PBS. La viabilidad de las células fue verificada como se describió en el apartado anterior (Canadian Council on Animal Care, 1984; Alberti et al., 2005; 2009).
Cultivo de las CEMO
Las CEMO fueron extraídas del fémur de las ratas, el que fue disecado. La médula ósea se obtuvo pasando medio de cultivo líquido (α-MEM) por uno de los extremos de la epífisis del fémur a través de una aguja de la jeringuilla de perfusión que se insertó en la cavidad medular para poder obtener el material celular. Las células no adherentes fueron removidas cuando el medio fue reemplazado por medio de cultivo fresco (α-MEM, suplementado con suero fetal bovino (SFB) 20%, L-glutamina 2mM y estreptomicina 100 mg/ml estéril). Finalmente, las células obtenidas (CEMO) fueron subcultivadas. La viabilidad celular fue verificado (Canadian Council on Animal Care, 1984; Alberti et al., 2005; 2009).
Preparación de las suspensiones celulares del estriado y corteza cerebral
Las células fueron aisladas de fetos de rata usando la metodología previamente descrita por Castillo et al. (2003) con algunas modificaciones. Brevemente, ratas en estado de gestación (17 días) fueron sacrificadas por dislocación cervical y el tejido embrionario de la corteza cerebral y el estriado fueron disecados y colectados en solución de PBS fría conteniendo glucosa al 6% (PBSg). El tejido fetal fue digerido con PBSg más tripsina al 0,1% durante 20 min a 37 oC. La suspensión de células fue obtenida por disociación mecánica con pipeta Pasteur en presencia de medio mínimo Eagle modificado por Dubecco DMEM, SFB al 10% y L-glutamina 2 mM. Las células fueron colectadas por centrifugación a 1000 rpm durante 5 min, y el precipitado celular fue resuspendido en el medio descrito anteriormente. Se usó tinción con azul tripán para verificar la viabilidad de las células.
Extracción de ADN genómico
Para la extracción del ADN genómico de las células de ratas se utilizó el método del fenol-cloroformo (Maniatis et al., 1990), en síntesis: 2x106 células colectadas se centrifugaron a 10 000 rpm por 10 min a 4 oC y el sedimento se resuspendió en 300 µl solución de lisis (Tris-HCl 50 mM pH 8,25; EDTA 25 mM, NaCl 25 mM, SDS al 1%). La mezcla se trató durante 2 h a 56 oC con 100 µg/µL de proteinasa K (Boehringer Mannheim, Alemania) y seguidamente se realizaron 2 extracciones de proteínas con igual volumen de fenol-cloroformo-alcohol isoamílico (25:24:1) (v/v/v) y cloroformo-alcohol isoamílico (24 :1) (v/v) con sus respectivas centrifugaciones a 10 000 rpm por 10 min a 4 oC. El material genético se precipitó con 2 volúmenes de etanol absoluto y 0,1 volumen de acetato de sodio 3 M pH 5,3 durante toda la noche a -20 oC. El precipitado de ácidos nucleicos que se obtuvo por centrifugación a 10000 rpm durante 20 min, se lavó con etanol al 70% y se secó a temperatura ambiente, siendo resuspendido finalmente en 50 µL de tampón tris-EDTA (TE) (Tris-HCl 1 mM; EDTA 1 mM pH 8,0). El ARN presente en la muestra se digirió con la adición de ARNasa H (10 µg/mL) (Boehringer Mannheim, Germany), incubándose durante 1 h a 37 oC. Seguidamente, se realizó la purificación del ADN empleando un volumen de cloroformo-alcohol isoamílico (24 :1) (v/v) y el sobrenadante se conservó a -20 oC.
La calidad del proceso de extracción del ADN se determinó mediante electroforesis en gel de agarosa al 0,8% en solución tampón tris borato EDTA (TBE 0,5x) (tris-borato 0,045 M; EDTA 0,001M) que contenía bromuro de etidio, un marcador de fluorescencia (0,5 mg/mL), la corrida se realizó a un voltaje constante de 150 V durante 30 min en la fuente (Pharmacia LKB Multidrive XL). La visualización se realizó mediante luz ultravioleta en un transiluminador (Macrovue 2011, LKB). La concentración de ADN se estimó espectrofotométricamente mediante la medición de su absorbancia a 260 nm (Maniatis et al., 1990).
Amplificación del ADN por la RAPD
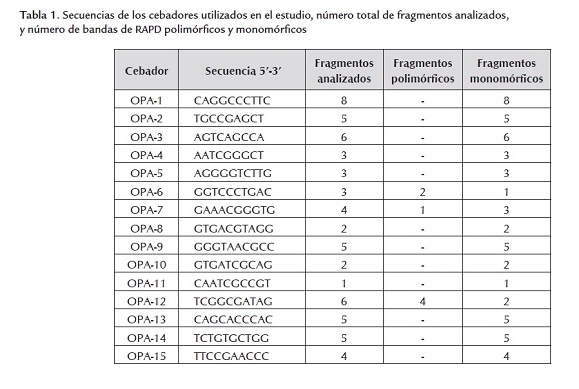
Para la amplificación del ADN de cada una de las células estudiadas se realizó una reacción de PCR con cada uno de los 15 cebadores OPA1 al OPA15 (Kit A, Operon Technologies Inc, California, EUA) cuyas secuencias se muestran en la tabla 1. Las condiciones de la reacción fueron evaluadas variando las concentraciones de los principales componentes de la reacción de PCR con el objetivo de optimizar la técnica: cebador (5, 10, 25, 50 y 75 pmol en 25 µL), Taq ADN polimerasa (0,5 U; 1 U; 1,5 U; 2,0 U; 2,5 U), MgCl2 (1, 1,5; 2; 2,5; 3; 4; 5 mM) y ADN molde (5, 10, 25, 50, 75, 100 ng). Posteriormente, la reacción de amplificación del ADN optimizada se realizó en un volumen final de 25 µL que contenían 2,5 µL de solución tampón de amplificación 10 x (Tris HCl 100 mM (pH 9,0 a 25 ºC), KCl 500 mM, Tritón 1 %) (CIGB, Cuba), 2,5 mM de MgCl2 (CIGB, Cuba), 200 µM de cada dinucleótido trifosfato (dNTP) (Amersham, EUA), 5 pmoles del cebador correspondiente, 2 U Taq ADN polimerasa (CIGB, Cuba) y 100 ng de ADN molde. Se incluyó un control negativo en cada ensayo, donde la mezcla de reacción contenía agua destilada estéril en lugar de ADN molde. La reacción de amplificación se realizó en un termociclador (Minicycler, MJ Research, EUA), con la siguiente secuencia: desnaturalización inicial a 94 oC por 5 min, seguido de 40 ciclos de tres pasos: desnaturalización a 94 oC por 1 min, hibridación a 35 oC por 1 min y extensión a 72 oC por 2 min, con una extensión final después del último ciclo a 72 oC por 15 min. Para la detección del producto de amplificación se analizaron 20 µL de cada mezcla resultante en electroforesis en gel de agarosa al 1,2%, preparado en tampón TBE 0,5x con bromuro de etidio 0,5 mg/mL. La corrida electroforética y la visualización de las bandas se realizaron tal como se explicó anteriormente.
Análisis del polimorfismo genético entre las células
Teniendo en cuenta que en los marcadores RAPD el fenotipo dominante de un locus se considera como la presencia de una banda, y el fenotipo recesivo es la ausencia de esa banda, los individuos se comparan fenotípicamente en cada locus. El análisis se realizó clasificando las bandas como presentes (1) o ausentes (0) para cada célula, a partir de lo cual se construyó una matriz binaria. El inverso del coeficiente de similitud de Jaccard (Sj, modificado por Sneath, 1957) se calculó de la manera siguiente: Sj= 1-a/(a+b+c) donde a representa el número de bandas compartidas entre las dos células, b representa el número de bandas presentes en la célula 1 y ausentes en el 2, c representa el número de bandas ausentes en la célula 1 y presentes en la 2, obteniéndose una matriz de distancia a partir de la cual, utilizando la estrategia de agrupaciones mediante el método de unión al vecino (Neighbor Joining) y de análisis no pareado de media aritmética (UPGMA del inglés, Unweighted Pair Group Method with Arihtmetical Averages), se construyeron los dendogramas, con el empleo de un paquete de programa FreeTree, versión 0.9.1.59 (Pavlicek et al., 1999) basados en los resultados obtenidos con los 15 cebadores. El análisis de bootstrap se realizó con 1000 réplicas.
Resultados
Las condiciones óptimas de cada uno de los componentes de la reacción de RAPD fueron: 2,5 mM de MgCl2, 5 pmoles del cebador, 2U Taq ADN polimerasa y 100 ng de ADN molde. Con estas cantidades se obtiene el mayor número de bandas con la mayor intensidad (datos no mostrados).
Al analizar el polimorfismo genético entre las CEMO, CMMO, células estriadas y de corteza, los 15 cebadores utilizados generaron 62 bandas de amplificación de RAPD reproducibles, dentro de las cuales 7 mostraron polimorfismo entre las células estudiadas y 55 estuvieron presentes en todas las células estudiadas. La cantidad media de fragmentos amplificados por cebador fue de 4,2 (tabla 1).
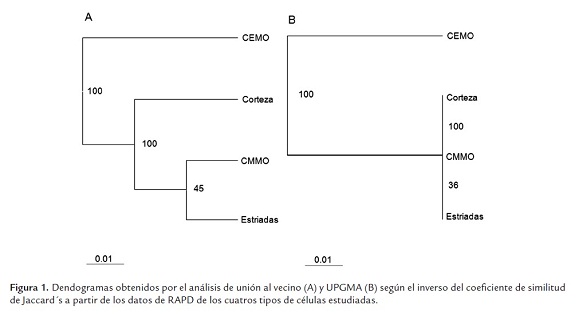
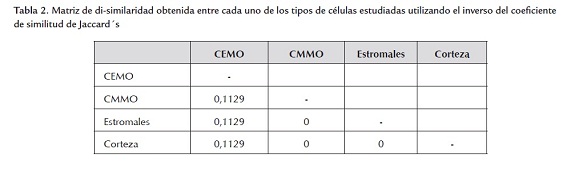
Los dendrogramas obtenidos a partir del patrón de bandas generadas por los 15 cebadores mediante la técnica de RAPD utilizando los métodos de distancia de unión al vecino y UPGMA se muestran en la figura 1. Según las distancias genéticas de las bandas amplificadas para cada tipo de células encontradas entre estas, se forman dos grupos bien definidos, por ambos métodos, que se corresponden con las CEMO (grupo I) y las células de la corteza, estriadas y CMMO (grupo II). Las CEMO se encuentran separadas del resto de las células estudiadas con una distancia genética de 0,1129 (Coeficiente de Similitud 88,7%). En la tabla 2 se muestra la distancia genética obtenida entre las células.
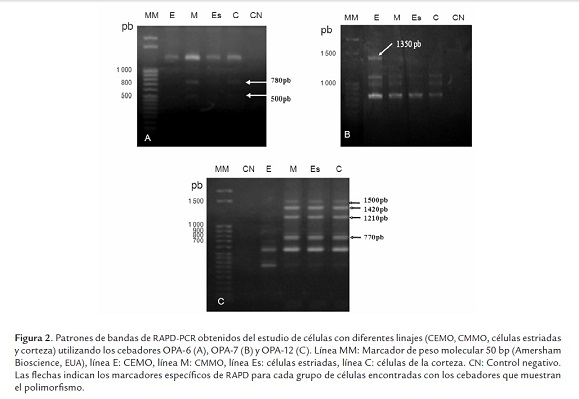
Con los cebadores OPA-6, 7 y 12 se mostró el polimorfismo genético entre las células estudiadas (figura 2), con el resto de los cebadores no se encontró variabilidad genética (datos no mostrados). Marcadores específicos de RAPD aparecen en cada grupo obtenido (figura 2). Con el cebador OPA-7, un marcador de RAPD de 1350 pb aparece en esta célula y no en el resto de las células estudiadas. De la misma forma, con el cebador OPA-6 (500 y 780 pb) y OPA 12 (770, 1210, 1420 y 1500 pb) aparecen marcadores de RAPD específicos para las CMMO, células estriadas y de corteza que no se encuentran en las CEMO. Pudiéndose diferenciar las CEMO de las CMMO, células embrionarias del estriado y de corteza.
Discusión
Hace varias décadas se consideraba que entre las células madre adultas presentes en la médula ósea solo un tipo de ellas, las hematopoyéticas y mesenquimales, conservaban la capacidad regenerativa. Sin embargo, gracias a la caracterización inmunomolecular de estas células hoy se sabe que la composición de la médula ósea es más compleja. Se ha identificado un grupo heterogéneo de células madre adultas que incluyen, además de las mencionadas con anterioridad, la denominada población lateral y las células progenitoras adultas multipotentes (Brazelton et al., 2000; García et al., 2004; Feron, 2007; Hernández et al., 2007; Roser et al., 2007; Hess y Borlongan, 2008).
La técnica del RAPD es una herramienta sumamente poderosa, la habilidad de detectar regiones altamente variables en el ADN a partir de un método rápido tiene una gran importancia en las investigaciones biomédicas, en la detección de varios tipos de daños y mutaciones en el ADN, en la caracterización de aislamientos, en el mapeo de genes, en el estudio de poblaciones, en la identificación de cepas, en estudios epidemiológicos y taxonómicos, así como en el análisis de rasgos simples y complejos del fenotipo como es el fenómeno de la resistencia (Savelkoul et al., 1999; Scott y Straus, 2000; Bardakci, 2001).
Para el análisis del polimorfismo genético entre las células estudiadas se utilizó el inverso del coeficiente de similitud de Jaccard’s para expresar de manera cuantitativa el parecido entre distintos genotipos, este es un tipo de coeficiente de asociación que mide las coincidencias y diferencias en los estados de los caracteres entre dos unidades taxonómicas operativas (OTU). Los dos coeficientes de asociación más ampliamente usados como medidas de similitud en análisis genéticos son: el índice de Nei (Nei y Li, 1979) y el índice de Jaccard (1908). En la extensa bibliografía de estudio para la caracterización genética se utilizan indistintamente ambos índices con datos provenientes de RAPD, AFLP, RFLP e isoenzimas. Los índices de Nei y de Jaccard se diferencian porque el primero le otorga un peso doble a la posibilidad de que ambas OTU presenten al carácter comparado. Por ello, se ha sugerido (Link et al., 1995; Piepho y Laidig, 1997) que es más apropiado utilizar para datos de RFLP e isoenzimas el índice de Nei, y para caracteres de RAPD y AFLP el índice de Jaccard. La razón descrita por Piepho y Laidig (1997) es que los marcadores RAPD y AFLP, al ser dominantes, determinan la presencia o ausencia de una banda en cierta posición, correspondiendo usualmente cada posición de banda a un locus. En cambio, los marcadores RFLP e isoenzimas, por ser codominantes, producen fragmentos de tamaños variables dados por diferentes alelos de un gen. Por tanto, en individuos idénticos, con marcadores codominantes, el locus se manifestará con solo una posición de banda en el gel, siendo conveniente darle un peso doble como lo expresa el índice de Nei.
En nuestro estudio los niveles de discriminación de esta técnica fueron demostrados al encontrar polimorfismo entre los tipos de células estudiadas. Además, esta variabilidad no fue encontrada solamente entre las mismas especies de células sino entre especies de células diferentes, proponiendo 3 cebadores (OPA-6, 7 y 12) que son capaces de demostrar el polimorfismo genético de las células estudiadas y cuyos patrones de RAPD permiten la identificación de los grupos de células obtenidos por el análisis del polimorfismo (CEMO y estriadas, corteza y CMMO). El análisis del polimorfismo genético de los linajes celulares estudiados permitió concluir que las células examinadas tienen una homogeneidad genética (88,7% de similitud), sin embargo, existen diferencias genéticas que permiten diferenciar a las CEMO del resto de las células estudiadas. Las CEMO se corresponden con células adherentes que apoyan la hematopoyesis y contribuyen a la formación de otros tejidos. Estos precursores no hematopoyéticos se conocen también como células madre mesenquimales (CMM o MSC, del inglés mesenchymal stem cells) (Alberti y García, 2003; Alberti et al., 2005; Hombach et al., 2008) y son células menos heterogéneas en cuanto a poblaciones de células que las CMMO que incluyen más poblaciones celulares, y que las del SNC donde podemos encontrar, además de neuronas, astrocitos y glias, células estas mucho más especializadas que las CEMO (Alberti y García, 2003; Lescaudron, 2003; García et al., 2004; Alberti et al., 2005; 2009). De ahí las diferencias genéticas encontradas, al menos para estos marcadores moleculares.
Es conocido que bajo ciertas condiciones de inducción, estas CEMO han diferenciado a neuronas, glias y astrocitos. Los cambios observados en estas células durante la diferenciación están posiblemente mediados por señales recibidas con probabilidad por células dañadas (Alberti y García, 2003; García et al., 2004; Hombach et al., 2008; Alberti et al., 2009). Por tanto, la identificación de esas señales que inducen a estos mecanismos de desdiferenciación, transdiferenciación, cambios epigenéticos o simplemente la fusión celular, son la llave para dilucidar los mecanismos moleculares que subyacen a estos fenómenos (Fernández et al., 2004; Hombach et al., 2008).
La amplificación del ADN con el cebador OPA 6 y 12 mostró seis bandas monomórficas para CMMO, células de corteza y del estriado. Estos hallazgos son interesantes si tenemos en cuenta que estas células mononucleadas pertenecen a un linaje celular diferente al de las del sistema nervioso. Los resultados del análisis del polimorfismo utilizando la técnica de RAPD sugieren que estas poblaciones celulares podrían estar relacionadas entre sí y compartir material genético similar. Este resultado nos plantea una importante interacción entre las células estudiadas y una relación filogenética cercana. Estudios filogenéticos más profundos llegarían a resultados más concretos.
Un aspecto crítico en la plasticidad de las células madres adultas es que están relacionadas a una variedad de mecanismos (desdiferenciación, transdiferenciación, fusión celular, etc.), que no las restringe a su misma línea germinal (Fernández et al., 2004; Hombach et al., 2008). Aunque muchos de estos eventos biológicos no son concluyentes, hay nuevas definiciones causadas por la fusión celular, resultando estas en la reprogramación nuclear y, por supuesto, en cambios importantes en la célula en cuanto a su actividad biológica. De acuerdo con los mecanismos mencionados anteriormente, les permite a estas células fusionarse y reprogramar nuevamente, como se ha observado con la CEMO y su fusión con tejido del hígado y neuronas, entre otras células, y que no pertenecen a su misma línea germinal (Hombach et al., 2008).
Algunos estudios han demostrado que varios factores afectan los perfiles de RAPD obtenidos, manifestándose en la presencia de bandas falsas y en la poca reproducibilidad del ensayo (Ellsworth et al., 1993, Carlton et al., 1995; Howard et al., 1996; Fraga et al., 2002), por lo que la optimización y el control interno de la técnica se hacen imprescindibles para obtener resultados confiables y una resolución consecuente entre reacciones de amplificación separadas o entre estudios (Welsh y Mc Clelland,1990; Bassam et al., 1992; Muralidharan y Wakeland, 1993; Micheli et al., 1994; Atienzar et al., 2000; Diakou y Dovas, 2001; Gutierrez et al., 2003; Pelayo et al., 2003; Fernández-Calienes et al., 2004; Fraga et al., 2005). En nuestro estudio encontramos patrones reproducibles al optimizar previamente la reacción de RAPD.
En la técnica de RAPD puede ser que fragmentos de tamaño similar no presenten homología y, sin embargo, en el análisis de los resultados se tiene en cuenta que las bandas con la misma masa molecular expresada en pares de bases e igual desplazamiento se consideraran como fragmentos idénticos, esto pudiera ser una desventaja de la técnica que se resuelve mediante el análisis de varios cebadores y un mayor número de bandas para analizar (Welsh y Mc Clelland, 1990; Bardakci, 2001; Diakou y Dovas, 2001). En nuestro estudio se analizó el polimorfismo de las células examinando 62 bandas obtenidas por la técnica de RAPD al utilizar 15 cebadores, lo que se considera un análisis suficiente para los resultados obtenidos.
La técnica del RAPD nos permitió detectar diferencias entre células genéticamente muy cercanas, generando bandas específicas que están relacionadas con el tipo de célula y que pudieran estarlo con diferentes fenotipos o características propias de estos linajes celulares. Estudios futuros permitirán dilucidar las características de los marcadores de RAPD encontrados para los grupos de células.
Conclusiones
Mediante la técnica de RAPD se demostró la variabilidad genética entre las CEMO de las CMMO, células estriadas y de corteza las cuales mostraron una homogeneidad genética mediante la técnica de RAPD. Se propusieron marcadores específicos de RAPD para cada grupo de células. Este es el primer estudio del polimorfismo genético de las CMO y del SNC de ratas. La técnica del RAPD se reporta como un método rápido, sensible y eficaz para mostrar el polimorfismo genético, aportando datos de interés sobre la composición genética, similitudes y diferencias de las células estudiadas, haciendo de esta una técnica muy atractiva para estudios polimórficos en poblaciones de células con diferente linaje, lo que reviste una gran importancia desde el punto de vista biológico y clínico, ya que al conocer las características moleculares de estas células podremos aportar datos de interés relacionados con el estadio de diferenciación de las mismas y su potencialidad en el empleo de protocolos de trabajo que incluyan diseños de tratamientos con estas células.
Referencias bibliográficas
1 Alberti, E., Los, M., García, R., Fraga, J., Serrano, T., Hernández, E. et al. 2009. Prolonged survival and expression of neural markers by Bone Marrow-Derived Stem Cells transplanted into brain lesions. Medical Science Monitor. 15: 47-52. [ Links ]
2 Alberti, E., García, R., Serrano, T., Blanco, L., Martínez, L., Mendoza, Y., Rosillo, J. C. 2005. Evaluación de la supervivencia de las células mononucleadas en un modelo de ratas con lesión estriatal por ácido quinolínico. Revista de Neurología, 40: 518-522. [ Links ]
3 Alberti, E., García, R. 2003. Células estromales de la medula ósea: una fuente terapéutica alternativa en la restauración de enfermedades degenerativas del sistema nervioso central. Revista de Neurología, 37: 752-758. [ Links ]
4 Atienzar, F., Evenden, A., Jha, A., Savva, D., Depledge, M. 2000. Optimized RAPD analysis generates high quality genomic DNA profiles at high annealing temperature. Biotechniques, 28: 52-54. [ Links ]
5 Bardakci, F. 2001. Random amplified polymorphic DNA (RAPD) markers. Turkish Journal of Biology, 25: 2185-2196. [ Links ]
6 Bassam, B. J. G., Caetano-Anolles, P. M., Gresshoff, P. M. 1992. DNA amplification fingerprinting of bacteria. Applied Microbiology and Biotechnology, 38: 70-76. [ Links ]
7 Brazelton, T. R., Fabio, M., Rossi, V., Keshet, G. L., Blau, H. M. 2000. From marrow to brain: Expression of Neuronal Phenotypes in adult mice. Science, 290: 1775-1179. [ Links ]
8 Canadian Council on Animal Care. 1984. Guide to the care and use of experimental animal. 2. [ Links ]
9 Carlton, J. M. R., Howarrd, J., Jense, J. B., Walliker, D. 1995. A rapid technique for the detection of DNA polymorphisms in Plasmodium. Experimental Parasitology, 80: 163-166. [ Links ]
10 Castillo, L., Martínez, L., Grygar, E., Hutter-Pair, B. 2003. Characterization and differentiation of EGF-responsive striatal and septal precursor cells. International Journal of Developmental Neuroscience, 21: 41-47. [ Links ]
11 Cushwa, W. T., Dodds, K. G., Crawford, A. M., Medrano, J. F. 1996. Identification and genetic mapping of random amplified polymorphic DNA (RAPD) markers to the sheep genome. Mammalian Genome, 7:580-585. [ Links ]
12 Diakou, A., Dovas, C. I. 2001. Optimization of random-amplified polymorphic DNA producing amplicons up to 8500 bp and revealing intraspecies polymorphism in Leishmania infantum isolates. Analytical Biochemistry, 288: 195-200. [ Links ]
13 Ellsworth, D. L., Rittenhouse, K. D., Honeycutt, R. L. 1993. Artifactual variation in Randomly Amplified Polymorphic DNA banding patterns. Biotechniques, 14: 214-217. [ Links ]
14 Fernández, C. I., Alberti, E., Mendoza, Y., Martínez, L., Collazo, J., Rosillo, J. C., Bauza, Y. 2004. Motor and cognitive recovery induced by Bone Marrow Stem Cells grafted to hippocampus of impaired aged rats. Functional and therapeutic considerations. Annals of the New York Academy of Sciences, 1019:48-52. [ Links ]
15 Fernández-Calienes, A., Fraga, J., Pointier, J. P., Yong, M., Sanchez, J., Coustau, C., Gutiérrez, A., Théron, A. 2004. Detection and genetic distance of resistant populations of Pseudosuccinea columella (Mollusca : Lymnaeidae) to Fasciola hepatica (Trematoda: Digenea) using RAPD markers. Acta Tropica, 92: 83-87. [ Links ]
16 Feron, F. 2007. Reparation du systeme nerveux central: les strategies actuelles de therapie cellulaire. Revue Neurolqique (Paris), 163: 323-330. [ Links ]
17 Fraga, J., Rodriguez, J., Fuentes, O., Fernández-Calienes, A., Castex, M. 2005. Optimization of Random Amplified Polymorphic DNA techniques for use in genetic studies of Cuban Triatominae. Revista do Instute de Medicina Tropical de São Paulo, 47: 295-300. [ Links ]
18 Fraga, J., Rojas, L., Sariego, I., Sarría, C.A. 2002. Optimization of Random Amplified Polymorphic DNA techniques for its use in genetic studies of Trichomonas vaginalis. Infection Genetics and Evolution, 2: 73-75. [ Links ]
19 García, R., Aguiar, J., Alberti, E., De La Cuetara, K., Pavón, N. 2004. Bone Marrow Stromal Cells Produce Nerve Growth Factor and Glial Cell Line derived Neurotrophic Factors. Biochemical and Biophysical Research Communication, 316: 753-754. [ Links ]
20 Gutierrez, A., Pointier, J. P., Fraga, J., Jobet, E., Modat, S., Peréz et al. 2003. Fasciola hepatica: identification and molecular markers for resistance and susceptible Pseudosuccinea columella snail hosts. Experimental Parasitology, 105: 211-218. [ Links ]
21 Hernández, P., Cortina, L., Artaza, H., Pol, N., Lam, R. M., Dorticós, E. 2007. Autologous bone-marrow mononuclear cell implantation in patients with severe lower limb ischaemia: A comparison of using blood cell separator and Ficoll density gradient centrifugation. Atherosclerosis, 194: 52-56. [ Links ]
22 Hess, D. C., Borlongan, C. V. 2008. Stem cells and neurological diseases. Cell Proliferation. 41: 94-114. [ Links ]
23 Hombach, K. S., Panigrahi, S., Rashedi, I., Seifert, A., Alberti, E., Pocar et al. 2008. Adult stem cells and their trans-differentiation potential - perspectives and therapeutic applications. Journal of Molecular Medicine, 86: 1301-1314. [ Links ]
24 Howard, J., Carlon, J. M. R., Walliker, D., Jensen, J. B. 1996. Use of random amplified polymorphic DNA (RAPD) technique in inheritance studies of Plasmodium falciparum. Journal of Parasitology, 82: 941-946. [ Links ]
25 Jaccard, P. 1908. Nouvelles resechers sur la distribution florale. Bulletinde la Société Vaudoise des Sciences Naturelles, 44: 223-270. [ Links ]
26 Lescaudron, L. 2003 Autologous adult bone marrow stem cell transplantation in an animal model of Huntington´s Disease: Behavioral and morphological outcomes. International Journal of Neuroscience, 113: 945-956. [ Links ]
27 Link, W., Dixkens, C., Singh, M., Schwall, M., Melchinger, A. E., Jaccard, P. 1995. Genetic diversity in European an Mediterranean faba bean germplas revealed by RAPD markers. Theoretical and Applied Genetics, 90: 27-32. [ Links ]
28 Mahmood, A., Lu, D., Yi, L., Chen, J. L., Chopp, M. 2001. Intracranial bone marrow transplantation after traumatic brain injury improving functional outcome in adult rats. Journal of Neurosurgeon, 94: 589-595. [ Links ]
29 Maniatis, T., Fritsch, E. F., Sambrook, J. 1990. Molecular cloning: a laboratory manual. New York: Cold Spring Harber Laboratory Press. Cold Spring Harber. [ Links ]
30 Mezey, E., Chandross, K. J., Harta, G., Maki, R. A., McKercher, S. R. 2000. Turning blood into brain: Cells bearing neuronal antigens generated in vivo from bone marrow. Science, 290: 1779-1782. [ Links ]
31 Micheli, M. R., Bova, R., Pascale, E., DAmbrosio, E. 1994. Reproducible DNA fingerprinting with the random amplified polymorphic DNA (RAPD) method. Nucleic Acids Research, 22: 1921-1922. [ Links ]
32 Muralidharan, K., Wakeland, E. K. 1993. Concentration of primer and template qualitatively affects products in random amplified polymorphic DNA-PCR. Biotechniques, 14: 362-364. [ Links ]
33 Nei, M., Li, W. H. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proccedings of the National Academic of Science. 76: 5269-5273. [ Links ]
34 Pavlícek, A., Hrdá, S., Flegr, J. 1999. FreeTree-freeware program for construction of phylogenetic trees on the basis of distance data and for bootstrap/jackknife analysis of the trees robustness. Application in the RAPD analysis of genus Freenkelia. Folia Biologica (Prague), 45: 97-99. [ Links ]
35 Pelayo, L., Fraga, J., Núñez, F. A., Mendoza, D., Torres, D., Finlay, C. M. 2003. Genetic characterization by random amplified polymorphic DNA analysis (RAPD) of 18 isolates of Giardia lamblia obtained from day care children. Experimental Parasitology, 104:162-166. [ Links ]
36 Piepho, H. P., Laidig, F. 1997. A review of methods for cluster analysis of marker data. UPOV document BMT/47/7. UPOV, Genf. [ Links ]
37 Rosser, A. E., Zietlow, R., Dunnett, S. B. 2007. Stem cell transplantation for neurodegenerative diseases. Current Opinion in Neurology, 20: 688-692. [ Links ]
38 Savelkoul, P. H. M., Aarts, H. J. M., Haas, J., Dijkshoorn, L., Duim, B., Otsen, M. et al. 1999. Amplified Fragment length Polymorphism Analysis: the state of an art. Journal of Clinical Microbiology, 37: 3083-3091. [ Links ]
39 Scott, J. A., Straus, N. A. 2000. A review of current methods in DNA fingerprinting. En Samson, R. A. y J. I. Pitt (eds.). Integration of Modern Taxonomic Methods for Penicilium and Aspergillus classification. Amsterdam: Harwood Academic Publisher. [ Links ]
40 Serrano, T., Alberti, E., Lorigados, L., Díaz Armesto, I., Morales Vallejo, A. 2005. Caracterización inmunofenotípica de las células de la médula ósea. Biotecnología Aplicada, 22: 246-249. [ Links ]
41 Sneath, P. H. A. 1957. Some thoughts on bacterial classification. Journal of General Microbiology, 17: 201-226. [ Links ]
42 Weissman, I. L., Anderson, D. J., Gage, F. 2001. Stem and progenitor cells: origins, phenotypes, lineage commitments and transdifferentiation. Annual Reveview of Cell and Derevelopmental Biology, 17: 387-403. [ Links ]
43 Welsh, J., Mc Clelland, M. 1990. Fingerprinting genomes using PCR with arbitrary primers. Nucleic Acids Research,18: 7213-7218. [ Links ]
44 Williams, J. G. K., Kubelik, A. R., Livak, K. J., Rafalski, J. A., Tingey, S. V. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetics markers. Nucleic Acids Research, 18: 6531-5. [ Links ]
45 Zhao, L. R., Duan, W. M., Reyes, M., Keene, D., Verfaillie, C. M., Low, W. C. 2002. Human bone marrow stem cells exhibit neural phenotypes and ameliorate neurological deficits after grafting into the ischemia brain of rats. Experimental Neurology, 174: 11-20. [ Links ]