Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Biotecnología
Print version ISSN 0123-3475
Rev. colomb. biotecnol vol.13 no.2 Bogotá July/Dec. 2011
ARTÍCULO DE INVESTIGACIÓN
Identificación de polimorfismos en RXam2, un gen candidato de resistencia a la bacteriosis vascular de yuca
Identification of polymorphisms in RXam2 a cassava bacterial blight resistance gene candidate
Elízabeth Contreras1 , Camilo López2
1BSc. Estudiante de Maestría. Grupo Manihot Biotec. Departamento de Biología. Universidad Nacional de Colombia, sede Bogotá.
2BSc. MSc. PhD. Profesor Asociado. Grupo Manihot Biotec. Departamento de Biología. Universidad Nacional de Colombia, sede Bogotá. celopezc@unal.edu.co
Recibido: abril 07 de 2011 Aprobado: noviembre 10 de 2011
Resumen
La yuca se constituye en la base de la alimentación para más de mil millones de personas en el mundo. Una de las principales enfermedades de la yuca y que podría comprometer la seguridad alimentaria es la bacteriosis vascular ocasionada por la bacteria Xanthomonas axonopodis pv. manihotis (Xam). El gen candidato de resistencia RXam2 codifica para una proteína con dominios NBS (Nucleotide Binding Site) y LRR (Leucine Rich Repeats) y colocaliza con un QTL (Quantitative Trait Loci) que explica el 61.6% de la resistencia a la cepa CIO151 de Xam. En este trabajo se secuenció parcialmente el gen RXam2 en tres variedades de yuca: MCOL2246, TMS60444 y SG107-35 con el objetivo de tener una visión preliminar del grado de polimorfismos tipo SNP (Single Nucleotide Polymorphism) que se presenta en este gen. La región secuenciada incluye 507 pb de la región promotora y 1309 pb de la secuencia codificante. Se logró identificar 5 y 31 SNPs al interior de las variedades MCOL2246 y TMS60444, respectivamente. Al mismo tiempo el número de SNPs entre las variedades fue de 44, 34 y 23 para MCOL2246-TMS60444, TMS60444-SG107-35 y MCOL2246-SG107-35, respectivamente. El mayor número de SNPs estuvieron localizados en la región -500 a -300 pb que corresponden a un fragmento de la región promotora del gen, aunque también se identificó un importante número de polimorfismos en la región codificante. Este estudio permitirá identificar el número de polimorfismos en este gen en un mayor grupo de variedades de yuca con el fin de asociar estos polimorfismos con el fenotipo de resistencia/susceptibilidad.
Palabras clave: yuca, resistencia, polimorfismos, SNP.
Abstract
Cassava is a staple food for more than a billion people. One of the major diseases of cassava which could compromise food security is known as cassava bacterial blight, caused by the bacterium Xanthomonas axonopodis pv. manihotis (Xam). The RXam2 resistance gene candidate encodes a protein with Nucleotide Binding Site and Leucine Rich Repeats domains, and colocalizes with a QTL (Quantitative Trait Loci) that accounts for 61.6% of the resistance to the Xam strain CIO151. In this work we sequenced 1816 bp corresponding to a partial sequence of this gene in three cassava varieties with the aim of having a preliminary overview of the degree of Single Nucleotide Polymorphisms (SNPs). The sequenced regions included 507 bp of the promotor and 1309 bp of the coding sequence. It was possible to identify five and 31 intravarietal SNPs in MCOL2246 and TMS60444, respectively. In addition, the number of SNPs between varieties was 44, 34 and 23 for MCOL2246-TMS60444, TMS60444-SG107-35 and MCOL2246-SG107-35, respectively. The largest number of SNPs was located in the promoter region -500 to -300 bp. This study may help to develop alternatives to identify SNPs in a more diverse group of cassava varieties, and possibly associate them with resistance/susceptible phenotypes.
Key words: Cassava, resistance, polymorphisms, SNP.
Introducción
El sistema inmune de las plantas puede dividirse en dos niveles. El primer nivel conocido como PTI (PAMP Trigger Immunity) se basa en la capacidad de reconocimiento de PAMPs (Pathogen Associated Molecular Patterns) y depende de receptores vegetales denominados PRRs (Pathogen Recognition Receptors) (Zipfel, 2009). Este reconocimiento activa una señalización que culmina con la reprogramación de la expresión de genes y en muchos casos con una respuesta hipersensible que evita la colonización del patógeno a tejidos no infectados (Chisholm et al., 2006). Los patógenos desarrollaron proteínas efectoras que al ser inyectadas al interior de las células de la planta suprimen la PTI. Las plantas, a su vez desarrollaron las proteínas de resistencia (R) que permiten el reconocimiento de las proteínas efectoras para activar el segundo nivel de inmunidad que se conoce como ETI (Effector Triggered Immunity) (Chisholm et al., 2006). Las proteínas R identificadas en diferentes especies de plantas poseen una alta similitud estructural entre sí, a pesar de estar implicadas en el reconocimiento de patógenos tan distintos como virus, bacterias, hongos o nematodos (Jones y Dangl, 2006). El grupo de proteínas de resistencia más común presenta en la región central un dominio de unión a nucleótido (NBS, Nucleotide-Binding Site), en el extremo C-terminal un dominio de repeticiones ricas en leucinas (LRR, Leucine Rich Repeat) y en la región N-terminal ya sea un dominio de tipo “coiled-coil” (CC), o dominios con similitud a las proteínas Toll de Drosophila y a las interleukinas IL-1 de mamíferos (dominios TIR) (Hammond-Kosack y Kanyuka, 2007).
La yuca es uno de los cultivos más importantes en las regiones tropicales, subtropicales y subsaharianas del mundo; es cultivada en más de 90 países y constituye la base de la alimentación para cerca de 1000 millones de personas en el mundo (Ceballos, 2002). La bacteriosis vascular es causada por la bacteria Xanthomonas axonopodis pv. manihotis (Xam), un patógeno foliar y vascular (López et al., 2006). Las pérdidas causadas por la bacteriosis pueden alcanzar el 80 o 100% de la cosecha (López et al., 2006). Hasta el momento no se ha clonado ningún gen de resistencia en yuca a esta enfermedad aunque importantes esfuerzos se han llevado a cabo con este fin (López et al., 2003; López et al., 2007). Mediante la estrategia de genes candidatos se pudo identificar una secuencia de tipo NBS que colocaliza con un QTL que explica el 61.6% de la resistencia a la cepa CIO151 de Xam (Lopez et al., 2007). Como una manera de obtener una validación funcional de la importancia de este gen de resistencia se planea estudiar los polimorfismos presentes en este gen para asociarlos con el fenotipo resistencia/susceptibilidad en diferentes variedades de yuca.
Materiales y métodos
Material vegetal y extracción de ADN
Plantas de yuca in vitro de las variedades SG10735, MCOL2246 y TMS60444 fueron obtenidas a través del banco de germoplasma de yuca del CIAT (Centro internacional de agricultura tropical). Las plantas fueron propagadas in vitro, endurecidas en suelo y mantenidas en invernadero a 28°C/21°C (día/noche), con fotoperíodo de 12 horas luz, 12 horas oscuridad. Hojas jóvenes fueron colectadas y maceradas en nitrógeno líquido para extraer el ADN genómico de acuerdo al protocolo descrito por Dellaporta et al. (1983). El ADN genómico se empleó como molde para realizar las amplificaciones del fragmento del gen RXam2.
Amplificación, clonación y secuenciación
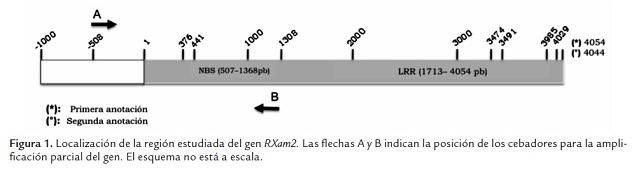
A partir de la secuencia disponible del gen RXam2 se diseñaron los cebadores RXam2-V.F y RXam2-V.R 5´-CTTAAACAGAACTCAATTTT-3´ y 5´-GGAATTCATAGTTTTTTGGA-3´, respectivamente. Los cebadores permiten amplificar 508 pb curso arriba del inicio de la transcripción del gen RXam2 asi como la región que codifica el extremo N-terminal de la proteína que incluye la región correspondiente aldominio NBS. La región de 508 pb se ha denominado como promotora aunque puede comprender una parte del 5´UTR (figura 1 ). Cada reacción de PCR se llevó a cabo en un volumen final de 50 µl que contenía 0.2 µM de cada cebador, 0.2 µM de dNTPs, 2 mM de MgSO4, 1X de buffer de PCR y una unidad de enzima DNA polimerasa (Proofreading Hifidelity, Invitrogen). Las reacciones de PCR se efectuaron en un termociclador MyCyclerTM BioRad® utilizando los siguientes ciclos de temperatura: apertura inicial a 94°C por 2 minutos, 35 ciclos de amplificación de 30 segundos de apertura a 94°C, 30 segundos de anillamiento a 51.6°C y 2 minutos de extensión a 68°C, con un ciclo final de extensión a 72°C por 5 minutos agregando en este último ciclo 1 U de polimerasa Dream taq (Fermentas), para adicionar la T en los extremos de los amplicones. El producto de la amplificación se verificó por electroforesis en gel de agarosa al 1.2% teñido con bromuro de etidio en buffer TAE 0.5X.
A partir del producto de PCR se realizó la elución con el kit Wizard SV Gel and PCR Clean-up System (Promega, Madison, WI, USA). Cada amplicón eluído se clonó en el vector pGEM-T-easy (Promega, Madison, WI, USA) siguiendo las instrucciones de la casa comercial. A continuación, cada producto de clonación fue transformado en células electrocompetentes de Escherichia coli DH5a por electroporación con el electroporador MicroPulserTM BioRad® siguiendo las condiciones de voltaje, resistencia y capacitancia recomendadas por la casa comercial. Las células se incubaron una hora a 37°C en 1ml de medio 2XYT con agitación constante. Las bacterias se sembraron en medio LB sólido con selección para ampicilina a 100 µg/µl y se incubaron a 37°C toda la noche. Se confirmó la presencia del inserto en algunos de los clones positivos, elegidos al azar, mediante PCR de colonia. A partir de los clones positivos se realizó el aislamiento del ADN plasmídico empleando el kit Wizard Plus SV Miniprep (Promega, Madison, WI, USA) utilizando 5 ml de un cultivo en medio LB líquido con ampicilina 100 µg/µl. Finalmente el producto del ADN plasmídico fue secuenciado empleando los cebadores del vector y/o del gen RXam2 en un centro de servicios certificados.
Resultados y discusión
Amplificación y secuenciación
Como parte de un estudio preliminar encaminado a identificar las regiones más polimórficas del gen RXam2, se amplificó una región parcial del gen en tres variedades distintas de yuca. Las variedades seleccionadas fueron SG10735, TMS60444 y MCOL2246, las cuales presentan un fenotipo resistente, susceptible y desconocido a la cepa de Xam CIO151, respectivamente. Se logró obtener suficiente cantidad de ADN de las tres variedades para poder continuar con el proceso de amplificación del gen RXam2.
Dado que la yuca es una planta con alta heterocigocidad y tetraploide es posible que dentro de un solo individuo se puedan presentar hasta cuatro alelos para un gen particular (Fregene et al., 1997). Con el fin de identificar todos los posibles alelos se procedió a amplificar y secuenciar directamente el producto de PCR de regiones seleccionadas en dos variedades. Sin embargo, por la naturaleza genética de la yuca, las secuencias fueron de baja calidad no pudiendo identificar verdaderos SNPs (Single Nucleotide Polymorphims) (datos no mostrados). Como alternativa se procedió a clonar el producto de PCR de cada uno de los fragmentos y secuenciar el mayor número de clones posibles obtenidos. De esta manera se lograron obtener secuencias de buena calidad para las tres variedades. Para las variedades TMS60444 y MCOL2246 se obtuvo información de secuencias para cinco clones y uno para la variedad SG10735.
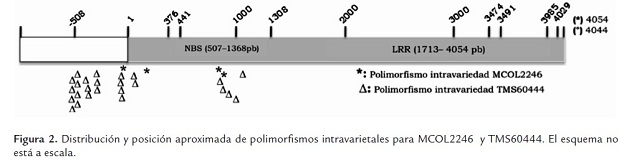
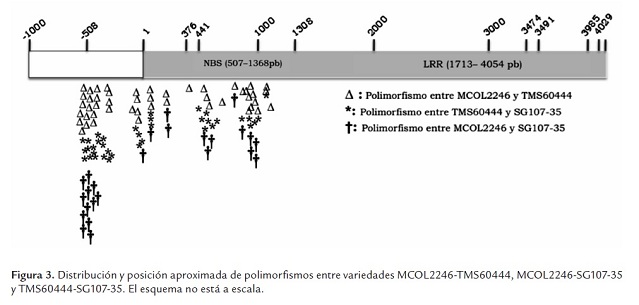
El genoma de la yuca fue recientemente liberado (octubre 2009) (www.phytozome.com) lo que permitió identificar la secuencia completa del gen RXam2. Sin embargo, recientemente se liberó una nueva versión (diciembre de 2010). Basados en los datos de la primera versión se pudo identificar que el gen RXam2 hacía parte del scaffold 10489, cubría la posición 36359-40412 y el gen denominado cassava 20457.m1. La anotación bioinformática predecía dos intrones (376-441 pb, 3474-4029 pb). Sin embargo, en la nueva versión, el gen aparece en el scaffold 07610 con el codón de inicio en la posición 91289 y el de parada en la posición 95332 y se anotó con un solo intrón (3491-3985 pb) (figura 1 , tabla 1 ). Se decidió realizar el análisis de polimorfismos bajo los dos modelos génicos predichos para calcular el porcentaje de cambios en las regiones codificantes y no codificantes.
Polimorfismos intravarietales
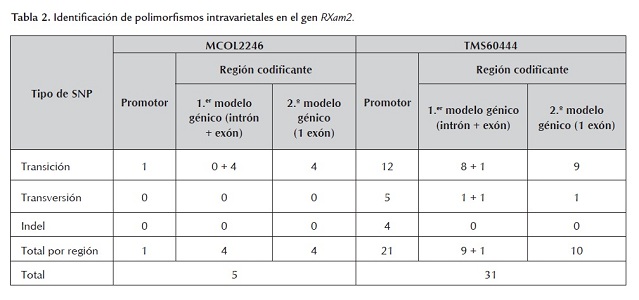
En la tabla 2 se incluyen los datos de la presencia de SNPs e inserciones/deleciones (indels) intravarietales. Para la variedad SG107-35 no se pudieron determinar los SNPs intravarietales debido a que solo se pudo obtener una secuencia de buena calidad para uno de los clones obtenidos. Varios intentos se realizaron para obtener clones de SG107-35, pero los clones resultantes no correspondían al gen, no presentaban el tamaño esperado o las secuencias fueron de baja calidad. La variedad con más SNPs intravarietales fue TMS60444, que registró 31 en comparación con 5 SNPs detectados en la variedad MCOL2246. Los datos no permiten determinar el número de alelos para cada variedad, pero el hecho de encontrar menos SNPs en la variedad MCOL2246 sugiere un nivel de ploidia menor con respecto a TMS60444, lo cual concuerda con observaciones hechas a través de SSRs que han demostrado que sobre algunas regiones del genoma de yuca se presentan dos alelos sugiriéndose un proceso de diploidización en yuca (Mba et al., 2001), el cual puede ser diferencial entre variedades de yuca. La mayoría de polimorfismos identificados en TMS60444 se ubicaron en la región promotora (tabla 2 , figura 2 ) y pocas diferencias se observaron al comparar los dos modelos génicos predichos según la primera y segunda versión del genoma de yuca (tabla 2 ).
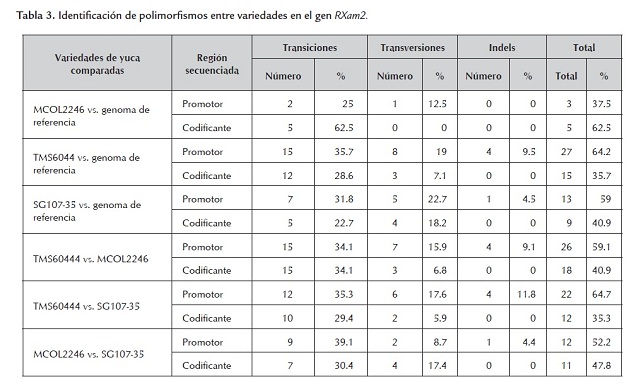
Polimorfismos entre variedades
Considerando la poca diferencia en los dos modelos génicos, para la comparación entre variedades se consideró únicamente el modelo génico predicho según la segunda versión del genoma de yuca. Cuando se compara el número de polimorfismos entre variedades se observa que en general el número es bastante similar (tabla 3 ). El número total de SNPs entre variedades fue de 44, 23 y 34 entre MCOL2246-TMS60444, MCOL2246-SG107-35 y TMS60444-SG107-35, respectivamente. Los porcentajes de transiciones en estas comparaciones fueron 68.2%, 69.5% y 64.7% mientras que los porcentajes obtenidos para transversiones fueron 22.7%, 26.1% y 23.5 % respectivamente, e indican valores muy homogéneos en los tres casos (tabla 3 ). Cuando se compara el nivel de polimorfismos de estas variedades con la secuencia del genoma de referencia, la cual es una línea autofecundada generada a partir de la variedad MCOL1505, se observa que el menor número de polimorfismos se presenta con la variedad MCOL2246, sugiriendo una relación evolutiva más cercana (tabla 3 ). Los porcentajes de transiciones (87.5%, 64.3% y 53.5%) también fueron mayores que los de transversiones (12.5%, 26.1% y 26.1% para MCOL2246, TMS6044 y SG107-35 vs. el genoma de referencia, respectivamente). Un mayor porcentaje de transiciones ha sido previamente reportado en un estudio de SNPs en un amplio grupo de genes de yuca (López et al., 2005).
En general se observa que un porcentaje mayor de SNPs, tanto intravarietales como entre variedades (37.5% - 64.7%), fueron identificados en la región promotora del gen (tabla 3 , figura 2 y 3). La región definida como promotor puede incluir el 5’UTR del gen, pero solo estudios de 5’RACE o análisis de ESTs permitirán definir precisamente el tamaño de esta región. En este estudio el número de indels fue relativamente bajo y este tipo de polimorfismo solo se identificó en la región promotora (tabla 3 ). El mayor número de polimorfismos (SNPs e indels) identificados en el promotor sugiere que esta región está sometida a menores presiones selectivas y puede mutar sin comprometer la función del gen. Sin embargo, polimorfismos en esta región pueden comprometer la expresión del gen y conferir susceptibilidad a las plantas como se ha demostrado en arroz y pimentón para los genes Xa27 y Bs3 respectivamente (Gu et al., 2005; Romer et al., 2007). Resultará valioso determinar en consecuencia el fenotipo de resistencia/susceptibilidad para estas variedades así como el perfil de expresión en respuesta a Xam.
Conclusiones
Se identificaron polimorfismos de tipo SNPs e indel en tres variedades de yuca en una región parcial del gen candidato de resistencia RXam2. Los polimorfismos pudieron ser divididos en intravarietales y entre variedades. El mayor porcentaje de polimorfismos se encuentra ubicado en la región promotora. No obstante, se identificó también un importante número de polimorfismos en la región codificante, por lo cual se hace necesario, en futuros estudios, la secuenciación de las dos regiones génicas.
Agradecimientos
Este proyecto fue financiado por la DIB (División de Investigación de la Universidad Nacional de Colombia, sede Bogotá).
Referencias bibliográficas
1 Ceballos H. 2002. La yuca en Colombia y el mundo: nuevas perspectivas para un cultivo milenario. En: CIAT (eds) La yuca en el Tercer Milenio: Sistemas modernos de producción, procesamiento, utilización y comercialización, 586. [ Links ]
2 Chisholm S., Coaker G., Day B., Staskawicz B. J. 2006. Host-microbe interactions: shaping the evolution of the plant immune response. Cell 124: 803-814. [ Links ]
3 Dellaporta S., Woody J., Hicks J. 1983. A plant DNA minipreparation: version II. Plant Molecular Biology Reporter 1: 19-21. [ Links ]
4 Fregene M., Ángel F., Gómez R., Rodríguez F., Chavarriaga P., Roca W., Tohme J., Bonierbale M. 1997. A molecular genetic map of cassava (Manihot esculenta Crantz). Theoret. Appl. Genet. 95: 431-441. [ Links ]
5 Gu K., Yang B., Tian D., Wu L., Wang D., Sreekala C., Yang F., Chu Z., Wang G. L., White F. F. et al. 2005. R gene expression induced by a type-III effector triggers disease resistance in rice. Nature 435: 1122-1125. [ Links ]
6 Hammond-Kosack K. E., Kanyuka K. 2007. Resistance genes (R Genes) in plants. In Encyclopedia of Life Sciences, J. W. Sons, ed. (London), pp. 1-21. [ Links ]
7 Jones J. D. G., Dangl J. L. 2006. The plant immune system. Nature 444: 323-329. [ Links ]
8 López C., Zuluaga A., Cooke R., Delseny M., Tohme J., Verdier V. 2003. Isolation of resistance gene candidates and characterization of a RGC cluster in cassava. Mol. Genet. Genomics. 269: 658-671. [ Links ]
9 López C., Piégu, B., Cooke R., Delseny M., Tohme J., Verdier V. 2003. Using cDNA and genomic sequences as tools to develop SNP strategies in cassava (Manihot esculenta Crantz). Theoret. Appl. Genet. 110: 425-431 [ Links ]
10 López C., Quesada-Ocampo L. M., Bohórquez A., Duque M. C., Vargas J., Tohme J. Verdier V. 2007. Mapping EST-derived SSRs and ESTs involved in resistance to bacterial blight in Manihot esculenta. Genome 50: 1078-1088. [ Links ]
11 López C., Restrepo S., Verdier V. 2006. Limitations of cassava bacterial blight: New advances. Acta Biol. Col. 11: 21-45. [ Links ]
12 Mba R. E. C., Stephenson P., Edwards K., Melzer S., Nkumbira J., Gullberg U., Apel K., Gale M., Tohme J., Fregene M. 2001. Simple sequence repeat (SSR) markers survey of the cassava (Manihot esculenta Crantz) genome: towards an SSR-based molecular genetic map of cassava. Theoret. Appl. Genet. 102: 21-31. [ Links ]
13 Romer P., Hahn S., Jordan T., StrauB T., Bonas U., Lahaye T. 2007. Plant-pathogen recognition mediated by promoter activation of the pepper Bs3 resistance gene. Science 318: 645-648. [ Links ]
14 Zipfel P. F. 2009. Complement and immune defense: from innate immunity to human diseases. Immunology letters. 126: 1-7. [ Links ]