Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Biotecnología
Print version ISSN 0123-3475
Rev. colomb. biotecnol vol.14 no.1 Bogotá Jan./June 2012
ARTÍCULO CORTO
Caracterización molecular de los genes pr1 y tpt1 en especies y variedades cubanas de tabaco (Nicotiana tabacum L.)
Molecular characterization of pr1 and tpt1 proteins in cuban tobacco species and varieties (Nicoatiana tabacum L.)
Sandra Pérez Alvarez, PhD1, Daniel Cabezas Montero, PhD1, Yoannys Dominguez Rodriguez, MSc 1, Orlando Coto Albelo, PhD 2, Humberto García Cruz, PhD 3.
1 Universidad Agraria de la Habana - C.P. 32700 - Habana - Cuba.
2 Instituto de Fruticultura Tropical - C. P. 11300 - Habana - Cuba.
3 Instituto de Investigaciones del Tabaco, San Antonio de los Baños, Habana, Cuba.
Recibido: enero 20 de 2011 Aprobado: abril 24 de 2012
Resumen
La identificación de genes nuevos relacionados con la respuesta de Nicotiana tabacum L. al estrés biótico y abiótico contribuye al mejoramiento genético del cultivo del tabaco en todo el mundo. El objetivo de este trabajo fue detectar la presencia de los genes tpt1 y pr1 en el genoma de algunas especies y variedades cubanas de tabaco. Se identificaron 265 etiquetas de secuencias expresadas (ESTs-expressed sequences tags) que nunca se habían informado con anterioridad en especies vegetales, y el gen que codifica para la proteína tumoral controlada durante la transcripción (Transcriptional Controlled Tumor Protein) (tpt1) nunca se había informado en N. tabacum, pues los estudios del mismo se han centrado en su mayoría en animales y humanos. Los resultados del microarreglo se confirmaron utilizando la RT-PCR cuantitativa en tiempo real. Los genes tpt1 y proteína relacionada con la patogénesis (pr1) se utilizaron para diseñar cebadores para la caracterización molecular de algunas especies y variedades de tabaco cubano. El gen tpt1 solamente se expresó en dos especies (N. glutinosa y N. tomentosiformis) y el pr1, después de la digestión, mostró diferentes bandas entre las variedades susceptibles y resistentes. La presencia de estos genes en una parte del germoplasma analizado constituye un paso inicial para la utilización de este conocimiento en el mejoramiento genético del tabaco.
Palabras clave: etiquetas de secuencias expresadas (ESTs), microarreglo, caracterización molecular.
Abstract
The identification of new genes related with Nicotiana tabacum L. response to biotic and abiotic stress contribute to the genetic improvement of tobacco plants around the world. The aim of this research was to identify tpt1 and pr1 genes in the genomic of some cuban tobacco varieties and species. The microarray technology has been used with ESTs for the construction of a cDNA library. 265 expressed sequences tags (ESTs) that have never been reported before in vegetables species were identified and the gene that codified for the Transcriptional Controlled Tumor Protein (tpt1), has never been reported before in N. tabacum, with the studies about this gene being focused on human and animals mostly. The microarray results were confirmed using quantitative RT- PCR. TCTP and pathogenesis-related protein 1 (PR-1) ESTs were used to design primers for the molecular characterization of some species and cuban tobacco varieties. The tpt1 gene was only expressed in two species (N. glutinosa y N. tomentosiformis) and pr1 gen, after a digestion, exhibited different bands for susceptible and resistant varieties. The detection of these genes in some species and cuban tobacco varieties represents one step to go deeply in the molecular characterization of cuban germoplasm.
Key words: expressed sequence tag (ESTs), cDNA microarray, molecular characterization.
Introducción
El tabaco es uno de los productos de mayor demanda en el mundo y es objeto de gran intercambio comercial. En Cuba, la producción tabacalera ocupa un lugar destacado en la economía por ser una de las principales fuentes de ingreso con un gran reconocimiento internacional al considerarse entre los mejores del mundo (Basulto, 2005).
El desarrollo de tecnologías moleculares permite un salto cualitativo en las investigaciones de la fisiología y la defensa de las plantas (Vidhyasekaran, 2008). El microarreglo es una novedosa tecnología molecular que posibilita el análisis de expresión con una alta sensibilidad al monitorear el genoma con un fragmento de ADN, por lo que se obtiene un mejor esquema de las interacciones simultáneas entre miles de genes y las etiquetas de secuencias expresadas (ESTs) son cadenas cortas de ADN (alrededor de 500-800 pares de bases) que forman parte de un ADNc (Adams et al., 1991). La secuenciación parcial de ADNc para generar ESTs se utiliza para la obtención rápida y eficaz de un perfil detallado de genes expresados en varios tejidos, tipos de células o fases de desarrollo. Los conocimientos sobre la biología de las plantas se enfocan en las respuestas fisiológicas y bioquímicas a nivel microscópico; sin embargo, aún es insuficiente lo que se conoce sobre los cambios en los niveles de expresión de los genes (Ranz y Machado, 2006; Gilad y Borevitz, 2006).
Desde el punto de vista genómico funcional, la combinación de las ESTs con los microarreglos, permiten la identificación de nuevos genes relacionados con las respuestas defensivas de las plantas (Lee y Hwang, 2006). El estudio de estos genes y de su expresión en el tiempo puede contribuir a la comprensión de la resistencia de la planta y es el punto de partida para su aplicación práctica en el control de enfermedades (Que et al., 2006).
Los genes tpt1 y pr1 codifican para la proteína tumoral controlada durante la traducción y la proteína relacionada con la patogénesis, respectivamente; estas proteínas intervienen en procesos como el crecimiento vegetal y la tolerancia a diferentes tipos de estrés (Pérez et al., 2006). Numerosos estudios han demostrado la presencia de estos genes en varias especies vegetales de importancia económica, por lo que este trabajo tiene como objetivo detectar la presencia de los genes tpt1 y pr1 en el genoma de algunas especies y variedades cubanas de tabaco.
Materiales y métodos
Se sembraron semillas de cuatro variedades de tabaco provenientes de la provincia de Yunnan (R. P China) en bolsas con suelo ferralítico rojo. Las variedades utilizadas fueron 'Hongda', (tabaco Negro) susceptible al TMV y 'NC82', 'YH05' y 'YUN201' (tabaco Burley) resistentes al virus (Comunicación personal Dong Haitao, 2005). Las plantas crecieron en un semillero a una temperatura de 25oC y una humedad relativa del 70% hasta los 45 días de edad. Luego, se tomaron las muestras (5g) provenientes de las hojas, tallos y raíces, y se trituraron en un mortero con nitrógeno líquido para la extracción y purificación del ácido ribonucleico total (ARN total).
La primera cadena de ADNc fue sintetizada mediante el Sistema de Síntesis RiboClone® ADNc (Promega 2005) con la pareja de cebadores oligo(dT)16 (5'-AACATATGCGGCCGCATTATGGCCTTT16-3' y 5'GATCTTCCACGCGTCGA C(T)163') y a partir de las condiciones de amplificación recomendadas en el prospecto del juego de reactivos Marathon TM . Para la síntesis de la segunda cadena de ADNc se combinaron 2 mg. mL-1 de la primera cadena de ADNc, 76 mL de H2O desionizada, 10 mg. mL-1 de solución de PCR 10x, una mezcla de 20 mM de dNTP 50x, 3 Pmol. mL-1 de cada cebador utilizado en la síntesis de la primera cadena de ADNc, 25 mM Mg2+ y 2 mg. mL-1 50 x mezcla de la enzima transcriptasa reversa AMV. Se utilizó el programa GeneAmp 9700 (Applied Biosystems, Foster City, CA) y se emplearon 5 mL del producto amplificado para la separación a través de la electroforesis en gel de agarosa al 1 % teñido con bromuro de etidio con el patrón de peso molecular 1 kb (Promega 2005).
Las secuencias se editaron con el programa SEQUENCE NAVIGATOR (Perkin Elmer/Applied Biosystems) y los programas BLASTX y BLASTN se utilizaron para buscar similitudes con las proteínas y los ácidos nucleicos en la base de datos dbEST del NCBI de los EUA. Las secuencias de los genes provenientes de los ADNc se depositaron en la base de datos Genbank de EST (www.ncbi.nlm.nih.gov/dbEST/species=tobacco).
El análisis de la expresión se realizó mediante el microarreglo de ADNc en membranas de nylon (Amersham Ltd.) según lo descrito por Pérez et al. (2006). La hibridación se realizó toda la noche a 60oC en un horno de hibridación. La validación de los resultados de la hibridación con microarreglos se realizó mediante la RT-PCR cuantitativa en tiempo real y se utilizó el método SYBR® Green (Invitrogen life technologies 2003).
Para determinar la presencia de los genes tpt1 y pr1 en las especies N. glutinosa L., N. tomentosiformis Goodsp., N. debneyi Domin., N, exigua H-M Wheeler, N. excelsior (J. M. Black) J. M. Black., N. megalosiphon van Heurck & Müll. Arg. y las variedades de N. tabacum L. Habana-2000, Corojo 99, Criollo 98, Habana 2.1.1, Burley Habana-13 (BH-13), BHmN, Corojo, San Luis 21 y Virginia Resistente -14 se sembraron semillas en casas de cultivo protegido, bajo condiciones semicontroladas, en el Instituto de Investigaciones del Tabaco (IIT). Se tomaron muestras de 5 g de hojas de cada especie y variedad en estudio y se realizó la extracción de ADN genómico mediante el método CTAB California según el protocolo de Sabater y Vilumara (1988) y se purificó siguiendo el protocolo de Ausubel (1995).
Los cebadores se diseñaron mediante el programa Vector NTI (Invitrogen) a partir de las secuencias identificadas en este trabajo y teniendo en cuenta los resultados del alineamiento, las secuencias de los cebadores son para tpt1 (5'-TGTCTGGTGGAGTGCTTCTG-3', 3'-TCATTTGCTGTGTCCCACAT-5') y para pr1 (5'-GGGAGTTTTGAGCTTTGGATGAG - 3', 3'-GACACCTAACGGGACTGCTTTC C - 5').
Los fragmentos de ADN genómico se amplificaron mediante la técnica PCR en un termociclador Tpersonal (Biometra®). El programa consistió en 5 min iniciales de desnaturalización a 92ºC, a continuación 36 ciclos de desnaturalización, alineamiento y extensión a 92oC por 30 s, 55ºC por 45 s, 72ºC por 45 s y 10 min de extensión final a 72ºC. La visualización de los resultados se realizó mediante electroforesis en gel de agarosa al 0.8% teñido con bromuro de etidio.
Resultados y discusión
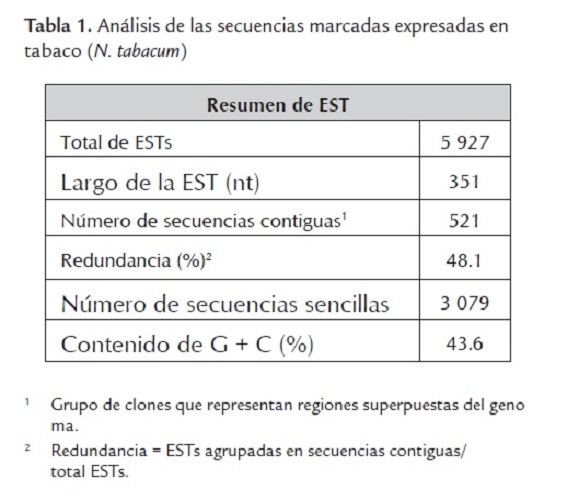
Se generaron 5927 secuencias desde ambos extremos de los clones seleccionados de la genoteca de ADNc de N. tabacum (tabla 1).
Las ESTs se utilizan para profundizar en los procesos moleculares que tienen lugar en las plantas y en otros organismos así como para la identificación de genes nuevos. Ji et al. (2006) realizaron un estudio en plantas de soya (G. soja ) para analizar posibles respuestas fisiológicas bajo condiciones de estrés; Jantasuriyarat et al. (2005) obtuvieron más información sobre los mecanismos de defensa de la planta de arroz (O. sativa) contra el hongo Magnaporthe grisea, al obtener ESTs de las cuales el 13% fueron secuencias únicas.
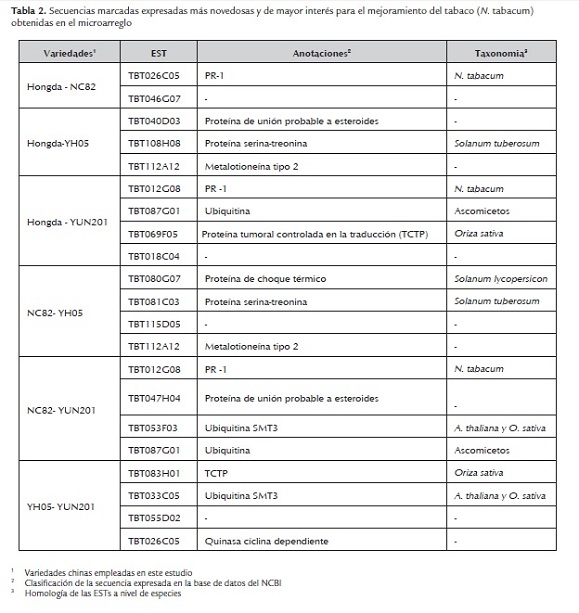
Como resultado del microarreglo construido en el presente trabajo, se obtuvieron 359 ESTs. En la tabla 2 se presentan las que resultaron ser novedosas y las que pueden tener mayor importancia o interés para el mejoramiento del tabaco.
La evaluación de la expresión a través del microarreglo de ADNc de las secuencias únicas identificadas permitió el examen simultáneo de un gran número de secuencias y el análisis de su relación con funciones biológicas.
Las 245 ESTs que resultaron ser novedosas al no encontrarse homología en la dbEST del NCBI, sugirieron la posibilidad de ser genes importantes específicos para N. tabacum. Análisis futuros de estas secuencias podrían contribuir a una mejor comprensión de la función y organización del genoma de esta planta.
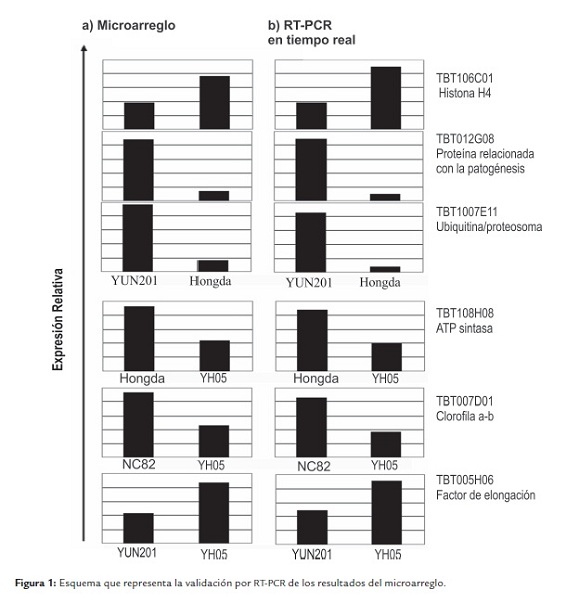
Los genes que codifican para la TCTP y la PR1 fueron seleccionados para detectar su presencia en variedades cubanas y especies de tabaco (N. tabacum), ya que en el caso del gen tpt1 no ha sido muy estudiado en plantas, y el pr1 es un gen relacionado con respuestas de defensa. La detección de estos genes en variedades y especies sería de gran utilidad para el mejoramiento genético del tabaco en Cuba. De las 359 secuencias obtenidas después del microarreglo, se seleccionaron seis para validar los resultados a través de la PCR cuantitativa en tiempo real ( figura 1). La Ub y el factor de elongación se utilizaron como controles internos por ser genes de mantenimiento y por participar en procesos celulares, como sostenimiento de la estructura celular o metabolismo primario (Radonic et al., 2004; Nicot et al., 2005). Según Vandesompele et al. (2002) deben utilizarse al menos dos o tres genes de mantenimiento como controles internos para la normalización, porque el uso de un solo gen pudiera conllevar a errores.
Como se observa en la figura 1, los valores globales de las expresiones de los genes entre el microarreglo y la cuantificación relativa mediante la amplificación con la RT-PCR cuantitativa en tiempo real, no variaron considerablemente, lo que demuestra la confiabilidad de los resultados obtenidos en el microarreglo construido.
La mayoría de las secuencias probadas mostraron un comportamiento similar entre un experimento y otro, solo la Ub y la PR-1 muestran una pequeña diferencia en la variedad Hongda y aunque la Ub se considera como un gen de mantenimiento estable en varias especies de plantas como O. sativa y A. thaliana (Czechowski et al., 2005; Jain et al., 2006) existen informes que señalan su expresión inestable en tres cultivares de O. sativa entre un experimento y otro (Caldana et al., 2007).
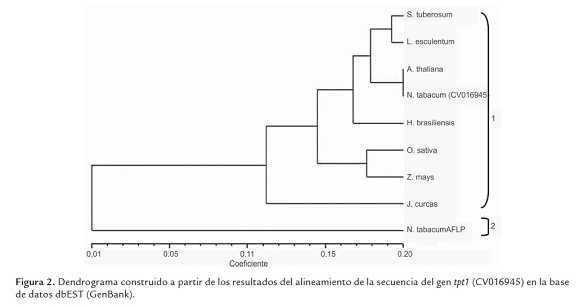
La figura 2 muestra el dendrograma construido a partir de los resultados del alineamiento de la secuencia del gen tpt1 en la base de datos dbEST (GenBank). En el árbol se definen dos grupos principales (1 y 2). El agrupamiento 1 incluye ocho secuencias identificadas en varias especies de angiospermas; estas fueron agrupadas en varios subgrupos; mientras que en el agrupamiento 2 solo se ubica una secuencia identificada en N. tabacum.
Los resultados obtenidos en el alineamiento confirmaron lo planteado por varios autores, que el gen tpt1 es un gen eucariótico altamente conservado (Arcuri et al., 2005; Thierry-Mieg y Thierry-Mieg, 2006). La secuencia en estudio (CV016945) se ubicó en uno de los subgrupos del agrupamiento 1 y resultó genéticamente similar a una secuencia homóloga identificada en A. thaliana . Esta observación resulta muy interesante, teniendo en cuenta que otras secuencias significativas fueron identificadas en especies de la familia Solanaceae (S. tuberosum y S. lycopersicon ). Estas dos secuencias son genéticamente similares entre sí, pero no lo son respecto a N. tabacum CV016945, pues aunque se encuentran en el mismo agrupamiento principal, se ubicaron en un subgrupo diferente.
Los resultados de la amplificación del gen tpt1 evidenciaron su presencia en las especies N. glutinosa y N. tomentosiformis ( figura 3). En estas especies se obtuvieron fragmentos de ~850 pb que se corresponden con el tamaño del fragmento esperado según la secuencia conocida (828 pb) a partir de la cual se diseñaron los cebadores.
Khan y Narayan (2007) plantearon que la reorganización de las secuencias de ADN, la pérdida de secuencias de bases, así como el aislamiento geográfico, fueron procesos fundamentales en el origen y diferenciación de las especies de Nicotiana . Al analizar los resultados obtenidos en la amplificación teniendo en cuenta este planteamiento, es posible que este gen haya sido eliminado del genoma de N. debneyi , N. exigua, N. excelsior y N. megalosiphon. Estas especies pertenecen a la sección Suaveolentes, la que según Chase et al. (2003) es de origen monofilético y ha evolucionado aisladamente en Australia y el Pacífico Sur.
Se demostró que la especie N. tabacum se originó por la hibridación natural de N. sylvestris y N. tomentosiformis por lo que resultó interesante que el gen tpt1 no se haya detectado en ninguna de las cuatro variedades de tabaco estudiadas.
La amplificación del gen pr1 mediante la técnica de la PCR, resultó en un fragmento de aproximadamente 600 pb en todas las variedades estudiadas; estos resultados coinciden con los planteados por Freitas et al. (2003) quienes realizaron estudios genómicos sobre estos genes pr1y no encontraron diferencias entre los distintos genotipos de trigo estudiados.
El estudio de este gen resulta importante ya que ofreció información que puede servir no solo para la especie en estudio, sino también para otras especies de plantas, sobre todo las pertenecientes a la familia Solanaceae . Con respecto a esto, algunos autores como Kyu et al. (2006), reportan que estos genes pr1 se identificaron en los genomas de diversas especies de organismos dentro de las que se incluyen además de algunas plantas, nemátodos y humanos.
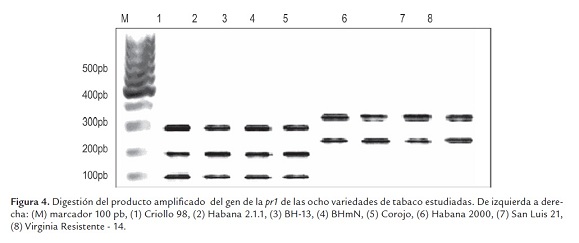
A pesar de observarse los mismos patrones de amplificación en las ocho variedades en estudio, estos presentan diversos comportamientos ante las enfermedades que atacan el cultivo de tabaco en Cuba; por tanto, se realizó a estos productos de amplificación una digestión con la enzima de restricción Hind III S-400 Matrix, para buscar polimorfismos, cuyos resultados se muestran en la figura 4.
Como se observa en la figura anterior, las dos bandas de restricción de las variedades Corojo, Habana 2000, San Luis 21, Virginia Resistente - 14 (5-8) presentan un tamaño aproximado de 350pb y 250pb, respectivamente. El producto de amplificación que fue sometido a la digestión enzimática, en el caso de estas variedades, contenía un solo sitio de restricción. Este resultado coincide con lo esperado, ya que la secuencia a partir de la cual se diseñaron los cebadores para la amplificación también contenía un solo sitio de restricción. Por otra parte, las tres bandas de restricción de las variedades Criollo 98, Habana 2.1.1, BH-13, BHmN (1-4) son de aproximadamente 100pb, 200pb y 300pb, respectivamente, mostrando este fragmento de amplificación dos sitios de restricción.
Estos resultados pudieran indicar que en las últimas variedades analizadas se amplificó un fragmento de ADN genómico que presenta una variación en su secuencia de nucleótidos, lo cual trajo como consecuencia que apareciera un nuevo sitio de restricción para la enzima Hind III. A su vez, estos resultados concuerdan con los obtenidos por Rowland et al. (2003), quienes trabajaron con Vaccinium ssp. Dichos autores encontraron polimorfismos al realizar amplificaciones a varios genes de interés y luego digerir estos fragmentos de amplificación con la enzima de restricción Alu I, obteniendo para algunos genotipos de esta especie, diferentes patrones de bandas de restricción.
Como aspecto práctico de las investigaciones de los genes pr1, a partir de su utilización se han encontrado variedades resistentes a diversas enfermedades (Edreva, 2005). Por estas razones se pudiera afirmar que es importante haber encontrado en el germoplasma analizado, la secuencia que pertenece al gen que codifica para la PR-1, aunque las variedades muestren características diferentes al ser tratados sus respectivos productos de amplificación con la enzima de restricción.
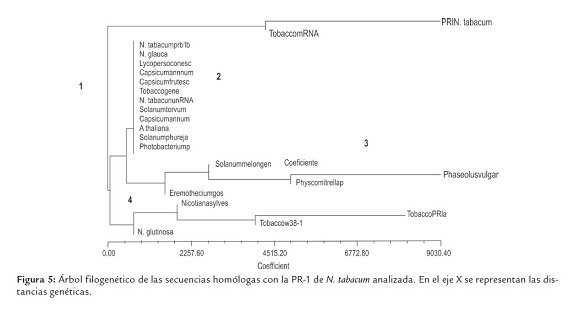
Se realizó además para esta secuencia (PR1) un análisis filogenético entre esta y secuencias afines en otros organismos, cuyos resultados se muestran en la figura 5.
Al analizar filogenéticamente la PR-1 de N. tabacum utilizada en esta investigación para el diseño de los cebadores, esta se ubicó en el primer grupo de los cuatro existentes, hallándose distante del resto de las PR-1 informadas. La mayor similitud fue con el ARNm del tabaco, aunque las distancias genéticas son muy diferentes (0.90 y 0.45, respectivamente). De manera general, se puede apreciar que todas las PR-1 son de especies dicotiledóneas coincidiendo este resultado con Liu y Xue (2006) quienes al analizar la PR-1 de Oriza sativa encontraron grupos de PR-1 para especies mono y dicotiledóneas, concluyendo que la divergencia de las proteínas PR fue subsecuente a la divergencia de las plantas mono y dicotiledóneas.
En el grupo 2 la mayoría de las PR son de plantas que pertenecen a la familia Solanaceae lo que coincide con los resultados publicados por Freitas et al. (2003) donde los dos agrupamientos observados estuvieron formados por proteínas pertenecientes a especies de la misma familia. La similitud de la PR de A. thaliana con las PR de especies de la familia Solanaceae también se informó por estos autores.
Concluisones
Se demostró la efectividad de los cebadores específicos diseñados para la amplificación de los genes tpt1 y pr1.
Se detectó la presencia del gen tpt1 en las especies N. glutinosa y N. tomentosiformis.
Se detectaron diferencias con respecto a la secuencia del gen pr1, que permitieron caracterizar ocho variedades utilizadas en el Instituto de Investigaciones del Tabaco en el programa de mejoramiento genético de este cultivo.
Bibliografía
1 Adams M., Kelley J., Gocayne J., Dubnick M., Polymero-poulos M., Xiao H., Merril R., Wu A., Olde B., Moreno R., Kerlavage A., McCombie W., Venter J.1991. Complementary DNA sequencing: expressed sequence tags and human genome project. Science. 252: 1651-1656. [ Links ]
2 Arcuri F., Papa S., Meini A., Carducci A., Romagnoli R., Bianchi L., Riparbelli MG., Sánchez J., Palmi M., Tosi P.,Cintorino M. 2005. The Translationally Controlled Tumor Protein Is a Novel Calcium Binding Protein of the Human Placenta and Regulates Calcium Handling in Trophoblast Cells. Biology of reproduction. 73:745-751. [ Links ]
3 Ausubel, FM. 1995. Protocolo de Biología Molecular. Published by Wiley, J & Sons, Inc. Third Edition, EUA, p 1-18. [ Links ]
4 Basulto O. 2005. Cuba acapara 68% del mercado mundial de tabacos Premium. Granma Internacional, 7 de Septiembre. [ Links ]
5 Caldana C., Scheible W-R., Mueller-Roeber B., Ruzicic S. 2007. A quantitative RT-PCR platform for high-throughput expression profiling of 2500 rice transcription factors. Plant Methods. 3:7. doi: 10.1186/1746-4811-3-7 [ Links ]
6 Chase N., Knapp S., Cox A., Clarkson J., Butsko Y., Joseph J., Savolainen V., Parokonny A. 2003. Molecular systematics, GISH and the origin of hybrid taxa in Nicotiana (Solanaceae). Annals of Botany. 92:107-127. [ Links ]
7 Czechowski T., Stitt M., Altmann T., Udvardi M., Scheible WR. 2005. Genome-wide identification and testing of superior reference genes for transcript normalization in Arabidopsis. Plant Physiology. 139:5-17. [ Links ]
8 Edreva A. 2005. Pathogenesis-related proteins: research progress in the last 15 years. Gen. Appl. Plant Physiology. 31(1-2):105-124. [ Links ]
9 Freitas L., Koehler-Santos P., Salzano F. 2003. Pathogenesis-related proteins in Brazilian wheat genotypes: protein induction and partial gene sequencing. Ciencia Rural. 33 (3). [ Links ]
10 Gilad Y., Borevitz J. 2006. Using DNA microarrays to study natural variation. Current Opinion in Genetics and Development. 16: 553-558. [ Links ]
11 Jain M., Nijhawan A., Tyagi A., Khurana J. 2006. Validation of housekeeping genes as internal control for studying gene expression in rice by quantitative real-time PCR. Biochemical and Biophysical Research Communications. 345:646-651. [ Links ]
12 Jantasuriyarat Ch., Gowda M., Haller K., Hatfield J., Lu G., Stahlberg E., Zhou B., Huameng L., HyRan K., Yeisoo Yu., Dean R., Rod A., Soderlund C., Guo-Liang Wang. 2005. Large-Scale Identification of Expressed Sequence Tags Involved in Rice and Rice Blast Fungus Interaction. Plant Physiology. 138(1): 105-115. [ Links ]
13 Ji W., Li Y., Li J., Dai C-H., Wang X., Bai X., Cai H., Yang L., Zhu Y-M. 2006. Generation and analysis of expressed sequence tags from NaCl-treated Glycine soja. BMC Plant Biology. 6(4). doi: 10.1186/1471-2229-6-4 [ Links ]
14 Khan M., Narayan R. 2007. Phylogenetic diversity and relationships among species of genus Nicotiana using RAPDs analyses. African Journal of Biotechnology. 6(2):148-162. [ Links ]
15 Kyu J., Lee ChS., Kook B. 2006. Activation of pepper basic PR-1 gene promoter during defense signaling to pathogen, abiotic and environmental stresses. Gene. 356:169-180. [ Links ]
16 Lee S., Hwang B. 2006. CASAR82A, a Pathogen-induced Pepper SAR8.2, Exhibits an Antifungal Activity and its Overexpression Enhances Disease Resistance and Stress Tolerance. Plant Molecular Biology. 61(1-2): 95-109. [ Links ]
17 Liu Q., Xue Q. 2006. Computational identification of novel PR-1-type genes in Oryza sativa. Journal of Genetic. 85(3):193-198. [ Links ]
18 Pérez S., Cabezas D., Haitao D. 2006. Large-scale identification of ESTs from Nicotiana tabacum by normalized cDNA library sequencing. Revista International Contributions to Tobacco Research. 22(1):114-124. [ Links ]
19 Que Y., Lin J., Zhang J., Gu Y., Ruan M., Chen T., Chen R., Xu L., Zhang M. 2006. Cloning and analysis of NBS class disease resistance gene analogous in sugarcane. Proc. Interl. Symp. On Technologies to improve Sugar Productivity in Developing Countries, Guilin, P. R of China. 623-628. [ Links ]
20 Radonic A., Thulke S., Mackay I., Landt O., Siegert W., Nitsche A. 2004. Guideline for reference gene selection for quantitative real-time PCR. Biochemistry Biophysics Communication. 313(4): 856-862. [ Links ]
21 Ranz J., Machado C. 2006. Uncovering evolutionary patterns of gene expression using microarrays. Trends in Ecology & Evolution. 21:29-37. [ Links ]
22 Rowland O., Ludwig A., Merrick C., Baillieul F., Tracy F., Durrant W. 2005. Functional analysis of Avr9 Cf-9 rapidly elicited genes identifies a protein kinase, ACIK1, that is essential for full Cf-9-dependent disease resistance in tomato. The Plant Cell. 17:295-310. [ Links ]
23 Sabater, J., Vilumara, A. 1988. Buenas Prácticas de Laboratorio (GLP): Principios básicos, Ed. Díaz de Santos S.A. [ Links ]
24 Vandesompele J., De Preter K., Pattyn F., Poppe B., Van Roy N., De Paepe A., Speleman, F. 2002. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biology. 3(7):RESEARCH0034. [ Links ]
25 Thierry-Mieg D., Thierry-Mieg J. 2006. Ace View: a comprehensive cDNA-supported gene and transcripts annotation. Genome Biology. 7(Suppl 1):S12. doi:10.1186/gb-2006-7-s1-s12. [ Links ]
26 Vidhyasekaran P. 2008. Fungal pathogenesis in plants and crops: molecular biology and host defence mechanism. 2a ed. CRC Press. p 536. [ Links ]