Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Colombiana de Biotecnología
versão impressa ISSN 0123-3475
Rev. colomb. biotecnol vol.15 no.1 Bogotá jan./jun. 2013
ARTÍCULO DE REVISIÓN
Formación de endosporas en Clostridium y su interacción con el proceso de solventogénesis
TÍTULO CORTO: Esporulación en Clostridium y su interacción con solventogénesis
Endospore formation in Clostridium and its interaction with solventogenesis
Ximena Pérez Mancilla1 y Dolly Montoya Castaño2.
1Microbióloga Industrial, M.Sc. Instituto de Biotecnología de la Universidad Nacional de Colombia. xcperezm@unal.edu.co
2Profesora Titular Universidad Nacional de Colombia. Ph.D. M.Sc. Instituto de Biotecnología de la Universidad Nacional de Colombia. dmontoyac@unal.edu.co. Autor de correspondencia
Recibido: septiembre 15 de 2012 Aprobado: junio 12 de 2013
Resumen
La solventogénesis y la esporulación son mecanismos de las células de Clostridium para resistir ambientes hostiles. Este segundo proceso ha sido estudiado utilizando como modelo lo que sucede con Bacillus, aunque se reconocen diferencias marcadas entre los dos géneros especialmente en el inicio, específicamente en los eventos por medio de los que se da la fosforilación del regulador maestro Spo0A. En la actualidad se ha avalado la teoría que afirma que tres histidin quinasas huérfanas, diferentes a las proteínas Spo0B (histidin quinasa) y Spo0F(fosfotransferasa) en Bacillus, son las encargadas de fosforilar en Clostridium de forma directa a Spo0A, que posteriormente activa la transcripción de diferentes factores sigma relacionados, de forma similar en los dos géneros bacterianos. La proteína Spo0A, perteneciente a la familia de reguladores de respuesta, se comporta como una entidad regulatoria global que tiene injerencia sobre los procesos de formación de esporas y solventes, modulando genes necesarios para que se produzcan acetona y butanol, además de genes de esporulación. El entendimiento de este proceso ha llevado a los investigadores a emplear diferentes técnicas moleculares que permitan incrementar la producción de solventes, así como eliminar la propiedad de las células de producir endosporas. Por tal razón, este escrito presenta un resumen de estas dos redes de expresión de genes, conectadas por el regulador maestro Spo0A.
Palabras clave: esporulación, solventogénesis, fosforilación, Clostridium, Spo0A.
Abstract
Solventogenesis and sporulation are mechanisms used by Clostridium cells to resist hostile environments. Sporulation has been studied using as a model what happens with Bacillus, but marked differences were recognized, particularly in the events that led the phosphorylation of the master controller Spo0A. Currently, a theory that claims that three orphan histidine kinases, different from Spo0B (histidin kinase) and spo0F (phosphotransferase) proteins in Bacillus, phosphorilate directly Spo0A in Clostridium activating transcription of different sigma factors which are similar in the two bacterial genera, has been supported. Spo0A protein, which belongs to the family of response regulators, behaves as an entity that has global regulatory interference on the processes of spore formation and solvents, modulating genes necessary to produce acetone and butanol. The understanding of this process has led researchers to employ different molecular techniques that increase the production of solvents, and remove the property of the cells to produce endospores. Reason why, this paper presents an updated summary of these two gene expression networks, connected by the master regulator spo0A.
Key words: sporulation, solventogenesis, phosphorylation, Clostridium, Spo0A
Introducción
Dentro de los mecanismos de diferenciación celular encontrados en las células procariotas, tal vez el más reconocido es la producción de endosporas, que ha sido estudiado principalmente en microorganismos Gram-positivos del género Bacillus y en menor escala en Clostridium. Los clostridios, que como característica fisiológica importante se consideran bacilos anaeróbicos, aparecieron como una clase separada de forma previa al evento de la gran oxidación (˜2.700 millones de años atrás), mientras que Bacillus, identificado como un bacilo Gram-positivo aerobio, apareció hace aproximadamente 2.300 millones de años como una clase divergente (de Hoon et al., 2010; Battistuzzi et al., 2004). La compleja red de expresión de genes que abarca la esporulación, ha sido ampliamente estudiada en este último grupo de microorganismos, y así mismo ha servido de modelo para comprender dicho sistema en otros procariotas, porque a pesar de los recientes progresos, el estudio de la genética de Clostridium permanece en un estado de desarrollo menor.
Dentro del género Clostridium existen varias especies de alto impacto en la industria de las biorefinerías, por su capacidad de producir los solventes Acetona, Butanol y Etanol (ABE) por medio de fermentación a partir de fuentes celulósicas, carbohidratos simples o glicerol (Patakova et al., 2012). El uso de ingeniería genética para mejorar este proceso fermentativo ha dedicado sus esfuerzos principalmente a incrementar la producción natural de butanol, a ampliar el rango de posibles sustratos utilizados y a incrementar la tolerancia a los solventes por parte de la célula (Papoutsakis, 2008). Se han empleado varias estrategias, dentro de las cuales está la expresión policistrónica de genes de producción de butanol en organismos heterólogos que no tienen la capacidad natural de esporular como Pseudomonas putida, que presentó un rendimiento máximo de producción de butanol a partir de glicerol puro de 122 mg/L (Nielsen et al., 2009). Otras herramientas como sistemas promotores inducibles, estrategias de supresión, microarreglos de ADN, mutagénesis por transposición, mutagénesis sitio dirigida por medio de la herramienta ClosTron II y tecnología de ARN antisentido, se han implementado hasta el momento en la búsqueda del aumento de los rendimientos de producción de solventes (Dürre, 2011a; Heap et al., 2010; Papoutsakis, 2008).
Con la tendencia actual de comprender globalmente la información que se puede obtener a partir de técnicas moleculares de punta y generar modelos predictivos de los organismos vivos a partir de la biología de sistemas, se considera fundamental entender las redes regulatorias y metabólicas para poder implementar estrategias de manipulación exitosas en microorganismos como Clostridium, que permitan incrementar su potencial biotecnológico (Lee et al., 2005). Es por esta razón que la presente revisión pretende discutir dos procesos metabólicos y fisiológicos de gran importancia en este microorganismo, la esporulación y la solventogénesis, que hasta el día de hoy se sabe están conectados principalmente por la proteína reguladora maestra Spo0A.
Esporulación de bacterias Gram -positivas
El fenómeno de la esporulación de bacterias Gram-positivas ha sido ampliamente investigado, especialmente en el género Bacillus y en menor escala en Clostridium. Estos microorganismos son capaces de producir una espora por célula altamente resistente a condiciones ambientales adversas a través de una intrincada red de interacciones ADN-proteína y proteína-proteína (De Hoon et al., 2010). La espora, que en términos morfológicos difiere significativamente de la célula vegetativa, está compuesta en la mayoría de los casos por una envoltura externa conocida como exosporium, seguida hacia el interior por las capas de proteína de la cubierta y la corteza, que está conformada por peptidoglicano. Después de esta estructura se encuentra el protoplasto de la espora, que comprende pared celular de la célula germinal, membrana y el llamado núcleo, en el que se encuentran enzimas, ribosomas, dipicolinato de calcio y ADN asociado a proteínas pequeñas ácido solubles (SASP por sus siglas en inglés) (Mitchell, 2001).
Estados morfológicos de la esporulación
Tanto los bacilos como los clostridios tienen siete estados morfológicos dentro del desarrollo de la esporulación. En la fase I el ADN se reconfigura adquiriendo forma de filamento axial, bajo un proceso reversible. Posteriormente en el segundo paso (fase II) se forma una doble membrana asimétrica, para continuar en la etapa III, donde se constituye la conocida pre-espora1. Entre las dos membranas que se encuentran en la célula se forma la corteza de la espora que está compuesta de peptidoglicano modificado (fase IV) (Atrih & Foster, 2001), después de la cual se genera la cubierta de la espora de naturaleza proteica rica en residuos de cisteína, en la etapa V(Labbé, 2005). Sin embargo, se ha encontrado que para algunos microorganismos como p.ej. Clostridium pasteurianum y C. botulinum, se forma inicialmente la cubierta y después la corteza (Labbé, 2005). Por último, en el paso VI se da el proceso de maduración de la espora con la consecuente síntesis de ácido dipicolínico, la incorporación de calcio y el desarrollo de la resistencia y se da lugar al estado VII, donde actúa una enzima lítica para liberar a la espora de la célula progenitora (Labbé, 2005). Es así como, se genera este tipo de célula diferenciada, que hasta el momento se reconoce como la forma viva más resistente.
Desarrollo de la esporulación
El inicio de la esporulación en los clostridios solventogénicos se da por eventos como el descenso dramático del pH extracelular o la exposición a oxígeno a diferencia de Bacillus donde se da por la limitación de nutrientes (Higgins & Dworkin, 2012; Paredes et al., 2005; Sauer et al., 1995). Los clostridios acumulan material de reserva similar a la amilopectina, llamado genéricamente granulosa, que servirá como fuente de carbono y energía durante la formación de la endospora. Esto induce un hinchamiento de la célula, conocido como el "estado clostridial", donde se observa la forma de cigarrillo, típica de esta forma transicional previa a la diferenciación (Dürre & Hollergschwandner 2004). Una vez la bacteria percibe las condiciones adversas que influencian el proceso, se fosforila el regulador maestro de la transcripción conocido como Spo0A, que activa o reprime la expresión de genes importantes para el proceso, al unirse a blancos específicos de ADN (Mitchell, 2001; Ravagnani et al., 2000).
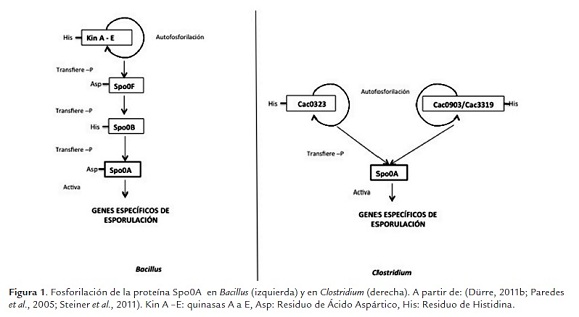
El proceso de fosforilación de Spo0A para iniciar el mecanismo de esporulación ha sido ampliamente estudiado en Bacillus subtilis y es reconocido como un sofisticado sistema de transducción de señales con múltiples componentes (Figura 1) (Paredes et al., 2005; Burbulys et al., 1991; Perego, 1998). Cabe destacar que, cuando se obtuvo la secuencia del genoma de C. acetobutylicum, no se encontraron genes ortólogos de la fosfotransferasa B (Spo0B) y de Spo0F, fundamentales para la fosforilación de Spo0A en Bacillus (Nölling & Breton, 2001). Debido a evidencias como esta, en conjunto con el análisis de genomas de otros organismos de este género, se han propuesto diferentes estrategias por medio de las cuales los clostridios pueden fosforilar a Spo0A, tales como el uso de quinasas huérfanas que fosforilen directamente al regulador maestro (Dürre & Hollergschwandner 2004). Steiner y colaboradores avalaron esta teoría en 2011, demostrando que por medio de histidin-quinasas huérfanas, existían dos posibles vías alternas a través de las cuales podía darse este fenómeno (Steiner et al. 2011). Se encontró que la proteína Cac0323 por una parte y las Cac0903 y Cac3319 por otra, podrían fosforilar directamente a Spo0A en Clostridium acetobutylicum (Burbulys et al., 1991; Dürre 2011a; Steiner et al., 2011).
Una vez fosforilado, Spo0A puede reprimir la expresión de genes como el que codifica para la proteína AbrB, que juega un papel fundamental en la transición entre el crecimiento vegetativo y la esporulación (Scotcher et al., 2005). Así mismo, induce la expresión de genes que contienen la información necesaria para producir factores sigma específicos del proceso de esporulación, regulando de forma eficiente y coordinada este mecanismo (Dürre, 2005). Se han encontrado en Clostridium genes homólogos de los factores sigma estudiados en Bacillus, mostrando que la arquitectura modular de la red de regulación de la esporulación está altamente conservada entre estas bacterias (De Hoon et al., 2010; Santangelo et al., 1998; Dürre & Hollergschwandner, 2004). El factor alternativo σH, que se expresa de forma constitutiva, permite en conjunto con Spo0A˜P, la expresión de los genes del operon spoIIA, que codifica a σF (factor sigma ubicado en la pre-espora), el factor anti-sigma SpoIIAB que mantiene a σF inactivo y el factor anti-anti sigma SpoIIAA que puede formar un complejo con SpoIIAB cuando este está unido a ADP, permitiendo a σF activarse. A su vez, σF permite la transcripción de los genes SpoIIGA y SpoIIGB, que codifican para una proteasa que permite la activación del factor σE, presente en la célula madre. Este factor a su vez permite la activación del factor σG en la pre-espora, que favorece después la expresión de σK en la célula madre (Dürre, 2005a; Dürre & Hollergschwandner, 2004; Hilbert & Piggot, 2004; Santangelo, 1998; Sauer et al., 1995). Este proceso de expresión consecutiva de genes se da de forma similar en los clostridios que hasta ahora se han estudiado. Sin embargo, en especies como C. beijerinckii, la expresión de los genes que producen σE y σG se da de forma más acelerada, ocasionando que la formación de la pre-espora y la endospora se den en menor tiempo (Shi & Blaschek, 2008).
Cuando las células de Clostridium entran en la fase estacionaria de crecimiento, en el momento en el que comienzan a diferenciarse, remodelan su metabolismo para dirigir tanto el flujo de carbono como de electrones ya no a la producción de biomasa, sino a la producción de solventes en un proceso conocido como solventogénesis (Amador-Noguez et al., 2011), el cuál le permite incrementar los niveles de pH externo. El proceso biotecnológico para producir acetona, butanol y etanol (ABE) por parte de clostridios solventogénicos se destacó durante la primera parte del siglo pasado por ser la segunda fermentación más importante después del etanol. Como tal, la producción microbiana de butanol fue reportada inicialmente por Louis Pasteur, pero fue Chaim Weizmann quien aisló el microorganismo Clostridium acetobutylicum y en 1915 patentó la fermentación, especialmente para producir acetona durante la primera guerra mundial. Posteriormente los esfuerzos se dirigieron a la producción de butanol como laca para automóviles (Jones & Woods, 1986). Actualmente, resurgió el interés de implementar nuevamente este tipo de biorefinerías debido a la aplicación de butanol como biocombustible, especialmente en China que cuenta con 11 plantas de producción y Brasil que cuenta con una planta. Sin embargo, todas estas instalaciones basan su producción en el uso de sustratos como melazas o almidones, que suelen tener altos costos para procesos de este tipo, sin contar con que compiten con suministros nutricionales (Dürre, 2011a) .
La fermentación acetobutílica se desarrolla en general siguiendo dos fases: acidogénica y solventogénica. En la primera el microorganismo crece activamente y los azúcares son convertidos en ácidos acético y butírico en mayor proporción, con la consecuente disminución de pH que ocasiona, en conjunto con otros factores, un cambio metabólico en la célula dando lugar a la segunda fase (solventogénica). En este punto los ácidos son reconsumidos y convertidos en acetona y butanol, permitiendo a la célula estar activa metabólicamente por más tiempo al incrementar el pH externo. Sin embargo, los solventes, en especial el butanol, también son tóxicos para la célula, por lo que la formación de endosporas comienza de forma simultánea con la solventogénesis, de modo que las redes regulatorias de los dos procesos están cercanamente relacionadas (Dürre, 2008).
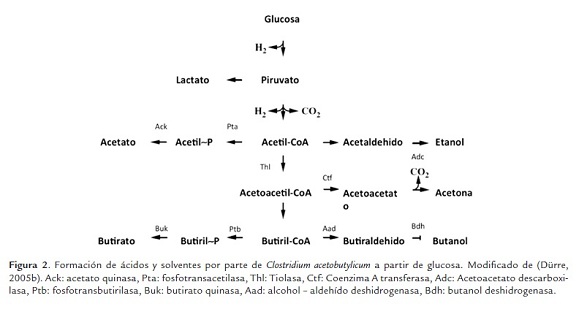
C. acetobutylicum toma los azúcares por medio del sistema de fosfotransferasas, y utiliza la ruta metabólica de Embden-Meyerhof (EMP) para obtener piruvato que es posteriormente oxidado a acetil-CoA por medio de la piruvato:ferredoxin oxidoreductasa (Ezeji et al., 2010; Jang et al., 2012). La ferredoxina reducida es una fuente de hidrógeno, mientras que el NADH generado durante la glicólisis es usado para producir ácido butírico (Buckel, 2005). Después que se producen dos moléculas de acetil-CoA, estas pueden convertirse ya sea a productos oxidados como la acetona, el acetato o CO2, o a metabolitos reducidos como el butanol, butirato o etanol (Gheshlaghi, Scharer, Moo-Young, & Chou, 2009). El proceso de acetogénesis genera una molécula de ATP por cada molécula de acetil- CoA por medio de la fosforilación a nivel de sustrato. En esta vía, el acetil-CoA se convierte en acetil-fosfato por la acción de una fosfotransacetilasa (Pta). Finalmente, la acetato quinasa (Ack) interviene en la producción de acetato y ATP. Por su parte, en la fermentación de butirato, el acetil-CoA obtenido a partir de la decarboxilación del piruvato acoplado con la reducción de la ferredoxina, se condensa por medio de la enzima tiolasa (Thl) para producir acetoacetil-CoA, que es reducido a butiril -CoA. Por medio de una fosfotransbutirilasa (Ptb) este se convierte en butiril- fosfato, y finalmente una quinasa (Buk) produce butirato y ATP (Figura 2) (Jones & Woods, 1986).
En la fase solventogénica actua la enzima acetoacetil- CoA: Acetato/butirato - coenzima A transferasa (Ctf) que cataliza la conversión de los ácidos producidos previamente en la fase exponencial en sus derivados de Acil CoA y acetoacetato. Después la enzima Acetoacetato descarboxilasa (Adc) convierte este último compuesto en CO2 y acetona. El Acetil CoA puede ser transformado en etanol, que se produce en bajas concentraciones de forma constante durante las dos fases de la fermentación, por medio de la enzima acetaldehído deshidrogenasa y una alcohol deshidrogenasa. Para producir butanol, la enzima butiraldehído/ butanol deshidrogenasa E (AdhE o Aad) transforma el butiril-CoA en butiraldehído, que posteriormente se transforma en butanol por medio de la butanol deshidrogenasa (BdhB o BdhII) (Figura 2) (Jones & Woods, 1986).
Los genes que codifican estas enzimas han sido ampliamente estudiados, especialmente aquellos que están organizados en los operones sol y adc, los cuales se localizan en un megaplásmido conocido como pSOL1. Particularmente el operon monocistrónico adc, tiene como gen estructural el que codifica para la enzima acetoacetato descarboxilasa, la cual cataliza el último paso durante la vía de formación de acetona, y por ende es necesario para remover el exceso de acetoacetato (Dürre et al., 2002). El operon sol contiene la información de las dos subunidades de la CoA transferasa (ctfA y ctfB), y de la aldehído/alcohol deshidrogenasa E (adhE) (Dürre et al., 2002; Cornillot et al., 1997; Fischer et al., 1993).
Relación de las redes de solventogénesis y esporulación en Clostridium
La solventogénesis es inducida por Clostridium con el fin de contrarrestar el efecto de la presencia de ácidos producidos, para otorgar a la célula tiempo para completar la formación de endosporas (Dürre et al., 2002). Cuando disminuye el pH, dichos ácidos se encuentran en su forma no disociada y son capaces de pasar la membrana celular por difusión, alterando el gradiente de protones alrededor de esta estructura celular y ocasionando en últimas la muerte de la célula. El reconsumo de ácidos por medio de la enzima acetoacetil- CoA: Acetato/butirato - coenzima A transferasa para su transformación en solventes, incrementa el pH externo, permitiendo a la célula garantizar su supervivencia por largos periodos al darle la posibilidad de esporular (Dürre, 2005b). De igual forma, el inicio de la producción de solventes y esporas está acoplado con la síntesis de granulosa, por lo que se han realizado diferentes estudios que sugieren mecanismos de regulación similares para estos 3 eventos fisiológicos (Ravagnani et al., 2000). Woolley y Morris mostraron que mutantes deficientes en las tres respuestas fueron capaces de revertir esta acción de forma espontánea, mostrando que la pérdida de capacidades era una consecuencia pleiotrópica de una lesión en algún gen regulatorio global (Woolley & Morris 1990). Ravagnani y colaboradores postularon en el año 2000 que es Spo0A el regulador maestro de estos procesos (Ravagnani et al., 2000).
Spo0A es una proteína perteneciente al grupo de reguladores de respuesta bacterianos, que tiene dos dominios conectados por una región variable. La región N- terminal o fosfoaceptora, contiene un residuo de ácido aspártico conservado que sirve como sustrato para la fosforilación, modulando la actividad de la proteína (Schmeisser & Brannigan, 2000). Por otra parte, la región C-terminal o efectora contiene un motivo hélice-vuelta-hélice de unión a ADN conservado entre homólogos de Spo0A de microorganismos formadores de endosporas de los géneros Bacillus y Clostridium (Gutierrez & Montoya 2009; Schmeisser et al., 2000). Esta región tiene afinidad por la secuencia consenso de ADN 5"-TGNCGAA-3" conocida como la "caja 0A", localizada en regiones regulatorias; afectando de forma directa o indirecta diferentes procesos característicos de la fase estacionaria de crecimiento (Ravagnani et al., 2000).
El gen adc es controlado por un único promotor que al parecer es inducido por Spo0A, ya que se han encontrado tres cajas 0A en la región regulatoria 5" del operon. Sin embargo, se ha demostrado que otros factores de transcripción también están implicados en la regulación de este promotor (Dürre, 2008; Dürre et al., 2002). Adicionalmente se sabe que el estado de relajación del ADN tiene un efecto inductor sobre el gen adc, es así como el desenrrollamiento del ADN en combinación con Spo0A y determinados factores de transcripción, promueven la inducción de adc (Dürre, 2005a; Dürre et al., 2002; Ravagnani et al., 2000). En un estudio del proteoma de cepas de C. acetobutylicum capaces e incapaces de formar esporas, se mostró que en el microorganismo con mutación knockout del gen que produce Spo0A, no se encontraba ni esta proteína ni la proteina Adc (Sullivan & Bennett, 2006). Por su parte, el operon sol tiene corriente arriba de su promotor, una caja 0A. Harris y colaboradores generaron en 2002 una cepa con Spo0A inactiva por mutación sitio-dirigida que mostró concentraciones mínimas de solventes respecto a la cepa patrón e incapacidad de formar endosporas, contrario a una cepa con sobreexpresión de Spo0A, que mostró una producción de butanol mayor que la cepa control y un proceso de esporulación acelerada, soportando la hipótesis de que este regulador de respuesta controla positivamente a las dos redes mencionadas (Harris et al., 2002).
Cuando Spo0A se encuentra fosforilado puede actuar igualmente como represor de la expresión de ciertos genes, tales como el que codifica para el regulador AbrB (gen abrB310 en Clostridium), que previene el inicio temprano de la esporulación, inhibiendo la transcripción de genes específicos de la esporulación en la fase exponencial de crecimiento (Higgins & Dworkin, 2012; Scotcher & Bennett, 2008). En una investigación realizada en 2005, Scotcher y colaboradores utilizaron ARN antisentido contra abrB310 en una cepa de C. acetobutylicum, y observaron disminución en la producción de solventes, sugiriendo inicialmente que podía ser este el regulador de la transición entre acidogénesis y solventogénesis (Scotcher et al., 2005). Scotcher y Bennett reportaron un nuevo estudio en 2008, donde hicieron uso de vectores reporteros de β- galactosidasa en cepas silvestres y en cepas con Spo0A defectuoso, y corroboraron la regulación negativa de Spo0A sobre abrB310 y otro gen homólogo conocido como abrB3647. En este mismo estudio reconocieron que AbrB310 es necesario más no suficiente para dar inicio al proceso solventogénico, soportando la hipótesis de que Spo0A actúa como controlador maestro de esta red metabólica (Scotcher & Bennett, 2008).
En los últimos años se han realizado varias aproximaciones para identificar el papel que juegan otros genes importantes en la regulación de la esporulación dentro de la solventogénesis. Una de estas investigaciones evaluó el papel de la fosfatasa SpoIIE, que se encarga de remover fosfato del factor anti-anti-sigma SpoIIAA, permitiendo al final que el factor σF sea activo en la pre-espora y se pueda continuar el proceso. Se concluyó que la sobreexpresión del gen spoIIE no afectaba de forma directa a la solventogénesis, así como que los cambios en el perfil de producción de solventes en las células con represión de la expresión del gen estaban mediados por los efectos ocasionados en la esporulación ( Scotcher & Bennett, 2005). En un estudio más reciente se estableció el papel de los factores σE y σG, concluyendo que este último factor no tiene rol alguno en la regulación de la solventogénesis, y que al inactivarse el gen sigE pueden producirse solventes dependiendo del estado fisiológico del inoculo, lo que sugiere que no hay un vínculo directo con la solventogénesis, pero que las dos redes pueden desacoplarse en un estado temprano de la diferenciación celular (Tracy et al., 2011).
Avances en ingeniería genética y metabólica relacionados con esporulación y solventogénesis
Tal y como se ha mencionado anteriormente, el regulador de respuesta Spo0A constituye el vínculo entre las redes de esporulación y solventogénesis. Sin embargo, ya que las esporas no son metabólicamente activas, se considera ideal tener células capaces de producir solventes sin sufrir el proceso de diferenciación, por lo que se han considerado diversas estrategias, desde sobreexpresar genes de solventogénesis en cepas que no esporulan, hasta desacoplar las dos redes. Cornillot y colaboradores re-introdujeron los genes de solventogénesis en 2 cepas degeneradas sin capacidad de formar esporas (M5 y DG1), restableciendo la capacidad de producir butanol, pero en concentraciones menores a la cepa de referencia (Cornillot et al., 1997). Posteriormente, el grupo de E. Papoutsakis intentó incrementar la producción de butanol usando como modelo la cepa M5, sobreexpresando genes de solventogénesis y realizando knockout de genes de acidogénesis, observando la necesidad de entender y controlar el flujo de electrones en la ruta metabólica, punto crucial para los microorganismos anaeróbicos (Sillers et al., 2008). Por otra parte, Alsaker y col. realizaron un estudio en 2004 donde evaluaron el efecto de la sobreexpresión de Spo0A sobre el estrés inducido por butanol (en una cepa modificada para tal efecto), que mostró una incrementada tolerancia a este solvente (Alsaker et al., 2004).
Para poder desacoplar de forma exitosa las dos redes y por tanto crear una cepa de C. acetobutylicum capaz de producir solventes más no de esporular, se ha evaluado la inactivación de la expresión de diferentes factores sigma y de SpoIIE, como se mencionó previamente (Relación de las redes de solventogénesis y esporulación en Clostridium). Al inactivar los factores σF y σE, que muestran su máximo nivel de expresión en la fase II, previo a la división asimétrica, los microorganismos son capaces de producir solventes, pero la productividad depende de la edad del inóculo. Cuando se interrumpe la expresión de SpoIIE o de σG, las cepas se comportan de la misma manera que la cepa patrón en cuanto a la solventogénesis (Tracy et al., 2011). Este tipo de estudios ha permitido tener una mirada comprensiva de los procesos que constituyen el ciclo de vida de los clostridios sacarolíticos, y reconocer que hace falta conocimiento sobre otros reguladores que controlan el cambio de acidogénesis a solventogénesis y cómo interactúan con el regulador maestro Spo0A, y así poder plantear estrategias de ingeniería metabólica exitosas para incrementar la producción de solventes (Tracy et al., 2012; Jones et al., 2011; Lütke-Eversloh & Bahl, 2011; Papoutsakis, 2008).
Conclusión
Clostridium genera esporas como un medio para sobrevivir ante condiciones adversas, tal y como lo hace Bacillus mediante un mecanismo altamente conservado, que presenta diferencias entre estos dos géneros especialmente en el inicio del sistema, controlado por la fosforilación del regulador maestro Spo0A. Es esta proteína, perteneciente al grupo de los reguladores de respuesta, el vínculo fundamental entre dos redes de expresión de genes de importancia en el ciclo de vida de la célula: esporulación y solventogénesis. La presencia de "cajas 0A" en regiones regulatorias de operones de genes relacionados con las dos rutas lo demuestran, así como los efectos demostrados experimentalmente sobre la esporulación y la solventogénesis en ausencia de esta proteína. Al ser la solventogénesis un bioproceso de alto impacto biotecnológico, se considera de gran valor el hecho de poder entender la regulación a nivel molecular de la obtención de solventes a partir de ácidos, para de esta manera poder plantear tácticas de ingeniería genética y metabólica que permitan incrementar de forma eficiente la rentabilidad de este proceso.
Referencias bibliográficas
1 Alsaker, K. V, Spitzer, T. R., & Papoutsakis, E. T. 2004. Transcriptional Analysis of spo0A Overexpression in Clostridium acetobutylicum and Its Effect on the Cell " s Response to Butanol Stress. Journal of bacteriology. 186(7): 1959-1971. doi:10.1128/JB.186.7.1959. [ Links ]
2 Amador-Noguez, D., Brasg, I. a, Feng, X.-J., Roquet, N., & Rabinowitz, J. D. 2011. Metabolome remodeling during the acidogenic-solventogenic transition in Clostridium acetobutylicum. Applied and environmental microbiology. 77(22): 7984-97. doi:10.1128/AEM.05374-11. [ Links ]
3 Atrih, a, & Foster, S. J. 2001. Analysis of the role of bacterial endospore cortex structure in resistance properties and demonstration of its conservation amongst species. Journal of applied microbiology. 91(2): 364-72. [ Links ]
4 Battistuzzi, F. U., Feijao, A., & Hedges, S. B. 2004. A genomic timescale of prokaryote evolution: insights into the origin of methanogenesis, phototrophy, and the colonization of land. BMC evolutionary biology. 4, 44. doi:10.1186/1471-2148-4-44. [ Links ]
5 Burbulys, D., Trach, K., & Hoch, J. 1991. Initiation of sporulation in B. subtilis is controlled by a multicomponent phosphorelay. Cell. 64, 545-552. [ Links ]
6 Cornillot, E, Nair, R. V, Papoutsakis, E. T., & Soucaille, P. 1997. The genes for butanol and acetone formation in Clostridium acetobutylicum ATCC 824 reside on a large plasmid whose loss leads to degeneration of the strain. Journal of bacteriology. 179(17): 5442-7. [ Links ]
7 De Hoon, M. J. L., Eichenberger, P., & Vitkup, D. 2010. Hierarchical evolution of the bacterial sporulation network. Current biology: CB. 20(17): R735-45. doi:10.1016/j.cub.2010.06.031. [ Links ]
8 Dürre, P. 2005a. Sporulation in Clostridia (Genetics). In Peter Dürre (Ed.), Handbook on Clostridia. pp. 659 - 666. Boca Ratón, Florida: Taylor & Francis Group. [ Links ]
9 Dürre, P. 2005b. Formation of Solvents in Clostridia. In Peter Dürre (Ed.), Handbook on Clostridia. pp. 671 - 685. Boca Ratón, Florida. [ Links ]
10 Dürre, P. 2008. Fermentative butanol production: bulk chemical and biofuel. Annals of the New York Academy of Sciences. 1125, 353-62. doi:10.1196/annals.1419.009. [ Links ]
11 Dürre, P. 2011a. Fermentative production of butanol--the academic perspective. Current opinion in biotechnology. 22(3): 331-6. doi:10.1016/j.copbio.2011.04.010. [ Links ]
12 Dürre, P. 2011b. Ancestral sporulation initiation. Molecular microbiology. 80(3): 584-7. doi:10.1111/j.1365-2958.2011.07628.x. [ Links ]
13 Dürre, P, Böhringer, M., Nakotte, S., Schaffer, S., Thormann, K., & Zickner, B. 2002. Transcriptional regulation of solventogenesis in Clostridium acetobutylicum. Journal of molecular microbiology and biotechnology. 4(3): 295-300. [ Links ]
14 Dürre, P, & Hollergschwandner, C. 2004. Initiation of endospore formation in Clostridium acetobutylicum. Anaerobe. 10(2): 69-74. doi:10.1016/j.anaerobe.2003.11.001. [ Links ]
15 Ezeji, T., Milne, C., Price, N. D., & Blaschek, H. P. 2010. Achievements and perspectives to overcome the poor solvent resistance in acetone and butanol-producing microorganisms. Applied microbiology and biotechnology. 85(6): 1697-712. doi:10.1007/s00253-009-2390-0. [ Links ]
16 Fischer, R. J., Helms, J., & Dürre, P. 1993. Cloning, sequencing, and molecular analysis of the sol operon of Clostridium acetobutylicum, a chromosomal locus involved in solventogenesis. Journal of bacteriology. 175(21): 6959-69. [ Links ]
17 Gheshlaghi, R., Scharer, J. M., Moo-Young, M., & Chou, C. P. (2009). Metabolic pathways of clostridia for producing butanol. Biotechnology advances, 27(6), 764-81. doi:10.1016/j.biotechadv.2009.06.002 [ Links ]
18 Gutierrez Escobar, A., & Montoya Castaño, D. 2009. Evolutionary Analysis for the Functional Divergence of the Spo0A Protein: The Key Sporulation Control Element. In silico biology, 9: 149-162. doi:10.3233/ISB-2009-0405. [ Links ]
19 Harris, L. M., Welker, N. E., Eleftherios, T., & Papoutsakis, E. T. 2002. Northern, Morphological, and Fermentation Analysis of Spo0A Inactivation and Overexpression in Clostridium acetobutylicum ATCC 824. Journal of bacteriology. 184(13): 3586-3597. doi:10.1128/JB.184.13.3586. [ Links ]
20 Heap, J. T., Kuehne, S. a, Ehsaan, M., Cartman, S. T., Cooksley, C. M., Scott, J. C., & Minton, N. P. 2010. The ClosTron: Mutagenesis in Clostridium refined and streamlined. Journal of microbiological methods. 80(1): 49-55. doi:10.1016/j.mimet.2009.10.018. [ Links ]
21 Higgins, D., & Dworkin, J. 2012. Recent progress in Bacillus subtilis sporulation. FEMS microbiology reviews. 36(1): 131-48. doi:10.1111/j.1574-6976.2011.00310.x. [ Links ]
22 Hilbert, D., & Piggot, P. 2004. Compartmentalization of Gene Expression during Bacillus subtilis Spore Formation. Microbiology and Molecular Biology. 68(2): 234 - 261. doi:10.1128/MMBR.68.2.234. [ Links ]
23 Jang, Y., Lee, J., Malaviya, A., Seung, D. Y., Cho, J. H., & Lee, S. Y. 2012. Butanol production from renewable biomass: rediscovery of metabolic pathways and metabolic engineering. Biotechnology journal. 7: 186-198. doi:10.1002/biot.201100059. [ Links ]
24 Jang, Y.-S., Lee, J., Malaviya, A., Seung, D. Y., Cho, J. H., & Lee, S. Y. 2012. Butanol production from renewable biomass: rediscovery of metabolic pathways and metabolic engineering. Biotechnology journal. 7(2): 186-98. doi:10.1002/biot.201100059. [ Links ]
25 Jones, D. T., & Woods, D. R. 1986. Acetone-butanol fermentation revisited. Microbiological reviews. 50(4): 484-524. [ Links ]
26 Jones, S. W., Tracy, B. P., Gaida, S. M., and Papoutsakis, E. T. 2011. Inactivation of F in Clostridium acetobutylicum ATCC 824 blocks sporulation prior to asymmetric division and abolishes E and G protein expression but does not block solvent formation. Journal of bacteriology. 193(10): 2429-40. doi:10.1128/JB.00088-11. [ Links ]
27 Labbé, R. 2005. Sporulation (Morphology) of Clostridia. In Peter Dürre (Ed.), Handbook on Clostridia. pp. 647 - 658. Boca Ratón, Florida: Taylor & Francis Group. [ Links ]
28 Lee, S. Y., Lee, D.-Y., & Kim, T. Y. 2005. Systems biotechnology for strain improvement. Trends in biotechnology. 23(7): 349-58. doi:10.1016/j.tibtech.2005.05.003 [ Links ]
29 Lütke-Eversloh, T., & Bahl, H. 2011. Metabolic engineering of Clostridium acetobutylicum: recent advances to improve butanol production. Current opinion in biotechnology. 22(5): 634-47. doi:10.1016/j.copbio.2011.01.011. [ Links ]
30 Mitchell, W. J. 2001. General Biology and Physiology. In H. Bahl & P. Dürre (Eds.), Clostridia: Biotechnology and Medical Applications. pp. 72 - 83. Weinheim: Wiley- VCH Verlag. [ Links ]
31 Nielsen, D. R., Leonard, E., Yoon, S.-H., Tseng, H.-C., Yuan, C., & Prather, K. L. J. Engineering alternative butanol production platforms in heterologous bacteria. Metabolic engineering. 11(4-5): 262-73. doi:10.1016/j.ymben.2009.05.003. [ Links ]
32 Nölling, J., & Breton, G. 2001. Genome Sequence and Comparative Analysis of the Solvent-Producing Bacterium Clostridium acetobutylicum. Journal of bacteriology. 183(16): 4823-4838. doi:10.1128/JB.183.16.4823. [ Links ]
33 Papoutsakis, E. T. 2008. Engineering solventogenic clostridia. Current opinion in biotechnology. 19(5): 420-9. doi:10.1016/j.copbio.2008.08.003. [ Links ]
34 Paredes, C. J., Alsaker, K. V, & Papoutsakis, E. T. 2005-. A comparative genomic view of clostridial sporulation and physiology. Nature reviews Microbiology. 3(12): 969-78. doi:10.1038/nrmicro1288. [ Links ]
35 Patakova, P., Linhova, M., & Rychtera, M. 2013. Novel and neglected issues of acetone-butanol-ethanol (ABE) fermentation by clostridia: Clostridium metabolic diversity, tools for process mapping and continuous fermentation systems. Biotechnology advances. 31(1): 58-67. doi:10.1016/j.biotechadv.2012.01.010. [ Links ]
36 Perego, M. 1998. Kinase-phosphatase competition regulates Bacillus subtilis development. Trends in microbiology. 6(9): 366-370. [ Links ]
37 Ravagnani, a, Jennert, K. C., Steiner, E., Grünberg, R., Jefferies, J. R., Wilkinson, S. R., Young, D. I., et al., 2000. Spo0A directly controls the switch from acid to solvent production in solvent-forming clostridia. Molecular microbiology. 37(5): 1172-85. [ Links ]
38 Santangelo, J. 1998. Sporulation and time course expression of sigma factor homologous genes in Clostridium acetobutylicum. FEMS microbiology letters: 161, 157-164. [ Links ]
39 Sauer, U., Santangelo, J., Treuner, A., Buchholz, M., & Dürre, P. 1995. Sigma factor and sporulation genes in Clostridium. FEMS microbiology reviews. 17: 331-340. doi:10.1111/j.1574-6976.1995.tb00216.x. [ Links ]
40 Schmeisser, F., & Brannigan, J. 2000. A new mutation in spo0A with intragenic suppressors in the effector domain. FEMS microbiology letters. 185, 1-6. doi:10.1111/j.1574-6968.2000.tb09049.x/full. [ Links ]
41 Scotcher, M. C., & Bennett, G. N. 2005. SpoIIE Regulates Sporulation but Does Not Directly Affect Solventogenesis in Clostridium acetobutylicum ATCC 824 SpoIIE Regulates Sporulation but Does Not Directly Affect Solventogenesis in Clostridium acetobutylicum ATCC 824. Journal of bacteriology. 187(6): 1930-1936. doi:10.1128/JB.187.6.1930. [ Links ]
42 Scotcher, M. C., & Bennett, G. N. 2008. Activity of abrB310 promoter in wild type and spo0A-deficient strains of Clostridium acetobutylicum. Journal of industrial microbiology & biotechnology. 35(7): 743-50. doi:10.1007/s10295-008-0341-x. [ Links ]
43 Scotcher, M., Rudolph, F., & Bennett, G. 2005. Effects of Decreased abrB310 Expression on the Transition from Acidogenesis to Solventogenesis , in Clostridium acetobutylicum ATCC 824 Expression on the Transition from Acidogenesis to Solventogenesis , in Clostridium acetobutylicum ATCC 824. Applied and environmental microbiology. 71(4): 1987-1995. doi:10.1128/AEM.71.4.1987. [ Links ]
44 Shi, Z., & Blaschek, H. P. 2008. Transcriptional analysis of Clostridium beijerinckii NCIMB 8052 and the hyper-butanol-producing mutant BA101 during the shift from acidogenesis to solventogenesis. Applied and environmental microbiology. 74(24): 7709-14. doi:10.1128/AEM.01948-08. [ Links ]
45 Sillers, R., Chow, A., Tracy, B., & Papoutsakis, E. T. 2008. Metabolic engineering of the non-sporulating, non-solventogenic Clostridium acetobutylicum strain M5 to produce butanol without acetone demonstrate the robustness of the acid-formation pathways and the importance of the electron balance. Metabolic engineering. 10(6): 321-32. doi:10.1016/j.ymben.2008.07.005. [ Links ]
46 Steiner, E., Dago, A. E., Young, D. I., Heap, J. T., Minton, N. P., Hoch, J. a, & Young, M. 2011. Multiple orphan histidine kinases interact directly with Spo0A to control the initiation of endospore formation in Clostridium acetobutylicum. Molecular microbiology. 80(3): 641-54. doi:10.1111/j.1365-2958.2011.07608.x. [ Links ]
47 Sullivan, L., & Bennett, G. N. 2006. Proteome analysis and comparison of Clostridium acetobutylicum ATCC 824 and Spo0A strain variants. Journal of industrial microbiology & biotechnology. 33(4): 298-308. doi:10.1007/s10295-005-0050-7. [ Links ]
48 Tracy, B. P., Jones, S. W., Fast, A. G., Indurthi, D. C., & Papoutsakis, E. T. 2012. Clostridia: the importance of their exceptional substrate and metabolite diversity for biofuel and biorefinery applications. Current opinion in biotechnology. 23(3):, 364-81. doi:10.1016/j.copbio.2011.10.008. [ Links ]
49 Tracy, B. P., Jones, S. W., & Papoutsakis, E. T. 2011. Inactivation of E and G in Clostridium acetobutylicum illuminates their roles in clostridial-cell-form biogenesis, granulose synthesis, solventogenesis, and spore morphogenesis. Journal of bacteriology. 193(6): 1414-26. doi:10.1128/JB.01380-10. [ Links ]
50 Woolley, R. C., & Morris, J. G. (1990). Stability of solvent production by Clostridium acetobutylicum in continuous culture: strain differences. Journal of Applied Microbiology. 69(5): 718-728. doi:10.1111/j.1365-2672.1990.tb01569.x. [ Links ]