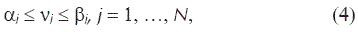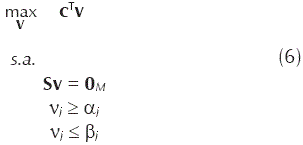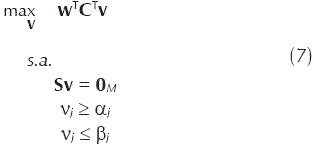Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Colombiana de Biotecnología
versão impressa ISSN 0123-3475
Rev. colomb. biotecnol vol.15 no.2 Bogotá jul./dez. 2013
https://doi.org/10.15446/rev.colomb.biote.v15n2.41263
http://dx.doi.org/10.15446/rev.colomb.biote.v15n2.41263
ARTÍCULO DE INVESTIGACIÓN
Balance de Flujos Metabólicos en Saccharomyces cerevisiae basado en Compartimentalización Intracelular
Metabolic Flows Balance in Saccharomyces cerevisiae based on Intracellular Compartmentalization
César Augusto Vargas García1, Carlos Eduardo García Sánchez2, Henry Arguello Fuentes3, Rodrigo Gonzalo Torres Sáez4
1 Msc Ingeniería de Sistemas e Informática, Universidad Industrial de Santander, caugusto.vargas@gmail.com.
2 PhD Ingeniería Química, Universidad Industrial de Santander, carlos.garcia6@correo.uis.edu.co.
3 PhD Electrical and Computer Engineering, Docente Asistente Escuela de Ingeniería de Sistemas e Informática, Universidad Industrial de Santander. henarfu@uis.edu.co.
4 PhD Bioquímica, Docente. Asistente Facultad de Ciencias Básicas, Universidad Industrial de Santander, rtorres@uis.edu.co.
Recibido: junio 15 de 2013. Aprobado: octubre 29 de 2013
Resumen
Una de las técnicas más utilizadas para la predicción de producción de bioproductos y distribución intracelular de flujos de microorganismos es el Análisis de Balance de Flujos - FBA por sus siglas en inglés. El FBA requiere de una función objetivo que represente el objetivo biológico del microorganismo estudiado. En este trabajo se propone un nuevo tipo de funciones objetivo basada en la combinación de objetivos de compartimentos físicos presentes en el microorganismo estudiado. Este tipo de funciones objetivo son examinadas junto con un modelo estequiométrico extraído de la reconstrucción iMM904 del microorganismo S. cerevisiae. Su desempeño se compara con la función objetivo más usada en la literatura, la maximización de biomasa, en condiciones experimentales anaeróbicas en cultivos continuos y aeróbicas en cultivos tipo lote. La función objetivo propuesta en este trabajo mejora las predicciones de crecimiento en un 10% y las predicciones de producción de etanol en un 75% respecto a las obtenidas por la función objetivo de maximización de biomasa, en condiciones anaeróbicas. En condiciones aeróbicas tipo lote la función objetivo propuesta mejora en un 98% las predicciones de crecimiento y en un 70% las predicciones de etanol con respecto a la función objetivo de biomasa.
Palabras clave: Análisis de Balance de Flujos, FBA, iMM904, S. cerevisiae, Función Objetivo basada en Compartimentos.
Abstract
Flux Balance Analysis - FBA - is one of the most used techniques in prediction of microorganism bioproducts. It requires an objective function that represents biological objective of the studied microorganism. This paper presents a new kind of objective functions based on individual physical compartment objetives in the studied microorganism. These kind of functions was tested with a stoichiometric model extracted from iMM904 reconstruction of S. cerevisiae and its performance is compared with the most used objective function in literature, growth maximization, in anaerobic and aerobic batch conditions. The presented objective function outperform growth predictions in 10% and ethanol predictions in 75% compared with obtained by maximization of growth objective function, in anaerobic conditions. In aerobic batch conditions the presented objective function outperforms in 98% growth preditions and 70% ethanol predictions compared with growth maximization.
Key words: Flux Balance Analysis, FBA, iMM904 reconstruction, Compartment based Objective Function.
Introducción
En los últimos años el Análisis de Balance de Flujos (FBA) ha permitido estudiar la obtención de bioproductos y la distribución intracelular de flujos; esta última a su vez ha facilitado la comprensión de la estructura biológica a escala genómica de microorganismos. El estudio de la distribución intracelular tiene aplicaciones que van desde la ingeniería metabólica hasta el descubrimiento de medicamentos (Gianchandani et al., 2010).
Uno de los desafíos actuales presentes en el FBA es la determinación de una función objetivo que permita reproducir con mayor exactitud resultados experimentales de microorganismos estudiados (Raman y Chandra, 2009). El microorganismo Escherichia coli cuenta con numerosos estudios sobre la mejor función objetivo que describe su comportamiento bajo diferentes condiciones experimentales, incluyendo anaeróbica y aeróbica, y con diferentes niveles de detalle en sus modelos estequiométricos, desde un conjunto reducido de reacciones hasta modelos extraídos de reconstrucciones a escala genómica (Feist y Palsson, 2010). En contraste el microorganismo Saccharomyces cerevisiae tiene solo un estudio que busca nuevas funciones objetivo y ninguno usando modelos a escala genómica (Feist y Palsson, 2010).
S. cerevisiae (conocida como levadura de cerveza) se utiliza para la producción de alimentos, químicos y combustibles, y ha sido pieza clave en el área de la biología eucariota debido a que conserva ciertas propiedades de organismos más complejos como el humano (Österlund et al., 2011). El estudio de reconstrucciones a escala genómica de S. cerevisiae ha servido como guía para la ingeniería metabólica en la mejora de producción de alimentos, químicos y combustibles, y como herramienta de interpretación biológica (Österlund et al., 2011).
La reconstrucción a escala genómica iMM904 es hasta ahora la más detallada y confiable en la reproducción de condiciones experimentales de S. cerevisiae (Mo et al., 2009). Supera en información genética a sus predecesoras iND750 (Duarte et al., 2004) e iFF708 (Förster et al., 2003).
La búsqueda de la mejor función objetivo del FBA se realiza de dos formas: a través de programación binivel (Terzer et al., 2009) y mediante evaluación sistemática de funciones (Feist y Palsson, 2010). En la primera, se eligen un conjunto de posibles reacciones cuya maximización o minimización puedan explicar una condición experimental y se determinan coeficientes que acompañan a dichas reacciones, a través de las soluciones obtenidas de un problema de programación binivel. El grupo de reacciones y los coeficientes que las acompañan se conocen como "función objetivo". Los problemas de optimización binivel se vuelven intratables computacionalmente en modelos a escala genómica (Gianchandani et al., 2008) debido a que su complejidad es NP-dura1 (Colson et al., 2007).
_____________________________
1 Problema de tiempo polinomial no deterministico. Implica que el tiempo que toma solucionar dicho problema incrementa de forma polinomial en relación a su tamaño (número de variables).
En la evaluación sistemática de funciones se elige un conjunto de funciones objetivo, ya conocidas y probadas, y mediante un criterio de selección se determina cuál de estas funciones obtiene la predicción más exacta para el modelo y condiciones experimentales estudiadas. Esta solución es computacionalmente menos costosa que la solución de problemas binivel debido a que el número de funciones objetivo probadas en la literatura es reducido (Feist y Palsson, 2010) y a que la prueba de una función objetivo consiste en resolver un FBA que a su vez consiste en resolver un problema de programación lineal.
Dentro de los trabajos de evaluación sistemática de funciones no hay registro de alguno que estudie la combinación de funciones objetivo conocidas con el fin de mejorar predicciones de producción de bioproductos como biomasa (o crecimiento) o etanol. Tampoco se ha estudiado que propiedades aporta la división espacial encontrada en microorganismos como S. cerevisiae a la predicción de bioproductos.
Hasta la fecha, la función objetivo que predice con la mayor exactitud la producción de biomasa (o crecimiento del microorganismo) y etanol de S. cerevisiae en condiciones experimentales aeróbicas tipo lote es la maximización de producción de biomasa (Gianchandani et al., 2008). Sin embargo, el estudio no se realizó sobre un modelo estequiométrico extraído de una reconstrucción a escala genómica.
El presente trabajo propone una nueva forma de crear funciones objetivo para el FBA basada en la división espacial del microorganismo S. cerevisiae. Esta división es conocida como compartimentalización. A cada compartimento se le asigna una función objetivo conocida. La función objetivo a utilizar en el FBA es la combinación de las funciones individuales de cada compartimento.
Este nuevo tipo de funciones es probada en el FBA utilizando un modelo estequiométrico extraído de la reconstrucción a escala genómica iMM904. Las condiciones experimentales a estudiar son anaeróbicas en cultivos continuos2 y aeróbicas tipo lote3 en fase exponencial4. Los resultados obtenidos con la mejor de estas funciones son comparados con los obtenidos usando la función objetivo maximización de biomasa, que es la sugerida por Gianchandani et al. (2008) y Feist y Palsson (2010) y usada en el FBA tradicional en condiciones aeróbicas y anaeróbicas.
_____________________________
2 En cultivos continuos el microorganismo estudiado está en estado de equilibrio estacionario. La tasa de crecimiento es constante así como el consumo de nutrientes y la producción de bioproductos.
3 El crecimiento del cultivo, la producción de bioproductos y la utilización de sustratos no se mantiene constante y termina después de un periodo de tiempo.
4 En fase exponencial las tasas de crecimiento, consumo de sustratos y formación de bioproductos es constante y está al máximo de su capacidad.
Este artículo está organizado de la siguiente forma: la sección de métodos explica el modelo estequiométrico y describe la técnica FBA. Además, esta sección presenta la forma de construir funciones objetivos basada en compartimentalización y el lugar que ocupa en la formulación FBA. La sección de resultados muestra el producto de aplicar la técnica FBA al modelo estequiométrico obtenido de la reconstrucción iMM904 utilizando las funciones objetivo propuestas en este trabajo y la función de maximización de biomasa.
Materiales y Métodos
Modelo Estequiométrico
En el modelo estequiométrico, la variación de la cantidad de un componente a través del tiempo 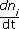 se define como una combinación lineal
se define como una combinación lineal
donde vj son las velocidades de reacción que lo afectan, Si,j es el coeficiente estequiométrico que indica la cantidad requerida o producida del componente nj en la reacción con velocidad vj , y N es el número de reacciones del sistema.
El conjunto de variaciones de todos los componentes nj donde i = 1, ..., M se representan por medio del sistema de ecuaciones lineales
donde n = [n1, ..., nM]T es el vector de cantidades de los componentes del sistema estudiado, S es una matriz de dimensiones M × N, la cual se conoce como la matriz estequiométrica, y v = [ν1, ..., νN]T es el vector de velocidades de reacción (vector de distribución de flujos o vector de flujos). En todo modelo estequiométrico el número de componentes es menor que el número de reacciones, es decir M < N.
Análisis de Balance de Flujos - FBA
Para estimar el comportamiento de un sistema usando el modelo estequiométrico, se asume estado de equilibrio estacionario del sistema el cual se representa por medio de la ecuación
donde 0M es un vector de longitud M cuyos elementos son todos cero. La ecuación matricial (3) define un sistema de ecuaciones lineales subdeterminado con infinitas soluciones. Para delimitar el espacio solución de la ecuación (3) se definen los límites de cada una de las velocidades de reacción νj del sistema mediante
donde αj y βj son los límites inferior y superior de νj. Por último, se establece una función objetivo
donde c = [c1,..., cN] es el vector que define el objetivo del sistema. Si la reacción νj está en la función objetivo ƒ entonces cj ≠ 0. En caso contrario cj ≠ 0.
Dados la matriz S, los vectores α[α1,..., αN], β = [β1,...βN] y c, se busca un vector v que sea solución del problema de programación lineal
La búsqueda de soluciones al problema (6) se conoce como FBA.
Funciones Objetivo Basadas en Compartimentos
Sea el compartimento P1 donde l = 1,...,L es un conjunto de índices j de v. Se define la combinación lineal C1Tv, donde C1 es un vector columna de dimensión N. Esta combinación se conoce como la función objetivo del compartimento P1. Si j  P1 y la velocidad ν1 no está incluida dentro de la función objetivo del compartimento P1 entonces Cj,l = 0. Por el contrario, si j
P1 y la velocidad ν1 no está incluida dentro de la función objetivo del compartimento P1 entonces Cj,l = 0. Por el contrario, si j  P1 y νj hace parte de la función objetivo del compartimento P1 entonces Cj,l ≠ 0. Por último Cj,l ≠ 0 si ƒ
P1 y νj hace parte de la función objetivo del compartimento P1 entonces Cj,l ≠ 0. Por último Cj,l ≠ 0 si ƒ 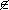 Pl.
Pl.
La figura 1 muestra un ejemplo de un sistema con 3 compartimentos: P1, P2 y P3. El conjunto P1 contiene las reacciones ν8, ..., ν14, P2, contiene las reacciones ν16, ..., ν18, P3 las reacciones ν20, ..., ν27. Las reacciones ν1, ..., ν7 no pertenecen a compartimento alguno y se conocen como reacciones de transporte externas del sistema. Las reacciones ν15 y ν19 representan el intercambio de componentes entre compartimentos y tampoco pertenecen a compartimento alguno. La figura 1 también muestra los vectores columna C1, C2 y C3 los cuales representan las funciones objetivo de los compartimentos P1, P2 y P3, respectivamente. Es importante aclarar que aunque las reacciones ν8, ..., ν14 pertenecen al compartimento P1, sólo las reacciones ν10, y ν13 se incluyen en la función objetivo C1Tv.
Dados S, α, β, la matriz C = [Cl1, ..., Clg] de dimensiones N × K y el vector w = [w1, ..., wK]T, el presente trabajo propone buscar el vector que es solución del problema de programación lineal
donde wk ≥ 0 con k = 1,...,K. El valor wk representa el aporte del objetivo del compartimento Plk al objetivo global del sistema. wTCT representa una nueva función objetivo obtenida de la combinación de las funciones objetivo de K compartimentos presentes en el sistema estudiado.
Simulaciones
La reconstrucción iMM904 cuenta con 1228 componentes internos y 1577 reacciones distribuidos en 8 compartimentos, disponible en formato SimPhenyTM. Cuenta con información sobre las capacidades del sistema representadas por los vectores α y β. Excluyendo algunas reacciones, los límites de νj, siendo PS el peso seco, son αj = -1000  para reacciones reversibles, αj = 0
para reacciones reversibles, αj = 0  para irreversibles y βj = 1000
para irreversibles y βj = 1000  . La función objetivo por defecto en esta reconstrucción es la maximización de biomasa o crecimiento del sistema (Feist y Palsson, 2010), representada por c, con cgw = 1 y cj = 0 para toda reacción diferente a la de crecimiento νgw. El modelo estequiométrico S1228×1577 y los vectores α y β se extrajeron de la reconstrucción iMM904 utilizando el paquete COBRA (Schellenberger et al., 2011) disponible para MatLab®.
. La función objetivo por defecto en esta reconstrucción es la maximización de biomasa o crecimiento del sistema (Feist y Palsson, 2010), representada por c, con cgw = 1 y cj = 0 para toda reacción diferente a la de crecimiento νgw. El modelo estequiométrico S1228×1577 y los vectores α y β se extrajeron de la reconstrucción iMM904 utilizando el paquete COBRA (Schellenberger et al., 2011) disponible para MatLab®.
Se utilizaron los compartimentos citosol, mitocondria y peroxisoma disponibles en la reconstrucción iMM904, los cuales se denominarán de aquí en adelante P1, P2 y P3. Para el compartimento P1 se propusieron 13 posibles funciones objetivo; para el compartimento P2, 4 posibles funciones objetivo; y para el compartimento P3, 5 posibles funciones objetivo. Estas funciones objetivo se denominarán de aquí en adelante C1, C2 y C3, respectivamente.
Para la construcción de las funciones C1 se eligió la maximización y minimización de las velocidades de reacción que corresponden a la producción y/o consumo de los componentes NADH, NADPH, ATP, etanol, dióxido de carbono, glicerol, succinato y acetato los cuales están presentes en el compartimento P1. Las funciones C2 consisten en la maximización y minimización de las velocidades de producción y/o consumo de los componentes NADH, NADPH y ATP del compartimento P2. Por último, para las funciones objetivo C3 se eligió la maximización y minimización de las reacciones que producen y/o consumen ácidos grasos, NADH, NADPH y ATP que se encuentran en el compartimento P3.
Estas funciones objetivo están relacionados principalmente con la producción y consumo de ATP y poder reductor, debido a que estos cofactores juegan un papel importante en la explicación del comportamiento celular. Otros objetivos considerados están asociados con la producción de biomasa, debido a la relevancia de este objetivo en el modelado FBA. La mayoría de los objetivos comparados en trabajos como Schuetz et al. (2007) y Knorr et al. (2007) quedan incluidos entre las combinaciones estudiadas en este trabajo. Su efectividad ha sido estudiada en la literatura de forma individual sin tener en cuenta compartimentos físicos del microorganismo estudiado (Feist y Palsson, 2010).
Se propuso un nuevo compartimento virtual representado por P4 cuyas funciones objetivo, denominadas C4 son la maximización y la minimización de biomasa. Este nuevo compartimento permite probar combinaciones de estas dos funciones y otros objetivos propuestos en el citosol. La tabla 1 lista las funciones objetivo propuestas en este trabajo para cada compartimento.
Se construyeron en total 1259 matrices C que corresponden a las posibles combinaciones de compartimentos y funciones objetivo de cada uno de estos. Por cada matriz C se construyó el vector w = 1K, donde 1K es un vector con todos sus elementos iguales a uno y con una longitud K igual al número de compartimentos incluidos en C.
Por cada matriz construida se formularon 12 instancias del problema (7) que representan 12 condiciones experimentales, divididas en 4 condiciones anaeróbicas en quimiostato (Nissen et al., 1997) y 8 aeróbicas tipo lote (Heyland et al., 2009). La condición anaeróbica significa que el medio en el que se encuentra el microorganismo carece de oxígeno y la aeróbica implica que hay presencia de oxígeno. El crecimiento se presentó por medio de la tasa específica de crecimiento. Esta tasa es igual a la tasa de dilución en cultivos continuos con unidades 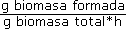 . En cultivos tipo lote, aunque no hay tasa de dilución, se conserva las mismas unidades que en el caso continuo. Las tablas 2 y 3 muestran las condiciones y componentes estudiados.
. En cultivos tipo lote, aunque no hay tasa de dilución, se conserva las mismas unidades que en el caso continuo. Las tablas 2 y 3 muestran las condiciones y componentes estudiados.
Cada una de las instancias del problema (7) formuladas para cada una de las matrices C se resuelve utilizando el software GLPK (Makhorin, 2011) con interfaz disponible para MatLab® a través del paquete COBRA, mediante un equipo con sistema operativo Linux. En total se resolvieron 12 × 1259 = 15108 instancias del problema (7).
Con el fin de facilitar la notación se define como la velocidad de reacción de nombre <rX> a la cual le corresponde la columna j de S, i.e. ν<rX> = νj. Los valores α<rX> y β<rX> definen los límites de la reacción ν<rX>. El valor c<rX> define el coeficiente que acompaña a ν<rX> en la función objetivo. La correspondencia entre nombres de reacción en la iMM904 y velocidades ν<rX> usados en las simulaciones del presente trabajo se muestran en la tabla 4. Las reacciones listadas son externas, salvo νgw y νATPM. Por convención, en la reconstrucción iMM904 todas las reacciones externas producen componentes externos.
Las condiciones experimentales anaeróbicas de Nissen et al. (1997) requieren modificación de algunas restricciones en el problema (7) que establece por defecto la reconstrucción iMM904. En el problema (7) la condición anaeróbica equivale a establecer αEX_o2(e) = βEX_o2(e) = 0. Debido al estado anaeróbico es necesario que el sistema tenga acceso a componentes que en ausencia de oxígeno no se pueden producir. Estos componentes son ergosterol, zymosterol y algunos ácidos grasos. Dicho acceso es permitido relajando las restricciones sobre 6 reacciones de intercambio; ν<EX_ergst(e)>, ν<EX_zymst(e)>, ν<EX_hdcea(e)>, ν<EX_ocdca(e)>, ν<EX_ocdcea(e)> y ν<EX_ocdcya(e)> ; de modo que pasen de ser irreversibles (sólo producción) a ser reversibles (consumo o producción). Por convención, en la reconstrucción iMM904, las reacciones correspondientes a estos componentes son irreversibles. Convertir en reversibles estas reacciones es equivalente a que el límite inferior sea α<rX> = -1000  en cada una de las velocidades de reacción de estos componentes.
en cada una de las velocidades de reacción de estos componentes.
La lista de restricciones por defecto de la iMM904 y las restricciones del problema (7) extraídas de Nissen et al. (1997) se presentan en la tabla 5. Esta lista no incluye todas las restricciones, sólo aquellas relevantes para el conjunto de datos experimentales de dicho trabajo.
En las condiciones aeróbicas tipo lote (Heyland et al., 2009) se modificó el límite inferior αEX_o2(e) de consumo de oxígeno por defecto de la reconstrucción iMM904 el cual pasó de -2  a -1000
a -1000  . Se cambió el límite inferior de consumo oxígeno para que los resultados obtenidos no fueran producto de esa restricción (Hjersted y Henson, 2009) si no del desempeño de las funciones objetivo. Además el consumo de oxígeno puede ser superior a 2
. Se cambió el límite inferior de consumo oxígeno para que los resultados obtenidos no fueran producto de esa restricción (Hjersted y Henson, 2009) si no del desempeño de las funciones objetivo. Además el consumo de oxígeno puede ser superior a 2  , ya que valores de hasta 10
, ya que valores de hasta 10  son comunes experimentalmente.
son comunes experimentalmente.
El mantenimiento interno de energía del sistema, representado en el modelo por νAPTM se conserva igual al provisto por la reconstrucción para todas las condiciones experimentales estudiadas. Para todas las condiciones experimentales se fijó el valor de consumo de glucosa νEX_glc(e) al correspondiente valor registrado. Las demás restricciones se mantuvieron igual a las suministradas por la reconstrucción iMM904.
Se midió el desempeño de la predicción de cada una de las matrices C en los dos grupos de condiciones experimentales. Este desempeño se representó en términos del promedio  gw del error en la predicción de crecimiento egw = |(νgw - νEgw)/νEgw|, con νgw como el valor de crecimiento predicho y νEgw su correspondiente valor experimental. También se determinó el promedio
gw del error en la predicción de crecimiento egw = |(νgw - νEgw)/νEgw|, con νgw como el valor de crecimiento predicho y νEgw su correspondiente valor experimental. También se determinó el promedio  EX_etoh(e) del error en la predicción de etanol eEX_etoh(e) = |(νEX_etoh(e)- νEEX_etoh(e))/νEEX_etoh(e)|, con νEX_etoh(e) como el valor predicho de etanol y νEEX_etoh(e) su correspondiente valor experimental.
EX_etoh(e) del error en la predicción de etanol eEX_etoh(e) = |(νEX_etoh(e)- νEEX_etoh(e))/νEEX_etoh(e)|, con νEX_etoh(e) como el valor predicho de etanol y νEEX_etoh(e) su correspondiente valor experimental.
Por último, se compararon las predicciones de etanol y crecimiento obtenidas por la matriz C con el menor error promedio en la predicción de crecimiento  gw, las predicciones de estos bioproductos obtenidas por la función objetivo del FBA tradicional, la maximización de biomasa (Feist y Palsson, 2010), y los resultados experimentales registrados en Nissen et al. (1997) y Heyland et al. (2009). Estas comparaciones se hicieron en términos del error promedio en la predicción de crecimiento
gw, las predicciones de estos bioproductos obtenidas por la función objetivo del FBA tradicional, la maximización de biomasa (Feist y Palsson, 2010), y los resultados experimentales registrados en Nissen et al. (1997) y Heyland et al. (2009). Estas comparaciones se hicieron en términos del error promedio en la predicción de crecimiento  gw y de etanol
gw y de etanol  EX_etoh(e).
EX_etoh(e).
Resultados
La tabla 6 muestra las diez matrices C que producen los promedios de error más bajos en la predicción de crecimiento egw, para cada una de las condiciones experimentales de las Tablas 1 y 2.
Las seis primeras matrices C usadas en condiciones anaeróbicas tienen promedios iguales egw (diferencias menores a 1 × 10-2). Las funciones objetivo C1 y C4 son la misma para estas seis matrices. La función objetivo C2 no aparece en alguna de estas. Esto significa que incluir alguna de las funciones del compartimento C2 no mejora la predicción de crecimiento. La diferencia en las primeras seis matrices radica en la función objetivo C3. Cada una de las alternativas de función objetivo C3 aparece en los seis primeros lugares. También existen una matriz C en la que no se incluye alguna función objetivo C3 (Fila 1 de resultados en condiciones anaeróbicas en la tabla 6). Lo anterior implica que el menor promedio de error de predicción de crecimiento se obtiene construyendo Cana con minimización de producción de NADH como función objetivo C1 y maximización de biomasa como función C4 . Cana reduce el error en la predicción de crecimiento egw en un 10% con respecto a la predicción obtenida con la función objetivo de maximización de biomasa. El menor valor de egw encontrado es de 0.19.
En condiciones aeróbicas en cultivos tipo lote se observa que son seis matrices las que obtienen el menor promedio egw, para las cuales el promedio es el mismo. Estas matrices difieren en la función objetivo C3. Lo anterior indica que incluir o no cualquier alternativa de la función objetivo C3 no afecta significativamente el valor de egw. Las seis matrices tienen como función objetivo C1 la minimización de la producción de NADH; en C2 la minimización de consumo de NADH y NADPH; y en C4 la maximización de la producción de biomasa. Por lo tanto la matriz Caer, compuesta por esta combinación de compartimentos y funciones objetivo, permite obtener la mejor predicción. El mínimo valor egw obtenido para este tipo de condiciones experimentales es 0.1.
En condiciones anaeróbicas, la función objetivo maximización de biomasa obtuvo un error de predicción promedio de crecimiento  gw de 0.21. La matriz Cana obtuvo un
gw de 0.21. La matriz Cana obtuvo un  gw de 0.19. Esto implica que se logró una reducción del % en el valor de . En cuanto a la producción de etanol, la función objetivo de maximización de biomasa obtuvo un error de predicción promedio de etanol
gw de 0.19. Esto implica que se logró una reducción del % en el valor de . En cuanto a la producción de etanol, la función objetivo de maximización de biomasa obtuvo un error de predicción promedio de etanol  EX_etoh(e) de 0.12. La matriz Cana obtuvo un
EX_etoh(e) de 0.12. La matriz Cana obtuvo un  EX_etoh(e) de 0.03, lo que significa una reducción del 75% en el valor de
EX_etoh(e) de 0.03, lo que significa una reducción del 75% en el valor de  EX_etoh(e). La figura 2 muestra el desempeño de la función objetivo maximización de producción de biomasa, la matriz Cana y el comportamiento experimental en condiciones anaeróbicas. Los valores obtenidos de egw y eEX_etoh(e) para cada condición anaeróbica son mostrados en la tabla 7.
EX_etoh(e). La figura 2 muestra el desempeño de la función objetivo maximización de producción de biomasa, la matriz Cana y el comportamiento experimental en condiciones anaeróbicas. Los valores obtenidos de egw y eEX_etoh(e) para cada condición anaeróbica son mostrados en la tabla 7.
En condiciones aeróbicas tipo lote, la función objetivo maximización de biomasa obtuvo un error de predicción promedio de crecimiento  gw de 4.04. La matriz Cana obtuvo un
gw de 4.04. La matriz Cana obtuvo un  gw de 0.1, logrando una reducción del 98% en el valor de
gw de 0.1, logrando una reducción del 98% en el valor de  gw. En cuanto a la producción de etanol, la función objetivo maximización de biomasa obtuvo un error de predicción promedio
gw. En cuanto a la producción de etanol, la función objetivo maximización de biomasa obtuvo un error de predicción promedio  EX_etoh(e) de Caer obtuvo un
EX_etoh(e) de Caer obtuvo un  EX_etoh(e) de 0.30, lo que significa una reducción del 70% en el valor de
EX_etoh(e) de 0.30, lo que significa una reducción del 70% en el valor de  EX_etoh(e). La figura 3 muestra el desempeño de la función objetivo maximización de producción de biomasa, Caer y el comportamiento experimental en condiciones aeróbicas tipo lote. Los valores obtenidos de egw y eEX_etoh(e) para cada condición aeróbica tipo lote son mostrados en la tabla 8.
EX_etoh(e). La figura 3 muestra el desempeño de la función objetivo maximización de producción de biomasa, Caer y el comportamiento experimental en condiciones aeróbicas tipo lote. Los valores obtenidos de egw y eEX_etoh(e) para cada condición aeróbica tipo lote son mostrados en la tabla 8.
Conclusiones
Combinar la maximización de producción de biomasa con la minimización de producción de NADH en el compartimento citosol de S. cerevisiae reduce el error promedio en la predicción de crecimiento en un 75% en las condiciones anaeróbicas de Nissen et al. (1997). Esta combinación también reduce en un % el promedio del error en la predicción de producción de etanol.
Si a la función objetivo encontrada en condiciones anaeróbicas se agrega la minimización de consumo de NADH y NADPH en el compartimento mitocondria de S. cerevisiae, se reduce el promedio del error en la predicción de crecimiento en un 98% en las condiciones aeróbicas tipo lote planteadas por Heyland et al. (2009). Esta nueva función objetivo reduce el promedio del error en la predicción de etanol en un 70% con respecto a la función de maximización de biomasa.
Agregar funciones objetivo de compartimentos de S. cerevisiae a la función objetivo de maximización de biomasa mejora las predicciones obtenidas por la función objetivo maximización de biomasa, propuesta en la literatura por Feist y Palsson (2010) y en la reconstrucción a escala genómica iMM904 (Mo et al., 2009), en condiciones experimentales anaeróbicas en cultivo continuo (Nissen et al., 1997) y aeróbicas tipo lote (Heyland et al., 2009).
Referencias bibliográficas
1 Colson B., Marcotte P., Savard G. 2007. An overview of bilevel optimization. Annals of Operations Research. 153(1): 235-256. [ Links ]
2 Duarte N.C., Herrgard M.J., Palsson B. Ø. 2004. Reconstruction and validation of Saccharomyces cerevisiae iND750, a fully compartmentalized Genome-Scale metabolic model. Genome Research. 14 (7): 1298-1309. [ Links ]
3 Feist A.M. y Palsson B.Ø. 2010. The biomass objective function. Current Opinion in Microbiology. 13(3): 344-349. [ Links ]
4 Förster J., Famili I., Fu P., Palsson B.Ø., Nielsen J. 2003. Genome-Scale reconstruction of the Saccharomyces cerevisiae metabolic network. Genome Research. 13(2): 244-253. [ Links ]
5 Gianchandani E.P., Oberhardt M., Burgard A., Maranas C., Papin J. 2008. Predicting biological system objectives de novo from internal state measurements. BMC Bioinformatics. 9(1): 43. [ Links ]
6 Gianchandani E.P., Chavali A.K., Papin J.A. 2010. The application of flux balance analysis in systems biology. Wiley Interdisciplinary Reviews: Systems Biology and Medicine. 2(3): 372-382. [ Links ]
7 Heyland J., Fu J., Blank L.M. 2009. Correlation between TCA cycle flux and glucose uptake rate during respiro-fermentative growth of Saccharomyces cerevisiae. Microbiology. 155(12): 3827-3837. [ Links ]
8 Hjersted J., Henson M. 2009. Steady-state and dynamic flux balance analysis of ethanol production by Saccharomyces cerevisiae. IET Systems Biology. 3(3): 167-179. [ Links ]
9 Knorr A.L., Jain R., Srivastava R. 2007. Bayesian-based selection of metabolic objective functions. Bioinformatics. 23(3): 351-357. [ Links ]
10 Makhorin A. 2011. GLPK (GNU Linear Programming Kit). v.4.47. http: //www.gnu.org/software/glpk. [ Links ]
11 Mo M.L., Palsson B.Ø., Herrgard M. 2009. Connecting extracellular metabolomic measurements to intracellular flux states in yeast. BMC Systems Biology. 3(1): 37. [ Links ]
12 Nissen T.L., Schulze U., Nielsen J., Villadsen J. 1997. Flux distributions in anaerobic, Glucose-Limited continuous cultures of Saccharomyces cerevisiae. Microbiology. 143(1): 203-218. [ Links ]
13 Raman, K., Chandra N. 2009. Flux balance analysis of biological systems: applications and challenges. Briefings in Bioinformatics. 10(4): 435-449. [ Links ]
14 Österlund T., Nookaew I., Nielsen J. 2011. Fifteen years of large scale metabolic modeling of yeast: Developments and impacts. Biotechnology Advances. 30(5): 979-988. [ Links ]
15 Schellenberger J., Que R., Fleming R.M., Thiele I., Orth J.D., Feist A.M., Zielinski D.C., Bordbar A., Lewis N.E., Rahmanian S., Kang J., Hyduke D.R., Palsson B.Ø. 2011. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox v2.0. Nature Protocols. 6(9): 1290-1307. [ Links ]
16 Schuetz R., Kuepfer L., Sauer U. 2007. Systematic evaluation of objective functions for predicting intracellular fluxes in Escherichia coli. Molecular Systems Biology. 3: 119. doi:10.1038/msb4100162. [ Links ]
17 Terzer M., Maynard. N.D., Covert M.W., Stelling J. 2009. Genome scale metabolic networks. Wiley Interdisciplinary Reviews: Systems Biology and Medicine. 1(3): 285-297. [ Links ]