Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Biotecnología
Print version ISSN 0123-3475
Rev. colomb. biotecnol vol.16 no.1 Bogotá Jan./June 2014
https://doi.org/10.15446/rev.colomb.biote.v16n1.44224
http://dx.doi.org/10.15446/rev.colomb.biote.v16n1.44224
ARTÍCULO DE INVESTIGACIÓN
Purificación y caracterización de lipopolisacáridos de Eikenella corrodens 23834 y Porphyromonas gingivalis W83
Purification and characterization of lipopolysaccharide from Eikenella corrodens 23834 and Porphyromonas gingivalis W83
Título corto: Metodología para el aislamiento e identificación de LPS de periodonto-patógenos
Diego Fernando Gualtero Escobar1, Jeimy Paola Porras Gaviria2, Sebastian Bernau Gutierrez3 , Diana Marcela Buitrago Ramírez4, Diana Marcela Castillo Perdomo5 y Gloria Ines Lafaurie Villamil6.
1 Candidato a Doctor en Biotecnología. Facultad de ciencias, Universidad Nacional de Colombia. Magister en Bioquímica. Lic. Biología y Química. Autor de correspondencia: gualterodiego@unbosque.edu.co
2 Lic. Química. porrasyeimi@unbosque.edu.co
3 Candidato a Magister en Ciencias Básicas Biomédicas, Facultad de ciencias, Universidad El Bosque. Biólogo. gutierrezsebastian@unbosque.edu.co
4 PhD. Ciencias Farmacéuticas. Bacterióloga. buitragodianam@unbosque.edu.co
5 Candidato a Doctor en Biotecnología, Facultad de ciencias, Universidad Nacional de Colombia. Magister en Microbiología. Bacterióloga. castillodiana@unbosque.edu.co
6 Directora Instituto UIBO. Magister en Epidemiología. Periodoncista. Odontóloga. investigaciones.odontologia@unbosque.edu.co
Unidad de Investigación Básica Oral (UIBO)-Laboratorio de biotecnología-División de Investigaciones-Facultad de Odontología-Universidad El Bosque. Av. Cra 9 No. 131A - 02. Edificio de rectoría 2 piso
Recibido: abril 12 de 2013 Aprobado: abril 15 de 2014
Resumen
La purificación de lipopolisacáridos (LPS) o endotoxinas y su caracterización es un aspecto esencial para estudios que buscan aclarar el papel de estas biomoléculas de bacterias Gram negativas presentes en la cavidad oral y su relación con enfermedades locales periodontales y sistémicas. Este estudio implementa una metodología para la extracción, purificación y caracterización de LPS a partir de bacteria completa de Eikenella corrodens 23834 y Porphyromonas gingivalis W83, utilizando técnicas previamente descritas. La extracción cruda de LPS se realizó con fenol-agua caliente; la purificación se realizó con tratamiento enzimático con nucleasas y proteasa, seguido de cromatografía de exclusión por tamaño (Sephacryl S-200 HR) con deoxicolato de sodio como fase móvil. La caracterización de los extractos purificados se realizó por barrido espectrofotométrico, pruebas bioquímicas de electroforesis SDS-PAGE, ensayo Purpald y la prueba cromogénica de LAL. Como control para la identificación y caracterización de los extractos purificados se utilizaron LPS comerciales de Escherichia coli, Salmonella typhimurium, Rodobacter sphaeroides y Porphyromonas gingivalis. La metodología implementada permitió la obtención de LPS de elevada pureza con la identificación de KDO o heptosas, un quimiotipo de LPS-S (liso) para E. corrodens y LPS-SR (semi-rugoso) para P. gingivalis W83. Ambos LPS purificados mostraron capacidad endotóxica a bajas concentraciones. La metodología implementada en este estudio para la purificación y caracterización de LPS a partir de bacteria completa fue eficiente al compararla con los LPS comerciales.
Palabras clave: endotoxinas, cromatografía, ácido 2-ceto-3-desoxioctulosónico (KDO), test LAL, periodontitis.
Abstract
Purification of lipopolysaccharide (LPS) or endotoxins and its characterization is an important aspect for studies aimed at clarify the role of these biomolecules from Gram negative bacteria present in the oral cavity and its relationship with periodontal and systemic diseases. This study describes an extraction, purification and characterization method of LPS from Eikenella corrodens 23834 and Porphyromonas gingivalis W83. LPS extraction was performed by using hot phenol-water; the purification was done with nuclease and protease enzymatic treatment, followed by size-exclusion chromatography (Sephacryl S-200 HR) with sodium deoxycholate as mobile phase. The characterization of the purified extracts was performed by spectrophotometric scanning, SDS-PAGE biochemical tests, Purpald assay and chromogenic LAL test. As control, commercial LPS from Escherichia coli, Salmonella typhimurium, P. gingivalis, and Rodobacter sphaeroides were used. The methodology mentioned above had allowed obtaining high purity LPS by identifying KDO or heptoses, a chemotype S-LPS (smooth) to E. corrodens; SR-LPS (semi-rough) for P. gingivalis W83. Both purified LPS showed endotoxic capacity at low concentrations. The methodology used in this study for purification and characterization of LPS from the whole bacteria was efficient when it was compared with commercial LPS.
Key words: endotoxin, 2-keto-3-deoxyoctonate (KDO), Chromatography, Limulus test (LAL), periodontitis.
Introducción
Estudios epidemiológicos han encontrado una asociación de riesgo entre enfermedades infecciosas, como la periodontitis, con enfermedad cardiovascular (Elkaim et al., 2008; Kebschull, Demmer, y Papapanaou, 2010). Se ha propuesto que esto sucede por el paso de bacterias viables y su degradación en plasma, con la posterior liberación de Lipopolisacáridos o endotoxinas (LPS), induciendo la secreción de citoquinas, las cuales han sido ampliamente involucradas en los procesos de enfermedad cardiovascular (Lafaurie et al., 2007; Marchesan et al., 2012; Pussinen et al., 2007). Sin embargo, aún falta evidencia clínica e investigativa que sustente este hecho (Thomopoulos et al., 2011).
Dentro de los factores de virulencia presentes en las bacterias Gram negativas se encuentran los LPS, polisacáridos de cápsula, fimbrias, hemaglutininas y proteasas (Curtis et al., 2005). Los LPS inducen respuestas inflamatorias sistémicas en el hospedero que pueden estar relacionadas con riesgo cardiovascular aterosclerótico (Kallio et al., 2008; Pussinen et al., 2007; Wiedermann et al., 1999).
Las bacterias Gram negativas sintetizan formas diversas de LPS denominadas Liso (S: smooth) y Rugoso (R: rough). LPS-S consiste de cadenas O-polisacáridas, que están constituidas de unidades repetidas de oligosacáridos, el núcleo de oligosacáridos y una región altamente conservada denominada el lípido A. Los LPS-R carecen de la cadena específica O y está conformada solo del núcleo de oligosacáridos y el lípido A. Existen cinco clases principales de LPS-R (Ra-Re) según la disminución en la constitución del núcleo de oligosacáridos (Raetz, y Whitfield, 2002). Los LPS-SR son aquellos que contienen el antígeno O pero en un menor grado de composición de polisacáridos (Hitchcock, y Brown, 1983). A pesar de las diferencias en estructura química y propiedades fisicoquímicas, los diferentes quimiotipos de LPS actúan como endotoxinas debido a que todos contienen la región del lípido A, la cual alberga el potencial de actividad biológica sobre las células.
Porphyromonas gingivalis es uno de los principales microorganismos que causa la progresión y severidad de la enfermedad periodontal. Es una bacteria anaerobia Gram negativa con forma de bacilo corto, que metaboliza principalmente péptidos y azúcares como fuente de energía (Lamont y Jenkinson, 1998). E. corrodens es un cocobacilo Gram negativo anaerobio facultativo de crecimiento difícil, habitante normal de la cavidad oral, con carácter de agente patógeno oportunista que se ha asociado con infecciones orales y no orales usualmente como parte de un consorcio microbiano (Jaramillo et al., 2006). E. corrodens está presente en placa subgingival de pacientes con enfermedad periodontal (Mayorga-Fayad et al., 2007), y pertenece al grupo de las HACEK que ocasiona al menos el 1,4 % de las endocarditis infecciosas (Posch et al., 2013).
Recientes estudios han identificado ácidos nucleicos de E. corrodens y P. gingivalis en lesiones ateroscleróticas de individuos con enfermedad cardiovascular y periodontitis (Padilla et al., 2007). Sin embargo, también se encuentran en venas saludables (Elkaim et al., 2007); por lo que no es claro el papel que desempeñan estos microorganismos en la inducción de enfermedades cardiovasculares. Cuando se relaciona un microorganismo con determinadas entidades clínicas se deben establecer los componentes por medio de los cuales es capaz de producir la enfermedad; es decir, los mecanismos de patogenia que se relacionan en forma directa con sus elementos estructurales, interacciones entre la misma especie y la presencia de un consorcio microbiano, lo que será útil no sólo para establecer si este microorganismo es realmente el agente causal de la enfermedad, sino también para aplicar una terapéutica eficaz (Jaramillo et al., 2006).
Varios métodos se han reportado para la extracción de LPS que van desde la utilización de fenol, trizol, etanol-MgCl2, butanol (Darveau, Hancock, 1983; Galanos et al., 1969; Morrison, Leive, 1975; Wu et al., 1987). Sin embargo, el método más utilizado es el de Westphal que utiliza el fenol en agua caliente para extraer los LPS de tipo R o S, con un bajo contenido de proteínas (Apicella, 2008; Darveau, & Hancock, 1983; Westphal, and Jann, 1965). Estos métodos de extracción siguen vigentes en los procesos de purificación de LPS (Posch et al., 2013).
Cuando se requiere valorar el potencial que tienen los LPS en inducir respuestas celulares específicas se necesita de procesos de purificación más rigurosos para eliminar posibles contaminaciones por otros factores de virulencia presentes en las bacterias Gram negativas (Curtis et al., 2005; Hirschfeld, 2000). La presencia de ácidos nucleicos o proteínas que forman complejos fuertes cuando se realiza el método convencional con fenol deben ser valorados en los procesos de purificación de LPS (Perdomo, Rolando, 2006). En el presente estudio se implementa una metodología para la obtención de LPS de elevada pureza utilizando técnicas convencionales previamente descritas para la extracción, purificación y caracterización de LPS de bacterias periondo-patógenas, cuyos LPS no han sido anteriormente descritos en términos de sus características bioquímicas.
Materiales y métodos
Cultivo celular bacteriano. Las bacterias de referencia Eikenella corrodens ATCC 23834 fueron adquiridas en la American Type Culture Collection y cultivadas en Laboratorio de Microbiología Oral de la Universidad El Bosque. Las cepas W83 de P. gingivalis y ATCC 23834 de E. corrodens fueron sembradas en agar Brucella (BBL Microbiology Systems, Cockeysville, Md.) enriquecido con 0,3% p/v de Bacto agar, 0,2% p/v de extracto de levadura, 5% v/v de sangre de cordero desfibrinada, 0,2% v/v de sangre hemolizada, 0,0005% p/v de hemina y 0,00005% p/v de menadiona e incubada en atmósfera de anaerobiosis con 9-13% de CO2 a concentraciones por debajo del 1% de oxígeno (Anaerogen, Oxoid, Hampshire, England) a 36°C durante 7 días. Posteriormente, se observaron las características macroscópicas de las colonias y se realizaron pruebas bioquímicas con el fin de confirmar la pureza del cultivo.
Porphyromonas gingivalis se confirmó con la prueba de luz ultravioleta negativa y la prueba de CAAM positiva (NCBZ-GLY-GLY-ARG clorhidrato de 7-amido-4-metil coumarina, para la detección de enzimas tipo tripsina), E. corrodens fue confirmada por la presencia de oxidasa, y la ausencia de catalasa y motilidad, así como el uso de sistemas de identificación comercial Rapid ANA II (RemelTM, Apogent). Se realizó una resiembra masiva de P. gingivalis con el fin de obtener 1,1 g de bacteria completa. Las colonias de E. corrodens fueron suspendidas en caldo ToddHewitt (Jaramillo et al., 2006), se incubaron en condiciones de anaerobiosis durante 7 días con el fin de obtener 3,5 g de la bacteria. Pasado el tiempo de incubación se confirmó la pureza del cultivo, se recogió la totalidad de las colonias presentes en el cultivo se pasaron a un tubo Falcon® estéril previamente pesado con el fin de conocer el peso seco de la bacteria. La purificación del LPS se realizó una vez se comprobó la pureza de la bacteria. Las bacterias se conservaron a -80°C hasta la purificación del LPS.
Purificación de LPS de E. corrodens ATCC 23834. El procedimiento de extracción y purificación de LPS de E. corrodens se realizó según el procedimiento reportado por Gualtero y cols., (2008). La caracterización de este LPS se realizó como se describe más adelante.
Extracción de lipopolisacáridos con fenol acuoso a partir de bacteria completa de Porphyromonas gingivalis W83. A partir de 1.1 g de bacteria completa en 5.5 mL de agua USP se realizó la extracción con 18 mL de fenol al 90 % v/v y 18 mL de agua USP, incubando 10 min a 68 ºC en agitación a 175 rpm (SHAKER BATH marca LaB - Line) e inmediatamente llevando a baño de hielo por 30 min. Posteriormente se centrifugó a 5000 g por 20 min a 6 ºC, recuperando la fase acuosa. La fase fenólica fue de nuevo sometida a re-extracción dos veces más. Para eliminar las trazas de fenol en la fase acuosa se llevó a cabo una diálisis con membrana de tamaño de poro 10 kDa (SnakeSkin™.Thermo Scientific, USA) en 1 L de agua estéril grado I (Direct-Q3., Millipore) a 4 ºC y con cambio de agua cada 8 horas por 3 días. El extracto obtenido fue centrifugado a 10000 g por 20 min, y al sobrenadante separado se le adicionó acetato de sodio a una concentración final 0.15 M. Para inducir precipitación se colocó la solución en baño de hielo, se adicionó etanol 96 % v/v gota a gota en agitación constante a una proporción 1:4 (muestra:etanol) con respecto a la cantidad de extracto resultante. La solución fue almacenada durante 24 h a - 20 ºC para continuar con el proceso de precipitación. Posteriormente se centrifugó a 4000 g por 5 min a 4 ºC, eliminando el sobrenadante y se determinó la masa del precipitado obtenido. Finalmente, se reconstituyó el precipitado en agua USP a una concentración final de 25 mg/mL y se liofilizó durante 24 h (HETO POWER DRY LL3000, Bomba Thermo Savant).
Purificación de lipopolisacáridos de bacteria Porphyromonas gingivalis W83. El procedimiento de purificación de LPS consistió en un tratamiento enzimático del extracto crudo, seguido de una separación por cromatografía de exclusión por tamaño.
Tratamiento enzimático del extracto crudo de Porphyromonas gingivalis W83. El procedimiento de digestión con nucleasas se realizó según lo reportado por Perdomo, & Rolando (2006). El extracto liofilizado se suspendió en 5 mL de buffer Tris 0.1 M - NaCl 0.15 M, pH 7.5, a una concentración final de 2,6 mg/mL. Se tomó una alícuota de 650 µL para análisis por espectrofotometría (BioRad, SamartSpec™ Plus. 200 nm - 400 nm). Consecutivamente se adicionó 500 µL de ribonucleasa A 0.5 mg/mL, 50 µL de desoxirribonucleasa 1 0.05 mg/mL, 100 µL de cloruro de magnesio 4 mM y se incubó a 60 ºC durante 3 h. Posteriormente se adicionó 100 µL de proteinasa K 0.05 mg/mL, 100 µL de cloruro de calcio 1 mM incubando a 37 ºC por 18 horas, se realizó diálisis y se liofilizó como se explicó anteriormente. Luego de cada proceso de digestión enzimática se tomó una alícuota para analizar su perfil espectrofotométrico.
Cromatografía de exclusión por tamaño. El liofilizado fue reconstituido a una concentración final de 5 mg/mL en 1 mL de buffer Tris 0.05 M HCl, 1.5 % p/v deoxicolato de sodio, EDTA 1 mM pH 9.5 y se adicionó a una columna para cromatografía de exclusión por tamaño (GE Healthcare HiPrep™ 16/60 Sephacryl™ S-200 HR) previamente equilibrada con 200 mL del buffer mencionado. Se colectaron fracciones de 2.5 mL (Fraction Collector Bio-Rad Model 2110) a una velocidad de flujo de 0.5 mL/min y se analizaron a 220 nm, 260 nm y 280 nm por espectrofotometría. Las fracciones de interés se determinaron en los picos o regiones con absorbancias más altas; posteriormente se realizó una precipitación con cloruro de sodio a una concentración final de 0.15 M, se agregó un volumen de etanol al 95 % cuatro veces mayor que el volumen de la fracción, a continuación se almacenaron las fracciones a - 20 ºC durante 24 horas y luego se realizó la diálisis y se liofilizó. Se determinó la masa del liofilizado y se reconstituyó en agua USP a una concentración de 1 mg/mL, para su posterior caracterización.
Caracterización de lipopolisacáridos de bacterias periodonto-patógenas. Los extractos purificados fueron caracterizados bioquímicamente por electroforesis SDS-PAGE, el test colorimétrico Purpald, y el test cromogénico de LAL.
Electroforesis SDS PAGE. Se preparó el gel de separación al 12 % y el gel de concentración al 4% (MiniPROTEAN® Tetra Cell. BioRad, USA). Se sembró 5 µg de lipopolisacárido de Porphyromonas gingivalis W83 y Eikenella corrodens ATCC 23834 en buffer Laemmli. También se sembraron como controles lipopolisacáridos comerciales de: Porphyromonas gingivalis ATTC 33277 (InvivoGen cat.#tlrl-3pelps), Rhodobacter sphaeroides DSM 158 (InvivoGen cat.#tlrl-rslps), Escherichia coli 0111:B4 (InvivoGen cat.#tlrl-3pelps), Escherichia coli 0127:B8 (Sigma cat.#L3129), Salmonella typhimurium ATCC 7823 (Sigma cat.#L7261). El gel de concentración se corrió 45 minutos a 0.02 A; el gel de separación se corrió 1 h:30 min a 0.03 A. El proceso de tinción se realizó según lo reportado por Tsai, Frasch, (1982).
Identificación de LPS en los extractos purificados por el ensayo Purpald. El ensayo de Purpald para la identificación de LPS en los extractos purificados de P. gingivales W83 y E. corrodens 23834 fue realizado según lo reportado por Lee & Tsai, (1999). Inicialmente se prepararon diluciones de KDO (ácido 2-ceto-3-desoxioctulosónico) de concentración 0.8 mM, 0.4 mM, 0.2 mM, 0.1 mM, 0.05 mM y 0.025 mM. Para lipopolisacáridos comerciales se prepararon concentraciones de 0,5 de: Salmonella tiphymurium ATCC 7823 (Sigma), P. gingivalis ATTC 33277 (Invivogen), Rhodobacter sphaeroides DSM 158 (Invivogen), E. coli 0111:B4 (Invivogen); y para E. coli 0127:B8 (Sigma) fue de 0.23 mg/mL. Con respecto a los LPS purificados de P. gingivalis W83 y de E. corrodens 23834 se prepararon a una concentración de 0.01 mg/mL. El blanco utilizado fue agua USP. Los datos fueron reportados en unidades de concentración milimolar (mM), teniendo en cuenta la transformación de datos de absorbancia con la curva de concentración conocida de KDO, lo que permitió la identificación del LPS y la cuantificación de KDO presente en las muestras analizadas.
Determinación de la capacidad endotóxica de los extractos purificados por LAL cromogénico. La prueba LAL (Lisado de amebocitos de Limulus) es una prueba sensible y específica para la identificación de endotoxinas. Para la cuantificación se utilizó un kit LAL cromogénico con diazoacoplamiento (Pyrochrome, Cape Cod, USA) con una sensibilidad de 0,005 U.E/mL, según las recomendaciones del fabricante. Se sembraron en una microplaca de 96 pozos (certificada libre de pirógenos, Dynex) las siguientes muestras: blanco (agua libre de pirógenos), curva de control de endotoxina estándar (CSE) de concentraciones 5 U.E/mL, hasta 0,078 U.E/mL, dilución 1:2 y las muestras de LPS a diferentes concentraciones. Se adicionó 50 µL de reactivo LAL pyrochrome suspendido en 3,2 mL de buffer Glucashield (inhibidor de beta glucanos) y se incubó a 37 ºC durante 25 minutos. Las absorbancias fueron registradas en un lector espectrofotométrico de microplacas a longitudes de onda de 570 nm. Cada una de las muestras fue valorada por duplicado y los datos fueron reportados como unidades de endotoxina por mililitro (U.E/mL) a partir de la transformación de datos de absorbancia de la curva de concentración conocida del CSE.
Resultados y discusión
Los cultivos de P. gingivalis W83 y E. corrodens ATCC 23834 se confirmaron por morfología de las colonias con ayuda del microscopio estereoscópico a 4,5 aumentos y con pruebas bioquímicas previamente mencionadas las cuales resultaron concordantes con las bacterias evaluadas. La morfología del cultivo de E. corrodens es característica de colonias tipo 2 (Figura 1A). Las colonias son circulares con un margen irregular y un diámetro menor a 3mm. La colonia contiene un perímetro rugoso, opaca no reflejada y un centro umbonado húmedo que parece hundirse dentro del agar, como ha sido reportado por Chen, Wilson, (1990).
Las colonias de P. gingivalis en agar Brucella suplementado se observan después de la incubación como colonias oscuras que pueden ir desde un marrón claro hasta negro después de más de 7 días de incubación, son convexas con forma de domo brillantes (Figura 1B).
Purificación de lipopolisacáridos. A partir de un peso húmedo de bacteria completa para P. gingivalis y E. corrodens se obtuvo un rendimiento porcentual del 0,7% p/p y 0,02 % p/p, respectivamente (tabla 1). Esto indica que el proceso de extracción y purificación de LPS para P. gingivalis fue más eficiente.
El proceso de purificación se realizó con tratamiento enzimático por nucleasas y proteasas. Posteriormente los extractos crudos eran purificados por cromatografía de exclusión molecular con una matriz de Sephacryl S-200 HR y una fase móvil de buffer de deoxicolato de sodio. El perfil cromatográfico a diferentes longitudes de onda (220, 260 y 280 nm) para los extractos de P. gingivalis y E. corrodens se observan en la figura 2. Para el extracto de P. gingivalis se observa un solo pico a un volumen de elución entre 50-70 mL aproximadamente (figura 2A). De igual manera se observa para el extracto de E. corrodens pero a diferente volumen de elución (80-120 mL, figura 2B). El cromatograma de P. gingivalis indica que el tratamiento enzimático fue mucho más efectivo en reducir contaminación por ácidos nucleicos y proteínas que para el de E. corrodens. Las lecturas de absorbancia a 220 nm se hacen para LPS debido a que el deoxicolato absorbe a la misma longitud de onda (200 nm).
El efecto del tratamiento enzimático para los extractos de P. gingivalis se siguió por barrido espectrofotométrico 200-400 nm como fue reportado por Perdomo, Rolando, (2006), figura 3. Se observa cómo el tratamiento enzimático para degradar RNA, DNA y proteínas permite la depuración del extracto (figuras 3A-3D). Se identifican 2 picos de máxima absorción: el primero a los 223 nm, cercano al espectro de absorción de LPS y el segundo cercano a los 260 nm, lo que sugiere una contaminación por presencia de ácidos nucleícos (figuras 3C y 3D). Luego de la cromatografía de exclusión molecular, esta contaminación se reduce observándose un solo pico (figura 4). Estos resultados concuerdan con lo reportado por otros investigadores, quienes observaron que el principal contaminante luego de la extracción con fenol son los ácidos nucleicos y en menor proporción por proteínas (Perdomo y Rolando 2006; Yi y Hackett, 2000). Además la cromatografía de exclusión molecular con Sephacryl S 200 HR junto con el deoxicolato como fase móvil, permiten aún más la elución de impurezas que se asocian comúnmente a LPS en membranas, como ha sido previamente reportado (Gualtero y cols., 2008; Shands and Chun, 1980; Wu et al., 1987).
Luego de la cromatografía las fracciones de interés fueron recolectadas y sometidas a eliminación del deoxicolato por precipitación con NaCl y Etanol. El extracto de E. corrodens precipitó adecuadamente mientras que el de P. gingivalis no, por lo que se debió realizar una evaporación, seguida de diálisis y liofilización. El precipitado del extracto de E. corrodens fue suspendido en agua LWR, dializado y liofilizado. Se obtuvieron 0,8 mg de E. corrodens y 7,6 mg de P. gingivalis (tabla 1). Estos resultados indican que el proceso de purificación fue más eficiente para P. gingivalis que para E. corrodens. Probablemente la precipitación con NaCl no es eficiente para volver insoluble la muestra en la solución. Con respecto a la precipitación de P. gingivalis, se realizaron además pruebas con acetato de sodio (1M) como agente caotrópico, sin embargo tampoco indujo precipitación (datos no mostrados). Los rendimientos obtenidos en la purificación de LPS con el método de extracción con fenol son esperados que estén por debajo del 2% p/p, y aunque nuestros resultados están por debajo de este valor estos puede deberse a que la bacteria completa fue pesada húmeda y no liofilizada. Actualmente nuevos enfoques están siendo implementados para mejorar el rendimiento de purificación de LPS a un 3%, sin embargo siguen siendo bajos (Rybka et al., 2008).
Los liofilizados fueron reconstituidos en agua LWR a una concentración de 1mg/mL y analizados por barrido espectrofotométrico (figura 4). Se registró los picos máximos de absorción a los 201 nm y 210 nm para P. gingivalis y E. corrodens, respectivamente que corresponde a la longitud de onda a la que absorben los LPS (Figura 4A y 4B). En ambos extractos no se registran picos a 260 y 280 nm, lo que sugiere que los extractos no contienen ácidos nucleicos y proteínas de consideración.
Caracterización de lipopolisacáridos a partir de extractos purificados. Los extractos purificados fueron sometidos a un proceso de caracterización bioquímica mediante electroforesis SDS PAGE, Purpald y test LAL.
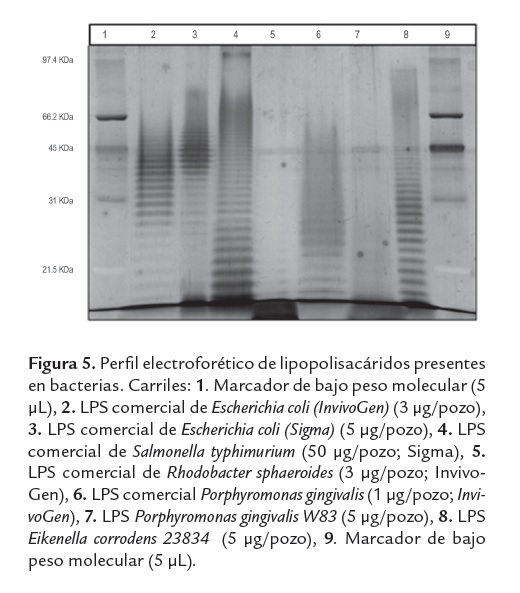
El perfil electroforético permite definir el quimiotipo del LPS (figura 5). Los LPS de tipo liso se presentan en forma de bandas sucesivas en escalera y aquellos que son de tipo semi-rugoso o rugoso pueden presentar bandas de menor peso o la ausencia completa del perfil en escalera (Tsai, Frasch, 1982; Wang et al., 2010).
Los lipopolisacáridos comerciales de E. coli y S. typhimurium presentan un perfil característico de LPS-S (liso), con bandas desde 66 a 14 kDa (carril 2-4, figura 5). El extracto purificado de E. corrodens presenta un perfil característico de LPS-S (carril 8). Por otro lado en el perfil del LPS comercial de R. sphaeroides hay ausencia de un patrón en escalera, pero si se observa una banda de bajo peso molecular por debajo de los 21 kDa. Este perfil es característico de LPS-R (rugosos), donde hay ausencia del antígeno O en la región polisacárido del LPS (Hitchcock & Brown, 1983). Un perfil similar también fue observado en el extracto purificado de P. gingivalis W83, con bandas tenues de bajo peso molecular (carril 7), lo que indica la presencia de LPS-SR (semi-rugoso).
Estos resultados son similares a los reportados previamente por Progulske y Holt, (1984) para LPS de E. corrodens de la cepa 23834. En ese estudio la extracción la hicieron a partir de bacteria completa con fenol-agua caliente y la purificación fue realizada por tratamiento enzimático con nucleasas y proteasas sin un procedimiento de separación de impurezas como la cromatografía o centrifugación. La caracterización del quimiotipo del LPS fue hecha por SDS-PAGE identificando la presencia de LPS-S en los purificados. A diferencia del estudio de Progulske & Holt, con la metodología implementada en este estudio se logró obtener LPS de elevada pureza libre de contaminantes como ácidos nucleicos y proteínas. En lo revisado en la literatura hasta el momento no existen estudios similares que determinen las propiedades bioquímicas y endotóxicas del LPS de E. corrodens.
Igualmente, no se han encontrado hasta el momento reportes en la literatura que describan las propiedades bioquímicas y endotóxicas de LPS de P. gingivalis W83. En un estudio realizado por Sismey-Durrant, Hopps, (1991), utilizó LPS de W83 para estimular fibroblastos in vitro. Sin embargo en ese estudio no se realizó una metodología que permitiera obtener LPS de elevada pureza así como tampoco se hizo la respectiva caracterización del purificado. El uso de métodos de extracción y purificación de LPS son un aspecto clave en la generación de extractos libres de impurezas que permitan su posterior uso en estudios en modelos celulares o in vivo (Hirschfeld et al., 2000; Posch et al., 2013)
La caracterización bioquímica del quimiotipo de LPS de P. gingivalis a partir de las cepas W83 y 33277 sugieren que estas cepas presentan diferencias en los procesos de biosíntesis de polisacáridos presentes en la región del antígeno O de los LPS, como ha sido reportado para la cepa W50 de P. gingivalis, observándose dos tipos de LPS, LPS-O y LPS-A con diferencian en la región polisacárida (Rangarajan et al., 2008). Debido a la importancia de estas moléculas como factores de virulencia, variaciones estructurales del LPS pueden estar relacionadas con procesos de evasión o atenuación del sistema inmune del hospedero (Matsuura M, 2013).
Para verificar la presencia de proteínas en los extractos purificados, se realizó una electroforesis SDS-PAGE y posteriormente teñido con PageBlue (ThermoScientific,USA), que utiliza Comassie G-250 para la tinción de proteínas (figura 6).
En las diferentes muestras, a excepción de la muestra del carril 8 (LPS P. gingivalis W83), se observa una banda con un peso aproximado de 45 kDa (figura 6). Se observa con diferente intensidad en LPS comerciales que han sido purificados por diferentes métodos, según lo reportado en sus insertos (ver número de catálogo en metodología), carriles 2-6. Incluso se observa esta banda en el LPS de E. coli 0111-B4 de InvivoGen® (cat.#tlrl-3pelps), el cual es ultrapurificado con fenol-trietilamina-deoxicolato (carril 3). También se observan otra banda en el LPS comercial de S. typhimurium entre los 31 a 21 kDa (carril 4). Por el contrario, esta banda se observa en menor intensidad en los extractos purificados de E. corrodens (carril 10), P. gingivalis W83 (carril 7), y P. gingivalis 33277 (carril 9), obtenidos en este y previos estudios (Gualtero y cols., 2008). Estos resultados sugieren que los LPS obtenidos con la metodología implementada en este estudio permite la obtención de lipopolisacáridos con pureza igual o superior a los obtenidos comercialmente.
La presencia de proteínas o lipopéptidos formando complejos con LPS purificados ha sido reconocida y reportada en varios estudios (Hirschfeld et al., 2000; Manthey , Vogel, 1994; Reddi et al., 1995). Sin embargo, el uso de fenol en el proceso de re-purificación es el que más elimina este tipo de contaminación mezclado con otros reactivos como la trietilamina (Hirschfeld et al., 2000; Yi, Hackett, 2000; Wang et al., 2010). También se han propuesto otros protocolos que no utilizan fenol para la re-purificación de LPS, eliminando los complejos proteínas-LPS asociados utilizando solventes no inflamables (Lin et al., 2005; Tirsoaga et al., 2007). Nosotros utilizamos liofilizado del extracto purificado de P. gingivalis W83 reconstituido en buffer LaemmLi 2X y desnaturalizado por medio de ebullición posteriormente, observando una disminución en la señal a la altura de 45 kDa (figura 6, carril 8). Probablemente el complejo LPS-proteína en la muestra fue disociada por la acción denaturante del buffer (LaemmLi, 1970).
Para cuantificar la concentración de proteína o péptidos presentes en los LPS comerciales y los extractos purificados de E. corrodens 23834 y P. gingivalis W83, se realizó una determinación por el método del ácido bicinconínico (Smith et al., 1985), utilizando un kit comercial con una curva estándar de albúmina desde 2000 µg/mL a 25 µg/mL (Thermo Scientific Pierce BCA Protein Assay Kit. USA). Las muestras fueron diluidas en agua libre de endotoxinas o con buffer DOC a una concentración de 400 µg/mL. Por esta metodología no se logró identificar y cuantificar proteína en ninguna de las muestras (datos no mostrados). Teniendo en cuenta estos resultados contradictorios no podemos asegurar la presencia de contaminación por péptidos o proteínas en las muestras de LPS comerciales y los extractos purificados. Se requiere de más experimentación que nos permita aclarar la naturaleza de las moléculas detectadas con una masa de 45 kDa por electroforesis SDS-PAGE.
Identificación de LPS en los extractos purificados por el Ensayo Purpald. Los LPS presentan en el núcleo de la región polisacárida, KDO y heptosas característicos en este tipo de glicocongujados (Lee and Tsai, 1999). Mediante el ensayo Purpald se realizó la identificación de LPS en los extractos purificados de E. corrodens y P. gingivalis (tabla 2). Se observa en los diferentes LPS (comerciales o purificados) que no hay una correspondencia en cuanto a la concentración de la muestra y la concentración relativa de LPS en relación al KDO. Estas diferencias se deben a variaciones en el número de glicoles vecinales sin sustituir (UTVG) presentes en el KDO y heptosas al interior de cada uno de los LPS analizados (Lee and Tsai, 1999).
De esta manera se identifica la presencia de LPS en los extractos purificados de E. corrodens 23834 y P. gingivalis W83, observándose una mayor concentración de KDO en P. gingivalis que en E. corrodens, a pesar que fueron colocados a una misma concentración del extracto en solución.
* Los valores de concentración milimolar relativa para cada lipopolisacárido fueron calculados a partir de una curva de calibración de KDO de concentración conocida.
Hasta el momento no se ha determinado a nivel molecular y estructural la región nuclear del LPS para P. gingivalis W83 y E. corrodens 23834. Sin embargo, se ha identificado por espectrometría de masas acoplada a cromatografía de gases (GC-MS), la presencia de KDO en LPS de P. gingivalis y en algunas cepas como W50, la ausencia de heptosas en la región nuclear, con LPS que presentan un quimiotipo liso (Kumada et al., 1993; Paramonov et al., 2009; Rangarajan et al.,). La presencia de KDO y heptosas presentes en LPS de E. corrodens 23834 fue identificada en baja concentración en estudios previos por el método ácido tiobarbitúrico y de Wright (Progulske and Holt, 1984). Esos resultados se correlacionan con los obtenidos en este estudio con el ensayo Purpald.
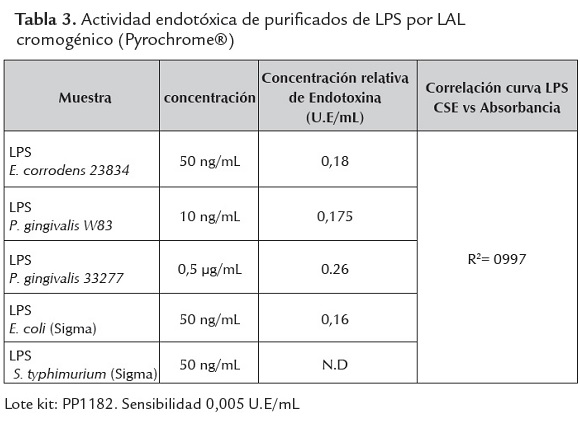
Por último se determinó la capacidad endotóxica de los lipopolisacáridos purificados de E. corrodens y P. gingivalis por la técnica cromogénica del lisado de amebocitos de Limulus (LAL; Pyrocrome., Cape Code. USA). Esta técnica es la más sensible y específica para la cuantificación de endotoxinas (Ide et al., 2004; Lindsay et al., 1989; Pussinen et al., 2007). Se observa que los LPS purificados de P. gingivalis W83 y E. corrodens 23834 tienen una mayor actividad endotóxica a bajas concentraciones, similar a las observadas por el LPS de E. coli (tabla 3). Mientras que, el LPS de P. gingivalis 33277 a elevadas concentraciones no presenta buena reactividad con este test. Sin embargo, deberá aumentarse el tamaño de muestras de la prueba para confirmar estos resultados estadísticamente.
En el caso del LPS de S. typhimurium la actividad no fue determinada debido a que la reacción estaba por encima del límite superior de la curva del control estándar de endotoxina, lo que sugiere una mayor potencia endotóxica para este LPS. Aunque se ha reportado que la actividad endotóxica para algunos LPS no es reflejada en el ensayo LAL (Dehus et al., 2006), estos resultados sugieren que los LPS purificados a partir de E. corrodens y P. gingivalis son endotoxinas y presentan actividad biológica.
Conclusiones
La metodología implementada en este estudio, utilizando diferentes técnicas previamente reportadas para la extracción, purificación y caracterización de LPS, permitió la obtención de LPS de elevada pureza a partir de cultivos de bacteria completa de P. gingivalis W83 y E. corrodens 23834. La caracterización bioquímica de los extractos purificados determinó la presencia de LPS-S para la cepa 23834 de E. corrodens y de LPS-SR para la cepa W83 de P. gingivalis. Ambos LPS purificados fueron positivos para KDO y presentaron actividad endotóxica.
Agradecimientos
Este trabajo fue financiado para la purificación de LPS de E. corrodens 23834 por la Fundación para la promoción de la Investigación y la Tecnología del Banco de la República (proyecto 2,706). Además fue financiado para la purificación de LPS de P. gingivalis W83 por el Departamento Administrativo de Ciencia Tecnología e Innovación COLCIENCIAS (proyecto: 130851928960). Queremos también agradecer a la División de Investigaciones de la Universidad El Bosque por la financiación y gestión administrativa para la ejecución de estos proyectos.
A la memoria del profesor Gerardo Pérez del laboratorio de Bioquímica, Departamento de Química de la Universidad Nacional de Colombia, por sus asesorías en la purificación por cromatografía del extracto de E. corrodens.
Conflicto de interés
Los autores expresamos que no existe ningún conflicto de interés en la publicación de este estudio.
Referencias bibliográficas
1 Apicella, M.A. 2008. Isolation and characterization of lipopolysaccharides. Methods in molecular biology (Clifton, N.J.). 431, pp. 3-13. [ Links ]
2 Chen, C. K., Wilson, M.E. 1990. Outer membrane protein and lipopolysaccharide heterogeneity among Eikenella corrodens isolates. The Journal of Infectious Diseases. 162(3): 664-671. [ Links ]
3 Curtis, M.A., Slaney, J.M., Aduse-Opuko, J. 2005. Critical pathways in microbial virulence. Journal of clinical periodontology. 32(6): 28-38. [ Links ]
4 Darveau, R.P., Hancock, R.E. 1983. Procedure for isolation of bacterial lipopolysaccharides from both smooth and rough Pseudomonas aeruginosa and Salmonella typhimurium strains. Journal of Bacteriology. 155(2): 831-838. [ Links ]
5 Dehus, O., Hartung, T., Hermann, C. 2006. Endotoxin evaluation of eleven lipopolysaccharides by whole blood assay does not always correlate with limulus amebocyte lysate assay. Journal of Endotoxin Research. 12(3): 171-180. [ Links ]
6 Elkaim, R., Dahan, M., Kocgozlu, L., Werner, S., Kanter, D., Kretz, J.G., and Tenenbaum, H. 2008. Prevalence of periodontal pathogens in subgingival lesions, atherosclerotic plaques and healthy blood vessels: A preliminary study. Journal of Periodontal Research. 43(2): 224-231. [ Links ]
7 Galanos, C., Lüderitz, O., Westphal, O. 1969. A new method for the extraction of R lipopolysaccharides. European Journal of Biochemistry. 9(2): 245-249. [ Links ]
8 Gualtero, D., Castellanos, J.E., Pérez, G., Lafaurie, G.I. 2008. Purificación de lipopolisacáridos de Porphyromonas gingivalis libre de polisacáridos utilizando cromatografía de alta resolución Sephacryl S-200. Acta Biológica Colombiana. 13(3): 147-160. [ Links ]
9 Hirschfeld, M., Ma, Y., Weis, J.H., Vogel, S.N., and Weis, J.J. 2000. Cutting edge: Repurification of lipopolysaccharide eliminates signaling through both human and murine toll-like receptor 2. Journal of Immunology. 165(2): 618-622. [ Links ]
10 Hitchcock, P.J., Brown, T.M. 1983. Morphological heterogeneity among Salmonella lipopolysaccharide chemotypes in silver-stained polyacrylamide gels. Journal of Bacteriology. 154(1): 269-277. [ Links ]
11 Ide, M., Jagdev, D., Coward, P.Y., Crook, M., Barclay, G.R., and Wilson, R.F. 2004. The short-term effects of treatment of chronic periodontitis on circulating levels of endotoxin, C-reactive protein, tumor necrosis factor-alpha, and interleukin-6. Journal of Periodontology. 75(3): 420-428. [ Links ]
12 Jaramillo, R., Suárez, P., Barraza, B., Lara, P., Teherán, L., Escamilla, J. 2006. Eikenella corrodens: Pathogenesis and clinic aspects. Colombia Médica. 37(3): 228-241. [ Links ]
13 Kallio, K.A., Buhlin, K., Jauhiainen, M., Keva, R., Tuomainen, A.M., Klinge, B., Pussinen, P.J. 2008. Lipopolysaccharide associates with pro-atherogenic lipoproteins in periodontitis patients. Innate Immunity. 14(4): 247-253. [ Links ]
14 Kebschull, M., Demmer, R.T., Papapanou, P.N. 2010. "Gum bug, leave my heart alone!"--epidemiologic and mechanistic evidence linking periodontal infections and atherosclerosis. Journal of Dental Research. 89(9): 879-902. [ Links ]
15 Kumada, H., Kondo, S., Umemoto, T., & Hisatsune, K. 1993. Chemical structure of the 2-keto-3-deoxyoctonate region of lipopolysaccharide isolated from Porphyromonas (bacteroides) gingivalis. FEMS Microbiology Letters. 108(1): 75-79. [ Links ]
16 LaemmLi, U.K. 1970. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227(5259): 680-685. [ Links ]
17 Lafaurie, G.I., Mayorga-Fayad, I., Torres, M.F., Castillo, D.M., Aya, M.R., Baron, A., and Hurtado, P.A. 2007. Periodontopathic microorganisms in peripheric blood after scaling and root planing. Journal of Clinical Periodontology. 34(10): 873-879. [ Links ]
18 Lamont, R.J., Jenkinson, H.F., 1998. Life below the gum line: Pathogenic mechanisms of Porphyromonas gingivalis. Microbiology and Molecular Biology Reviews: MMBR, 62(4): 1244-1263. [ Links ]
19 Lee, C.H., Tsai, C.M., 1999. Quantification of bacterial lipopolysaccharides by the Purpald assay: Measuring formaldehyde generated from 2-keto-3-deoxyoctonate and heptose at the inner core by periodate oxidation. Analytical Biochemistry. 267(1): 161-168. [ Links ]
20 Lin, M.F., Williams, C., Murray, M.V., and Ropp, P.A. 2005. Removal of lipopolysaccharides from protein-lipopolysaccharide complexes by nonflammable solvents. Journal of Chromatography. B, Analytical Technologies in the Biomedical and Life Sciences. 816(1-2): 167-174. [ Links ]
21 Lindsay, G.K., Roslansky, P.F., Novitsky, T.J. 1989. Single-step, chromogenic limulus amebocyte lysate assay for endotoxin. Journal of Clinical Microbiology. 27(5): 947-951. [ Links ]
22 Manthey, C.L., Vogel, S.N. 1994. Elimination of trace endotoxin protein from rough chemotype LPS. Journal of Endotoxin Research. 1(2): 84-91. [ Links ]
23 Marchesan, J.T., Morelli, T., Lundy, S.K., Jiao, Y., Lim, S., Inohara, N., Giannobile, W.V. 2012. Divergence of the systemic immune response following oral infection with distinct strains of Porphyromonas gingivalis. Molecular Oral Microbiology. 27(6): 483-495. [ Links ]
24 Matsuura, M. 2013. Structural modifications of bacterial lipopolysaccharide that facilitate Gram-negative bacteria evasion of host innate immunity. Frontiers in immunology. 4: 109. [ Links ]
25 Mayorga-Fayad, I., Lafaurie, G.I., Contreras, A., Castillo, D.M., Baron, A., and Aya Mdel, R. 2007. Subgingival microbiota in chronic and aggressive periodontitis in Bogotá, Colombia: An epidemiological approach. Microflora subgingival en periodontitis crónica y agresiva en Bogotá, Colombia: un acercamiento epidemiológico. Biomedica: Revista Del Instituto Nacional De Salud. 27(1): 21-33. [ Links ]
26 Morrison, D.C., Leive, L. 1975. Fractions of lipopolysaccharide from Escherichia coli O111:B4 prepared by two extraction procedures. The Journal of Biological Chemistry. 250(8): 2911-2919. [ Links ]
27 Padilla, E.C., Lobos, G.O., Jure, O.G., Matus, F.S., Descouvieres, C.C., Hasbun, A.S., Nunez, F.L. 2007. Isolation of periodontal bacteria from blood samples and atheromas in patients with atherosclerosis and periodontitis. Aislamiento de bacterias periodontopaticas desde hemocultivos y ateromas obtenidos de pacientes con aterosclerosis y periodontitis Revista Médica De Chile. 135(9): 1118-1124. [ Links ]
28 Paramonov, N.A., Aduse-Opoku, J., Hashim, A., Rangarajan, M., Curtis, M.A. 2009. Structural analysis of the core region of O-lipopolysaccharide of Porphyromonas gingivalis from mutants defective in O-antigen ligase and O-antigen polymerase. Journal of Bacteriology. 191(16): 5272-5282. [ Links ]
29 Perdomo R., Montero, V. 2006. Purificación de lipopolisacáridos de E. coli 055:B5 por cromatografía de exclusión molecular. Biotecnología Aplicada. 23:117-123. [ Links ]
30 Posch, G., Andrukhov, O., Vinogradov, E., Lidner, B., Messner, P., Holst, O. and Schaffer, C., 2013. Structure and immunogenicity of the rough-type lipopolysaccharide from the periodontal pathogen Tannerella forsythia. Clinical and vaccine immunology: CVI. 20(6): 945-953. [ Links ]
31 Progulske, A., Holt, S.C. 1984. Isolation and characterization of the outer membrane and lipopolysaccharide from Eikenella corrodens. Infection and Immunity. 43(1): 166-177. [ Links ]
32 Pussinen, P.J., Tuomisto, K., Jousilahti, P., Havulinna, A.S., Sundvall, J., and Salomaa, V., 2007. Endotoxemia, immune response to periodontal pathogens, and systemic inflammation associate with incident cardiovascular disease events. Arteriosclerosis, Thrombosis, and Vascular Biology. 27(6): 1433-1439. [ Links ]
33 Raetz, C.R. and Whitfield, C. 2002. Lipopolysaccharide endotoxins. Annual Review of Biochemistry. 71:635-700. [ Links ]
34 Rangarajan, M., Aduse-Opoku, J., Paramonov, N., Hashim, A., Bostanci, N., Fraser, O.P., Curtis, M.A. 2008. Identification of a second lipopolysaccharide in Porphyromonas gingivalis W50. Journal of Bacteriology. 190(8): 2920-2932. [ Links ]
35 Reddi, K., Poole, S., Nair, S., Meghji, S., Henderson, B., and Wilson, M. 1995. Lipid A-associated proteins from periodontopathogenic bacteria induce interleukin-6 production by human gingival fibroblasts and monocytes. FEMS Immunology and Medical Microbiology. 11(2): 137-144. [ Links ]
36 Rybka, J., Grycko, P., Francisco, J.D.C., Gamian, A. and Dey, E.S. 2008. Application of supercritical carbon dioxide (scCO2) for the extraction of lipopolysaccharides (LPS) from Salmonella enterica subsp. enterica PCM 2266. The Journal of Supercritical Fluids. 45(1): 51-56. [ Links ]
37 Shands, J.W.,Jr, Chun, P.W. 1980. The dispersion of gram-negative lipopolysaccharide by deoxycholate. Subunit molecular weight. The Journal of Biological Chemistry. 255(3): 1221-1226. [ Links ]
38 Sismey-Durrant, H.J., Hopps, R.M. 1991. Effect of lipopolysaccharide from Porphyromonas gingivalis on prostaglandin E2 and interleukin-1-beta release from rat periosteal and human gingival fibroblasts in vitro. Oral Microbiology and Immunology. 6(6): 378-380. [ Links ]
39 Smith, P.K., Krohn, R.I., Hermanson, G.T., Mallia, A.K., Gartner, F.H., Provenzano, M.D., Klenk, D.C. 1985. Measurement of protein using bicinchoninic acid. Analytical Biochemistry. 150(1): 76-85. [ Links ]
40 Thomopoulos, C., Tsioufis, C., Soldatos, N., Kasiakogias, A., and Stefanadis, C. 2011. Periodontitis and coronary artery disease: A questioned association between periodontal and vascular plaques. American Journal of Cardiovascular Disease. 1(1): 76-83. [ Links ]
41 Tirsoaga, A., Novikov, A., Adib-Conquy, M., Werts, C., Fitting, C., Cavaillon, J.M., Caroff, M. 2007. Simple method for repurification of endotoxins for biological use. Applied and Environmental Microbiology. 73(6): 1803-1808. [ Links ]
42 Tsai, C.M., Frasch, C.E. 1982. A sensitive silver stain for detecting lipopolysaccharides in polyacrylamide gels. Analytical Biochemistry. 119(1): 115-119. [ Links ]
43 Wang, X., Zhang, C., Shi, F., Hu, X., 2010. Purification and characterization of lipopolysaccharides. Sub-Cellular Biochemistry. 53: 27-51. [ Links ]
44 Wetsphal O, Jann K. 1965. Bacterial lipopolysaccharides. Extraction with phenol-water and further aplication of the procedure. in: In New York: Academic Press (Ed.), Methods in carbohydrate chemistry. pp. 83-91 [ Links ]
45 Wiedermann, C.J., Kiechl, S., Dunzendorfer, S., Schratzberger, P., Egger, G., Oberhollenzer, F., Willeit, J., 1999. Association of endotoxemia with carotid atherosclerosis and cardiovascular disease: Prospective results from the bruneck study. Journal of the American College of Cardiology. 34(7): 1975-1981. [ Links ]
46 Wu, L.H., Tsai, C.M., Frasch, C.E. 1987. A method for purification of bacterial R-type lipopolysaccharides (lipooligosaccharides). Analytical Biochemistry. 160(2): 281-289. [ Links ]
47 Yi, E.C., Hackett, M. 2000. Rapid isolation method for lipopolysaccharide and lipid A from gram-negative bacteria. The Analyst. 125(4): 651-656. [ Links ]