Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Colombiana de Biotecnología
versão impressa ISSN 0123-3475
Rev. colomb. biotecnol vol.16 no.1 Bogotá jan./jun. 2014
https://doi.org/10.15446/rev.colomb.biote.v16n1.44287
http://dx.doi.org/10.15446/rev.colomb.biote.v16n1.44287
ARTÍCULO DE REVISIÓN
Rol biológico y aplicaciones de los miRNAs en cáncer de seno
Biological role and applications of miRNAs in breast cancer
Yeimy Viviana Ariza Márquez1, Ángela Patricia Beltrán López2, Ignacio Briceño Balcázar3 y Fabio Ancizar Aristizabal4.
1 Bacterióloga y laboratorista clínica UCMC, cPhD biotecnología Universidad Nacional de Colombia, yvarizam@unal.edu.co
2 Bacterióloga y laboratorista clínica UCMC, cPhD biotecnología Universidad Nacional de Colombia, apbeltranl@unal.edu.co
3 Profesor titular Universidad de la Sabana y profesor Pontificia Universidad Javeriana, ibriceno@javeriana.edu.co
4 Director Instituto de Biotecnología de Universidad Nacional de Colombia, faaristizabalg@unal.edu.co
Recibido: abril 24 de 2013 Aprobado: abril 4 de 2014
Resumen
Los miRNAs son pequeños RNAs que participan en diversos procesos de regulación génica, mediante ribointerferencia y juegan un papel clave en diversos procesos biológicos, tales como proliferación celular, diferenciación y apoptosis. En consecuencia, la expresión alterada de miRNAs contribuye a la enfermedad humana, incluyendo cáncer. En esta revisión, nos centraremos en los recientes hallazgos de miRNAs que inciden en el desarrollo de cáncer y particularmente en cáncer de seno, simultáneamente evaluaremos sus mecanismos de regulación, su clasificación, su uso como marcadores de invasión tumoral, de sensibilidad a fármacos y adicionalmente exploraremos la utilidad de los miRNAs en el diagnóstico, seguimiento y tratamiento individualizo. Finalmente encontramos que los miRNAs representan una gran alternativa para entender las bases moleculares de los procesos tumorales implícitos en cáncer de seno y una vez se conozcan todas sus dianas, será posible dilucidar al menos en parte este proceso complejo y multigénico, ayudado mediante herramientas como la generación de bases de datos, para reportan la expresión diferencial de miRNAs, elementos que nos permitirá realizar medicina preventiva y mejorar la calidad de vida de los pacientes y sus familias.
Palabras clave: cáncer de seno, miRNAs, anti-oncomir, oncomir, regulación post-transcripcional, RNAm.
Abstract
MiRNAs are small RNAs that are involved in various processes of gene regulation by RNAi and play a key role in various biological, such as cell proliferation, differentiation and apoptosis processes. Consequently, the altered expression of miRNAs contribute to human disease, including cancer. In this review, we will focus on the recent findings of miRNAs that affect the development of cancer, particularly breast cancer, simultaneously evaluate their regulatory mechanisms , their classification , their use as markers of tumor invasion, drug susceptibility and further explore the utility of miRNAs in the diagnosis, monitoring and individualize treatment. Finally found that miRNAs represent a great alternative for understanding the molecular basis of implicit tumor processes in breast cancer and once all targets are known, it will be possible to elucidate at least in part this complex and multigenic process, aided by tools such as generation of databases, to report the differential expression of miRNAs, elements that allow us to preventive medicine and improve the quality of life of patients and their families.
Key words: breast cancer, miRNAs, anti-oncomir, Oncomir, post-transcriptional regulation, RNAm.
Introducción
En octubre de 2001 tres publicaciones de la revista "Science", indicaron el rol de pequeños ncRNAs o sRNAs (non coding o small RNAs, RNAs no codificantes o pequeños, por sus siglas en inglés) en el desarrollo del nemátodo Caenorhabditis elegans (Lagos-Quintana et al., 2001; Lau et al.,2001; Lee y Ambros 2001). Posteriormente el Premio Nobel de Fisiología o Medicina de 2006 fue concedido a Andrew Fire y Craig Mello por su descubrimiento de los RNAs de interferencia (RNAi) dentro de los que identificaron los siRNAs, (small interfering RNA, en español ARN pequeño de interferencia o ARN de silenciamiento), los micro ARN (miARN o miRNA por sus siglas en inglés), y los ARNs asociados a Piwi (Piwi-ARNs o piRNAs por sus siglas en inglés), de los tres se identificó que su mecanismo de acción es el silenciamiento génico mediado por RNAs de cadena sencilla, que siendo complementarios a un RNA mensajero (RNAm), conducen habitualmente a la degradación de éste último. En esta revisión exploraremos la biogénesis, características y dianas terapéuticas de los miRNAs, enfatizando en cáncer de seno.
Los miRNAs son moléculas claves en la regulación de diversos procesos celulares dentro de los que es posible citar; proliferación, diferenciación, movilidad, invasión, muerte celular, entre otras, (Li et al., 2010) debido a esto, se ha generado un interés particular en la investigación del rol que pueden desempeñar en cáncer. Junto a esto se han identificado como un mecanismo clave del control post-transcripcional dentro de las redes de regulación génica. En consecuencia ellos surgen como importantes mediadores en la tumorogénesis, dando lugar a un cambio de paradigma en oncología donde se busca identificar posibles dianas para encaminarnos hacia una medicina personalizada, donde el uso de microarrays de miRNA ha permitido la identificación de un gran número de miRNAs, como biomarcadores para cáncer. En esta vía es interesante reconocer que los miRNAs tienen una estabilidad notable, en tejido embebido en parafina y muestras de sangre con lo cual se perfilan como marcadores tumorales para ser usados en medicina personalizada.
Múltiples estudios han evidenciado que la expresión anormal de uno o más miRNAs, está asociado con la patogénesis de enfermedades humanas incluyendo tumores sólidos y hematopoyéticos. (Calin y Croce 2006). El patrón de expresión de los miRNAs y perfil global de expresión de miRNAs es conocido como "miRNoma" (Yong y Anindya 2009), y a partir de este, se ha identificado que ciertos miRNAs pueden actúan como oncogenes, genes supresores de tumor, incidir en procesos metástasicos e incluso pueden actuar como moduladores de células madre. En presencia de una regulación negativa de los miRNAs se les ha encontrado actividad como supresores de tumor, en contraste la acumulación de subconjuntos de estos, los involucra en funciones como oncogenes. Lo cual ha sido evidenciado en diferentes estudios, como por ejemplo la baja regulación de let- 7 en cáncer de pulmón (Takamizawa et al., 2004), eliminación de miR-15 y miR-16 en leucemia linfocítica crónica (LLC) (Cimmino et al., 2005), y el hallazgo de miR-17-5p y miR-20a en el control de la muerte celular y proliferación (O'Donnell et al., 2005; Croce CM 2009; Shimono et al., 2009). De igual modo se ha reportado regulación transcripcional o epigenética de miRNAs en cáncer (Lujambio et al., 2007; Saito et al., 2006). En las investigaciones actuales no sólo se identifican miRNA como biomarcadores, sino que adicionalmente se determina el gen que tienen como diana y los mecanismos subyacentes que pueden estar involucrados. El rápido descubrimiento de muchos de los blancos de los miRNA y sus vías, ha contribuido al desarrollo de productos terapéuticos basados en miRNA, pero los avances en el desarrollo de fármacos a partir de secuencias antisentido o siRNA se ha visto obstaculizado por la estabilidad, la especificidad y los problemas de entrega al RNAm diana.
En la última década, el número de genes humanos que se sabe son regulados por miRNAs ha aumentado rápidamente (He y Hannon 2004; Chen C. Z., 2005), esto mediante un número creciente de estudios que han demostrado que la expresión de miRNAs se correlaciona con patrones diferenciales que se ven afectados por el tejido implicado y grado de especialización, del mismo modo se ha determinado que existe un patrón de expresión diferencial entre los diversos tipos de cáncer mediante estudios de cohortes que incluyen casos y controles. (Lu et al., 2005). Hasta la fecha más de 24.521 miRNAs precursores han sido registrados en miRBase (Versión miRBase 20) y se prevé que el 30% de estos miRNAs, regulan genes codificantes de proteínas en el genoma humano (Kent y Mendell 2006).
En cáncer de seno se ha establecido que existen patrones de expresión de miRNAs asociados al fenotipo de los diferentes subtipos de cáncer, donde el análisis de los patrones de expresión de miRNA ha llevado a la identificación de firmas que pueden diferenciarse del tumor de los tejidos normales (Lorio et al., 2005 y Blenkiron et al., 2007). Análisis de RNAm objetivo de miRNAs con expresión diferencial en los tejidos normales y tumorales de seno, indica que su expresión aberrante impacta la regulación de las redes celulares importantes conocidas para conducir a cáncer de seno (O'Day E, Lal A 2010). Esto se sustenta en la observación de que varias características de tumor de seno clínicamente relevantes, como el tamaño del tumor, la afectación ganglionar, invasión vascular, los receptores hormonales y el estado de HER2, también están relacionados con la expresión de microRNAs particulares (Lorio et al., 2005 ; Blenkiron et al., 2007 y Lowery et al., 2009]. Además, los microARNs podrían ser utilizados como marcadores de la potencial metastásico de los tumores primarios de seno. (Tavazoie et al., 2008).
El cáncer de seno es una enfermedad genética de fenotipo complejo, que representa a nivel mundial el cáncer más común en mujeres (Veronesi et al., 2005; Alvarez RH. 2010) no obstante actualmente se cuenta con herramientas que garantizan un mejor pronóstico debido a los avances en el diagnóstico, mediante el examen clínico de la glándula, el autoexamen, la mamografía, en la misma línea el manejo de las pacientes en el tratamiento mediante radioterapia, quimioterapia, hormonoterapia, acompañado del estudio de la enfermedad para el entendimiento de las bases genéticas de esta, con el fin de generar una medicina personalizada, donde se evalué en cada paciente su carga genética y susceptibilidad asociada, además factores que puedan desencadenar riesgo a presentar la enfermedad están permitiendo mejorar el pronóstico en dichas pacientes e incidir en el mejoramiento de la calidad de vida de las mismas.
Esta revisión recopila las publicaciones científicas originales más importantes emitidas a la fecha, que reportan la participación de miRNAs en cáncer, centrándose en su incidencia en cáncer de seno, explorando su potencial como blancos terapéuticos y retos futuros.
Definición de microRNA
miRNAs son una clase de RNAs no codificantes que participan en la regulación de la expresión génica. Los miRNAs son fragmentos cortos de RNA, de 19 a 25 nucleótidos que se unen al RNAm objetivo donde inhiben su traducción o causan la degradación de este (Bentwich et al., 2005). De acuerdo a la base de datos de secuencias miRBase (publicada en junio de 2013, versión 20) más de 24.521 miRNAs precursores han sido registrados, de los cuales 30424 son productos maduros de miRNAs, de 206 especies. Se prevé que el 30% de estos miRNAs, regulan genes codificantes de proteínas en el genoma humano. (Kent y Mendell 2006). A pesar de este gran número de miRNAs identificados, el RNAm destino y la función biológica se ha atribuido a sólo unos de ellos. Con estos lineamientos ha sido claramente demostrado que algunos miRNAs, influyen en el desarrollo y progreso de cáncer (Calin y Croce 2006). Por lo tanto, la identificación de la expresión diferencial de miRNAs, estableciendo sus funciones asociadas con cáncer y el desarrollo de formas para modular sus actividades pueden producir poderosas estrategias terapéuticas contra el cáncer.
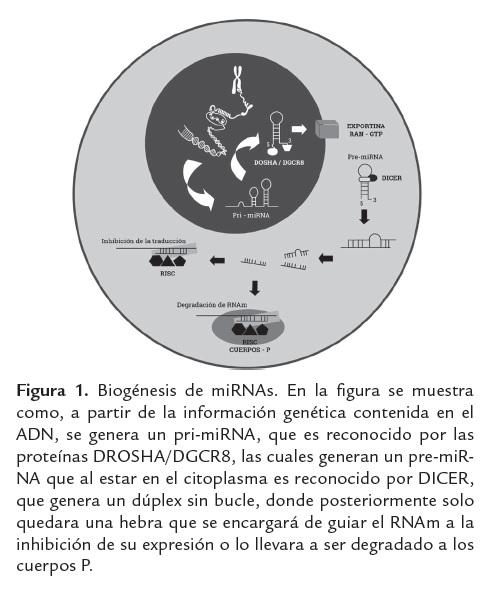
Los miRNAs son secuencialmente transformados en moléculas precursoras que son codificadas por genes miRNA (Bartel DP 2004) (figura 1). Los genes que generan los miRNA están conservados evolutivamente y pueden estar situadas dentro de intrones o exones de genes que codifican proteínas (70%) o en áreas intergénicas (30%) (Rodriguez et al., 2004). La mayor parte de los miRNAs intrónicos o exónicos se orientan en paralelo con su gen anfitrión, lo que sugiere que se transcribe de forma simultánea, mientras que el grupo restante de miRNAs se transcribe de las regiones intergénicas o de genes que componen unidades independientes de transcripción (Rodriguez et al., 2004).
Los genes miRNA se denominan con el mismo nombre (mir) escrito en cursiva para distinguirlos de los miRNA correspondientes al producto maduro (denominado miR) seguido de un número, por ejemplo, miR-1, los miRNAs de similares secuencia se distinguen por una letra adicional (a, b, c...) seguido del número del miRNA (por ejemplo, miR-125b). Un miRNA de secuencia madura idéntica puede aparecer en varios loci del genoma con secuencias de diferentes precursores. En este caso, los genes miRNA diferentes se distinguen por la adición de otro número (1, 2, 3...) al final (por ejemplo miR-125b-1).
Biogénesis de los microRNAs.
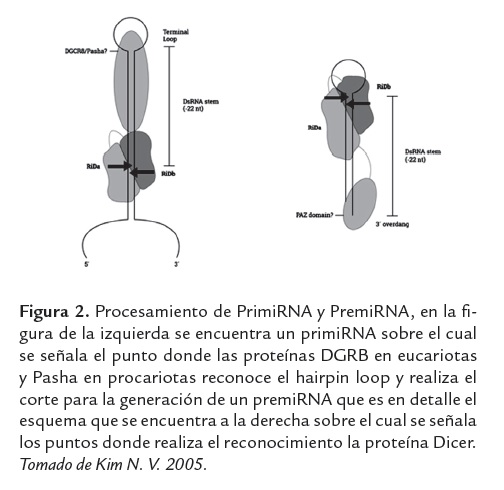
Los miRNAs son procesados a partir de un transcrito de alrededor de 70 a 90 nucleótidos, el cual tiene una estructura de "hairpin", "hairpin loop" o "Stem-loops" (Hutvanger y Zamore 2002) por la acción de la RNA polimerasa II. Los Stem-loops son RNA de cadena sencilla, con una secuencia de nucleótidos que se pliegan sobre sí mismos para formar una doble hélice con una región en la secuencia que presenta complementariedad imperfecta, formando un lazo abierto en uno los extremos, con un casquete (7-metilguanosina) en el extremo 5´ y una cola poli (A) en el extremo 3´. Los transcritos primarios que contienen una sola estructura Stem-loops se les denominan monocistrónicos y aquellos que poseen más de un hairpin se les conocen como policistrónico. Sin embargo, otras vías pueden generar un conjunto menor de miRNAs, en especial de repeticiones genómicas, por ejemplo, la RNA polimerasa III que es responsable de la transcripción de miRNAs en las repeticiones Alu. (Borchert et al., 2009). Tras la síntesis, del pri-miRNA este se transforma inicialmente en el interior del núcleo por la RNAsa II (Drosha) y el DGCR8 (proteína de unión a RNA de cadena doble, región crítica DiGeorge) donde se da paso al precursor miRNA (pre-miRNA) de ~ 70 nucleótidos figura 2a. (Lee et al., 2003). Pri-miRNA es transportado al citoplasma por el factor de exportación nuclear (Exportin-5/Ran-GTP), donde se procesa a través de una serie de cortes por la endonucleasa RNAsa III, Dicer TRBP / y Ago2 para generar un dúplex miRNA maduro de 17-25 pb, figura 2b.(Bentwich I, 2005). El duplex miRNA es procesado por la helicasa A quedando un miRNA de cadena simple llamada hebra guía y degradándose la segunda hebra*, luego la hebra guía entra al complejo RISC que dirige el proceso, produciendo el silenciamiento del RNAm diana. (Berezikov et al., 2006). Es comúnmente aceptado que los miRNAs maduros regulan la expresión génica mediante la unión a la región 3' no traducida (3'-UTR) del RNAm diana, provocando la degradación del RNAm o la inhibición de su traducción. Figura 1,(Bentwich I 2005; Berezikov et al., 2006; Bernstein et al., 2001; Hutvagner et al., 2001). El miRNA maduro es parcialmente complementario a uno o varios RNAm (Jackson et al., 2007; Pillai et al., 2007), la especificidad, en la elección de destino de los miRNAs, se define principalmente por la complementariedad de la secuencia entre los sitios de destino del RNAm y la secuencia de nucleótidos de la posición 2-8 en el extremo 5' del miRNA, conocido como " Región seed" (Bernstein et al., 2001; Brennecke et al., 2005).
En eucariotas la degradación del RNAm puede ocurrir mediante dos vías, donde cada una de ellas es iniciada por un acortamiento gradual de la cola poli A del RNAm. En el primer caso, la secuencia del RNAm puede ser degradado en sentido 3´- 5´ por una progresiva descomposición, por exosoma, o en el segundo caso por extracción de la caperuza seguido por la degradación de la hebra en sentido 5´- 3´, catalizada por la exonucleasa XRN1 (Filipowicz et al., 2008). Los niveles de RNAm son controlados por ribonucleoproteinas (RNPm) a través del reclutamiento de los componentes de la maquinaria de degradación, lo que lleva a la deadenilación y pérdida de la caperuza del RNAm. En lo que concierne a la degradación o por lo menos sus pasos finales, se cree que ocurre en las estructuras celulares llamadas cuerpos-P, estructuras que poseen enzimas catabólicas del RNAm (GW182 y RCK/p54) y represores trasduccionales.
Los niveles de proteína del gen objetivo, por consiguiente, se pueden ver reducidos, mientras que los niveles de RNAm pueden o no estar disminuidos. En los seres humanos, principalmente los miRNAs inhiben la traducción de proteínas de los genes objetivos y sólo con poca frecuencia causa la degradación o la división del RNAm (Bartel D P. 2004). Teniendo en cuenta que el cáncer es en última instancia, una consecuencia de la expresión génica desordenada, se puede inferir que los miRNAs contribuyen al desarrollo del cáncer (Caldas et al., 2005; Lu et at., 2005).
Mecanismos de silenciamiento y regulación de los miRNAs.
Los miRNAs se suelen unir a la región 3' UTR del transcrito, donde inician la degradación del mRNA o inhiben la traducción. El silenciamiento mediado por miRNAs se lleva a cabo mediante inhibición de la traducción o por degradación dependiente del grado de complementariedad entre el miRNA y la región 3'UTR del transcrito.
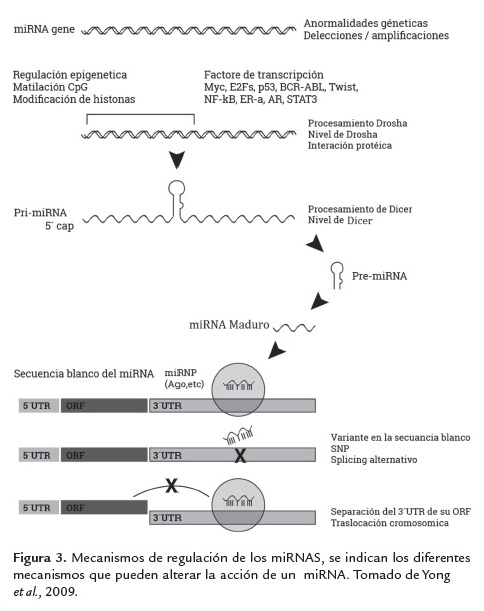
Mediante los enfoques de la bioinformática, diversos programas como miRanda, Pictar, Targetscan, Tarbase entre otros, puede predecir miles de genes que son potencialmente regulados por miRNAs (Shirdel et al., 2011) basados en la complementariedad de secuencias, si bien existe un gran número de falso positivos actualmente se está investigando para reducirlos mediante el refinamiento de dichas herramientas, y en base en estas, también se han generado base de datos robustas que contienen esta información y están a disposición de los investigadores para ser consultadas como miRbase, MirWalk, microRNA.org, miRDB, mir2disease.org y miRTarBase. Los mecanismos subyacentes de por qué y cómo se liberan los miRNAs en gran medida son desconocidos aunque por otra parte los mecanismos que los pueden modificar se han logrado dilucidar y se muestran en la figura 3.
La pérdida de la función de un miRNA es debido a varios mecanismos, incluyendo la eliminación genómica, mutaciones, silenciamiento epigenético, y/o alteraciones en el procesamiento del miRNA (Calin et al., 2002; Calin et al., 2005; Saito et al., 2006; Nakamura et al., 2007). Como muestra de ello, dos estudios proporcionaron información acerca de los mecanismos de regulación de miRNAs donde establecieron que la interrupción de la interacción de un solo miRNA y su RNAm objetivo puede producir un fenotipo anormal en células de mamífero. Además, existe evidencia que los miRNAs están regulados indirectamente a través del control de sus enzimas de procesamiento. (Mayr et al., 2007; Lee y Dutta 2007),
De modo similar Thomson et al., 2006 demostraron que una regulación negativa de miRNAs en el cáncer humano no se asocia con una reducción de los niveles de los transcritos primarios de miRNA. Los autores en tanto, sugieren que la regulación de miRNAs puede estar dada en posteriores pasos de procesamiento figura 3, por ejemplo, a través de la función alterada de la enzima Drosha (Thomson et al., 2006).
En este mismo sentido la transcripción de un gen miRNA, el mir-124a, ha demostrado ser inactivado por hipermetilación de su promotor en diferentes tumores humanos. Este proceso de silenciamiento epigenético es un mecanismo conocido para inactivar genes codificantes de proteínas en células de cáncer y puede aplicarse de modo similar a los miRNAs. De modo similar el gen miRNA mir- 127 se expresa en las células normales, pero no en las células con cáncer, Saito y colaboradores en 2006, demostraron que miR-127 está presente en cultivos de células de cáncer humano, después del tratamiento con drogas desmetilantes, lo que sugiere que está sujeto a silenciamiento epigenético por hipermetilación del promotor.
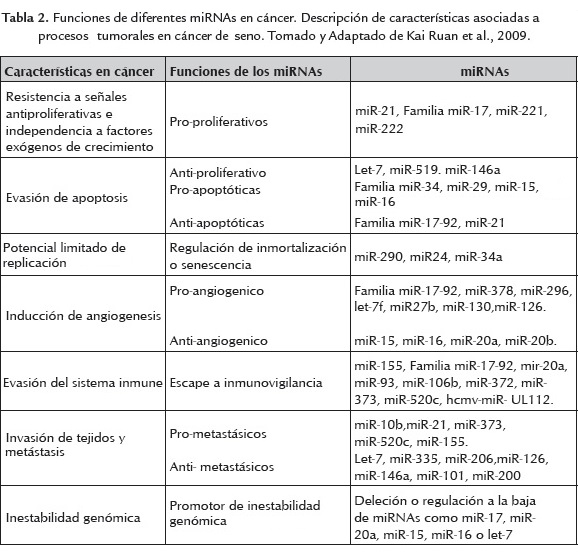
En la tabla 1 se resumen la expresión diferencial de miRNAs en cáncer de seno, centrándose en el potencial de miRNAs, como biomarcadores para el diagnóstico, tratamiento y apoyo en la búsqueda de nuevos blancos.
Dependiendo del efecto que generen los miRNAs en el gen diana, se les ha asignado una clasificación y denominación:
MicroRNAs oncogénicos (oncomirs)
Los "oncomirs" son miRNAs que promueven el desarrollo de tumores, regulando e inhibiendo genes supresores de tumor / o controlando los genes que intervienen en la diferenciación celular o apoptosis. Los oncomirs están significativamente sobre expresados en varios tumores debido a mecanismos epigenéticos o desregulación a nivel transcripcional figura 3 (Brennecke et al., 2005; Cho WC.2007).
Dentro de los miRNAs identificados como oncomirs se encuentra el miR-21 que está involucrado en varios procesos relacionados con cáncer, como invasión y migración. Varios grupos han observado la sobre expresión de miR-21 en tumores de seno en comparación con tejidos sanos (Yang et al., 2004; Sempere et al., 2007; Si et al., 2007; Frankel et al., 2008). Al suprimir la expresión endógena de miR-21 en células de cáncer de seno, se observó un aumento en la expresión de 58 proteínas, lo que implica que podrían ser blancos potenciales de miR-21. La validación de 12 de estos objetivos candidatos fueron realizados mediante ensayos de luciferasa, donde mostraron que seis de ellos eran blancos directos de miR-21. En general, los resultados demuestran que el miR-21 afecta la expresión de muchos de sus objetivos a través de inhibición de la traducción y pone de manifiesto la utilidad de la proteómica para la identificación de los objetivos de miRNA. (Yang et al., 2009). En la evaluación genómica de las células MCF-7 y los potenciales blancos del miR-21 se identificaron algunos RNAm reguladores de p53, en los que se incluyen FAM3C, ACTA2, APAF1, BTG2, FAS, CDKN1A (p21), PDCD4 y SESN1, sugiriendo un vínculo funcional entre el miR-21 y la vía supresora de tumor de p53 (Frankel et al., 2008; Hui et al., 2009). La generación de MCF-7 knock down de miR-21 inhibe el crecimiento de células in vitro y suprime el xenoinjerto en modelo murino con células MCF-7. (Frankel et al., 2008). Adicionalmente el supresor de tumor tropomiosina 1 (TPM1) fue identificado como otro de los objetivos de miR-21 en células MCF-7.
Por otra parte, miR-27a fue reportado en cáncer de seno como un oncomiR que regula el gen ZBTB10 (zinc finger and BTB domain containing 10), un represor de proteínas especificas oncogenicas (Sp). La inactivación de miR-27a en células MDA-MB-231 induce expresión de ZBTB10 y disminuye la expresión de los genes Sp. miR-27a suprimió la expresión de cdc2/ciclina B inhibidor de Myt-1 en células MDA-MB-231, lo que aumenta la actividad de la cdc2/ciclina B e induce la proliferación de células de cáncer de seno. De este modo es evidente que los miRNAs regulan distintas cascadas de señalización en células de cáncer de seno.
MicroRNAs Supresores de tumor (Anti-oncomirs)
Los miRNAs pueden inhibir la tumorogénesis por represión de oncogenes. Lo cual se ha evidenciado al estudiar la interacción de miRNA con proteínas de la familia ErbB. La familia ErbB juega un papel importante en el desarrollo de organismos, proliferación celular y sobrevida de tumores epiteliales humanos. (Yarden y Sliwkowsk 2001) Scott y colaboradores en el 2007 encontraron que ErbB2 está sobre expresado en cerca del 20-30% de cáncer de seno humano y se identificó al miR-125a como el regulador de este, en células de cáncer de seno. Adicionalmente, miR-125a y miR-125b están sobre expresados en células SKBR3, produciendo disminución de la proteína ErbB2 en un 40% a 65% y la disminución de ErbB3 entre el 60% a 80%. En las células SKBR3 que sobre expresan el miR-125a o miR-125b se altera su crecimiento dependiente de anclaje, migración, y las capacidades para invadir. (Scott et al., 2007; Li et al., 2009).
Hay varios miRNAs que se consideran como supresores de tumor, a saber, se han identificado let-7, miR-17-5p, miR-29, miR-34 y miR-127 (Zhang et al., 2007; Takamizawa et al., 2004). De hecho, los primeros miRNAs identificados como asociados a tumor (miR-15 y miR-16) son de esta clase (Calin et al., 2002), y ahora experimentalmente se ha demostrado su actividad como anti-oncogénicos (Calin et al., 2008). Además, el gen supresor de tumor BRCA1 (Breast Cáncer1) también ha sido identificado como un blanco de miR-15a y miR-16 (Zhu et al., 2009).
La localización genómica del clúster de miR-17/20, está en el cromosoma 13q31 y está asociado con pérdida de heterocigosidad en diferentes tipos de cáncer, incluyendo cáncer de seno. El miR-17/20 reprime la expresión de la ciclina D1, suprime la proliferación de células MCF-7 e inhibe la transición de la fase G1/S en el ciclo celular. (Yu et al., 2008). En líneas de cáncer de seno, la disminución de la expresión de miR17/20 esta inversamente correlacionada con la alta expresión de la ciclina D1, en comparación con tejido normal de seno. La deleción dirigida del clúster miR17/20 en modelo de ratón, ha mostrado que se genera la muerte poco después del nacimiento (Ventura et al., 2008).
Los estrógenos, que se unen al receptor ER- α, son un factor de riesgo para el desarrollo de cáncer de seno. El ER-α está sobre expresado en aproximadamente 75% de los cánceres primarios de seno. Se informó que la expresión de miR-206 está fuertemente inhibida por agonistas de ER-α, y los niveles de miR-206 son más altos en células ER-α negativas de la línea MB-MDA-231 al ser contrastadas con las células MCF-7 ER-α positivas (Adam et al., 2007; Kondo et al., 2008). El miR-206 reduce la sobre-expresión del ER-α a nivel de las células MCF-7, lo que indica un circuito de retroalimentación entre la autorregulación de ER-α y miR-206. La disminución de la expresión de miR- 206 se produce en tejido humano ER-α positivo, de cáncer de seno. El miR-206 y el nivel de expresión de proteínas ER-α están inversamente correlacionados con cáncer de seno. De manera similar el miR-205 regula la expresión del receptor HER3, inhibiendo de este modo las señales de proliferación mediadas por HER (Lorio et al., 2009)
Por otro lado, la evidencia ha mostrado que Ubc9 (enzima transportadora de ubiquitina- 9), y E2-conjugada (enzima de conjugación de ubiquitina 2) afectan el crecimiento celular y desarrollo de cáncer, adicionalmente se someten a la regulación post-transcripcional por miR-30e en el cáncer de seno. La expresión ectópica que suprimen el crecimiento en las células, por miR-30e puede ser parcialmente revertido por Ubc9 (Wu et al., 2009).
De modo similar se ha encontrado que la sobreexpresión de miR-29a en el cáncer de seno suprime la expresión de TTP (tristetraprolina), una proteína implicada en la degradación de los 3´UTR de RNAms con regiones ricas en UA, evento que conduce a la EMT y metástasis en cooperación con la señalización del oncogén Ras (sarcoma de rata) (Gebeshuber et al., 2009).
MicroRNAs como Marcadores de invasión tumoral
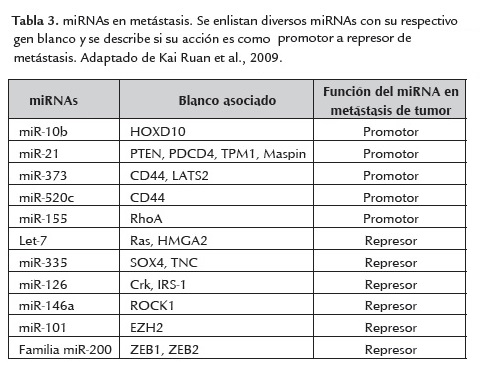
Un evento de metástasis representa un proceso complejo mediante el cual células de un tumor sólido primario invaden tejido adyacente y crecen en un tumor secundario (Tavazoie et al., 2008). Las células de cáncer de seno tienen el potencial de diseminarse a casi cualquier parte del cuerpo donde continúan con su crecimiento y multiplicación. La expresión de gen CD44 está implicado en la adhesión celular y diferentes componentes de la matriz extracelular, se ha encontrado que su expresión está reducida durante la progresión de cáncer de seno a fenotipo metastático (López et al., 2005). Los miRNA, miR-373 y miR-520c estimulan la migración e invasión de células de cáncer de seno humano mediante la supresión de la expresión de CD44 (Huang et al., 2008), también el miR-21 promueve en las células de cáncer de seno los procesos de invasión y metástasis, por reconocimiento de múltiples genes supresores de tumor (Zhu et al., 2008). Análogamente el miR-10b promueve la migración celular in vitro e inicia la invasión de tumor de seno in vivo mediante la regulación del gen HOXD10. (Ma et al., 2007). El miR-10b está sobre regulado en células de cáncer de seno comparado con células epiteliales primarias de glándula mamaria humana o células MCF-10A y miR-10b está directamente regulado por TWIST mediante regulación transcripcional (Ma et al., 2007).De modo similar TWIST promueve EMT y metástasis del tumor mamario (Yang et al., 2004). La familia de miR-200 y el miR-205, regulan la EMT, que como es sabido es un paso fundamental en la metástasis de un tumor, y se ha establecido que la expresión de estos miRNAs esta disminuida en líneas celulares de cáncer de seno invasivo con un fenotipo mesenquimal.
En contraste los miRNAs, miR-126, miR-206 y miR-335 han sido identificados como miRNAs supresores de metástasis en cáncer de seno. (Tavazoie et al., 2008) La expresión de miR-126 y miR-335 en tumores mamarios humanos esta inversamente asociado con recaída metastásica. La metástasis de cáncer de seno y migración son inhibidas por miR-335 que tiene como blanco el factor de transcripción SOX4, que se sabe regula el desarrollo celular y la migración (Tavazoie et al., 2008; Romero et al., 2012).
miRNAs en cáncer
La primera evidencia directa de la participación de miRNAs en cáncer, la hizo Calin y colaboradores en el 2002, durante el estudio de una deleción en el cromosoma 13, la cual se presenta frecuentemente en LLC, donde encontraron 2 genes de miRNAs dentro de esta deleción de 30 Kb, el mir-15 y mir-16. Al evaluar muestras de sangre de pacientes con LLC, encontraron que estos dos miRNAs estaban ausentes o regulados negativamente en la mayoría de los casos (68%), en comparación con linfocitos de pacientes normales. Este hallazgo sugirió que estos dos miRNAs están causalmente involucrados en la patogénesis de la LLC. Poco después el mismo grupo identifico que el 50% de los genes miRNAs se ubican en regiones genómicas asociadas con cáncer, tales como sitios frágiles, regiones de pérdida de heterocigosidad mínima, en genes HOX, regiones de mínima amplificación y regiones comunes de rompimiento (Calin et al., 2004). En soporte a esta evidencia, Cimmino y colaboradores en 2005, mostraron que el factor antiapoptótico Bcl2 está directamente regulado por miR-15 y miR16-1, donde funcionalmente comprobaron que estos miRNAs pueden ser considerados como nuevos reguladores de genes supresores de tumor. Además la pérdida o la baja regulación de miRNAs con potencial oncogénico, puede causar una sobrexpresión de los blancos a los cuales están dirigidos y en último promover la aparición de cáncer. Tal es el caso de miR-205 que fue reportado como inhibidor del potencial clonogénico de las células en cáncer de seno que mostraban un aumento de la respuesta a agentes anticancerígenos, consecuentes con la disminución en la regulación de ErbB3, miembro de la familia EGFR (Lorio et al., 2009). Otros estudios han indicado que el miR-205 también regula negativamente la transición epitelio mesenquimal (EMT), evento clave en metástasis del cáncer. (Gregory et al., 2008; Gandelli et al., 2009).
En 2005, tres informes plantearon mecanismos, de cómo los miRNAs podría contribuir en la progresión tumoral. Dos estudios independientes, describieron la relación entre un grupo de miRNA, mir-17-92 y la vía oncogénica de Myc (He et al., 2005; O'Donnell et al., 2005). Un tercer informe demostró una interacción entre el miRNA let-7 y el protooncogen RAS (Johnson et al., 2005).
Además de la evidencia de los miRNAs como supresores de tumor o como oncogenes, también se elucido el rol de los miRNAs en la fisiología normal del seno, en un estudio realizado por Avril-Sassen y colaboradores en el 2009 se examinó la expresión de miRNAs en la glándula mamaria de ratones en edad juvenil y adulta, evaluando diferentes etapas como la de embarazo, lactancia e involución de la glándula (Avril-Sassen et al., 2009). El hallazgo clave fue el encontrar miRNAs que se expresan en 7 grupos probablemente co-regulados durante el desarrollo de la glándula mamaria, hecho que conecta a varias familias de los miRNAs en distintas fases del ciclo de la glándula mamaria. Curiosamente, varios miRNAs (incluyendo miR-25 y el grupo miR-17-92) que previamente habían sido asociados con cáncer de seno agresivo basal también se encontraron sobre expresados durante la pubertad y la gestación, los períodos del pico fisiológico ductal y proliferación alveolar, lo que sugiere que pueden formar parte de un programa fisiológico de proliferación y también incidir en el crecimiento generado por tumores. Además, los miRNAs asociados con el fenotipo diferenciado final (por ejemplo, Let-7) aumentan en la pubertad y la gestación, correlacionando con un aumento en la proporción de epitelio maduro. A diferencia de miR-29 que alcanza su máxima expresión durante la involución causada por la post-lactancia, con una reducción en la expresión de sus RNAm previstos como objetivos, hecho que favorece a los genes relacionados con la adhesión focal, lo que sugiere un papel del miR-29 en la remodelación de la glándula mamaria. Otro estudio identifico marcadores de células madre de cultivo de células epiteliales mamarias de ratón (Ibarra et al., 2007), donde encontraron que Let-7 presenta deleción en células progenitoras, mientras que miR-205 esta (en este sistema) como un marcador de fenotipo de células madre. Tales estudios de la función fisiológica de los miRNAs son esenciales para comprender cómo su desregulación influye en el fenotipo de cáncer de seno.
En la misma línea ahora se está investigando en la identificación de los blancos de los miRNAs con el fin de identificar las redes sobre las cuales inciden los miRNAs y de este modo entender como el sistema biológico se está comportando frente a cada cambio, con el fin de poder determinar en qué medida cada miRNA puede estar regulando un solo blanco o simultáneamente, más de una diana y que esta, a su vez pueda estar generando toda una cascada de señales que desemboquen en un fenotipo tumoral (Kim et al., 2012).
Gracias al conocimiento sobre la participación de miRNAs en la iniciación, progresión y metástasis tumoral, y al descubrimiento de muchos miRNAs que imparten efectos supresores de tumor, al restaurar su expresión y pretender producir efectos terapéuticos se espera que estos emerjan como una opción terapéutica eficaz para el tratamiento de Cáncer (Romero et al., 2012). Una clara síntesis fue expuesta por Liu en un artículo de revisión en el 2012, del cual se toma una de las gráficas con el fin entender mucho más concretamente los diferente miRNAs asociados con los diferentes estadios del seno y el proceso carcinogénico (figura 4).
Estos supresores MIR inhibidores de tumor y oncomiRs promotores de tumor son prometedores candidatos para la intervención terapéutica como terapia de reemplazo como Mir o antagomirs.
La lista de biomarcadores miRNA del panel inferior, codificados como MIR sobre regulados (en formato normal) y los MIR sub- regulados (subrayados), que mostraron expresión diferencial en MASCs normales / glándulas mamarias en comparación con los tumores primarios de mama. En el panel MaSC se expresan diferencialmente, let- 7b / c , miR- 205 , -22 , -93 y -200 en comparación con las células epiteliales diferenciadas; miR- 138 , -431 , y -133a /b se relacionan con las glándulas mamarias durante el embarazo y la lactancia, en comparación sin embarazo y en involución de la glándula. El panel Br Ca (cáncer de seno) presenta expresados diferencialmente MIR en cáncer de seno en comparación con el tejido normal de seno. El panel MIR de ER / PR relacionado con el estado de receptores de estrógenos ER o estado receptores de progestágenos PR (ER + o PR + tumores vs ER - o PR- tumores, respectivamente). Panel de Invasión muestra MIR regulados en los tumores con metástasis ganglionar (let- 7 y miR -9 -3) o invasión vascular (miR-10b, miR-123, miR-29a, miR-205, etc). El panel incluye DRFS arriba y MIR -regulado y relacionados con alto riesgo o mal pronóstico de pacientes ER + y ER- en tumor mama. Panel de resistencia a los medicamentos se enumeran MIR (algunos muestran los genes sobre los que actúan directamente) relacionado a resistencia a los medicamentos de las células de cáncer de seno o tumores primarios, tales como la resistencia a tamoxifeno de células MCF- 7 y tumores HER2 + / ER- ( miR-221/222 ), resistencia de células MDA- MB- 435 y 468 células a paclitaxel (miR-221/222 , - 125b) , resistencia a la doxorrubicina de células MCF-7 (miR- 127 , 34a, 27b, 206 , 106a , 21 , 214 , y let-7) , y la resistencia a cisplatino de células MCF-7( miR- 146 , -10a / b , -221 / 222 , -29a / b , -206 , - 200b / c , -345 , -126 , -205 y -342 ).
De modo similar miRNAs específicos han mostrado una expresión alterada en células tumorales resistentes a diferentes medicamentos, un ejemplo es mostrado en un estudio donde evaluaron la línea MCF-7, una variante resistente a fármacos que mostró estar asociada con disminución de los niveles de miR-451, miR-328 y miR-326 que regulan la expresión del gen de Resistencia a múltiples drogas (MDR1), la glicoproteína-P de resistencia a cáncer de seno (BCRP/ABCG2) y la proteína 1 asociada a resistencia a múltiples drogas (MRP-1/ABCC1), respectivamente. La reconstrucción de la expresión de los miRNAs sensibilizó las células a los efectos antiproliferativos de la doxorubicina (miR-451), mitoxantrone (miR-328) y VP-16(miR-326) en cada uno de los casos (Pan et al., 2009 and Wang et al., 2012).
Perspectivas
Teniendo en cuenta las limitaciones de las actuales pruebas de detección de cáncer, el uso de miRNAs como marcadores tumorales para el diagnóstico y el pronóstico ha despertado interés en investigación. La detección y el tratamiento de cáncer es facilitado en gran medida por las tecnologías ómicas (Cho WC 2010b; Cascione et al., 2013). Aunque se hacen necesarias más investigaciones que evalúen los efectos de miRNAs mediante ensayos clínicos para superar las barreras de la presentación efectiva al RNAm blanco, mejorar la eficacia, estabilidad y minimizar toxicidades fuera del blanco. El interés en los miRNAs es ahora mayor que nunca y la literatura se está nutriendo rápidamente con los informes sobre los nuevos miRNAs, sus genes diana validados y el desarrollo de productos terapéuticos basados en miRNA que pretenden realizar un abordaje terapéutico basado en miRNA y la clínica de cada paciente.
Microarrays de miRNA pueden identificar la expresión de varios cientos de genes en la misma muestra a la vez, mientras que requiere solamente pequeñas cantidades de RNA total (Yin et al., 2008). Estas tecnologías no sólo ayudan a identificar potenciales miRNAs supresores tumorales y oncogénicos, sino también ayudan a proporcionar una comprensión más completa de sus vías y los mecanismos base, de miRNAs específicos que están asociados con cáncer, células madre, metástasis y sensibilidad a fármacos. Los miRNAs pueden complementar otros biomarcadores genómicos y proteómicos para el diagnóstico y pronóstico del cáncer (Cho WC, 2007b) teniendo en cuenta que cada miRNA puede controlar a cientos de genes diana.
Aunque el desarrollo reciente de medios farmacológicos más eficaces para modular las actividades miRNA proporciona nuevas oportunidades terapéuticas, como la introducción de oligonucleótidos antisentido para reducir la expresión de miRNAs que pueden generar desvío de los efectos indeseables (Kota et al., 2009). Una muestra de ello es el estudio publicado por Kim y colaboradores en el 2011, donde mediante vectores virales se demuestra el efecto antitumoral del miR-145 en células de cáncer de seno, hecho que abre el camino hacia el uso de miR-145 como terapia antitumoral para el tratamiento de pacientes de cáncer de seno. Adicionalmente en un estudio se muestra que un agregado exógeno de miRNA (miR- 141) tiene un ritmo más lento de degradación y permanecía detectable por períodos más largos en placenta, lo que sugiere que los miRNAs tienen una mayor estabilidad que los RNAm incluso en ausencia de protección y estando sometido a la actividad nucleasa en el plasma, hechos que podrían potenciarse (por ejemplo, a través de asociación con partículas) (Chim et al., 2008).
Al surgir las implicaciones terapéuticas de miRNA, las estrategias basadas en miRNA pueden formular, antagonizar o restaurar las funciones de miRNAs. La tecnología de oligonucleótidos antisentido ya se está desarrollando utilizando la vía de RNAi para la terapia génica. Las aplicaciones de RNAi se basan en la sincronización entre un oligonucleótido y un RNAm. En la práctica, una falta de coincidencia solo puede conducir a una completa pérdida de silenciamiento (Brummelkamp et al., 2002) y por "los efectos fuera del blanco" está comprometida la especificidad de la identidad de la secuencia, que al realizar reconocimiento al azar de RNAm reprimen la expresión de genes no esperados. Si múltiples siRNAs se utilizan para la selección de múltiples hits, múltiples efectos fuera del objetivo pueden suceder. Las más recientes investigaciones han demostrado que mediante el diseño adecuado de la siRNAs, así como el uso de nucleótidos modificados, los efectos no específicos puede ser minimizados (Enders et al., 2009). Estos oligonucleótidos deben ser modificados químicamente para permitir la estabilidad en el suero y la captación celular. Dos estudios han aplicado con éxito 2'-O-Metil modificado, a RNA antisentido para inhibir la función de miRNA en cultivos celulares (Hutvagner et al., 2004; Meister et al., 2004).
La entrega de los inhibidores del miRNA o miRNAs sintéticos en las células es uno de los mayores desafíos para el desarrollo de esta solicitud. A pesar de los avances de los últimos años, es necesaria más investigación para obtener la aplicación sistemática de inhibidores de miRNA a su sitio de acción. Un trabajo reciente de Krutzfeldt et al., 2005 demostró que el RNA antisentido conjugado modificado con colesterol, designado "antagomirs" podría efectivamente inhibir la función de miRNA in vivo en ratón adulto. Los autores aplicaron tres inyecciones al día por vía intravenosa de antagomirs y la inhibición de miRNAs lograda fue efectiva durante un período de cuatro semanas en la mayoría de los tejidos, excepto cerebro (Krutzfeldt et al., 2005).
Incluso el uso de la tecnología de LNA (ácidos nucleicos bloqueados) ha presentado un gran avance, del cual es ejemplo los ensayos clínicos con LNA-antimiR-122 en fase I que ya se ha iniciado sobre la base de interesantes datos preliminares en primates no humanos (Elmen et al., 2008). Además, se han logrado avances en los sistemas de entrega y la administración sistémica de miRNA mediante virus adeno-asociados (AAV) que han sido probados en modelos preclínicos (Kota et al., 2009). Del mismo modo, los liposomas catiónicos o formulaciones de nanopartículas a base de polímeros, se pueden desarrollar para lograr la entrega de miRNA mímicos y aumentar la eficacia terapéutica.
Aunque los sistemas de vectores virales ofrecen opciones adicionales para presentación eficiente y órgano-específica, este enfoque debe superar las reservas sobre la base de la experiencia negativa con la terapia génica. La inhibición específica de un único gen objetivo es por lo general el intento a realizar, y la experiencia en campo ha demostrado que secuencias antisentido pueden, en determinadas circunstancias, ser insuficiente para enfermedades complejas como el cáncer.
Por el contrario, los miRNAs afectan muchos RNAm objetivo, de modo que una regulación más completa se puede lograr con la inhibición de un solo miRNA. Por lo tanto, profundizar en el conocimiento del papel funcional de miRNAs abrirá nuevas oportunidades en el desarrollo revolucionario de estrategias terapéuticas para la quimioprevención del cáncer (Enders et al., 2009; Lu et al., 2009) generando un perfil de expresión de miRNA, que podría clasificar el cáncer humano, generando micro-perfiles de expresión de RNA claramente diferenciados en tipos de cáncer humano según su origen en el desarrollo. Un subgrupo de productos de los tumores gastrointestinales que surgen de endodermo, se distinguió por patrones de expresión miRNA. Además, los tumores en un linaje de células individuales, como la leucemia linfoblástica aguda donde se diferenciaron de acuerdo con su subyacente anormalidad genética en el BCR / ABL de tumores positivos, las células T tumorales, y aquellos con reordenamiento del gen MLL (Enders et al., 2009).
Los miRNAs son útiles para el diagnóstico clínico, su ventaja clave podría ser su gran estabilidad. A diferencia de la mayoría de RNA mensajeros, los miRNAs son de larga duración in vivo (Lim et al., 2005; Xi et al., 2007) y muy estable in vitro (Tang et al., 2006), lo que podría permitir el análisis de muestras embebidas en parafina para aplicaciones de diagnóstico de rutina.
Un enfoque se informó recientemente por Ebert et al., 2010, ellos desarrollaron inhibidores del miRNA que puede ser expresada de forma transitoria en cultivos de células de mamíferos. Estos inhibidores competitivos denominados "esponjas miRNA" donde la esponja contienen RNA complementario a sitios de unión a un miRNA de interés, y son producidos a partir de transgenes en las células. Como con la mayoría de los genes miRNA, los sitios de unión de una esponja son específicos para el miRNA en la región "seed", lo que les permite bloquear una familia entera de miRNAs asociados. Este enfoque transgénico ha demostrado ser una herramienta útil para investigar las funciones del miRNA en una variedad de sistemas experimentales (Ebert et al., 2010).
Una dirección diferente fue tomada por Tsuda et al., 2005. Los autores diseñaron miRNAs sintéticos cuyo objetivo fue sobre expresar proteínas de tumor, como la proteína HER-2. Un miRNA sintético orientado al RNAm de HER-2 inhibió con éxito la expresión de la proteína HER-2 en las células de cáncer de ovario (Tsuda et al., 2005). En conjunto, estos estudios tienen como promesa establecer miRNAs como futuras dianas terapéuticas. Una de las limitaciones de las terapias antisentido de RNA es el número limitado de células que pueden ser objeto. Cualquier enfoque para deletar un miRNA antisentido particular, sólo dará lugar a una respuesta parcial. Esta puede representar una limitación para terapias contra el cáncer. Queda por ver, si los efectos indirectos en las células de cáncer que no han sido blanco directo, puede superar esta limitación.
Con interés cada vez mayor, los avances de la investigación en las funciones de miRNA y los adelantos tecnológicos, la terapéutica basada en miRNA pueden crear un cambio de paradigma en la medicina y la industria farmacéutica Por lo tanto, con el aumento de conocimientos sobre los miRNAs asociadas a subtipos moleculares y las características clínico-patológicas de cáncer, se cree que los miRNAs puede resultar útiles como herramientas de diagnóstico y pronóstico en el futuro cercano.
Por el contrario, un efecto de función parcial puede ser de valor terapéutico en enfermedades neurodegenerativas, como el Parkinson o la enfermedad de Alzheimer. Una restauración parcial de la producción de dopamina por la terapia antisentido puede dar lugar a una mejoría clínica significativa en pacientes de Parkinson. Del mismo modo, una reducción parcial de las proteínas que causan la enfermedad de Alzheimer puede llevar a una mejoría clínica y podría ser alcanzable por bases genéticas de RNA o terapia miRNA. (Sassen et al., 2008)
Por otro lado, como las células madre pueden regenerar y diferenciarse a distintos tipos celulares, son siempre un foco intenso de interés en investigación. Se ha informado que los miRNA juega un papel regulador en la vía de división de células madre, si este mecanismo contribuye a prevenir y tratar el cáncer será digno de estudio (Hatfield et al., 2005)
Conclusiones
En los últimos años, los miRNAs se han convertido en actores principales en las complejas redes de regulación génica y han sido implicados en diversos aspectos de las enfermedades humanas. La investigación de los miRNAs en cáncer de seno, han mejorado significativamente nuestra comprensión de la patogénesis de este cáncer. La atención se ha centrado en el estudio de oligonucleótidos antisentido y los métodos para inhibir la función de miRNA con tecnología de pequeños RNAi, para la sustitución de miRNAs y explorar su potencial como agentes terapéuticos. En esta línea diversos estudios han puesto de manifiesto que la caracterización de oncomiRs y antioncomiRs pueden generar clasificaciones más precisas en cáncer y predecir resultados a terapias de los pacientes, con alta exactitud.
Nuevo conocimiento sobre el papel funcional de oncomiRs está revolucionando la biología del cáncer y abrirá nuevas oportunidades en investigación biomédica. El análisis e identificación de transcritos diana en combinación de genómica, proteómica y miRomica podrán ayudar a delimitar el espectro de blancos que son regulados por miRNAs, dejando abierto el campo de investigación para la generación de conocimiento, que permita mejorar la calidad de vida de nuestra población. Siempre habrá obstáculos en la batalla contra el cáncer, sin embargo, la identificación de miRNAs proporcionara interesantes oportunidades en el diagnóstico, pronóstico y la terapia personalizada del cáncer de seno. En un futuro próximo se podrían convertir en marcadores de blancos y su implementación será útil como una herramienta de investigación.
Referencias bibliográficas
1 Adams BD., Furneaux H., White BA. 2007. The micro-ribonucleic acid (miRNA) miR-206 targets the human estrogen receptor-alpha (ERalpha) and represses ERalpha messenger RNA and protein expression in breast cancer cell lines. Mol. Endocrinol. 21:1132-1147. [ Links ]
2 Alvarez R.H. 2010. Present and future evolution of advanced breast cancer therapy. Breast Cancer Res. 12 (Suppl. 2) S1. [ Links ]
3 Avril-Sassen S., Goldstein L.D., Stingl J., Blenkiron C., Le Quesne, J., Spiteri I., Karagavriilidou K., Watson C.J. 2009. Characterisation of microRNA expression in post-natal mouse mammary gland development. BMC Genomics. 10, 548 . [ Links ]
4 Bartel, D. P. 2004. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116(2): 281-297. [ Links ].
5 Bentwich I. 2005. Prediction and validation of microRNAs and their targets. FEBS Lett. 579:5904-5910. [ Links ]
6 Bentwich, I., Avniel, A., Karov, Y., Aharonov, R., Gilad, S., Barad, O., Barzilai A., Einat P., Einav U., Meiri E., Sharon E., Spector Y., and Bentwich, Z. 2005. Identification of hundreds of conserved and nonconserved human microRNAs. Nature genetics. 37(7), 766-770. [ Links ]
7 Berezikov, E., Cuppen, E., and Plasterk, R. H. 2006. Approaches to microRNA discovery. Nature genetics. 38, S2-S7. [ Links ]
8 Bernstein E., Caudy AA., Hammond SM., Hannon GJ. 2001. Role for a bidentate ribonuclease in the initiation step of RNA interference. Nature. 409:363-366. [ Links ]
9 Blenkiron C, Goldstein LD, Thorne NP, Spiteri I, Chin SF, Dunning MJ, Barbosa-Morais NL, Teschendorff AE, Green AR, Ellis IO, Tavaré S, Caldas C, Miska EA. 2007 MicroRNA expression profiling of human breast cancer identifies new markers of tumor subtype. Genome Biol. 8(10): R214. [ Links ]
10 Borchert GM., Dance M., Weber M., and Cavaillé J. 2009. C19MC microRNAs are processed from introns of large Pol-II, non-protein-coding transcripts. Nucleic Acids Res. 37(10): 3464-3473. [ Links ]
11 Brennecke J., Stark A., Russell RB., Cohen SM . 2005. Principles of microRNA-target recognition. PLoS Biol. 3:e85. [ Links ]
12 Brummelkamp T.R., Bernards R., and Agami R. 2002. "A system for stable expression of short interfering RNAs in mammalian cells," Science. 296: 550-553. [ Links ]
13 Caldas C. and Brenton JD. 2005. Sizing up miRNAs as cancer genes. Nat Med. 11:712-714. [ Links ]
14 Calin, G. A.; Cimmino, A.; Fabbri, M.; Ferracin, M.; Wojcik, S. E.; Shimizu, M., Taccioli C, Zanesi N, Garzon R, Aqeilan RI, Alder H, Volinia S, Rassenti L, Liu X, Liu CG, Kipps TJ, Negrini M, and Croce, C. M. 2008. MiR-15a and miR-16-1 cluster functions in human leukemia. Proceedings of the National Academy of Sciences. 105(13): 5166-5171. [ Links ]
15 Calin GA., Croce C M. 2006. MicroRNA signatures in human cancers. Nat Rev Cancer. 6:857-866. [ Links ]
16 Calin, G. A.; Dumitru, C. D.; Shimizu, M.; Bichi, R.; Zupo, S.; Noch, E.; Aldler H, Rattan S, Keating M, Rai K, Rassenti L, Kipps T, Negrini M, Bullrich F and Croce, C. M. 2002. Frequent deletions and down-regulation of micro-RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia. Proceedings of the National Academy of Sciences. 99(24): 15524-15529. [ Links ]
17 Calin, G. A.; Ferracin, M.; Cimmino, A.; Di Leva, G.; Shimizu, M.; Wojcik, S. E.; Iorio MV, Visone R, Sever NI, Fabbri M, Iuliano R, Palumbo T, Pichiorri F, Roldo C, Garzon R, Sevignani C, Rassenti L, Alder H, Volinia S, Liu CG, Kipps TJ, Negrini M and Croce, C. M. 2005. A MicroRNA signature associated with prognosis and progression in chronic lymphocytic leukemia. New England Journal of Medicine. 353(17): 1793-1801. [ Links ]
18 Calin GA, Sevignani C, Dumitru CD, Hyslop T, Noch E, Yendamuri S, Shimizu M, Rattan S, Bullrich F, Negrini M, Croce CM. 2004. Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers. Proceedings of the National Academy of Sciences of the United States of America. 101(9): 2999-3004. [ Links ]
19 Cascione L, Gasparini P, Lovat F, Carasi S, Pulvirenti A, Ferro A, Alder H, He G, Vecchione A, Croce CM, Shapiro CL, Huebner K. 2013. Integrated microRNA and mRNA signatures associated with survival in triple negative breast cancer. PloS one. 8(2): e55910. [ Links ]
20 Chen, C.-Z., 2005. MicroRNAs as oncogenes and tumor suppressors. The New England Journal of Medicine. 353: 1768- 1771. [ Links ]
21 Chen L, Li Y, Fu Y, Peng J, Mo MH, Stamatakos M, Teal CB, Brem RF, Stojadinovic A, Grinkemeyer M, McCaffrey TA, Man YG, Fu SW. 2013. Role of deregulated microRNAs in breast cancer progression using FFPE tissue. PloS one. 8(1): e54213. [ Links ]
22 Chim, S. S.; Shing, T. K.; Hung, E. C.; Leung, T. Y.; Lau, T. K.; Chiu, R. W.; and Lo, Y. D. 2008. Detection and characterization of placental microRNAs in maternal plasma. Clinical Chemistry. 54(3):482-490. [ Links ]
23 Cho WC. 2007. OncomiRs: the discovery and progress of microRNAs in cancers. Mol Cancer . 6(1):60. [ Links ]
24 Cho WC. 2007b. Contribution of oncoproteomics to cancer biomarker discovery. Mol Cancer. 6(25):25-27. [ Links ]
25 Cho, W. 2010. MicroRNAs: potential biomarkers for cancer diagnosis, prognosis and targets for therapy. The International Journal of Biochemistry & Cell Biology. 42(8): 1273-1281. [ Links ]
26 Cho WC. 2010b. Omics approaches in cancer research. In: Cho WC, editor. An omics perspective on cancer research. New York/Berlin: Springer; 1-9. [ Links ]
27 Cimmino A, Calin GA, Fabbri M, Iorio MV, Ferracin M, Shimizu M, Wojcik SE, Aqeilan RI, Zupo S, Dono M, Rassenti L, Alder H, Volinia S, Liu CG, Kipps TJ, Negrini M, Croce CM. 2005. miR-15 and miR- 16 induce apoptosis by targeting BCL2, Proceedings of the National Academy of Sciences of the United States of America, 102, 39. 13944-13949. [ Links ]
28 Croce CM. 2009. Causes and consequences of microRNA dysregulation in cancer. Nat Rev Genet. 10: 704-714. [ Links ]
29 Ebert Margaret S. y Sharp Phillip A. 2010.MicroRNA sponges: Progress and possibilities. RNA November 16: 2043-2050. [ Links ]
30 Elmén J, Lindow M, Schütz S, Lawrence M, Petri A, Obad S, Lindholm M, Hedtjärn M, Hansen HF, Berger U, Gullans S, Kearney P, Sarnow P, Straarup EM, Kauppinen S. 2008. LNA-mediated microRNA silencing in non-human primates. Nature. 452(7189):896-899. [ Links ]
31 Filipowicz, Witold, Bhattacharyya, Suvendra N., Sonenberg, Nahum. 2008. Mechanisms of post-transcriptional regulation by microRNAs: are the answers in sight? Nat Rev Genet. 9:102-114. [ Links ]
32 Frankel LB., Christoffersen NR., Jacobsen A., Lindow M., Krogh A., Lund AH. 2008. Programmed cell death 4 (PDCD4) is an important functional target of the microRNA miR-21 in breast cancer cells. J Biol Chem. 283:1026-1033. [ Links ]
33 Gandellini P, Folini M, Longoni N, Pennati M, Binda M, Colecchia M, Salvioni R, Supino R, Moretti R, Limonta P, Valdagni R, Daidone MG, Zaffaroni N. 2009. miR-205 excerts tumor-suppressive functions in human prostate through down-regulation of protein kinase cepsilon. Cancer Rea. 69. 2287-95. [ Links ]
34 Gebeshuber CA., Zatloukal K., Martinez J. 2009. miR-29a suppresses tristetraprolin, which is a regulator of epithelial polarity and metastasis. EMBO 10(4):400-5. [ Links ]
35 Gregory PA, Bert AG, Paterson EL, Barry SC, Tsykin A, Farshid G, Vadas MA, Khew-Goodall Y, Goodall GJ. 2008. The miR-200 family and miR-205 regulate epithelial to mesenchymal transition by targeting ZEB1 and SIP1. Nature Cell Biology. 10(5): 593-601. [ Links ]
36 Hatfield, S. D.; Shcherbata, H. R.; Fischer, K. A.; Nakahara, K.; Carthew, R. W.; and Ruohola-Baker, H. 2005. Stem cell division is regulated by the microRNA pathway. Nature. 435(7044), 974-978. [ Links ]
37 He L.; Hannon, G. J. 2004. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5(7): 522-531. [ Links ]
38 He L., Thomson JM., Hemann MT., Hernando-Monge E., Mu D., Goodson S., Powers S., Cordon-Cardo C., Lowe SW., Hannon GJ., Hammond SM. 2005. A microRNA polycistron as a potential human oncogene. Nature. 435:828-833. [ Links ]
39 Huang Q., Gumireddy K., Schrier M., le Sage C., Nagel R., Nair S., Egan DA., Li A., Huang G., Klein-Szanto AJ., Gimotty PA., Katsaros D., Coukos G., Zhang L., Pure E., Agami R. 2008. The microRNAs miR-373 and miR-520c promote tumour invasion and metastasis. Nat Cell Biol. 10:202-210. [ Links ]
40 Hui AB, Shi W, Boutros PC, Miller N, Pintilie M, Fyles T, McCready D, Wong D, Gerster K, Waldron L, Jurisica I, Penn LZ, Liu FF. 2009. Robust global micro-RNA profiling with formalin-fixed paraffin-embedded breast cancer tissues. Laboratory investigation. 89(5): 597-606. [ Links ]
41 Hutvagner G., McLachlan J, Pasquinelli AE., Balint E., Tuschl T., Zamore PD. 2001. A cellular function for the RNA-interference enzyme Dicer in the maturation of the let-7 small temporal RNA. Science. 293:834-838. [ Links ]
42 Hutvagner G., Simard MJ., Mello CC., Zamore PD. 2004. Sequence-specific inhibition of small RNA function. PLoS Biol. 2:E98. [ Links ]
43 Hutvagner G. and Zamore P D. 2002. RNAi: nature abhors a double strand. Curr. Opin. Genet. Dev. 12, 225-232. [ Links ]
44 Ibarra I., Erlich Y., Muthuswamy S.K., Sachidanandam R., Hannon G.J. 2007. A role for microRNAs in maintenance of mouse mammary epithelial progenitor cells. Genes Dev. 21: 3243. [ Links ]
45 Lorio MV, Casalini P, Piovan C, Di Leva G, Merlo A, Triulzi T, Ménard S, Croce CM, Tagliabue E. 2009. microRNA-205 regulates HER3 in human breast cancer. Cancer research. 69(6): 2195-2200. [ Links ]
46 Jackson RJ., Standart N. 2007. How do microRNAs regulate gene expression? Science Signaling. 2007(367), re1. [ Links ]
47 Johnson SM., Grosshans H., Shingara J., Byrom M., Jarvis R., Cheng A., Labourier E., Reinert KL., Brown D., Slack FJ. 2005. RAS is regulated by the let-7 microRNA family. Cell. 120:635-647. [ Links ]
48 Kai Ruan, Xiaoguang Fang, Gaoliang Ouyang. 2009. MicroRNAs: Novel regulators in the hallmarks of human cancer. Cancer Letters. 285(2): 116-126. [ Links ]
49 Kent O. A. y Mendell J. T. 2006. A small piece in the cancer puzzle: MicroRNAs as tumor suppressors and oncogenes. Oncogene. 25(46): 6188-6196. [ Links ]
50 Kim Junil, Choi Minsoo, Kim Jeong-Rae, Jin Hua, Narry Kim V. and Cho Kwang-Hyun. 2012. The co-regulation mechanism of transcription factors in the human gene regulatory network. Nucleic Acids Research. 40(18): 8849-8861 [ Links ]
51 Kim S.J., Oh J.S., Shin JY., Lee KD., Sung KW., Nam SJ., Chun KH. 2011. Development of microRNA-145 for therapeutic application in breast cancer. Journal of Controlled Release. 155: 427-434. [ Links ]
52 Kim, V. N. 2005. MicroRNA biogenesis: coordinated cropping and dicing. Nature reviews Molecular cell biology, 6(5), 376-385. [ Links ]
53 Kondo, N.; Toyama, T.; Sugiura, H.; Fujii, Y.; and Yamashita, H. 2008. miR-206 Expression Is Down-regulated in Estrogen Receptor ά-Positive Human Breast Cancer. Cancer research. 68(13): 5004-5008. [ Links ]
54 Kota J, Chivukula RR, O'Donnell KA, Wentzel EA, Montgomery CL, Hwang HW, Chang TC, Vivekanandan P, Torbenson M, Clark KR, Mendell JR, Mendell JT. 2009. Therapeutic microRNA delivery suppresses tumorigenesis in a murine liver cancer model. Cell. 137(6): 1005-1017. [ Links ]
55 Krutzfeldt J., Rajewsky N., Braich R., Rajeev KG., Tuschl T., Manoharan M., Stoffel M . 2005. Silencing of microRNAs in vivo with 'antagomirs'. Nature. 438:685-689. [ Links ]
56 Lagos-Quintana M, Rauhut R, Lendeckel W, Tuschl T. 2001. Identification of novel genes coding for small expressed RNAs. Science. 294:853-858. [ Links ]
57 Lau N.C., Lim L. P., Weinstein E. G., Bartel D. P. 2001. An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans. Science. 294: 858-862. [ Links ]
58 Lee R.C., Ambros V. 2001. An extensive class of small RNAs in Caenorhabditis elegans. Science. 294: 862-864. [ Links ]
59 Lee Y., Ahn C., Han J., Choi H., Kim J., Yim J., Lee J., Provost P., Radmark O., Kim S., Kim VN. 2003. The nuclear RNase III Drosha initiates microRNA processing. Nature. 425:415-419. [ Links ]
60 Lewis BP., Burge CB., Bartel D.P. 2005. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets. Cell. 120:15-20. [ Links ]
61 Lewis, B. P.; Shih, I. H.; Jones-Rhoades, M. W.; Bartel, D. P.; and Burge, C. B. 2003. Prediction of mammalian microRNA targets. Cell. 115(7): 787-798. [ Links ]
62 Li Mengfeng, Li Jun, Ding Xiaofan, He Mian and Cheng Shi-Yuan. 2010. microRNA and Cancer. AAPS Journal. 12: 309-317. [ Links ]
63 Li W., Duan R., Kooy F., Sherman SL., Zhou W., Jin P. 2009. Germline mutation of microRNA- 125a is associated with breast cancer. J Med Genet. 46:358-60. [ Links ]
64 Lim LP., Lau NC., Garrett-Engele P., Grimson A., Schelter JM., Castle J., Bartel DP., Linsley PS., Johnson JM. 2005. Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs. Nature. 433:769-773. [ Links ]
65 Liu Huiping. 2012. MicroRNAs in breast cancer initiation and progression. Cellular and Molecular Life Sciences. 69(21): 3587-3599. [ Links ]
66 Lopez JI., Camenisch TD., Stevens MV., Sands BJ., McDonald J., Schroeder JA. 2005. CD44 attenuates metastatic invasion during breast cancer progression. Cancer Res. 65:6755-6763. [ Links ]
67 Lorio, M.V., Ferracin, M., Liu, C.-G., Veronese, A., Spizzo, R., Sabbioni, S., Magri, E., Pedriali, M., Fabbri, M., Campiglio, M., Ménard, S., Palazzo, J.P., Rosenberg, A., Musiani, P., Volinia, S., Nenci, I., Calin, G.A., Querzoli, P., Negrini, M., Croce, C.M. 2005. MicroRNA gene expression deregulation in human breast cancer. Cancer Research. 16: 7065-7070. [ Links ]
68 Lowery AJ, Miller N, Devaney A, McNeill RE, Davoren PA, Lemetre C, Benes V, Schmidt S, Blake J, Ball G, Kerin MJ 2009. MicroRNA signatures predict oestrogen receptor, progesterone receptor and HER2/neu receptor status in breast cancer. Breast Cancer Res. 11(3):R27. [ Links ]
69 Lu J., Getz G., Miska EA., Alvarez-Saavedra E., Lamb J., Peck D., Sweet-Cordero A., Ebert BL., Mak RH., Ferrando AA., Downing JR., Jacks T., Horvitz HR., Golub T,. 2005. MicroRNA expresión profiles classify human cancers. Nature. 435:834-838. [ Links ]
70 Lujambio A., Ropero S., Ballestar E., Fraga MF., Cerrato C., Setien F., Casado S., Suarez-GauthierA., Sanchez-Cespedes M., Gitt A., Spiteri I., Das PP., Caldas C., Miska E, Esteller M. 2007. Genetic unmasking of an epigenetically silenced microRNA in human cancer cells. Cancer Res. 67:1424-1429. [ Links ]
71 Lu L, Katsaros D, Shaverdashvili K, Qian B, Wu Y, de la Longrais IA, Preti M, Menato G, Yu H. 2009. Pluripotent factor lin-28 and its homologue lin-28b in epithelial ovarian cancer and their associations with disease outcomes and expression of let-7a and IGF-II. European Journal of Cancer. 45(12): 2212-2218. [ Links ]
72 Ma L., Teruya-Feldstein J., Weinberg RA. 2007. Tumour invasion and metastasis initiated by microRNA-10b in breast cancer. Nature. 449:682-688. [ Links ]
73 Mayr C., Hemann MT., Bartel DP. 2007. Disrupting the pairing between let-7 and Hmga2 enhances oncogenic transformation. Science. 315:1576-1579. [ Links ]
74 Meister G., Landthaler M., Dorsett Y., Tuschl T. 2004. Sequence specific inhibition of microRNA- and siRNA-induced RNA silencing. RNA. 10:544-550. [ Links ]
75 Nakamura T., Canaani E., Croce CM. 2007. Oncogenic All1 fusion proteins target Drosha-mediated microRNA processing. Proc. Natl. Acad. Sci. USA 104:10980-85. [ Links ]
76 Ng, E. K.; Wong, C. L.; Ma, E. S.; and Kwong, A. 2009. MicroRNAs as new players for diagnosis, prognosis, and therapeutic targets in breast cancer. Journal of oncology. 30, 542-6. [ Links ]
77 O'Day E, Lal A. 2010. Review MicroRNAs and their target gene networks in breast cancer. Breast Cancer Res. 12(2):201. [ Links ]
78 O'Donnell KA, Wentzel EA, Zeller KI, Dang CV, Mendell JT. 2005. c-Myc-regulated microRNAs modulate E2F1 expression. Nature. 435:839-843. [ Links ]
79 Pan YZ., Morris ME., Yu AM. 2009. MicroRNA-328 negatively regulates the expression of breast cancer resistance protein (BCRP/ABCG2) in human cancer cells. Mol Pharmacol. 75:1374-9. [ Links ]
80 Pillai RS, Bhattacharyya SN, Filipowicz W. 2007. Repression of protein synthesis by miRNAs: how many mechanisms? Trends Cell Biol. 17:118-126. [ Links ]
81 Rajewsky N . 2006. microRNA target predictions in animals. Nat Genet. 38 Suppl: S8-S13. [ Links ]
82 Rodriguez, A.; Griffiths-Jones, S.; Ashurst, J. L.; and Bradley, A. 2004. Identification of mammalian microRNA host genes and transcription units. Genome research. 14(10a): 1902-1910. [ Links ]
83 Romero-Cordoba Sandra, Rodriguez-Cuevas Sergio , Rebollar-Vega Rosa, Quintanar-Jurado Valeria, Maffuz-Aziz, Jimenez-Sanchez Antonio, Bautista-Piña Veronica, Arellano-Llamas Rocio, and Hidalgo-Miranda Alfredo.2012. Identification and Pathway Analysis of microRNAs with No Previous Involvement in Breast Cancer. PLoS One. 7(3): e31904. [ Links ]
84 Saito Y, Liang G, Egger G, Friedman JM, Chuang JC, Coetzee GA, Jones PA. 2006. Specific activation of microRNA-127 with downregulation of the proto-oncogene BCL6 by chromatinmodifying drugs in human cancer cells. Cancer Cell. 9:435-443. [ Links ]
85 Sassen, S.; Miska, E. A.; and Caldas, C. 2008. MicroRNA—implications for cancer. Virchows Archiv. 452(1): 1-10. [ Links ]
86 Scott G. K., Goga A., Bhaumik D., Berger C. E., Sullivan C. S., y Benz C. C., 2007. Coordinate suppression of ERBB2 and ERBB3 by enforced expression of micro-RNA miR-125a or miR-125b. The Journal of Biological Chemistry. 282( 2): 1479-1486. [ Links ]
87 Sempere LF, Christensen M, Silahtaroglu A, Bak M, Heath CV, Schwartz G, Wells W, Kauppinen S, Cole CN.2007. Altered microRNA expression confined to specific epithelial cell subpopulations in breast cancer. Cancer Research. 67(24): 11612-11620. [ Links ]
88 Shirdel EA., Xie W., Mak TW., Jurisica I. 2011. NAViGaTing the micronome-using multiple microRNA prediction databases to identify signalling pathwayassociated microRNAs. PloS one 6: e17429. [ Links ]
89 Shimono Y, Zabala M, Cho RW, Lobo N, Dalerba P, Qian D, Diehn M, Liu H, Panula SP, Chiao E, Dirbas FM, Somlo G, Pera RA, Lao K, Clarke MF. 2009. Downregulation of miRNA-200c links breast cancer stem cells with normal stem cells. Cell. 138: 592-603. [ Links ]
90 Si ML., Zhu S., Wu H., Lu Z., Wu F., Mo YY. 2007. miR-21-mediated tumor growth. Oncogene. 26: 2799-2803. [ Links ]
91 Takamizawa J., Konishi H., Yanagisawa K. 2004. "Reduced expression of the let-7 microRNAs in human lung cancers in association with shortened postoperative survival," Cancer Research. 64(11): 3753-3756. [ Links ]
92 Tang F., Hajkova P., Barton SC., Lao K., Surani MA. 2006. MicroRNA expression profiling of single whole embryonic stem cells. Nucleic Acids Res. 34:9. [ Links ]
93 Tavazoie SF., Alarcon C, Oskarsson T, Padua D, Wang Q, Bos PD, Gerald WL, Massague J. 2008. Endogenous human microRNAs that suppress breast cancer metastasis. Nature. 451:147-152. [ Links ]
94 Thomson JM., Newman M., Parker JS., Morin-Kensicki EM., Wright T., Hammond SM. 2006. Extensive post-transcriptional regulation of microRNAs and its implications for cancer. Genes Dev. 20:2202-2207. [ Links ]
95 Tsuda N., Kawano K., Efferson CL., Ioannides CG. 2005. Synthetic microRNA and double-stranded RNA targeting the 3 untranslated region of HER-2/neu mRNA inhibit HER-2 protein expression in ovarian cancer cells. Int J Oncol. 27:1299-1306. [ Links ]
96 Ventura A., Young AG., Winslow MM., Lintault L., Meissner A., Erkeland SJ., Newman J., Bronson RT., Crowley D., Stone JR., Jaenisch R., Sharp PA., Jacks T. 2008. Targeted deletion reveals essential and overlapping functions of the miR-17 through 92 family of miRNA clusters. Cell. 132:875-886. [ Links ]
97 Veronesi U., Boyle P., Goldhirsch A., Orecchia R., Viale G. 2005. Breast cancer, Lancet. 365 :1727-1741. [ Links ]
98 Wang H, Tan G, Dong L, Cheng L, Li K, Wang Z, Luo H. 2012. Circulating MiR-26b as a Marker Predicting Chemoresistance in Breast Cancer. PLoS ONE. 7(4). [ Links ]
99 Wu F., Zhu S., Ding Y., Beck WT., Mo YY. 2009. MicroRNA-mediated regulation of Ubc9 expression in cancer cells. Clin Cancer Res. 15: 1550-7. [ Links ]
100 Xi, Y.; Nakajima, G.; Gavin, E.; Morris, C. G.; Kudo, K.; Hayashi, K.; and Ju, J. 2007. Systematic analysis of microRNA expression of RNA extracted from fresh frozen and formalin-fixed paraffin-embedded samples. Rna. 13(10): 1668-1674. [ Links ]
101 Yan, L.-X., Huang, X.-F., Shao, Q., Huang, M.-Y., Deng, L., Wu, Q.-L., Zeng, Y.-X., Shao, J.-Y. 2008. MicroRNA miR-21 overexpression in human breast cancer is associated with advanced clinical stage, lymph node metastasis and patient poor prognosis. Rna. 11: 2348-2360. [ Links ]
102 Yang J., Mani SA., Donaher JL., Ramaswamy S., Itzykson RA., Come C., Savagner P., Gitelman I., Richardson A., Weinberg RA. 2004. Twist, a master regulator of morphogenesis, plays an essential role in tumor metastasis. Cell. 117:927-939. [ Links ]
103 Yang Y., Chaerkady R., Beer MA., Mendell JT., Pandey A. 2009. Identification of miR-21 targets in breast cancer cells using a quantitative proteomic approach. Proteomics. 9:1374-84. [ Links ]
104 Yarden Y., Sliwkowski MX. 2001: Untangling the ErbB signalling network. Nat Rev. 2:127-137. [ Links ]
105 Yin JQ., Zhao RC., Morris KV. 2008. Profiling microRNA expression with microarrays. Trends Biotechnol. 26:70-6. [ Links ]
106 Yong Sun Lee and Anindya Dutta, 2009. Micro RNAs in Cancer. Annu. Rev. Pathol. Mech. Dis. 4:199-227. [ Links ]
107 Yu Z., Wang C., Wang M., Li Z., Casimiro MC., Liu M., Wu K., Whittle J., Ju X., Hyslop T., McCue P., Pestell RG. 2008. A cyclin D1/microRNA 17/20 regulatory feedback loop in control of breast cancer cell proliferation. J Cell Biol. 182:509-517. [ Links ]
108 Zhang B., Pan X., Cobb GP., Anderson TA. 2007. microRNAs as oncogenes and tumor suppressors. Dev Biol. 302(1):1-12. [ Links ]
109 Zhu S., Wu H., Wu F., Nie D., Sheng S., Mo YY. 2008. MicroRNA-21 targets tumor suppressor genes in invasion and metastasis. Cell Res. 18:350-359. [ Links ]
110 Zhu, J. Y.; Pfuhl, T.; Motsch, N.; Barth, S.; Nicholls, J.; Grässer, F.; and Meister, G. 2009. Identification of novel Epstein-Barr virus microRNA genes from nasopharyngeal carcinomas. Journal of virology. 83(7): 3333-3341. [ Links ]