Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Colombiana de Biotecnología
versão impressa ISSN 0123-3475
Rev. colomb. biotecnol vol.18 no.2 Bogotá jul./dez. 2016
https://doi.org/10.15446/rev.colomb.biote.v18n2.61514
DOI: http://dx.doi.org/10.15446/rev.colomb.biote.v18n2.61514
ARTÍCULO DE INVESTIGACIÓN
PCR en tiempo real: una metodología útil para la detección y cuantificación de granulovirus
Real Time PCR: a useful methodology for detection and quantitation of granulovirus
Gloria Barrera**, Jazmín Murcia*, Jorge Cerón*, Paola Cuartas**, Cristian Guzmán*, Laura Villamizar**
* Bacteriólogo. Corpoica. Km 14 vía Mosquera, Colombia.
** Ph.D. Corpoica. Km 14 vía Mosquera, Colombia.
gbarrera@corpoica.org.co.
(autor para correspondencia), lvillamizar@corpoica.org.co, pcuartas@corpoica.org.co
Recibido: abril 5 de 2016 Aprobado: octubre 3 de 2016
Resumen
El uso de baculovirus como agentes de control biológico de insectos plaga, se ha convertido en una estrategia efectiva que se ha implementado gradualmente en diferentes sistemas productivos a nivel mundial. Para el desarrollo de un bioplaguicida a base de baculovirus, es necesario contar con una metodología para determinar el título viral en el producto en proceso y terminado. Para tal fin, en este trabajo se diseñó y optimizó una técnica de cuantificación viral (Betabaculovirus) mediante PCR cuantitativo (q-PCR). Se utilizó una sonda TaqMan diseñada sobre el gen de granulina, altamente conservado. Para la técnica de q-PCR se determinó la especificidad, sensibilidad y reproducibilidad, encontrando que puede detectar y cuantificar aislamientos del género Betabaculovirus provenientes de cinco especies diferentes de insectos (granulovirus de Tecia solanivora, Phthorimaea operculella,Erinnyis ello, Tuta absoluta y Spodoptera frugiperda) incluso de diferente origen geográfico, pero no detecta aislamientos del género Alphabaculovirus (nucleopoliedrovirus de Spodoptera ornithogalli, Diatraea saccharalis o S. frugiperda). El límite mínimo de detección de la técnica fue de 6,4 x 10-4 ng de ADN, lo que equivale a 1,25 x 105 copias del gen. Así mismo, la variación intra e inter ensayos fue mínima, demostrando la reproducibilidad de la misma. La aplicabilidad de la técnica fue evaluada para la detección de granulovirus en muestras de larva, suelo, y para determinar la concentración viral en un bioplaguicida formulado como concentrado emulsionable. En conclusión, la técnica de q-PCR desarrollada fue reproducible, sensible y específica, con aplicabilidad en estudios de persistencia viral en campo, control de infecciones en crías de insectos y control de calidad de bioplaguicidas a base de betabaculovirus.
Palabras clave: Granulovirus, PCR cuantitativa, persistencia viral, control de calidad.
Abstract
The use of baculovirus as a biocontrol agent is an effective strategy, which has been gradually implemented in different production systems worldwide. For the development of a biopesticide based on baculovirus, it is necessary to have a methodology to determine the viral concentration in the process and in the finished product. In this study, a technique for viral quantification by quantitative PCR (q-PCR) was designed and optimized; therefore we used a TaqMan probe designed on granulin gene which is highly conserved. The specificity, sensitivity and reproducibility of the technique were determined. The q-PCR was able to detect and quantify isolates from the genus Betabaculovirus from five different insects species (granulovirus from Tecia solanivora, Phthorimaea operculella, Erinnys ello, Tuta absoluta and Spodoptera frugiperda) even from different geographic origins, while other isolates of baculovirus as from the genus Alphabaculovirus (nucleopolyhedrovirus from Spodoptera ornithogalli, Diatraea saccharallis or S. frugiperda) were not detected. The minimum detection limit of the technique was 6.4 x 10-4 ng /µl of DNA, equivalent to 1.25 x 103 gene copies. Additionally, intra- and inter-assays variation was minimal, demonstrating the reproducibility of technique. The applicability of the technique was evaluated for detecting granulovirus from samples of larva and soil, and to determine the virus concentration in the biopesticide formulated as emulsifiable concentrate. In conclusion, quantitative PCR was a technique reproducible, sensitive and specific to allow viral persistence studies in field, viral infection control in rearing and quality control of a biopesticide based on betabaculovirus.
Key words: Granulovirus, quantitative PCR, viral persistence, quality control.
Introducción
Dentro de los virus entomopatógenos, uno de los grupos más estudiados es la familia Baculoviridae, la cual se encuentra dividida en cuatro géneros: Alphabaculovirus donde se clasifican nucleopoliedrovirus (NPV), Betabaculovirus donde se clasifican granulovirus (GV), ambos patógenos de insectos del orden Lepidoptera, Gammabaculovirus y Deltabaculovirus que agrupan GV y NPV patógenos de insectos del orden Hymenoptera y Diptera, respectivamente (ICTV, 2012). La mayoría de baculovirus han sido aislados de insectos lepidópteros y algunos pocos de dípteros e himenópteros (Martínez et al., 2012), a pesar de que existen aproximadamente 7600 especies de hospederos descritas para estos virus (Martignoni & Iwai, 1981).
Dentro de la estructura de los GVs se encuentran los cuerpos de inclusión (OBs del inglés Occlusion Bodies) con forma de gránulo, que contienen los viriones (partículas infecciosas per os) dentro de una matriz de proteína que los protege de factores medioambientales deletéreos (Rorhman, 2010). La granulina es la proteína mayoritaria en estos gránulos y posee un rango de tamaño entre 26.000 y 28.000 daltons. Esta proteína es codificada por el gen gran (747 pb), altamente conservado en los genomas de los GVs, por lo que es ampliamente utilizado en estudios filogenéticos (Rohrman, 1986; Zanotto et al., 1993; De Moraes & Maruniak, 1997).
A nivel mundial, se ha demostrado ampliamente la efectividad y el alto potencial de los GVs para el control biológico de diversas plagas de insectos (Valicente & Da Costa, 1995; Batista et al., 2001; Barreto et al., 2005; Villamizar et al., 2005). Estos virus a base de GVs poseen un estrecho rango de hospederos, lo cual representa una estrategia segura para la protección de otros insectos no blanco y organismos benéficos. De acuerdo con lo anterior, la Corporación Colombiana de Investigación Agropecuaria-CORPOICA desarrolló un bioplaguicida a base de un granulovirus de Tecia solanivora, formulado como un concentrado emulsionable (CE) para el control de la polilla guatemalteca de la papa (Tecia solanivora, Povolny, 1973) en campo. Este bioplaguicida demostró su potencial reduciendo la población de larvas en un 96% en experimentos bajo condiciones controladas en casa malla y con una eficacia del 83% en campo (Gómez et al., 2011).
Como parte del desarrollo de un bioplaguicida a base de baculovirus, es necesario contar con metodologías para la identificación molecular y cuantificación del microorganismo (ingrediente activo) en diferentes puntos del proceso, metodologías que conforman parte del control de calidad y que son requeridas para adelantar también los estudios toxicológicos de las formulaciones indispensables para el proceso de registro (Guillon, 2003). Estas metodologías también son aplicables en estudios de bioprospección que incluyan la búsqueda de nuevos aislamientos así como en estudios de persistencia del virus en ensayos de campo.
Actualmente, para la determinación y cuantificación de granulovirus se utilizan varias metodologías, entre ellas algunas cualitativas como la microscopia óptica de campo oscuro, pruebas de reproducción de síntomas virales, electroforesis en geles denaturantes de poliacrilamida y ELISA (Enzyme-Linked ImmunoSorbent Assay) (Parola et al., 2003). Además, existen metodologías de carácter cuantitativo como la espectrofotometría (Zeddam et al., 2003) que es poco específica y posee baja sensibilidad, además de presentar limitaciones para cuantificar virus en muestras complejas como formulaciones o muestras de suelo (Graham et al., 2015).
Las metodologías basadas en determinación de ADN son específicas y sensibles, especialmente la técnica de q-PCR. Algunos sistemas utilizan "agentes intercaladores fluorescentes" de la doble cadena de ADN, sin embargo, existe la posibilidad de producir falsos positivos si aparecen productos de PCR inespecíficos o dímeros de cebadores (Costa, 2004). Los sistemas que emplean diversos tipos de sondas fluorescentes (Taqman®, «molecular beacons" y "scorpions") poseen la ventaja de hibridar únicamenteen la secuencia del ADN diana. Particularmente, la sonda Taqman® es un oligonucleótido con fluorocromos en los dos extremos que hibrida en regiones internas y específicas de los productos de PCR (Didenko, 2001).
Teniendo en cuenta la importancia de una técnica sensible y específica para la detección y cuantificación de granulovirus, el objetivo del presente trabajo fue diseñar una metodología basada en q-PCR para la búsqueda de cepas, seguimiento del virus en suelo y manufactura de un bioplaguicida. Para demostrar tres ejemplos de posibles aplicaciones de la técnica, se realizaron tres ensayos de detección, en larvas de la cría T. solanivora, en el bioplaguicida formulado a base de este virus y en muestras de suelo con aplicación del bioplaguicida.
Materiales y métodos
Virus
Se emplearon suspensiones virales puras de los aislamientos utilizados TesoGV (GV de Tecia solanivora), PhopGV (GV de Phthorimaea. operculella), EeGV (GV de Erinis ello), SfGVBra (GV de Spodoptera frugiperda de Brasil), SfGVCol (GV de S. frugiperda de Colombia), TuabGV (GV de Tuta absoluta), SoNPV (NPV de Spodoptera ornithogalli), DsNPV (NPV de Diatraea saccharalis) y SfMNPV (NPV de S. frugiperda). La cuantificación de los cuerpos de inclusión por mL de los granulovirus (CI/mL) se realizó por espectrofotometría, empleando una curva de calibración previamente estandarizada (Gómez et al., 2009) y la cuantificación de CI/ml de los nucleopoliedrovirus se realizó mediante recuento directo en cámara de Neubauer en un microscopio óptico (40 X) (Caballero et al., 2001).
Extracción de ADN
Para la extracción de ADN a partir de virus puro se utilizó el protocolo descrito por Caballero et al. (2001), con algunas modificaciones. Para tal fin, 100 µL de una suspensión viral ajustada a 108 CI/mL se mezcló con 100 µL de Na2CO3 (0,5 M) y 50 µL de SDS al 10%, a continuación se incubó durante 10 min a 60°C. El sobrenadante que contenía los viriones fue tratado con 50 µL de proteinasa K (10 mg/mL) durante 30 min a 50°C. La extracción de ADN viral se realizó mediante dos pases con fenol saturado y uno con cloroformo, utilizando centrifugaciones intermedias a 3000 rpm; la precipitación del ADN se realizó con etanol y acetato de sodio 3 M.
Para la extracción de ADN a partir de larvas infectadas, previamente se realizó la purificación viral mediante maceración y homogenización de larvas con SDS al 0,1% (p/v). A partir de esta suspensión, se realizó la purificación de los cuerpos de inclusión virales mediante filtración por muselina para retirar el tejido grueso del insecto y centrifugaciones diferenciales utilizando un gradiente discontinuo de sacarosa (Espinel-Correal et al., 2010). El ADN se extrajo como se describió anteriormente. Para las muestras de suelo, la extracción se realizó mediante el Kit comercial Power Soil de MOBIO (No. catálogo: 12888-50). Para esto, los viriones presentes en la muestras fueron liberados de los CI mediante la adición de Na2CO3 (0,5 M), después de la extracción de los ácidos húmicos con el Kit. La recuperación de ADN se realizó en membrana y la elución con Tris 10 mM.
Para la extracción de ADN a partir del bioplaguicida a base del granulovirus VG003 formulado como un concentrado emulsionable (CE), primero se realizó la eliminación de los excipientes de formulación mediante la adición de hexano y posteriormente se realizó la extracción de ADN con cloroformo y etanol como se mencionó anteriormente (Caballero et al., 2001). La concentración de ADN fue estimada mediante espectrofotometría en un equipo Nanodrop 1000 (Thermo Fisher Scientific).
Cebadores y sonda
La sonda y los cebadores se diseñaron basados en la secuencia del gen de granulina de un aislamiento nativo, teniendo en cuenta su alineamiento con secuencias consignadas en el GENBANK, mediante la herramienta BLAST (Basic Local Alignment Search Tool) y seleccionando una región completamente homóloga entre las secuencias analizadas. Para el diseño de los cebadores y la sonda Taqman en la región seleccionada, se utilizó la herramienta SciTools Primer Quest de la empresa Integrated ADN Technologies IDT. Se seleccionó la opción con mejores características de temperatura de anillaje, estabilidad y porcentaje de GC en los cebadores. La sonda (5'/ 56-FAM /TCTCAAGAGTTTGGGTTCGGTGCT/ 3IABlk_FQ /3') se diseñó con el 6-carboxi fluoresceína en el extremo 5', como fluoróforo reportero y un Quencher Iowa Black (diseñado por IDT) en el extremo 3', el cual posee un espectro de absorbancia entre 420 a 620 nm, ideal para fluoresceína. Los cebadores fueron: Cebador reverso: 5' TCCTCGGGTACCATGTACTG 3' y Cebador directo: 5' CACTTGCGTCATCGACAA 3', los cuales amplifican un segmento de 137 pb del gen de granulina.
Construcción de controles positivos y curvas estándar
Para amplificar el gen completo de granulina se utilizaron cebadores degenerados (Barrera et al., 2009):
Gran-F: 5'-ATGGGATAYAAYAAAWCDYT-3'
Gran-R: 5'-TYARTANGCBGGDCCVGTRAA-3'
Los productos de PCR se ligaron directamente a un plásmido vector pCR 2.1-TOPO utilizando el Kit TOPO TA (Invitrogen K4500-01). La transformación se realizó en células de Escherichia coli quimiocompetentes Top10 (Invitrogen K4500-40). Las bacterias se crecieron en medio Luria Bertani-LB con ampicilina (50 µg/ml) a 37°C por 14 horas. Los clones positivos se seleccionaron y se confirmaron por secuenciación mediante el método de Sanger. El ADN obtenido de los plásmidos se utilizó para los controles positivos de la técnica q-PCR y para determinar la sensibilidad de la misma. El número de copias del gen por µL para las diluciones de ADN plasmídico se estimó mediante la fórmula descrita por Whelan y colaboradores (2003). Las curvas estándar se construyeron a partir de un ciclo umbral (threshold cycle ct) calculado para log10 copias del gen.
PCR en tiempo real
Para la optimización de las condiciones de q-PCR se utilizó un sistema convencional comercial con Taq Polimerasa, dinucleótidos trifosfatos (dNTPs) y cloruro de magnesio (MgCl2) marca Promega. Se probaron diferentes condiciones de amplificación (gradiente de temperatura entre 48,7°C y 55,6°C); asimismo diferente número de ciclos de 30, 35 y 40, con modificaciones en las concentraciones de cebadores y sonda (0.24µM, 0.36µM y 0.48µM). Para el desarrollo de este ensayo, se utilizó ADN viral obtenido a partir de las suspensiones virales purificadas. La reacción de amplificación consistió en un primer paso de 3 min a 95°C, seguido por 35 ciclos así: 10 segundos a 95°C, 45 segundos a 52°C y 30 segundos a 72°C. La amplificación, la detección de la señal y el análisis de datos se realizaron en el equipo ICycler IQ (BioRad).
Análisis de especificidad, sensibilidad y reproducibilidad
Para determinar la especificidad de la técnica de PCR en tiempo real, se utilizaron muestras de diferentes aislamientos de baculovirus: un aislamiento nativo de granulovirus de T. solanivora, un aislamiento peruano del granulovirus de P. operculella y un aislamiento colombiano de nucleopoliedrovirus de S. frugiperda. La sensibilidad de la PCR se determinó usando diluciones seriadas de ADN plasmídico con un factor de dilución 1/5, desde 1,96 x109 hasta 1,25 x 105 copias del gen por reacción. Este ensayo se repitió tres veces en el tiempo para demostrar la reproducibilidad de la técnica con tres réplicas intra-corrida de cada estándar.
Aplicaciones de la técnica de PCR en tiempo real
Cría de insectos: Se analizaron tres muestras de larvas sin infección aparente, provenientes de la cría del laboratorio de Control Biológico de CORPOICA.
Cuantificación de granulovirus en bioplaguicida
Se evaluaron dos lotes de producción del concentrado emulsionable a base de granulovirus de T. solanivora, elaborado siguiendo la metodología descrita por Gómez et al. (2011). Cada lote fue preparado con 3,5 L de suspensión viral con una concentración de 1,2 x 109 CIs/mL. La extracción de ADN de cada concentrado emulsionable se realizó como se describió anteriormente. La cuantificación del ingrediente activo se realizó por triplicado en cada lote.
Seguimiento del virus en campo: Se utilizó una parcela experimental (1.411 m2 aprox.) en una finca productora de papa, en la vereda El Alizal, municipio de Carmen de Carupa (Cundinamarca), sembrada con la variedad Parda Pastusa. La parcela fue manejada según las recomendaciones agronómicas generales, sin aplicación de plaguicidas de síntesis química para el manejo de la polilla guatemalteca. Quince días después del aporque se realizó la aplicación del concentrado emulsionable a base del GV de T. solanivora en una concentración de 1 x 108 CI/mL utilizando un volumen de aplicación de 400 L/ha. El bioplaguicida fue reconstituido en agua y aplicado por aspersión con bomba convencional de espalda (20 L) de cono hueco, dirigido a la base de las plantas (30 mL aprox. por planta), y abarcando un área circular con un radio aproximado de 25 cm alrededor del tallo. Se tomaron muestras de aproximadamente 8 g de suelo superficial con ayuda de una brocha, alrededor del tallo de tres plantas seleccionadas al azar. Los muestreos se realizaron una hora después de la aplicación y pasados 5, 10 y 15 días. Las mezclas se trituraron hasta obtener un polvo fino, se mezclaron y se tomó una muestra para la extracción de ADN según la metodología descrita anteriormente.
Resultados y discusión
Análisis de especificidad, sensibilidad y reproducibilidad
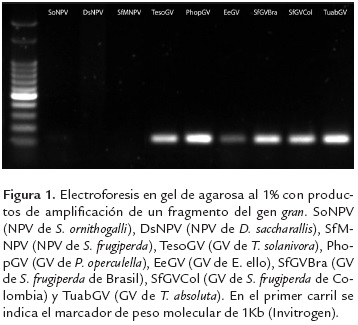
Los cebadores diseñados en este trabajo para amplificar un fragmento del gen de granulina de 137 pb, permitieron obtener amplicones utilizando ADN genómico de granulovirus provenientes de cinco especies diferentes de insectos, uno de T. solanivora, uno de P. operculella, uno de E. ello, uno de T. absoluta y dos de S. frugiperda (figura 1). Por otra parte, no se observaron fragmentos de amplificación en muestras de NPVs de S. ornithogalli, D. sacharallis o S. frugiperda.
Todos los aislamientos pertenecientes al género Betabaculovirus se caracterizan por presentar la proteína mayoritaria granulina en sus cuerpos de inclusión. El gen que codifica esta proteína presenta muy poca variabilidad entre los granulovirus (Garavaglia et al., 2012), por lo cual la metodología fue capaz de amplificar muestras de diferentes especies dentro del género.
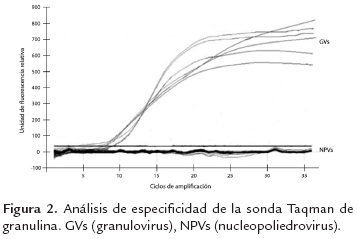
En los resultados de la PCR en tiempo real se observan las curvas de amplificación dadas por el crecimiento exponencial de las copias del gen (fluorescencia) ubicadas en el eje de las ordenadas versus el número de ciclos de la reacción ubicado en el eje de las abscisas. La especificidad de la sonda Taqman se observó en la amplificación por PCR en tiempo real, donde se utilizaron ADNs de los nueve aislamientos de GVs empleados en la PCR convencional (figura 2). Se observó señal de fluorescencia para los aislamientos de GVs mientras que la señal fue nula para los aislamientos de NPVs.
La PCR en tiempo real o cuantitativa (q-PCR), es una herramienta capaz de amplificar y simultáneamente cuantificar de forma absoluta el producto de ADN debido a la fluorescencia, la cual está correlacionada con la cantidad. A mayor número de copias iniciales de la muestra de ADN, más rápido se detectará un incremento en la fluorescencia dada en la q-PCR, como consecuencia de la acumulación de productos de PCR. De esta forma, es posible realizar la cuantificación de ADN de una forma exacta y reproducible. Sin embargo, existen algunos factores inherentes al desarrollo de la PCR que pueden influir en la especificidad de la técnica, los cuales se encuentran asociados con los niveles de astringencia de la misma; componentes como el cloruro de magnesio, el tipo de polimerasa y la concentración de las sondas son críticos (Espy, 2006), por lo cual la optimización de estos factores fue clave en los resultados obtenidos.
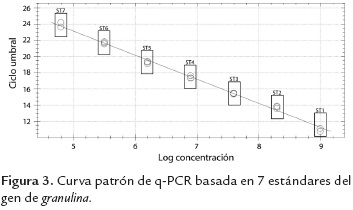
Para la cuantificación absoluta del gen gran por PCR en tiempo real se utilizaron diluciones seriadas del ADN plasmídico con un inserto del gen completo, con las cuales se graficó una recta estándar o patrón (figura 3). A partir de esta recta se determinó la mínima dilución en la cual se observó amplificación, la cual correspondió a 1,25 x 105 copias del gen correspondientes a 0,00064 ng de ADN.
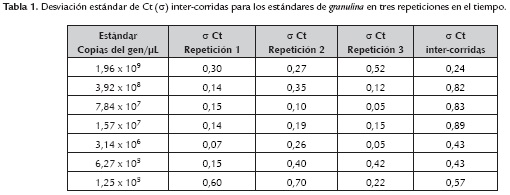
La reproducibilidad de la técnica fue evaluada realizando 3 ensayos de amplificación en diferentes tiempos, además de tres réplicas de cada ADN patrón en cada prueba. La desviación estándar (σ) intra e inter-ensayos (para cada ADN patrón) fue baja y nunca mayor de 1 (tabla 1), lo cual demostró que la técnica fue reproducible. La variabilidad entre las repeticiones de una muestra en el sistema Taqman es muy baja y generalmente se aumenta cuando se trabaja con concentraciones muy bajas o con amplicones de gran tamaño (superiores a 150 pb) (Bustin, 2000).
También se debe considerar que la reproducibilidad de la técnica de PCR en tiempo real se afecta por pequeñas variaciones en las concentraciones de los reactivos y errores de manipulación (Bustin, 2002), por lo cual la validación con estándares basados en el gen clonado contribuye a asegurar la calidad y los resultados de la cuantificación de las muestras (Epsy et al., 2006). Otras variables son importantes para la validación de la metodología, el coeficiente de correlación R2, la eficiencia y la pendiente de la curva patrón (Scott, 2006). Las curvas de calibración en cada corrida de muestras fueron verificadas mediante el valor de coeficiente de correlación superior a 0,99 y eficiencias mayores a 95%, según lo recomendado por Whelan y colaboradores (2003).
Aplicaciones de la metodología de PCR en tiempo real
Cría de insectos: Las crías de insectos utilizadas para evaluación de la actividad insecticida y para la multiplicación de diferentes aislamientos de granulovirus, son mantenidas en condiciones controladas de laboratorio. Los individuos que se utilizan para el establecimiento de cada cría y para su posterior recambio provienen de muestras de campo. Una metodología de control para introducir individuos libres de infección consiste en mantener estos individuos en cuarentena por una o dos generaciones y la posterior identificación de larvas libres de síntomas de enfermedad, lo que hace dispendioso y demorado el proceso de introducción de individuos a las colonias. El análisis por q-PCR de individuos de la cría de CORPOICA no mostró amplificación del gen de granulina en ninguna de las muestras analizadas. Esta metodología es altamente sensible, pues detecta mínimas cantidades de copias del gen que puedan estar presentes en una larva, aún en concentraciones subletales o latentes.
Las infecciones subletales son transmitidas verticalmente de parentales a su descendencia y son utilizadas como mecanismo de supervivencia viral cuando la infección horizontal es limitada; sin embargo, estas pueden llegar a ser letales en condiciones de estrés por cambios medioambientales o por la infección de otros patógenos (Cabodevilla et al., 2011). En el caso de uso de las crías para ensayos de actividad insecticida, las infecciones subletales se pueden reactivar y ser letales, causando resultados poco repetibles o generando falsos positivos.
Cuantificación de granulovirus en bioplaguicida: En el presente trabajo se determinó la concentración viral en dos lotes de un bioplaguicida formulado como un concentrado emulsionable a base de un aislamiento nativo de granulovirus. La cuantificación del ingrediente activo en el bioplaguicida formulado por la técnica de q-PCR mostró concentraciones similares a la concentración teórica esperada en producto final (1,6 x 109 CIs/mL), la cual se calcula teniendo en cuenta la concentración de la suspensión viral, la cantidad de sólidos adicionados al producto y la humedad final del mismo. Este resultado demuestra que la técnica es útil para hacer el control de calidad de la concentración del virus durante el proceso de manufactura de un bioplaguicida, tanto al producto en proceso como al producto terminado. Los resultados obtenidos para los dos lotes demuestran además, que no existen pérdidas importantes de principio activo durante el proceso de formulación.
Seguimiento del virus en campo: Los resultados de las cuantificaciones realizadas en las muestras de suelo, mostraron una disminución progresiva de las concentraciones del virus en el tiempo (tabla 2). La disminución de granulovirus durante el tiempo es debida posiblemente a un proceso natural de degradación o posiblemente al movimiento en el suelo debido a procesos de lixiviación por la lluvia. Estudios de persistencia de baculovirus mediante ensayos de eficacia, se han realizado en cultivos de tomate, algodón y soja para el control de Helicoperva armigera y en cultivos de manzana para el control de Cydia pomonella, evidenciando la presencia viral hasta 14 días post-aplicación (Tamez et al., 2000; Arthurs & Lacey, 2004). Sin embargo, técnicas más sensibles basadas en ADN podrían determinar la presencia viral en concentraciones más bajas y tiempos más prolongados.
El suelo representa el mayor reservorio de virus en el medio ambiente. Los cuerpos de inclusión pueden persistir en suelos ácidos o neutrales durante meses o años antes que sean transportados por el impacto de las gotas de lluvia, corrientes de aire, o mediante el movimiento de los artrópodos de la superficie del suelo (Williams, 2015). Para el análisis de la presencia de Baculovirus en suelos se ha utilizado la infección de larvas (Murillo et al., 2006), sin embargo, esta metodología no permite cuantificar la concentración de virus presente, ni permite la realización de estudios de seguimiento del virus posterior a su aplicación.
Conocer la persistencia del granulovirus en condiciones de cultivos de papa es importante para generar recomendaciones relacionadas con la forma y la frecuencia de aplicación del bioplaguicida, así como las concentraciones que se deben utilizar para el manejo integral de la plaga. Los análisis de persistencia con la metodología de q-PCR deben estar acompañados de estudios de eficacia, debido a que la técnica detecta la presencia de ADN del virus, lo cual no está directamente relacionado con su actividad biológica, que puede ser modificada por diferentes factores como luz ultravioleta, suelos alcalinos, etc.
Conclusiones
La metodología de q-PCR basada en el gen de granulina detecta betabaculovirus aislados de diferentes especies de insectos, debido a la alta conservación de este gen dentro del género viral. Este método de detección es de gran utilidad para la detección y cuantificación de granulovirus en diferentes tipos de muestras, incluyendo aquellas obtenidas a partir de tejido larval, suelo y bioplaguicidas a base de este virus. Lo anterior representa una ventaja sobre otras técnicas ampliamente utilizadas donde se requieren muestras de virus purificados para su cuantificación. La q-PCR desarrollada en este trabajo, se puede incluir en diferentes etapas del desarrollo de un bioplaguicida a base de granulovirus, desde la búsqueda de aislamientos con potencialidad para el control biológico hasta el control de calidad del proceso de manufactura del bioplaguicida.
Agradecimientos
Los autores expresan su agradecimiento a Colciencias por el apoyo financiero del presente trabajo.
Referencias bibliográficas
Arthurs, S.P., & Lacey, L.A. (2004). Field evaluation of commercial formulations of the codling moth granulovirus: persistence of activity and success of seasonal applications against natural infestations of codling moth in Pacific Northwest apple orchards. Biological Control, 31(3), 388-397. [ Links ]
Barrera, G.P., Cuartas, P., & Villamizar, L. (2009). Comparative analysis of a granulin fragment of Colombian granulovirus isolated from Tecia solanivora. IOBC/wprs Bulletin, 45, 129-132. [ Links ]
Barreto, M., Guimaraes, C., Texeira, F., Paiva, E., Valicente, F. (2005). Effect of Baculovirus spodoptera isolates in Spodoptera frugiperda (J.E. Smith) (Lepidoptera: Noctuidae) larvae and their characterization by RAPD. Neotropical Entomology, 34(1), 67-75. [ Links ]
Batista, A., Alves, S.B., Augusto, N., Pereira, R.M., & Alves, L. (2001). Stability and persistence of two formulations containing Anticarsia gemmatalis nuclear polyhedrovirus (AgMNPV). Neotropical Entomology, 30(3), 411-416. [ Links ]
Bustin, S. (2000). Absolute quantification of RNAm using real-time reverse transcription polymerase chain reaction assays. Journal of Molecular Endocrinology, 25, 169-193. [ Links ]
Bustin, S. (2002). Quantification of mrna using real-time reverse transcription PCR (RT-PCR): trends and problems. Journal of Molecular Endocrinology, 29, 23-39. [ Links ]
Caballero, P., Williams, T., & López-Ferber, M. (Ed). (2001). Los baculovirus y sus aplicaciones como bioinsecticidas en el control biológico de plagas. Valencia. España: Phytoma S.A. [ Links ]
Cabodevilla, O., Ibañez, I., Simón, O., Murillo, R., Caballero, P., & Williams, T. (2011). Occlusion body pathogenicity, virulence and productivity traits vary with transmission strategy in a nucleopolyhedrovirus. Biological Control, 56(2), 184-192. [ Links ]
Costa, J. (2004). Reacción en cadena de la polimerasa (PCR) a tiempo real. Enfermedades Infecciosas y Microbiología Clínica, 22(5), 229-305. [ Links ]
de Moraes, R.R., & Maruniak, J.E. (1997). Detection and identification of multiple baculoviruses using the polymerase chain reaction (PCR) and restriction endonuclease analysis. Journal of Virology Methods, 63, 209-217. [ Links ]
Didenko, V.V. (2001). DNA probes using fluorescence resonace energy transfer (FRET). Desings and applications. BioTechniques, 31, 1106-1121. [ Links ]
Espinel-Correal, C., Léry, X., Villamizar, L., Gómez, J., Zeddam, J.L., Cotes, A.M., & López-Ferber, M. (2010). Genetic and biological analysis of Colombian Phthorimaea operculella granulovirus isolated from Tecia solanivora (Lepidoptera: Gelechiidae). Applied and Environmental Microbiology, 76(22), 7617-25. [ Links ]
Espy, M.J., Uhl, J.R., Sloan, L.M., Buckwalter, S.P., Jones, M.F., Vetter, E.A., Yao, J.D., Wengenack, N.L., Rosenblatt, J.E., Cockerill III, F.R., & Smith, T.F. (2006). Real-Time PCR in clinical microbiology: Applications for routine laboratory testing. Clinical Microbiology Reviews, 19(1), 165-256. [ Links ]
Garavaglia, M.J., Miele, S.A.B., Iserte, J.A., Belaich, M.N., & Ghiringhelli, P.D. (2012). The ac53, ac78, ac101, and ac103 genes are newly discovered core genes in the family Baculoviridae. Journal of Virology, 86 (22), 12069-79. [ Links ]
Gómez, J., Moreno, C.A., Vega, K., Cotes, A.M., & Villamizar, L. (2011). Formulation effect over insecticidal activity of Phthorimaea operculella granulovirus VG003 for controlling Tecia solanivora. IOBC/wprs Bulletin. 66, 441-445. [ Links ]
Gómez, J., Villamizar, L., Espinel, C., & Cotes, A.M. (2009). Comparación de la eficacia y la productividad de tres granulovirus nativos sobre larvas de Tecia solanivora (Povolny) (Lepidoptera: Gelechiidae). Revista Corpoica - Ciencia y Tecnología Agropecuaría, 10(2), 152-158. [ Links ]
Graham, R.I., Tummala, Y., Rhodes, G., Cory, J.S., Shirras, A., Grzywacz, D., & Wilson, K. (2015). Development of a Real-Time qPCR assay for quantification of covert baculovirus infections in a major african crop pest. Insects, 6, 746-759. [ Links ]
Guillon, M.L. (2003). Regulation of biological control agents in Europe. En: Roettger, U.; Reinhold, M.(eds). International symposium on biopesticides for developing countries. CATIE, Turrialba. pp. 143-147. [ Links ]
ICTV. (2012). International Committee on Taxonomy of viruses. [ Links ]
Martínez, A.M., Pineda, S., Figueroa, J.I., Chavarrieta, J.M., & Williams, T. (2012). Los baculovirus como bioinsecticidas: evaluación de un nucleopoliedrovirus para el combate de Spodoptera frugiperda (Lepidoptera: Noctuidae) en México y Honduras. Ciencia Nicolaita, 56, 35-47. [ Links ]
Murillo, R., Muñoz, D., & Caballero, P. (2006). El potencial de los baculovirus como agentes de control biológico de plagas; Phytoma. 179, 51-63. [ Links ]
Parola, A.D., Sciocco-Cap, A., Glikmann, G., & Romanowski, V. (2003). An immunochemical method for quantitation of Epinotia aporema granulovirus (EpapGV). Journal of Virological Methods, 112, 13-21. [ Links ]
Rohrmann, G.F. (1986). Polyhedrin structure. Journal of General Virology, 67, 1499-1513. [ Links ]
Rohrmann, G.F. (2010). Baculovirus molecular biology. National Library of Medicine National Center for Biotechnology Information, Bethesda, MD. [ Links ]
Scott, P. (2006). Data analysis and reporting. En: Real-Time PCR. Editor Tevfik, D. p. 332. Oxford, England. Taylor & Francis. [ Links ]
Tamez-Guerra, P., McGuire, M.R., Behle, R.W., Hamm, J.J., Sumnern, H.R., & Shasha, B.S. (2000). Sunlight persistence and rainfastness of spray-died formulations of baculoviruses isolated from Anagrapha falcifera (Lepidoptera: Noctuidae). Journal of Economic Entomology, 93, 210-218. [ Links ]
Valicente, F.H., & Costa, E.F. (1995). Control of fall armyworm, Spodoptera frugiperda (J.E. Smith) with baculovirus spodoptera throught irrigation water. Anais Da Sociedade Entomológica Do Brasil, 24, 61-67. [ Links ]
Villamizar, L., Zeddam, J.L., Espinel, C., & Cotes, A.M. (2005). Implementación de técnica de control de calidad para la producción de un bioplaguicida a base del granulovirus de Phthorimaea operculella PhopGV. Revista Colombiana de Entomología, 31(2), 127-132. [ Links ]
Williams T. (2015). Biology and Ecology of Baculoviruses. Recuperado de http://www.trevorwilliams.info/Ecology_baculoviruses.htm. [ Links ]
Whelan, J., Rusell, N., & Whelan, M. (2003). A method for the absolute quantification of cDNA using real-time PCR. Journal of Immunological Methods, 278, 261-269. [ Links ]
Zanotto, P.M., Kessing, B.D., & Maruniak, J.E. (1993). Phylogenetic interrelationships among baculoviruses: evolutionary rates and host associations. Jornal of Invertebrate Pathology, 62, 147-164. [ Links ]
Zeddam, J.L., Vasquez, R.M., Vargas, Z., & Lagnaoui, A. (2003). Producción viral y tasas de aplicación del granulovirus usado para el control biológico de las polillas de la papa Phthorimaea operculella y Tecia solanivora (Lepidoptera: Gelechiidae). Boletin de Sanidad Vegetal. Plagas, 29, 659-667. [ Links ]