Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Biotecnología
Print version ISSN 0123-3475
Rev. colomb. biotecnol vol.18 no.2 Bogotá July/Dec. 2016
DOI: http://dx.doi.org/10.15446/rev.colomb.biote.v18n2.61520
ARTÍCULO DE INVESTIGACIÓN
Clonación, expresión y caracterización de una nueva esterasa derivada de metagenomas de suelos agrícolas colombianos
Cloning, expression and characterization of a novel esterase derived from colombian agricultural soils metagenomes
Carolina Villamil*, Patricia Del Portillo**, Alvaro Monguí**
* Bcs., Corporación CorpoGen Cr5 No 66a -34, Bogotá-Colombia.
cvillamil@corpogen.org
** PhD, Biotecnología Molecular, Corporación CorpoGen Cr5 No 66a -34, Bogotá-Colombia.
alvaro.mongui@corpogen.org
Recibido: diciembre 12 de 2015 Aprobado: octubre 6 de 2016
Resumen
El presente trabajo tuvo como objetivo la bioprospección de ADN metagenómico derivado de comunidades microbianas asociadas a un agroecosistema de importancia nacional. Este análisis permitió realizar la producción, expresión, purificación y caracterización de una enzima novedosa con actividad esterasa. Esta enzima, denominada LipM, había sido previamente identificada en clones metagenómicos derivados de suelos dedicados al cultivo de papa criolla (Solanum pureja), mediante secuencia de nueva generación y análisis bioinformáticos. La secuencia codificante de la enzima fue clonada en el vector pBADgiii y expresada en E. coli como sistema de expresión, lo que permitió optimizar el proceso de producción recombinante y su posterior purificación.
Funcionalmente la enzima presentó una mayor afinidad por sustratos de p-nitrofenil con ácidos grasos de cadena corta (<C8). LipM mostró completa funcionalidad a temperaturas entre 30 – 37 °C y en valores de pH cercanos al fisiológico (entre 7.0 y 8.0). Igualmente, esta enzima exhibió buena estabilidad en presencia de varios iones metálicos, inhibidores y 0.1% (p/v) de SDS. Su alto nivel de estabilidad en presencia de iones metálicos e inhibidores, así como su particular especificidad en cuanto a sustratos, la hacen una enzima óptima para utilización en diferentes aplicaciones biotecnológicas.
Palabras clave: Metagenómica, enzima esterasa, caracterización, suelos, Lipasa/esterasa.
Abstract
The present work had as a main objective to bioprospect metagenomic DNA from microbial communities associated with an agro-ecosystem of national importance. This analysis allowed the production, expression, purification and characterization of a novel enzyme with esterase activity. This enzyme, named here as LipM, was previously identified in metagenomic clones derived from soils dedicated to creole potato (Solanum pureja) crops by means of next-generation sequencing and bioinformatics analyses. The coding sequence of the enzyme was cloned into pBADgiii vector and expressed in E. coli as an expression system, allowing to optimize its recombinant production process and its further purification.
The enzyme functionally showed a greater affinity for p-nitrophenyl substrates with short-chain fatty acids (<C8). LipM showed full functionality at temperatures between 30 - 37°C and close to physiological pH (between 7.0 and 8.0). This enzyme also exhibited proper stability in the presence of various metal ions, inhibitors and at 0.1% (w/v) of SDS. Its high level of stability in the presence of inhibitors, metal ions and its particular specificity of substrates make it optimal for use in various biotechnology applications.
Key words: Metagenomics, esterase enzyme, characterization, soil, lipase/esterase.
Introducción
La metagenómica corresponde al aislamiento y estudio del material genético total (metagenoma) asociado a un determinado ambiente natural. La importancia de la metagenómica, en comparación con la secuenciación de genomas individuales y el método tradicional de cultivo, radica en que permite analizar la información genética de la gran mayoría de microorganismos que no son cultivables en condiciones de laboratorio (hasta el 99%) (Staley & Konopka, 1985). Los metagenomas derivados de ambientes naturales, como es el caso específico de suelos, tienen ventajas adicionales ya que debido a las condiciones ambientales de este tipo de ecosistema, la comunidad microbiana que allí se encuentra es sumamente rica y diversa (Torsvik & Ovreas, 2002). Esto convierte a la metagenómica en una herramienta poderosa para explorar el potencial biocatalítico de las comunidades microbianas y/o sus moléculas provenientes de los ambientes edáficos (Lee & & Lee, 2013).
Los ensayos realizados sobre librerías metagenómicas, tanto funcionales como aquellos basados en secuencia de ácidos nucleicos y análisis bioinformáticos, permiten identificar clones bacterianos con actividades de interés. Posteriormente, los genes asociados a dichos fenotipos pueden ser caracterizados e insertados en algún hospedero, con el fin de producir de forma recombinante las respectivas enzimas o metabolitos (Uchiyama & Miyazaki, 2009). Dentro de las diferentes clases de enzimas caracterizadas de metagenomas ambientales, las hidrolasas son altamente atractivas no solo por su importancia industrial, sino por tener una elevada especificidad de sustrato y una considerable estabilidad en solventes orgánicos (Hasan et al., 2006; Sharma et al., 2001; Uwe & Kazlauskas, 2006).
Las hidrolasas de origen bacteriano (EC 3.1.1.3) son aquellas que catalizan la hidrólisis y la síntesis de acilgliceroles de cadenas largas o cortas. Pertenecen a un amplio y diverso grupo denominado esterasas (EC 3.1.1.x) que incluye las carboxilesterasas (EC 3.1.1.1), las lipasas verdaderas (EC 3.1.1.3) y las fosfolipasas (Arpigny & Jaeger, 1999). Las lipasas y esterasas comparten un dominio de estructura característico conocido como el plegamiento α/β de las hidrolasas, que incluye un sitio activo altamente conservado correspondiente al triado catalítico Ser-Asp-His (Arpigny & Jaeger, 1999). En general estas enzimas juegan un papel importante a nivel industrial, ya que están involucradas principalmente en los procesos de producción de detergentes, en el procesamiento de alimentos y en la síntesis orgánica estero-especifica (Hasan et al., 2006). Adicionalmente, las lipasas y las esterasas están relacionadas con la bioconversión de lípidos en diferentes procesos, como es el aprovechamiento de fuentes de carbono, la modificación o el reciclaje de las membranas celulares la síntesis de biopolímeros, la síntesis del diesel y la producción en general de productos agroquímicos, saborizantes y fármacos (Hasan et al., 2006).
La demanda actual del mercado por lipasas/esterasas ha favorecido, durante los últimos años, su exploración en el contexto de librerías metagenómicas. De esta forma, ensayos funcionales y bioinformáticos con librerías metagenómicas derivadas de una amplia variedad de ambientes naturales, como son sedimentos marinos (Zhu et al., 2013), lodos activados (Zhang & Han, 2009), aguas termales (López et al., 2014), manglares (Andreote et al., 2012) y compost (Kim et al., 2010), entre otros, han favorecido el descubrimiento de genes novedosos codificantes para lipasas y esterasas con alto potencial de aplicación biotecnológico e industrial.
En estudios previos, nuestro grupo de investigación identificó un gen codificante para una lipasa/esterasa putativa, empleando métodos de secuencia masiva y análisis bioinformáticos, a partir de librerías metagenómicas de suelos de vocación agrícola (Calderón et al., manuscrito en preparación). En el presente artículo reportamos la clonación, expresión y caracterización de la actividad enzimática de la esterasa denominada LipM, usando para ello diferentes sustratos y evaluando el efecto del pH, la temperatura, diferentes cofactores e inhibidores, con el fin de determinar su rango y actividad relativa.
Materiales y Métodos
Clonación de un gen con dominio α/β hidrolasa de un metagenoma ambiental
El gen LipM, con dominio α/β hidrolasa fue amplificado de su clon metagenómico parental usando los iniciadores pBADgiiiA-LipM directo 5'- CG TCT AGA GCC TGT CGA TCA GCC AAC -3' y pBADgiiiA-LipM reverso 5'- CG TCT AGA ACC AGG TGC GCC CTC -3'. Para la amplificación por PCR se inició con un paso de desnaturalización a 95 °C por 5 min, seguido por 35 ciclos de: (a) desnaturalización a 95 °C por 45 s, (b) anillaje a 56 °C por 45 s y (c) extensión a 72 °C por 1 min. Finalmente se incluyó una extensión final de 8 min a 72 °C. El producto de PCR obtenido fue purificado, completamente digerido con la enzima XbaI e insertado utilizando la enzima T4 ligasa, en el vector de expresión pBADgiiiA del kit pBADgiii_man (Thermo Fisher Scientific, Massachusetts, EEUU), previamente digerido con la misma enzima de restricción y siguiendo las recomendaciones del fabricante. La correcta inserción de la secuencia codificante de LipM en el vector de expresión fue confirmada por secuenciación Sanger. El plásmido resultante, pBADgiiiA-LipM, fue transformado por electroporación en la bacteria Escherichia coli TOP10 (Thermo Fisher Scientific), siguiendo el protocolo del fabricante.
Expresión y purificación
La bacteria E. coli TOP10 con el plásmido pBADgiiiA-LipM fue incubada en agitación (200 rpm) a 37 °C en medio de cultivo Luria Bertani (LB) suplementado con ampicilina 50 mg/mL, hasta alcanzar una OD600 de 0.4. Posteriormente, la expresión de la proteína LipM recombinante (rLipM) se llevó a cabo por inducción con 0.2 % (p/v) de L-arabinosa. Después de 4 horas adicionales de incubación, el cultivo se centrifugó a 4600 x g por 25 minutos a 4 °C. El pellet celular resultante fue resuspendido en buffer Tris 10mM, 6M urea, 100mM NaH2PO4 e imidazol 15mM a pH 8.0 (García et al., 2013; Lara, 2011). La lisis celular se llevó a cabo con 1/3 (v/v) de perlas de 0.1mm de zirconia/silica en FastPrep-24 (MP Biomedicals California, USA) siguiendo un protocolo de 4 ciclos de lisis por acción mecánica a 60 m /min por 40 s y luego una etapa de enfriamiento a 4 º C por 5 min. Las muestras fueron luego centrifugadas a 15700 x g por 15 min y el sobrenadante resultante se acopló con la resina Ni-NTA (Qiagen Duesseldorf, Alemania) en agitación suave durante 4 h a temperatura ambiente, siguiendo las indicaciones del fabricante para llevar a cabo la purificación proteica por cromatografía de afinidad. Posteriormente, el extracto acoplado a la resina fue lavado con el mismo buffer de lisis, con el propósito de remover todas las proteínas no retenidas en la resina. La proteína rLipM fue eluida utilizando buffer de lisis con diferentes concentraciones de Imidazol (60-500 mM). Las fracciones resultantes se sometieron a electroforesis en gel de poliacrilamida-dodecil sulfato de sodio (SDS-PAGE) al 12%, en condiciones reductoras y desnaturalizantes, durante 2 h a 80 voltios. Posteriormente, las proteínas se transfirieron a una membrana Hybond-polivinildifluoruro (PVDF) (Merckmillipore Massachusetts, EEUU), en cámara semiseca (Laboratorios Bio-Rad, California, EEUU) durante 4 h a 10 voltios. La membrana fue sometida a bloqueo en una solución de PBS, Tween 20 (0,05 %) y leche descremada (5% p/v) durante una hora y posteriormente incubada con un anticuerpo anti-Histidinas conjugado con peroxidasa (Sigma-Aldrich, Saint Louis, USA). La detección de las proteínas se realizó utilizando un estuche para revelado con sustrato de peroxidasa (Laboratorios Vector, California, EEUU). Las fracciones de elución que incluyeron la proteína rLipM purificada fueron dializadas usando membranas de nitrocelulosa de poro 12-14 kDa contra PBS a 4 º C por 18 h.
Ensayo de actividad enzimática
La actividad enzimática de rLipM se evaluó por ensayo de actividad esterasa utilizando 12.5 µL de enzima purificada, 100 μL de buffer TRIS-HCl pH 7.5, para una concentración final de 50 mM y 12.5 μL de sustrato p-nitrofenil butirato (C4) (Sigma-Aldrich) para una concentración de 1 mM a 37 °C por 30 min. La cantidad de p-nitrofenol liberado se determinó por absorbancia a 410 nm en NanoDrop (Thermo Fisher Scientific). La reacción con Tris-HCl y PBS y sin enzima fue usada como blanco de ensayo y la Lipasa B de Candida antarctica (CalB) (Sigma-Aldrich) fue incluida como control positivo de la actividad. Las unidades enzimáticas se calcularon usando para ello una curva estándar de p-nitrofenol en un rango de concentración de 5 µM a 450 µM. Una unidad enzimática se definió como la cantidad de enzima necesaria para liberar 1µmol de sustrato por minuto bajo cada condición de ensayo (Zhu et al., 2013). Todos los ensayos fueron realizados por triplicado y los resultados se graficaron en unidades relativas tomando el valor del resultado máximo para cada ensayo como el 100%.
Especificidad de sustrato
La especificidad de sustrato de la enzima rLipM purificada se analizó utilizando ésteres de p-nitrofenil (p-NP-) con cadenas de ácidos grasos de diferentes longitudes: p-nitrofenil butirato (C4), octanoato, (C8) decanoato (C10), dodecanoato (C12), miristato (C14), palmitato (C16), y estearato (C18) previamente reconstituidos en acetonitrilo. Para los ensayos de actividad, 1 mM de cada uno de los sustratos en buffer Tris-HCl 50 mM pH 7.5, junto con 12.5 µL de enzima purificada, fueron incubados a 37 °C durante 30 minutos. Para los sustratos pNP- >C4 se adicionó 0.1 % (v/v) de Triton X-100 como agente emulsificante (Gawlicka et al., 2000).
Efecto de diferentes variables sobre la actividad esterasa
La temperatura óptima de reacción para rLipM se evaluó incubando la enzima a diferentes temperaturas en un rango de 4 °C a 65 °C por 30 min y usando solo p-nitrofenil butirato como sustrato.
El efecto del pH sobre la actividad esterasa de rLipM se evaluó midiendo la actividad de la enzima a 37 º C por 30 min, después de incubación con buffers a diferentes valores de pH: buffer fosfato (pH 7.0), buffer Tris-HCl (pH 8.0) buffer glicina-NaOH (pH 9.0), buffer bicarbonato (pH 10.0), buffer NaH2PO4-NaOH (pH 11.0) (Zhu et al., 2013).
Para evaluar el efecto de varios iones metálicos sobre la actividad esterasa de LipM, la enzima purificada se incubo con 1mM de KCl, LiCl, CaCl2, MgCl2, BaCl2, ZnCl, CuCl2, MnCl2, CoCl2 en buffer Tris-HCl pH 7.5 a 37 °C por 30 min.
Para evaluar los efectos de diferentes detergentes e inhibidores sobre la actividad esterasa de rLipM, la enzima purificada se incubó con los detergentes sodio dodecilsulfato (SDS), Tween 20 y Triton X-100. Por su parte, los inhibidores empleados fueron el fluoruro de fenilmetilsulfonilo (PMSF), ácido etilendiaminotetraacético (EDTA) ditiotreitol (DTT) y bromuro de cetiltrimetilamonio (CTAB). Tanto los detergentes como los inhibidores fueron evaluados a una concentración final de 0.1% y/o 1% (p/v) o (v/v) o 10 mM en 50 mM de tampón Tris-HCl (pH 7.5) a 37 º C por 30 min (Zhu et al., 2013). La actividad basal en cada caso se midió como en el ensayo estándar mencionado anteriormente, es decir, midiendo la actividad de degradación de cada uno de los buffers incluyendo el detergente o el inhibidor en ausencia de la enzima recombinante. Cada muestra se realizó por triplicado y el resultado graficado correspondió al promedio de los experimentos. En cada caso se incluyó la desviación estándar.
Resultados y Discusión
El análisis bioinformático sobre insertos de ADN metagenómico provenientes de un suelo dedicado al cultivo de papa criolla (Calderón et al., manuscrito en preparación), permitió realizar la identificación de una secuencia codificante de 591 pares de bases (pb) para una enzima lipasa putativa con dominio α/β hidrolasas (PFAM: PF07859.7). Esta enzima de 317 aminoácidos (aa), denominada LipM, mostró un peso teórico de 42 kDa y punto isoeléctrico (pI) de 5.8. El análisis de la secuencia de nucleótidos de este gen en la base de datos del NCBI (http://blast.ncbi.nlm.nih.gov), no evidenció identidad con ninguna secuencia reportada hasta ahora. Por su parte, el análisis por BLASTp evidenció que la proteína LipM presentó una identidad de 49% en su secuencia de aminoácidos con una proteína lipasa de Mycobacterium thermoresistibile ATCC 19527 (gbEHI10768.1) y de 47 % con la lipasa/esterasa de Mycobacterium abscessus (gbEIU36003.1). La secuencia GDSAGG en esta proteína corresponde al motivo conservado GXSXG de las α/β hidrolasas (Holmquist, 2000). Según la secuencia de este dominio conservado, se sugiere que esta enzima podría pertenecer al grupo V de la clasificación de lipasas/esterasas (Arpigny & Jaeger, 1999).
Expresión y purificación de LipM
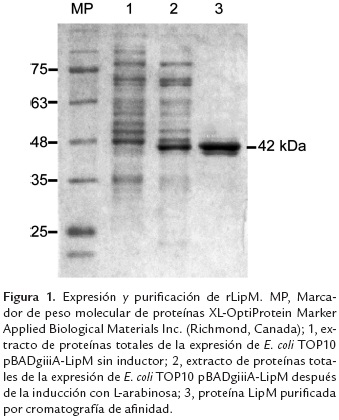
Con el fin de realizar la caracterización enzimática, la secuencia codificante de la enzima putativa LipM fue amplificada y subclonada en un vector de expresión. La expresión in vitro de la enzima rLipM en E. coli y su posterior purificación por cromatografía de afinidad permitió evidenciar la integridad de la enzima por SDS-PAGE (figura 1). Este análisis con el extracto total recombinante y la proteína rLipM purificada mostró una sola banda cerca de un tamaño aproximado de 48 kDa, la cual coincide con el tamaño esperado de la proteína incluyendo la cola de histidinas para su reconocimiento y purificación.
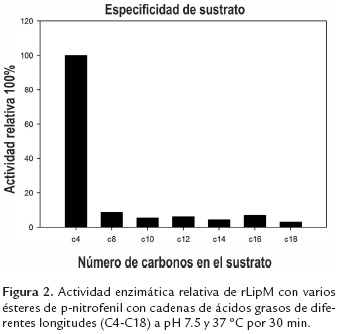
Determinación de la especificidad de sustrato
La determinación en la especificidad de sustrato de la enzima rLipM, fue evaluada empleando sustratos de p-nitrofenil con cadenas de ácidos grasos de diferente longitud. La enzima purificada mostró una alta afinidad por ácidos grasos de cadenas cortas (<C6) y menos afinidad por ácidos grasos de cadenas largas (>C8) (figura 2). La máxima actividad de rLipM, en este ensayo, fue observada para el sustrato p-NP-C4 seguido por p-NP-C8, a una temperatura de 37 °C y pH de 7.5. Estos resultados clasifican a LipM como una esterasa (E.C EC 3.1.1.x) más que una lipasa (E.c. EC 3.1.1.3). Esta especificidad de sustrato se ha observado también en otras esterasas derivadas de metagenomas ambientales (Zhang & Han, 2009; Zhu et al., 2013), así como también en esterasas aisladas de microorganismos, como es el caso de la enzima esterasa TLE del hongo Thermomyces lanuginosus DSM 10635 (Li et al., 2014) y recientemente la esterasa termoestable Est-gela de Bacillus gelatini KACC 12197 (Kim et al., 2015). La máxima actividad de la enzima con p-nitrofenil butirato (C4) se consideró como el 100 % de actividad relativa.
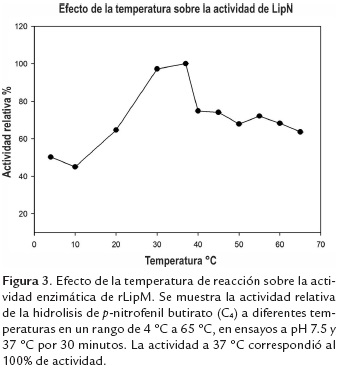
Efecto de la temperatura sobre la actividad enzimática
Adicionalmente, al determinar el efecto de la temperatura en la actividad enzimática de rLipM se observó que la temperatura óptima de actividad para esta enzima estuvo entre 30°C – 37°C (figura 3). También es importante resaltar que esta enzima conservó más de un 60% de su actividad en temperaturas que van entre 37°C y 60°C. Adicionalmente, a bajas temperaturas, en un rango de 4°C – 10ºC, rLipM siguió conservando valores significativos de actividad (40 – 50%), lo que en general la hace una enzima muy estable y por ende con mucho potencial industrial.
Estos resultados se relacionan con la amplia variabilidad en la actividad enzimática de lipasas/esterasas aisladas de metagenomas, con algunos ejemplos de enzimas termorresistentes (Chow et al., 2012; Fan et al., 2011; Yang et al., 2013) y algunos casos de enzimas que exhiben mayor actividad a temperatura ambiente o en el rango de 25 – 37ºC (Dong et al., 2015; Fang, Wang, Liu, & Jiao, 2015).
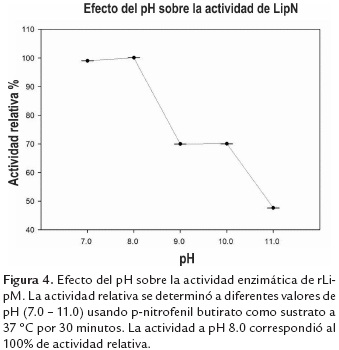
Efecto del pH sobre la actividad enzimática
Debido a que la actividad catalítica de muchas enzimas es sensible a cambios de pH, el efecto de esta variable sobre la actividad enzimática de rLipM fue evaluado (figura 4). No se probaron pH ácidos debido a que el p-nitrofenol no se puede cuantificar a pH bajos. Este análisis evidenció que el nivel máximo de actividad de rLipM se alcanzó a pH 8.0, es decir a niveles fisiológicos (pH 7.0 – 8.0). Otros estudios también demuestran que un número importante de lipasas/esterasas tienen mayor actividad a pHs cercanos al fisiológico (Li et al., 2014; Luo, Ding, Xu, Zheng & Zhong, 2015). Por el contrario, otro estudio de una enzima esterasa proveniente de un ambiente extremo en Colombia, tuvo máxima actividad a 55ºC y pH alcalino (9.0) (López et al., 2014).
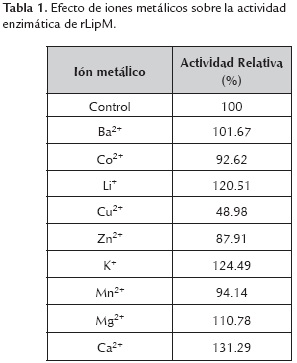
Efecto de cofactores sobre la actividad enzimática
Los cofactores inorgánicos, como es el caso de los iones metálicos, en muchos casos son esenciales para el adecuado funcionamiento de las enzimas. Por lo tanto, el sustrato enzimático con frecuencia tiene que estar unido a un activador que usualmente es un ión metal divalente (Eisenthal & Danson, 2002). La tabla 1 muestra el efecto de varios iones metálicos sobre la actividad de la enzima rLipM. La reacción se llevó a cabo por medio de la adición del sustrato y de determinados cofactores a concentración final de 1 mM, tomando la actividad enzimática sin cofactor como 100% de actividad relativa. De estos ensayos se observó que la actividad enzimática fue ligeramente aumentada por los iones K+ (124%), Li+ (120%), Mg2+ (110%) y en mayor medida por Ca2+ (131%) (tabla 1). El papel del Ca2+ como molécula de señalización celular y como activador de enzimas por medio de la estabilización ya ha sido reportada, más aún, su papel específico en enzimas lipolíticas ha sido reportado desde hace varias décadas (Gregory, Martin, Cheetham, & Rees, 1993); Kim et al., 1997; Posner & Morales, 1972; Simons et al., 1999). Sin embargo, cabe aclarar que la actividad enzimática de algunas lipasas y esterasas no se ve incrementada en presencia de iones Ca2+ (Kim et al., 2010; Zhu et al., 2013). Los iones Ba2+, Co2+, Zn2+ no tuvieron influencia sobre la actividad de rLipM. El ión Mn2+ en este caso tampoco modificó la actividad de la enzima, en contraste con lo que se ha reportado para otras esterasas (López et al., 2014). Por otro lado, la actividad enzimática fue inhibida ligeramente por Zn2+ (88%) y en mayor proporción por Cu+ (49%), siendo este último el ión que más efecto negativo tuvo sobre la actividad enzimática. El efecto negativo de este ión también ha sido reportado para otras esterasas obtenidas de diferentes muestras ambientales, como es el caso de unas enzimas provenientes de un metagenoma de lodos activados (Zhang & Han, 2009), la enzima EstEP16, derivada de un metagenoma de sedimento marino (Zhu et al., 2013) y la esterasa 499EST, proveniente de aguas termales colombianas (López et al., 2014).
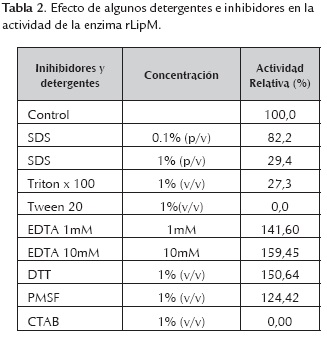
Efecto de detergentes e inhibidores sobre la actividad enzimática
El efecto de algunos inhibidores y detergentes en la actividad de la enzima LipMr se muestran en la tabla 2. En concordancia con varios artículos de caracterización de lipasas o esterasas, se encontró una inhibición completa con la presencia de Tween 20 y un efecto negativo por la adición de Triton X-100 (Dong et al., 2015; Rahman et al., 2016; Zhu et al., 2013). Sin embargo, a diferencia de otros estudios en presencia de SDS a una concentración de 0.1% (v/v) la actividad solo disminuyó en un 18% y adicionalmente a concentraciones de 1% (v/v) de SDS, la enzima retuvo una actividad residual mayor al 25%.
El inhibidor EDTA no influyó en la actividad de la enzima indicando que ésta no requiere de cationes divalentes para ser funcional.
En presencia del inhibidor PMSF la enzima conserva su actividad y se mantiene estable. Generalmente por poseer un sitio activo con serina se esperaría que hubiera una inhibición sin embargo, la enzima puede poseer una estructura de tapa que evite el efecto de inhibición de PMSF como en otros estudios (Chu, He, Guo, & Sun, 2008; Zhang & Han, 2009).
Conclusiones
En esta investigación reportamos la caracterización de la enzima LipM proveniente de un suelo de dedicación agrícola en Colombia. Esta enzima fue aislada a partir de una librería metagenómica y pertenece a la familia V de la clasificación de lipasas/esterasa. Presentó una mayor afinidad por sustratos de p-nitrofenil de ácidos grasos de cadena corta con un amplio rango de actividad a diferentes temperaturas y pH, teniendo mayor actividad a temperaturas entre 30 °C – 37 °C y en valores de pH cercanos al fisiológico (entre 7.0 y 8.0). Igualmente presentó buena estabilidad en presencia de varios iones metálicos y algunos inhibidores. Con base en los resultados de actividad reportados en esta investigación, consideramos que rLipM es una enzima con potencial biotecnológico e industrial, debido a que a 65 °C esta enzima conservó alrededor del 60% de su actividad, además de incrementar de forma significativa su actividad en presencia de CaCl2. Adicionalmente, rLipM sigue siendo parcialmente estable a concentraciones de 0.1-1% (p/v) de SDS. Este estudio también demostró que el enfoque metagenómico es muy útil para ampliar el conocimiento de la diversidad de enzimas, especialmente para esterasas bacterianas. El acceso a librerías metagenómicas ambientales es una fuente inmediata de nuevos biocatalizadores o enzimas con buen rendimiento que pueden, por un lado, ser aptas para optimizar los procesos de producción al introducir por ejemplo vectores de expresión citoplasmática o extracelular como el pBADgIII y/o cultivo en sistema de bioreactor y, por otro lado, pueden también ser modificadas por ingeniería genética para cumplir determinados propósitos de la industria. Este tipo de enzimas se podrían producir y comercializar en Colombia para procesos con mercado abierto, como el de transesterificación química de compuestos altamente contaminantes, producción de biodisel o como suplemento alimenticio de animales monogástricos.
Agradecimientos
Este trabajo fue financiado por COLCIENCIAS bajo la financiación No. 0142-2013. También agradecemos al Ministerio de Agricultura y Desarrollo Rural de Colombia (MADR) por apoyar el Consorcio de Investigación en Metagenómica Agrícola (CIMA).
Referencias bibliográficas
Andreote, F. D., Jiménez, D. J., Chaves, D., Dias, A. C. F., Luvizotto, D. M., Dini-Andreote, F., Fasanella, C. C., López, M. V., Baena, S., Taketani, R. G., & de Melo, I. S. (2012). The microbiome of Brazilian mangrove sediments as revealed by metagenomics. PloS One, 7(6), e38600. [ Links ]
Arpigny, J. L., & Jaeger, K. E. (1999). Bacterial lipolytic enzymes: classification and properties. The Biochemical Journal, 343(1), 177-183. [ Links ]
Calderon, D., Peña, L., Suarez, A., Villamil, C., Anzola, J., García-Betancur, J-C., Cepeda-Hernandez, M-L., Uribe-Vélez, D., Del Portillo, P., & Mongui-Cruz., A. (2016). A novel platform for the recovery of hidden soil enzymes in metagenomic clones. Manuscrito en preparación. Corporación CorpoGen, Bogotá-Colombia. [ Links ]
Chow, J., Kovacic, F., Dall, A. Y., Krauss, U., Fersini, F., Schmeisser, C., Lauinger, B., Bongen, P., Pietruszka, J., Schmidt, M., Menyes, I., Bornscheuer, U. T., Eckstein, M., Thum, O., Liese, A., Mueller-Dieckmann, J., Jaeger, K. E., & Streit, W. R. (2012). The metagenome-derived enzymes LipS and LipT increase the diversity of known lipases. PloS One, 7(10), e47665. [ Links ]
Chu, X., He, H., Guo, C., & Sun, B. (2008). Identification of two novel esterases from a marine metagenomic library derived from South China Sea. Applied Microbiology and Biotechnology, 80(4), 615-625. [ Links ]
Dong, J., Zhao, W., Gasmalla, M. A. A., Sun, J., Hua, X., Zhang, W., Han, L., Fan, Y., Feng, Y., Shen, Q., & Yang, R. (2015). A novel extracellular cold-active esterase of Pseudomonas sp. TB11 from glacier No.1: Differential induction, purification and characterisation. Journal of Molecular Catalysis B: Enzymatic, 121, 53-63. [ Links ]
Eisenthal, R., & Danson, M. J. (2002). Enzyme assays: A practical approach. Second Edition. New York: Oxford university Press. p 200-223. [ Links ]
Fan, X., Liu, X., Wang, K., Wang, S., Huang, R., & Liu, Y. (2011). Highly soluble expression and molecular characterization of an organic solvent-stable and thermotolerant lipase originating from the metagenome. Journal of Molecular Catalysis B: Enzymatic, 72(3-4), 319-326. [ Links ]
Fang, Y., Wang, S., Liu, S., & Jiao, Y. (2015). Discovery a novel organic solvent tolerant esterase from Salinispora arenicola CNP193 through genome mining. International Journal of Biological Macromolecules, 80, 334-340. [ Links ]
García, J., Santana, Z., Zumalacárregui, L., Quintana, M., González, D., Furrazola, G., & Cruz, O. (2013). Estrategias de obtención de proteínas recombinantes en Escherichia coli. VacciMonitor, 22(2), 30-39. [ Links ]
Gawlicka, A., Parent, B., Horn, M. H., Ross, N., Opstad, I., & Torrissen, O. J. (2000). Activity of digestive enzymes in yolk-sac larvae of Atlantic halibut (Hippoglossus hippoglossus): Indication of readiness for first feeding. Aquaculture, 184(3-4), 303-314. [ Links ]
Gregory, D. S., Martin, A. C., Cheetham, J. C., & Rees, A. R. (1993). The prediction and characterization of metal binding sites in proteins. Protein Engineering, 6, 29-35. [ Links ]
Hasan, F., Shah, A. A., & Hameed, A. (2006). Industrial applications of microbial lipases. Enzyme and Microbial Technology, 39(2), 235-251. [ Links ]
Holmquist, M. (2000). Alpha/Beta-hydrolase fold enzymes: structures, functions and mechanisms. Current Protein & Peptide Science, 1(2), 209-35. [ Links ]
Kim, J., Deng, L., Hong, E., & Ryu, Y. (2015). Cloning and characterization of a novel thermostable esterase from Bacillus gelatini KACC 12197. Protein Expression and Purification, 116, 90-97. [ Links ]
Kim, K. K., Song, H. K., Shin, D. H., Hwang, K. Y., & Suh, S. W. (1997). The crystal structure of a triacylglycerol lipase from Pseudomonas cepacia reveals a highly open conformation in the absence of a bound inhibitor. Structure, 5, 173-185. [ Links ]
Kim, Y. H., Kwon, E. J., Kim, S. K., Jeong, Y. S., Kim, J., Yun, H. D., & Kim, H. (2010). Molecular cloning and characterization of a novel family VIII alkaline esterase from a compost metagenomic library. Biochemical and Biophysical Research Communications, 393(1), 45-9. [ Links ]
Lara, A. (2011). Producción de proteínas recombinantes en Escherichia coli. Revista Mexicana de Ingeniería Química, 10(2), 209-223. [ Links ]
Lee, M. H., & Lee, S. W. (2013). Bioprospecting potential of the soil metagenome: novel enzymes and bioactivities. Genomics & Informatics, 11(3), 114-20. [ Links ]
Li, X-J., Zheng, R.-C., Wu, Z-M., Ding, X., & Zheng, Y-G. (2014). Thermophilic esterase from Thermomyces lanuginosus: molecular cloning, functional expression and biochemical characterization. Protein Expression and Purification, 101, 1-7. [ Links ]
López, G., Chow, J., Bongen, P., Lauinger, B., Pietruszka, J., Streit, W. R., & Baena, S. (2014). A novel thermoalkalostable esterase from Acidicaldus sp. strain USBA-GBX-499 with enantio selectivity isolated from an acidic hot springs of Colombian Andes. Applied Microbiology and Biotechnology, 98(20), 8603-16. [ Links ]
Luo, Z. H., Ding, J. F., Xu, W., Zheng, T. L., & Zhong, T. H. (2015). Purification and characterization of an intracellular esterase from a marine Fusarium fungal species showing phthalate diesterase activity. International Biodeterioration & Biodegradation, 97, 7-12. [ Links ]
Posner, I., & Morales, A. (1972). Mechanisms of enzyme and substrate activation by lipoprotein lipase cofactors. I. A specific requirement of physiological concentrations of calcium for enzyme activity. The Journal of Biological Chemistry, 247(8), 2255-65. [ Links ]
Rahman, M. A., Culsum, U., Tang, W., Zhang, S. W., Wu, G., & Liu, Z. (2016). Characterization of a novel cold active and salt tolerant esterase from Zunongwangia profunda. Enzyme and Microbial Technology, 85, 1-11. [ Links ]
Sharma, R., Chisti, Y., & Banerjee, U. C. (2001). Production, purification, characterization, and applications of lipases. Biotechnology Advances, 19(8), 627-662. [ Links ]
Simons, J. W. F. A., Van Kampen, M. D., Ubarretxena-Belandia, I., Cox, R. C., Alves Dos Santos, C. M., Egmond, M. R., & Verheij, H. M. (1999). Identification of a calcium binding site in Staphylococcus hyicus lipase: Generation of calcium-independent variants. Biochemistry, 38, 2-10. [ Links ]
Staley, J. T, & Konopka, A. (1985). Microorganisms in Aquatic and Terrestrial Habitats. Annual Review of Microbiology, 39, 321-346. [ Links ]
Torsvik, V., & Ovreas, L. (2002). Microbial diversity and function in soil: from genes to ecosystems. Current Opinion in Microbiology, 5(3), 240-245. [ Links ]
Uchiyama, T., & Miyazaki, K. (2009). Functional metagenomics for enzyme discovery: challenges to efficient screening. Current Opinion in Biotechnology, 20(6), 616-22. [ Links ]
Uwe, B., & Kazlauskas, J. (2006). Protein sources and optimization of biocatalyst performance. In Hydrolases in Organic Synthesis Regio and Stereoselective Biotransformations. Second Edition. Weinheim: Wiley-VCH Verlag GmbH & Co. p 200-215. [ Links ]
Yang, Z., Zhang, Y., Shen, T., Xie, Y., Mao, Y., & Ji, C. (2013). Cloning, expression and biochemical characterization of a novel, moderately thermostable GDSL family esterase from Geobacillus thermodenitrificans T2. Journal of Bioscience and Bioengineering, 115(2), 133-137. [ Links ]
Zhang, T., & Han, W.J. (2009). Gene cloning and characterization of a novel esterase from activated sludge metagenome. Microbial Cell Factories, 8, 67. [ Links ]





![First report of a begomovirus presents in yellow passionfruit [Passiflora edulis f. flavicarpa (Degene)] in Valle del Cauca, Colombia](/img/en/next.gif)