Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Colombiana de Biotecnología
versão impressa ISSN 0123-3475
Rev. colomb. biotecnol vol.18 no.2 Bogotá jul./dez. 2016
https://doi.org/10.15446/rev.colomb.biote.v18n2.61530
DOI: http://dx.doi.org/10.15446/rev.colomb.biote.v18n2.61530
ARTÍCULO DE REVISIÓN
Actualización en caracterización molecular de levaduras de interés industrial
Update on molecular characterization for yeast of industrial interest
Jorge Alberto Vásquez C*, Mauricio Ramírez Castrillón**, Zulma Isabel Monsalve F.***
* Grupo de Investigación e Innovación en Biotecnología (BITI); Centro de Biotecnología Industrial, SENA - Palmira, Valle del Cauca, Colombia. Correspodencia:
jorvasco1@misena.edu.co, jorvasco1@gmail.com
** Centro de Biotecnología, Universidade Federal do Rio Grande do Sul. Porto Alegre - Rio Grande do Sul. Brasil.
mauriciogeteg@gmail.com
*** Grupo de Investigación Agrobiotecnología, Facultad Ciencias Exactas y Naturales Universidad de Antioquia. Medellín. Colombia.
zmonsalve@gmail.com
Recibido: enero 23 de 2016 Aprobado: octubre 21 de 2016
Resumen
Las levaduras, además de ser un modelo de la investigación biomédica, tienen diversas aplicaciones en la industria alimentaria, en agricultura y la producción de etanol combustible. Dado que la calidad y la cantidad del producto dependen de la dinámica y la frecuencia de los microorganismos presentes en la fermentación, el uso de herramientas de caracterización molecular se ha incrementado y popularizado en las industrias que emplean levaduras. Estas técnicas se basan en la amplificación o análisis por enzimas de restricción de una porción del ADN genómico de levadura y se clasifican de acuerdo a su capacidad de resolución taxonómica para discriminar a nivel inter o intra-específica. La primera parte de la revisión incluye pruebas interespecíficas tales como, análisis de restricción o RFLP para las regiones ITS2, ITS1-5.8, D1 / D2 de los genes 26S ribosomal DNA. La segunda parte incluye, pruebas de uso común para caracterización nivel de cepa, tales como: la amplificación aleatoria del ADN polimórfico (RAPD), análisis cromosómico por electroforesis en gel de campo pulsado (PFGE), análisis de restricción del ADN mitocondrial (ADNmt- RFLP) análisis por mini / micro satélites y la huella genética de ADN por amplificación de regiones interdelta de los transposones Ty. Esta revisión describe y discute los detalles técnicos de los métodos más utilizados para la caracterización molecular de las levaduras y algunos ejemplos de sus aplicaciones en el contexto industrial.
Palabras clave: Levaduras, caracterización molecular, identificación intraespecífica especies, Saccharomyces cerevisiae.
Abstract
Yeasts, besides being a model of biomedical research, have various applications in the food industry, in agriculture and the production of ethanol fuel. Since the quality and quantity of the product depend on the dynamics and frequency of microorganisms present in the fermentation, the use of molecular characterization tools has been increased and popularized in the industries that use yeast. These techniques are based on amplification or analysis by restriction enzyme of a portion of yeast genomic DNA and are classified according their ability of taxonomic resolution to discriminate at inter- or intra-specific level. The first part of this review includes interspecific tests such as, restriction analysis or RFLP for the ITS2 regions, ITS1-5.8, D1 / D2 of ribosomal DNA 26S genes. The second part includes, tests commonly used for characterization at strain level, such as random amplified DNA polymorphism (RAPD), Chromosome analysis by pulsed gel field electrophoresis (PFGE), restriction analysis of mitochondrial DNA (ADNmt- RFLP), analysis of the mini / micro satellites and DNA fingerprinting by amplifying interdelta regions of Ty transposons. This review describes and discuses technical details of the most commonly used methods for molecular characterization of yeast and some examples of their applications in the industrial context.
Key words: Yeasts, molecular characterization, Saccharomyces cerevisiae.
Introducción
En la actualidad, el impacto del uso de las levaduras en la producción de alimentos y bebidas va mucho más allá de la noción popular del pan, la cerveza y la producción de vino usando Saccharomyces cerevisiae. En la industria de alimentos las levaduras se utilizan como fuente de obtención de vitaminas del complejo B, pigmentos, cofactores, extractos y producción de biomasa, entre otros. Algunas levaduras presentan un fuerte control antifúngico que facilita su uso como controladores del deterioro de alimentos. S. cerevisiae se ha venido usando ampliamente como suplemento alimenticio en vacunos, porcinos y aves, generando un incremento en el peso y talla de los ejemplares suplementados (Broadway et al., 2015). Entre sus aplicaciones, también se encuentra la producción de etanol para su uso como combustible, la cual se considera una industria emergente y quizás una de las de mayor proyección económica entre las que usan levaduras como catalizadores de este bioproceso (Tesfaw et al., 2014).
Para aprovechar de manera eficiente todas las propiedades de las levaduras a escala industrial se requiere identificar tanto sus características fisico-químicas, asi como la identidad taxonómica de la cepa. Actualmente, las principales técnicas de identificación de levaduras están basadas en métodos de biología molecular, aplican la PCR (Reacción en cadena de la polimerasa) la cual las hace más específicas, pueden ser extremadamente sensibles y los resultados se obtienen en corto tiempo. Esto, permite considerarlos como una alternativa a los métodos bioquímicos tradicionales, ya que se analiza una porción del genoma, el cual no depende del estado fisiológico de la levadura o del medio del cultivo del cual se aisle, reduciendo la aleatoriedad (Kopecka et al., 2013).
Dentro de las técnicas para identificación molecular se encuentran los análisis de las regiones ribosomales (5.8S, 18S y 26S), los espaciadores internos transcritos ITS y las regiones externas (ETS), la PCR en tiempo real, microchips, análisis de cromosomas por electroforesis en campo pulsado, análisis de restricción del ADN mitocondrial, los RAPD's (Amplificación aleatoria de ADN polimórfico), PCR de regiones repetitivas del genoma (microsatélites y minisatélites), la amplificación de secuencias δ y los AFLP(amplificación de fragmentos de longitud polimórfica), (Baselga et al., 2015).
En este análisis, se describe la pertinencia de cada una de estas técnicas dependiendo del contexto y del nivel de especificidad al cual se pretende llegar, lo que permite establecer diferencias a nivel inter e intraespecífico en la caracterización de levaduras con fines industriales.
Métodos para identificación de especies de levadura
La sistemática de un grupo taxonómico a cualquier nivel, idealmente debe ser monofilético (es decir, provenientes de un ancestro común) y cualquier sistema de identificación se basa en el rastreo de la historia filogenética y/o evolutiva. Esta historia es dificil de descubrir usando caracteres morfológicos, ya que ésta, al igual que la fisiología, evolucionan muchas veces en el tiempo de forma independiente en el mismo organismo. Por otra parte, los tratamientos realizados durante una clave taxonómica, en ocasiones, no permite distinguir fácilmente especies cercanas. En este sentido, la inferencia evolutiva de una especie usando herramientas moleculares permite soportar una mayor fidelidad de técnicas basadas en ADN para el rastreo y posicionamento filogenético de una especie de levadura (Kurtzman et al., 2015).
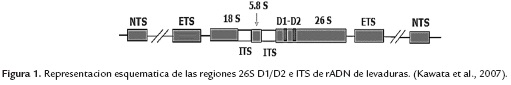
Los genes ribosomales que codifican las subunidades 5.8S, 18S, y 26S están dispuestos en tándem formando unidades de transcripción que se repiten en el genoma de las levaduras entre 100 y 200 veces (figura 1). En cada unidad de transcripción existen otras dos regiones, los espaciadores internos (ITS) y los externos (ETS), que se transcriben pero son procesadas y no forman parte de la molécula de rARN final. A su vez, las unidades codificantes están separadas por los espaciadores intergénicos IGS también llamados NTS. El gen 5.8S no se incluye en la unidad de transcripción previamente descrita pero aparece adyacente en la misma unidad de repetición en tandem en el caso de las levaduras (Alvares, 2011).
Entre ellas, dos regiones ribosomales son secuenciadas en levaduras: El dominio D1/D2 de la Subunidad Grande Ribosomal (LSU, proveniente de sus siglas en inglés) para identificación de levaduras ascomicetas (Kurtzman & Robnett, 2014) y la región que comprende los espaciadores transcritos internos y el gen ribosomal 5.8S (ITS1-5.8S rADN-ITS2) tanto para levaduras ascomicetas como basidiomicetas. La región ITS fue propuesta como "Barcode" para hongos y es aceptado para identificación de levaduras (Schoch et al., 2012).
Actualmente, el sistema de identificación de levaduras a nivel de especie que ofrece mayor fiabilidad es la secuenciación de la región LSU (Kurtzman & Robnett, 1998; Kurtzman et al., 2015). Cuando se encuentra una identidad igual o superior al 99% con secuencias depositadas en bases de datos como Genbank o Mycobank, una levadura es identificada a nivel taxonómico de especie (Belda et al., 2014; Kurtzman et al., 2015).
Para llevar a cabo la secuenciacion del dominio se han usando los cebadores: NL–1 (5'–GCA TAT CAA TAA GCG GAG GAA AAG–3') y NL–4 (5'–GGT CCG TGT TTC AAG ACG G–3') (Kurtzman & Robnett, 1997; Kopsahelis et al., 2009; Clavijo & Calderon, 2011). La disponibilidad en la base de datos Genbank (NCBI) de las secuencias de la mayoría de especies, descritas y por describir, hacen que esta técnica sea muy útil para asignar una levadura desconocida a una especie concreta. La comparación con las bases de datos se lleva a cabo usando el algoritmo MEGABLAST contrastando contra el material tipo de cada especie (Kurtzman et al., 2015).
En algunos clados específicos, es posible asociar inferencias evolutivas con potencial biotecnológico a nivel industrial. Por ejemplo, Kawata et al. (2007), utilizaron los cebadores NL1, NL4, ITS5, ITS4, ITS3 e ITS2 para dividir las levaduras industriales en clases (figura 1). Como resultado, encontraron polimorfismos en dos nucleótidos de la secuencia ITS2 y cuatro posiciones nucleotídicas en ITS 1, entre un grupo de 30 cepas de S. cerevisiae. Esto permitió la diferenciación de cepas de origen industrial en cuatro grupos: las cepas japonesas (sake yeast y shochu yeast), una cepa de panadería, un grupo de cepas vinícolas y un cuarto grupo de cepas wiskeras, cerveceras y de panadería, originarias también del Japón.
En algunos grupos taxonómicos la secuenciación de solo una región de ADN no permite la separación de especies diferentes, por lo que estrategias de análisis multigénicas son adoptadas, incluyendo regiones ribosomales y proteinas codificantes, tanto nucleares como mitocondriales. Varios ejemplos son reportados en la literatura, para diferenciar especies dentro de Candida, Kuraishia (Kurtzman & Robnett, 2014), Nakazawaea (Kaewwichian & Limtong, 2014), Cystobasidium (Yurkov et al., 2015), Kazachstania (James et al., 2015), entre otros géneros.
Métodos moleculares de caracterización intraespecífica
Si bien algunas similitudes fenotípicas son indicativas de que los aislados pertenecen a la misma especie, las técnicas moleculares proporcionan mayor confiabilidad en los resultados cuando se trata de caracterizar al nivel de cepa. Por ejemplo, mediante técnicas microbiologicas clásicas es imposible diferenciar cepas pertenecientes a la especie S. cerevisiae en el mosto de fermentación (Querol et al., 1993), sin embargo, usando análisis de secuencias de varias regiones mitocondriales y nucleares es posible distinguir poblaciones de diferentes cepas pertenencientes la especie S. cerevisiae (Badotti et al., 2014; Wolters et al., 2015).
A pesar que los RFLP's de la región ITS1-5.8S-ITS2 se utilizan para identificar levaduras, esta técnica se limita a un grupo reducido de levaduras asociadas a alimentos (Osorio-Cadavid et al., 2008; Lopez-Arboleda et al., 2010), por lo que requiere de la secuenciación para la confirmar la mayoría de agrupamientos realizados. Sin embargo, en algunos casos específicos -como en la confirmación de la presencia de S. cerevisiae-, los RFLP's son de gran utilidad (Tofalo et al., 2014). Esta técnica permite diferenciar cepas aisladas de sitios geográficamente distantes y cuyas aplicaciones industriales pueden diferir notablemente. La similitud de los patrones generados permite establecer correlaciones filogenéticas entre especies y cepas; así, la existencia de patrones únicos permite la diferenciación (Egli & Henick-Kling, 2001; Esteve-Zarzoso et al., 1999).
Algunas técnicas han sido desarrolladas para análisis intraespecífico y, por lo tanto, como agrupamiento o discriminación de cepas de levadura pertenecientes a una misma especie. Entre ellas se encuentran las técnicas denominadas "fingerprinting" basadas en regiones mini/microsatélite, sitios de "splicing" de intrones, RAPDs, AFLPs, RFLPs, regiones interdelta y SSCP (Polimorfismo de Conformación de Cadena Simple).
RAPD: Amplificación aleatoria de ADN polimórfico
Mediante la tecnica PCR se amplifica el ADN genomico en presencia de un iniciador (10 -14pb) de secuencia arbitraria, posteriormente se evalúan varios iniciadores de los cuales solo algunos presentarán un polimorfismo moderado. La temperatura de hibridacion entre el oligonucleótido y el ADN se facilita a 37°C, generando la amplificacion aleatoria de diversos fragmentos de ADN distribuidos a lo largo de todo el genoma, cuyos productos son visualizados mediante electroforesis usualmente de acrilamida.
El uso de RAPD's permite obtener las "huellas digitales" que son las diferencias de número y tamaño en los fragmentos del ADN amplificado o patrón de productos amplificados con diferentes perfiles moleculares, las cuales pueden ser características distintivas de especies e incluso de cepas (Capece, 2010).
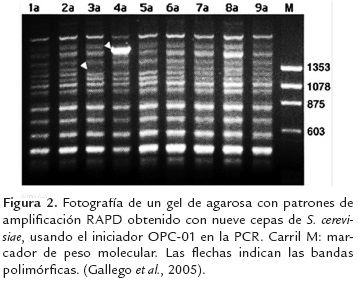
Los RAPD–PCR pueden usarse para aislar, seleccionar y caracterizar cepas de levaduras Saccharomyces nativas de fermentaciones espontáneas que no usan cepas comerciales, con el fin de seleccionar la levadura autóctona más adecuada para la vendimia. Los cebadores que se han propuestos para este fin son: OPA–1 (5'–CAG GCC CTT C–3'), OPA–10 (5'–GTG ATC GCA G–3'). La cepa S. cerevisiae J4 ha sido seleccionada por sus propiedades enologicas y de fermentacion (Rodriguez et al., 2013). La búsqueda de mejores cepas para enología, se puede complementar por medio de estudios filogenéticos representados con dendrogramas para determinar las distancias genéticas a partir de matrices proporcionadas por el algoritmo de Dice (1945). Este coeficiente se utiliza para agrupar datos según el método UPGMA, apoyándose en programas como NTSYSpc y TFPGA (Gallego et al., 2005), (figura 2).
El RAPD-PCR se ha usado también para el análisis de la variabilidad genética de los miembros del grupo Saccharomyces sensu stricto (S. bayanus, S. cerevisiae, S. paradoxus y S. pastorianus). La técnica ha permitido delimitar entre estas especies estrechamente relacionadas, así como grupos intraespecíficos como los dos existentes en la especie S. bayanus. Debido a que este marcador es de naturaleza multilocus permite determinar también el origen híbrido de la especies de levaduras tales como S. pastorianus y S, bayanus, a partir del análisis de la fracción de bandas mostradas por cada híbrido y sus cepas parentales (Nardi et al., 2006).
Esta técnica se caracteriza por no necesitar informacion sobre la secuencia del diseño del iniciador y a diferencia de otras técnicas moleculares empleadas, permite analizar la variabilidad a lo largo de todo el genoma revelando un mayor polimorfismo. Sin embargo, la baja temperatura de hibridación utilizada, genera perfiles de amplificación poco reproducibles siendo necesario llevar a cabo varias repeticiones para cada muestra partiendo de distintas extracciones de ADN. Para lograr un buen nivel de diferenciación polimórfica es necesario combinar los resultados de la amplificación con varios oligonucleótidos. A pesar de numerosas desventajas que la técnica presenta, es ampliamente reportada (de Almeida et al., 2015; Corte et al., 2015; Ozturk & Sagdic 2014; Wang et al., 2015).
Análisis cromosómico por Electroforesis en Campo Pulsado (PGFE)
El ADN genómico de levaduras, se digiere con una enzima de corte poco frecuente y se resuelve en un gel de agarosa en una cámara electroforética que emite pulsos eléctricos desde diferentes ángulos. La aplicación alterna de dos campos eléctricos transversos provoca que los cromosomas cambien continuamente su dirección de migración, evitando que queden retenidos en el entramado del gel de agarosa y permitiendo separar los fragmentos de ADN mayores a 225 Kb (Lee et al., 1989).
La existencia de reordenamientos cromosómicos (translocaciones, delecciones y amplificaciones de regiones cromosómicas) se basan en el alto nivel de polimorfismo cromosómico que se puede encontrar en las levaduras, característica que se aprovecha en el análisis por electroforesis en gel de campo pulsado (PFGE), el cual demuestra un alto polimorfismo de longitud entre los cromosomas pertenecientes a diferentes cepas de levadura. Tales variaciones en el tamaño de los cromosomas resultan de los reordenamientos de los cromosomas (GCR). De hecho, la caracterizacion de la estructura de los cromosomas de varias cepas del vino han revelado la existencia de translocaciones o delecciones, posiblemente por recombinación entre las secuencias repetidas Ty intercaladas por todo el genoma, lo cual se ha demostrado como una de las causas más importantes de la translocacion cromosomica (Blondin et al., 2009).
Se ha observado la aplicación de la técnica en diversos estudios, entre ellos, se resalta, el análisis sobre la diversidad genética de las poblaciones de S. cerevisiae en diez productores de cachaça en el sur Estado de Minas Gerais, Brasil. Un total de 106 aislamientos fueron identificados por PCR usando el cebador SCREC114, especifico para S. cerevisiae, mediante PFGE y por el análisis de ADN mitocondrial ADNmt–RFLP (Bernardi et al., 2008). Para asegurar la trazabilidad de los perfiles en los geles se usan stándares del cromosoma de S. cerevisiae ya disponibles comercialmente.
En otro estudio, Ferreira y colaboradores, (2009) evaluaron el potencial de gabiroba Campomanesia pubescens (DC) en la producción de una bebida fermentada usando levaduras seleccionadas y silvestres a partir de una fermentacion espontánea. La diversidad y dinámica de la población de levaduras durante la fermentacion fue observada por el análisis PFGE el cual mostro cinco cariotipos diferentes en los primeros días de la fermentacion. Después del séptimo día hubo una mayor frecuencia de un perfil similar al de S. cerevisiae.
La cariotipificación-PGFE, también se aplica en la identificación de cepas de interés en las fermentaciones vínicas (Puig et al., 2000). Existen varios trabajos centrados en la caracterización de levaduras mediante el análisis de polimorfismos. Esta técnica se puede utilizar para tipificar cepas de D. bruxellensis aisladas de vino (Mitrakul et al., 1999) y prevenir las grandes pérdidas económicas que deja el deterioro del vino por esta levadura. Por otra parte, en cepas de S. cerevisiae el polimorfismo obtenido es resultado de la adición o eliminación de largos fragmentos de ADN en cromosomas homólogos durante la evolución del genoma de las levaduras (Casaregola et al., 1998).
La cariotipificación de cromosomas de levaduras resulta muy útil para generar perfiles específicos para la caracterización de cepas, sin embargo los polimorfismos presentes entre diferentes especies, restringe su uso en el análisis de un gran número de levaduras contaminantes; además, la complejidad y larga duración del proceso, junto con el equipamiento especializado necesario, limitan aún más su aplicabilidad para la caracterización rutinaria en levaduras (El Hage & Houseley, 2013).
Análisis de restricción del ADN mitocondrial (RFLP - ADNmt)
El análisis de restricción del ADNmt se ha desarrollado en S. cerevisiae basado en las diferencias en el contenido de G+C existentes entre el ADN nuclear (nADN) y el ADNmt, ya que que en el nADN este es de un 40%, mientras que en el mtADN es solo de un 20%. Esto permite que si al realizar un análisis de restricción al ADN total, utilizando enzimas específicas para reconocer regiones ricas en G+C, tales como SacII y SstII cuya secuencia blanco es CCGCGG, el nADN sufre una sobredigestión generándose numerosos fragmentos de muy pequeño tamaño, los cuales son indetectables en un gel de agarosa. De esta manera, en una electroforesis de ADN total digerido con estas endonucleasas solo se observan los fragmentos correspondientes al ADNmt ubicados según su tamaño y mostrando un perfil específico de especie. Varios autores han introducido una variante que reduce el tiempo de duración del método original en 52,5 h, por disminución del tiempo de incubación para la digestión del ADN de 12 h a 33 min en un horno de microondas, y del crecimiento celular a 36 h (Orberá, 2004).
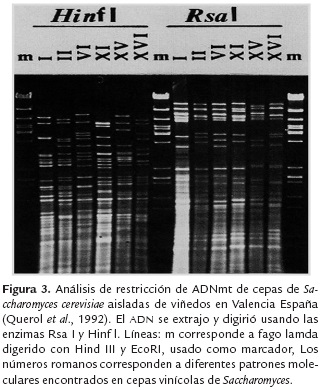
El análisis de polimorfismos de los fragmentos de restricción del ADN mitocondrial, ha sido ampliamente utilizado como método de caracterización de cepas de Saccharomyces estrechamente relacionadas de origen vínico y cervecero (Vezinhet et al., 1990; Muñoz et al., 2009). Investigadores de la Universidad de Valencia, España, demostraron la utilidad de la técnica para determinar la autenticidad de las cepas comerciales que se usan en los viñedos (Esteve-Zarsoso et al., 2001; Fernandez-Espinar et al., 2003), así como también para realizar el seguimiento a las fermentaciones y diferenciar las cepas comerciales de la flora nativa contaminante (figura 3).
Secuencias de mini/microsatélites como cebadores (MSP-PCR Fingerprinting)
Esta técnica se basa en el uso de la Reacción en Cadena de la Polimerasa (PCR) y consiste en la amplificación de fragmentos con cebadores de oligonucleótidos específicos para secuencias simples repetitivas que varían entre 10 y 100 pb presentes en el ADN, denominadas microsatélites, mientras que los minisatélites tienen una longitud inferior a 10 pb. Entre los más utilizados se encuentran (GTG)5, (GACA)4, Fago ADN M13 y la secuencia del M13 GAGGGTGGCGGTTCT. Esta técnica difiere del RAPD, en que la temperatura de hibridación del cebador es mayor (entre 50 y 55 °C), en lugar de de 37 °C, por lo que la probabilidad de hibridizar en zonas específicas del genoma aumenta, logrando una mayor reproducibilidad. Se ha empleado en la identificación de especies del género Zygosaccharomyces, contaminantes de salsas de mayonesa y otros aderezos para ensaladas (Orberá, 2004).
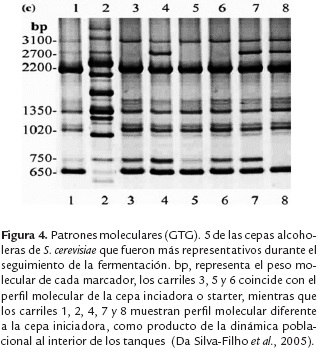
Da Silva-Filho et al., (2005) usaron el fingerprinting con el microsatélite (GTG)5, para hacer el seguimiento de los patrones moleculares de la microbiota presente en una misma fermentación en una destilería para la producción de etanol carburante en el nordeste de Brasil, encontrándo diferentes patrones moleculares que coincidieron con cambios en la población de levaduras en el mosto generadas por colonización con cepas nativas provenientes de la melaza o jugo de caña de azúcar, o tambien por las por condiciones de estrés del sustrato que favorecen el crecimiento de alguna cepa tolerante a estos cambios (figura 4).
Ramírez-Castrillón et al. (2014), advierten sobre una posible subestimación en análisis de diversidad obtenidos con perfiles genotípicos MSP-PCR Fingerprinting, debido a la gran cantidad de falsos positivos que pueden encontrarse usando esta técnica. Así, analizando dos conjuntos de datos de levaduras asociadas a vinos (considerada de baja diversidad) y sustratos asociados a viñedos (considerado de alta diversidad), pudo establecerse que los agrupamientos realizados con los perfiles, al ser secuenciados, representaban aislados de especies diferentes (por ejemplo, Dekkera bruxellensis y S. cerevisiae). Sin embargo, esta técnica es usada con frecuencia para descripción de especies nuevas (Valente et al., 2012; Praet et al., 2015) y caracterización intraespecífica tales como Torulaspora delbrueckii (Canonico et al., 2015) y Candida milleri (Vigentini et al., 2014).
AFLPs (Amplified Fragment Length Polymorphism)
Al igual que en los RAPDs esta técnica, no requieren información previa sobre la secuencia para el diseño del iniciador y aporta un gran número de bandas para el análisis, pero es mas reproducible que este último. Posterior a la restriccion del ADN genómico, se realiza la ligación de adaptadores oligonucleótidos, para la amplificación no selectiva de los fragmentos restringidos con iniciadores complementarios al adaptador. Los fragmentos obtenidos en la primera reacción de PCR, se reamplifican de manera selectiva usando entre una y tres bases diferenciales en el extremo 3', finalmente, la separación de los amplicones se realiza en geles de poliacrilamida. Típicamente se detectan entre 50 y 100 fragmentos de restricción por genoma de levadura en un gel desnaturalizante de poliacrilamida.
Los AFLPs son una técnica útil para discriminar levaduras a nivel cepa-específico (de Barros et al., 1999), el analisis molecular en levaduras mediante esta tecnica puede ser superior a los RAPDs. En un estudio comparativo, Gallego et al., (2005) evaluaron la utilidad de tres técnicas moleculares para el analisis genético de las cepas de S. cerevisiae: los RAPDs, AFLPs y los SSRs (microsatelites). Estas tres técnicas moleculares fueron usadas para caracterizar 27 cepas de levaduras vinicas de la "Denominacion de Origen Vinos de Madrid" España. Basados en la amplificacion de los fragmentos de cada genotipo, los AFLPs y los SSRs mostraron un poder discriminatorio similar y superior al de los RAPDs. El análisis con AFLPs y SSR permitió discriminar una combinacion total de 23 cepas de 27 evaluadas.
Esta técnica es ampliamente usada, por su poder de discernimiento, se usa como confirmatoria para caracterizar aislamientos de levaduras salvajes procedentes de los tanques de almacenamiento de melaza de caña de azúcar. Vásquez et al. (2015), realizaron la caracterización intraespecífica de cepas tolerantes a vinazas usando marcadores interdelta 12-21 y confirmando con AFLP se obtuvo cinco grupos con distancias genéticas que oscilaron entre 15 y 30%, además por pruebas bioquímicas se determinó que el 85% de los aislamientos pertencian al género S. cerevisiae.
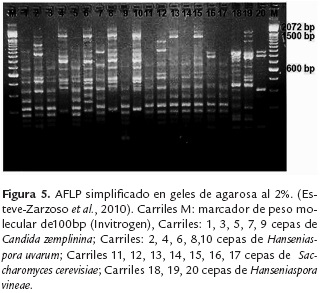
El uso de los AFLP's no se ha popularizado en los laboratorios microbiológicos de la industria debido al tiempo necesario para desarrollar la técnica, su alto nivel de complejidad, por la gran cantidad de reactivos y el tamaño de los geles de acrilamida. Sin embargo, ya se han propuesto modificaciones que podrían simplificar y permitir el ahorro en reactivos como revelar los AFLPs en gel de agarosa al 2%, en buffer tris borato EDTA al 1%. Este ejercicio facilitó la caracterización de la biota de levaduras, aisladas durante el seguimiento de una fermentación espontánea de mosto de uva, en la región de Taragona España (figura 5) (Esteve-Zarzoso et al., 2010).
Elementos delta del transposon Ty 1 de levaduras
Las secuencias delta son elementos de 330pb que flanquean los retrotransposones Ty1 y Ty2 de S. cerevisiae (Cameron et al.,1979). Estas regiones se pueden encontrar separadas de los transposones y dispersos por el genoma, en cuyo caso suelen llamarse solo elementos delta. Su número y posición en el genoma de las levaduras son específicos de cada cepa y estables aproximadamente durante 50 generaciones. En el genoma están presentes cerca de 5 familias de elementos Ty que contienen 50 sitios activos potenciales para traslocación del retrotransposon completo y también existen aproximadamente 300 elementos delta independientes como marcas de eventos insersionales pasados. Se ha reportado que los elementos delta están concentrados en regiones genómicas contiguas a los genes ARNt (Eigel & Feldmann, 1982).
La variabilidad intraespecífica que otorga el número y la localización de estos elementos fue aprovechada por Ness et al., (1993) para diseñar iniciadores específicos (δ1 y δ2) para la amplificación por PCR de las regiones delta, útiles para diferenciar cepas de S. cerevisiae. Estos autores muestran que la estabilidad de los elementos δ es suficiente para aplicar esta técnica como método de identificación de cepas de S. cerevisiae a nivel industrial, afirmaciones que fueron confirmadas posteriormente por numerosos autores. Algunos de estos trabajos muestran la gran variabilidad que revela esta técnica entre aislados de la especie S. cerevisiae con respecto a otras técnicas muy resolutivas como el análisis de restricción del ADNmt y la electroforesis de cromosomas (Fernández-Espinar et al., 2001; Pramateftaki et al., 2000).
En el 2003, Legras & Karst optimizaron la técnica mediante el diseño de dos iniciadores (δ12 y δ21) que se localizan muy próximos a δ1 y δ2. Su uso combinado revela un mayor polimorfismo que se refleja con la aparición de un mayor número de bandas. Schuller et al., (2004) lo confirmaron posteriormente mostrando que la combinación de δ2 y δ12 identificaba el doble de cepas que el juego de iniciadores diseñado por Ness et al., (1993).
Varios inconvenientes que presentaba esta técnica eran la poca reproducibilidad debido al efecto de la concentración de ADN y a la baja temperatura (42°C) para la hibridación de los primers (Fernández-Espinar et al., 2001; Schuller et al., 2004), estos problemas fueron superados con la estandarización de la concentración de ADN para todas las muestras, y el aumento de la temperatura de hibridación (55°C), logrando obtener perfiles de amplificación mucho más estables aunque con un menor número de bandas (Ciani et al., 2010).
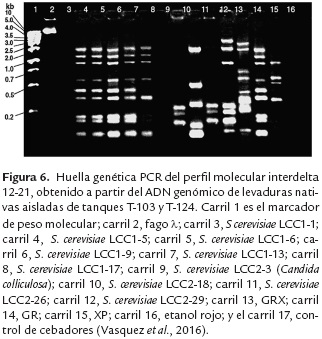
En las aplicaciones más recientes, se usó la huella interdelta, para comparar los perfiles moleculares de varias cepas nativas alcoholeras aisladas del ambiente de dos destilerías del Valle de Cauca en Colombia, logrando determinar que un 25% de las cepas aisladas de los tanques de almacenamiento de mieles, presentan un patrón molecular diferente al de las cepas comerciales usadas convencionalmente en la fermentación. Se observa que la huella genética interdelta presenta un alto polimorfismo entre cepas diferentes, dado su practicidad y bajo costo, es útil para la caracterización intraespecífica (figura 6), (Vasquez et al., 2015).
Discusion
Algunas técnicas moleculares han sido propuestas para la caracterización de especies y monitoreo de cepas industriales. Las primeras de ellas incluían el análisis electroforético del cariotipo, la amplificacion aleatoria del ADN polimórfico usando la reaccion en cadena de la polimerasa (RAPD-PCR) y la ribotificacion-PCR, las cuales han sido empleadas por diferentes autores para la identificacion de especies de levaduras del vino (Raspor et al., 2006). A partir del uso masivo de la reacción en cadena de la polimerasa para evaluar el polimorfismo en la longitud de los fragmentos de restricción (PCR-RFLP) y la secuenciación de ADN, se establece el consenso de dos regiones de ADN como estándar para la determinación de especies: LSU y la región ITS1-rADN 5.8S-ITS2 (Kurtzman et al., 2015). Por ejemplo, el uso de secuencias de ADN para identificar levaduras permitió duplicar el número de especies identificadas en los últimos diez años (Kurtzman et al., 2011).
La mayor base de datos de secuencias depositada en Genbank para identificación rápida de levaduras, o detección de linajes desconocidos, es la región LSU, seguido de la región ITS. Los resultados de Kawata et al., (2007) sugieren a los ITS como posibles marcadores polimórficos para el estudio de la diversidad interespecífica de levaduras. Cuando se pretende comparar de forma rápida la secuencia (editada, ensamblada) con cepas tipo depositadas en Genbank de identidad igual o superior a 99% con una especie descrita, esta técnica permite la identificación de una cepa a nivel taxonómico de especie. Si no es posible realizarse con LSU, se procede a comparar con la región ITS de la misma forma. Nótese que pueden haber excepciones al criterio de identidad dependiendo del clado filogenético. No obstante, la región ITS fue propuesta como código de barras (Barcode, en inglés) para identificación de hongos (Schoch et al., 2012), debido a que la región LSU no permite la identificación en algunos grupos de hongos no levaduriformes (para definición de levadura reciente ver Kurtzman et al., 2011).
A nivel industrial el monitoreo e identificación sirven para demostrar que la especie de levadura inoculada al inicio y al final de un proceso biotecnológico es el mismo y que la especie que domine una fermentación, por ejemplo, sea la esperada por el productor. También, es posible evaluar cambios adaptativos de una cepa industrial, al demostrar que cepas originales y "adaptadas" proceden de un ancestro común y que han "evolucionado" con su uso en cada proceso industrial (Dragosits & Mattanovich, 2013). Un reflejo de esto son los estudios fisiológicos (Oda & Ouchi, 1989) que demuestran que las cepas de S. cerevisiae cerveceras pueden presentar deficiencias en el levante o leavening, característica fundamental de las cepas de S. cerevisiae, de uso en panadería y que demuestran el grado evolutivo y posiblemente una mejor adaptación a un ambiente en particular.
A nivel intraespecífico, varias técnicas también fueron desarrolladas (sin involucrar secuenciación). Entre ellas, sondas de APN – Péptido Ácido Nucleico- (Stender et al., 2001), RAPD/AFLP (Azumi et al., 2001; de Barros Lopes et al., 1999), Microsatelite-primed PCR (Da Silva-Filho et al., 2005; Ramirez-Castrillón et al., 2014), PCR en tiempo real (Casey & Dobson 2004), electroforesis en Gel de Gradiente desnaturalizante –DGGE- (Prakitchaiwattana et al., 2004), Citometria de flujo (Jespersen et al., 1993). Su aplicación y sus campos de acción va desde el monitoreo de poblaciones de levaduras industriales hasta el análisis de diversidad de levaduras asociadas a bebidas y alimentos.
La baja reproducibilidad de los RAPDs implica la necesidad de hacer muchas repeticiones para obtener el patrón estándar y después incluir en el análisis solo la banda que aparece en todas las amplificaciones, esto la hace poco útil para uso rutinario. Adicionalmente, debido a la baja temperatura de hibridación utilizada (37°C) los perfiles de amplificación incluyen productos inespecíficos siendo necesario llevar a cabo varias repeticiones para cada muestra partiendo de distintas extracciones de ADN. Los microsatélites difieren del RAPD en que la temperatura de hibridación del iniciador es mayor (55 °C), en lugar de la de 37 °C, por lo que estos hibridan en zonas específicas del genoma, haciendo mayor su reproducibilidad, por lo cual esta técnica se ha convertido una de las más usadas para la caracterización intraespecífica de levaduras.
Ademas, la PCR-fingerprinting empleando cebadores individuales (SPAR), cebadores aleatorios y secuencias microsatelites puede usarse para la discriminacion inter y/o intra-especifica de levaduras. La PCR-fingerprinting se basa en la deteccion de secuencias repetitivas hipervariables (mini y microsatelites) favorecida para mostrar la variabilidad genética de los individuos estrechamente relacionados (Suranska et al., 2012).
Por otra parte, la técnica de electroforesis en gel de campo pulsado (PFGE), es una de las que ofrecen un mayor poder discriminatorio. Sin embargo, la complejidad y larga duración del proceso, junto con el equipamiento especializado necesario, limitan su aplicabilidad para la caracterización rutinaria de las levaduras (Rodriguez et al., 2010; Cordero-Bueso, 2016).
La huella genética por regiones delta, es una técnica rápida que puede distinguir dos levaduras incluso a nivel intraespecífico y resulta ideal para la utilización en la industria, por su simplicidad, sin embargo se requiere tener estandarizado el método de extracción y cuantificación del ADN genómico, así como también a la disposición equipos como el termociclador.
El uso del perfil de restricción del ADN mitocondrial resulta también ideal para la utilización en la industria por su rapidez, seguridad y economía, ya que no requiere material sofisticado, ni personal muy especializado, además es eficiente para diferenciar cepas de levaduras, ya que se basa en la existencia de un importante polimorfismo del ADN mitocondrial en las distintas cepas (Benitez et al., 1996), permitiendo seguir tanto la implantación de la cepa inoculada como la dinámica de crecimiento del resto de cepas presentes en el mosto (Querol et al., 1993).
Recientemente, la identificación basada en MALDI-TOF/MS ha revolucionado la identificación microbiana, incluyendo levaduras (Quian et al., 2008), acelerando el proceso de identificación de días (como el caso de la secuenciación) a horas (generalmente un turno) (Borneman et al., 2011). Actualmente es ampliamente usado en levaduras de interés clínico, con la expectativa de ser usado prontamente a nivel industrial, donde se requiere una base de datos creciente junto con el mejoramiento de la técnica (Kurtzman et al., 2015).
En el momento de la caracterización intraespecífica con técnicas moleculares, es importante realizar al menos dos técnicas diferentes para reducir las variables que impiden la reproducibilidad entre laboratorios e incluso entre ensayos.
En términos generales, las técnicas moleculares, se constituyen en una herramienta eficiente para la caracterización de microorganismos, siendo esta, una etapa indispensable para determinar la identidad genética de las cepas, en especial cuando se planea un uso industrial de los aislamientos. Las técnicas de caracterización morfológica y bioquímicas han predominado hasta ahora, por su relativa sencillez y porque en términos generales no requieren equipos sofisticados para llevarlas a cabo, no obstante es indispensable identificar la identidad genética del microorganismo en los casos en que se requiera determinar la inocuidad del cultivo específico para las cepas comerciales, o para realizar el seguimiento de la inocuidad de la fermentación y especialmente proteger la propiedad industrial.
Referencias bibliográficas
Álvarez, J. (2011). Aislamiento y caracterización genética y enológica de levaduras vínicas autóctonas de uva Prieto Picudo y caracterización aromática de sus vinos (DO «Tierra de León»). (Tesis doctoral). Recuperado de: http://buleria.unileon.es/xmlui/handle/10612/1882. [ Links ]
Asaregola, S., Nguyen, H. V., Lepingle, A., Brignon, P., Gnedre, F., & Gaillardin, C. (1998). A family of laboratory strains of Saccharomyces cerevisiae carry rearrangements involving chromosomes I and III. Yeast, 14, 551-561. [ Links ]
Azumi, M., & Goto-Yamamoto, N. (2001). AFLP analysis of type strains and laboratory and industrial strains of Saccharomyces sensu stricto and its application to phenetic clustering. Yeast, 18, (12), 1145-1154. [ Links ]
Badotti, F., Vilaça, S. T., Arias, A., Rosa, C. A., & Barrio, E. (2013). Two interbreeding populations of Saccharomyces cerevisiae strains coexist in cachaça fermentations from Brazil. FEMS Yeast Research, 14 (2), 289-301. DOI: 10.1111/1567-1364.12108. [ Links ]
Baselga, I., Zafra, O., Pérez L. E., Francisco-Álvarez, R., Rodriguez-Tarduchy, G., & Santos, C. (2016). An AFLP based method for the detection and identification of indigenous yeast in complex must samples without a microbiological culture. International Journal of Food Microbiology, doi:10.1016/j.ijfoodmicro.2016.09.014. [ Links ]
Belda, I., Navascues, E., Alonso, A., Marquina, D., & Santos, A. (2014). Microbiología del proceso de vinificación: selección de levaduras Saccharomyces cerevisiae autóctonas con óptimas propiedades enológicas. Reduca (Biologia). Serie Microbiologia,7(1), 1-14. [ Links ]
Benitez, T., Martinez, P., & Codon, C. (1996). Genetic constitution of industrial yeast. Microbiología SEM, 12, 371-384. [ Links ]
Bernardi, T.L., de Melo Pereira, V., Gomes, P. C., Souza, E. D., & Schwan, R. F. (2008). Saccharomyces cerevisiae strains associated with the production of cachaça: identification and characterization by traditional and molecular methods (PCR, PFGE and mtDNA-RFLP). World Journal Microbiology Biotechnology, 24, 2705-2712. [ Links ]
Blondin, B., Dequin, S., Querol, A., & Legras, J. L. (2009). Genome of Saccharomyces cerevisiae and Related Yeasts. Biology of Microorganisms on Grapes, in Must and in Wine. König (ed). 20, 361-376. [ Links ]
Borneman, A. R., Desany, B. A., Riches, D., Affourtit, J. P., Forgan, A. H., Pretorius, I.S., Egholm, M.l, & Chambers, P. J. (2011). Whole-Genome Comparison Reveals Novel Genetic Elements That Characterize the Genome of Industrial Strains of Saccharomyces cerevisiae. PLoS Genetics, 7 (2): e1001287. doi:10.1371/journal.pgen.1001287. [ Links ]
Broadway, P. R., Carroll, J. A., & Burdick, N. C. (2015). Live Yeast and Yeast Cell Wall Supplements Enhance Immune Function and Performance in Food-Producing Livestock: A Review. Microorganisms, 3, 417-427. doi:10.3390/microorganisms3030417. [ Links ]
Cameron, J. R., Loh, E., & Davis, R. W. (1979). Evidence for transposition of dispersed repetitive DNA families in yeast. Cell, 16, 739-751. [ Links ]
Canonico, L., Comitini, F., & Ciani, M. (2015). TdPIR minisatellite fingerprinting s a useful new tool for Torulaspora delbrueckii molecular typing. International Journal of Food Microbiology, 4, (200), 47-51. doi: 10.1016 /j.ijfoodmicro. 2015.01.020. [ Links ]
Capece, A., Romaniello, R., Siesto, G., Pietrafesa, R., Massari, C., Poeta, C., & Romano P. (2010). Selection of indigenous Saccharomyces cerevisiae strains for Nero d'Avola wine and evaluation of selected starter implantation in pilot fermentation. International Journal of Food Microbiology, 144, 187-192. [ Links ]
Casey, G. D., &. Dobson, A. D. (2004). Potential of using real-time PCR-based detection of spoilage yeast in fruit juice a preliminary study. International Journal of Food Microbiology, 91, 327-335. [ Links ]
Ciani, M., Comitini, F., Mannazzu, I., & Domizio, P.(2010). Controlled mixed culture fermentation: a new perspective on the use of non-Saccharomyces yeasts in winemaking. FEMS Yeast Research, 10, (2), 123-33. [ Links ]
Clavijo, A., & Calderon, I. L. (2011). Yeast assessment during alcoholic fermentation inoculated with a natural ''pied de cuve'' or a commercial yeast strain. World Journal Microbiology Biotechnology, 27, 1569-1577. [ Links ]
Cordero-Bueso, G., Esteve-Zarzoso, B., Gil-Díaz, M., García, M., Cabellos, J. M., & Arroyo T. (2016) Improvement of Malvar Wine Quality by Use of Locally-Selected Saccharomyces cerevisiae Strains. Fermentation, 2, 7, doi:10.3390/fermentation2010007www.mdpi.com/journal/fermentation. [ Links ]
Corte, L., di Cagno, R., Groenewald, M., Roscini, L., Colabella, C., Gobbetti, M., & Cardinali, G. (2015). Phenotypic and molecular diversity of Meyerozyma guilliermondii strains isolated from food and other environmental niches, hints for an incipient speciation. Food Microbiology, 48, 206-15. DOI: 10.1016/j.fm.2014.12.014. [ Links ]
Da Silva-Filho, E. A., Brito Dos Santos, S. K., Do Monte Resende, A., Falca˜o de Morais, J. O., de Morais Jr .M. A., & Simo˜es D. A. (2005). Yeast population dynamics of industrial fuel-ethanol fermentation process assessed by PCR-fingerprinting. Antonie van Leeuwenhoek, 88,13-23. [ Links ]
de Almeida, A. A., Nakamura, S. S., Fiorini, A., Grisolia, A. B., Svidzinski, T. I. E., & de Oliveira, K. M. P. (2015). Genotypic variability and antifungal susceptibility of Candida tropicalis isolated from patients with candiduria. Revista Iberoamericana de Micología, 32, (3), 153-158. [ Links ]
de Barros Lopez M., Rainiere S., Henschje P. A., & Langridge, P. (1999). AFLP fingerprinting for analysis of yeast genetic variation. International Journal Systematic Bacteriology, 49, 915-924. [ Links ]
Diaz, C., Molina, A.M., Nâhring, J., & Fischer, R. (2013). Characterization and dynamic behavior of wild yeast during spontaneous Wine Fermentation in steel tanks and amphorae. BioMed Research International, 1- 13. Article ID 540465, doi:10.1155/2013/540465. [ Links ]
Dice, L. R. (1945). Measures of theamountof ecologicassociation between species. Ecology, 26, 297-302. [ Links ]
Dragosits, M., & Mattanovich, D. (2013). Adaptive laboratory evolution - principles and applications for biotechnology. Microbial Cell Factories, 12 (64), 6-17. doi: 10.1186/1475-2859-12-64. [ Links ]
Egli, C. M., & Henick-Kling, T. (2001). Identification of Brettanomyces/Dekkera species based on polymorphism in the rRNA internal transcribed spacer region. American Journal of Enology and Viticulture, 52 (3), 241-247. [ Links ]
Eigel, A., & Feldmann, H. (1982). Ty1 and delta elements occur adjacent to several tRNA genes in yeast. EMBO Journal, 1, 1245-1250. [ Links ]
El Hage, A., & Houseley, J. (2013). Resolution of budding yeast chromosomes using pulsed-field gel electrophoresis. Methods in Molecular Biology, 1054, 195-207. [ Links ]
Esteve-Zarzoso, B., Bellochl, C.F., & QueroI, A. (1999). Identification of yeasts by RFLP analysis of the 5.85 rRNA gene and the two ribosomal internal transcribed spacers. International Journal of Systematic Bacteriology, 49, 329-337. [ Links ]
Esteve-Zarzoso, B., Peris, M. J., Maique, E. G., Uruburu, F., & Querol A. (2001). Yeast population dynamics during the fermentation and biological aging of sherry wines. Applied and Environmental Microbiology, 67 (5), 2056-2061. [ Links ]
Esteve-Zarzoso, B., Hierro, N., Mas, A., & Guillamón, J.. (2010). A new simplified AFLP method for wine yeast strain typing. LWT. Food Science and Technology, doi:10.1016/j.lwt..05.016. [ Links ]
Fernández-Espinar, M. T., López, V., Ramón, D., Bartra, E., & Querol, A. (2001). Study of the authenticity of commercial wine yeast strains by molecular techniques. International Journal of Food Microbioly, 70, 1-10. [ Links ]
Fernández-Espinar, M. T., Barrio, E., & Querol A. (2003). Genetic variability among species of the Saccharomyces sensu stricto. Yeast. 20, 1213-1226. [ Links ]
Ferreira, D. W., Ribeiro, D. D., de Melo Pereira, V. G., Gervasio, I.M., & Schwan, I. M. (2009). Indigenous and inoculated yeast fermentation of gabiroba (Campomanesia pubescens) pulp for fruit wine production. Journal of Industrial Microbiology & Biotechnology, 36 (4), 557-569. [ Links ]
Gallego, F.J., Peres, M. A., Nuñes, Y., & Hidalgo P. (2005). Comparison of RAPDs,AFLPs and SSR markers for the genetic analysis of yeast strains of Saccharomyces cerevisiae. Food Microbiology, 22, 561-568. [ Links ]
James, S., Carvajal Barriga, E., Portero Barahona, P., Nueno-palop, C., Cross, K., Bond, C., & Roberts I. (2015). Kazachstania yasuniensis sp. nov., a novel ascomycetous, yeast species found in mailand Ecuador and on the Galápagos. International Journal of Systematic and Evolutionary Microbiology, 65, (4), 1304-1309. doi:10.1099/ijs.0.000102. [ Links ]
Jespersen, L., Lassen, S., & Jakobsen, M. (1993). Flow cytometric detection of wild yeast in lager breweries, International Journal of Food Microbiology, 17, 321-328. [ Links ]
Kaewwichian, R., & Limtong, S., (2014). Nakazawaea siamensis f.a., sp. nov., a yeast species isolated from phylloplane. International Journal of Systematic and Evolutionary Microbiology, 64, (1), 266-270. doi: 10.1099/ijs.0.057521-0. [ Links ]
Kawata, M., Tsutomu, F., & Harayuki, I. (2007). Intraespecies diversity of the Industrial yeast strains Saccharomyces cerevisiae and Saccharomyces pastorianus based on analysis of the sequences of the internal transcribed spacer (ITS) Regions and the D1/D2 Region of 26S rDNA. Bioscience, Biotechnology, and Biochemistry, 71 (7), 1616-1620. [ Links ]
Kopecka, J., Matoulkova, D, Jelinkova, M., Felsberg, J., & Nemec, M. (2013). Molecular characterization of brewing and wine yeast strains in the Czech Republic. In 26th International Conference on Yeast Genetics and Molecular Biology. http://onlinelibrary.wiley.com/doi/10.1002/yea.v30.S1/issuetoc. [ Links ]
Kopsahelis, N., Nychas, G., Nisiotou, A., Kanellaki, M., Kourkoutas, Y., & Panas, P. (2009). Molecular characterization and molasses fermentation performance of a wild yeast strain operating in an extremely wide temperature range. Bioresource Technology, 100, 4854-4862. [ Links ]
Kurtzman, C. P., & Robnett, C. J. (1998). Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit (26S) ribosomal DNA partial sequences. Antonie van Leeuwenhoek, 73, 331-371. [ Links ]
Kurtzman, C. P., & Robnett, C. J. (2014). Description of Kuraishia piskuri f.a., sp. a new methanol assimilating yeast and transfer of phylogenetically related Candida species to the genera Kuraishia and Nakazawaea as new combinations. FEMS Yeast Research, 14 (7), 1028-1036. [ Links ]
Kurtzman, C. P., Quintilla, M., Kolecka, A., Theelen, B., Robert, V., & Boekhout, T. (2015). Advances in yeast systematics and phylogeny and their use as predictors of biotechnologically important metabolic pathways. FEMS Yeast Research., 15 (6): doi: http://dx.doi.org/10.1093/femsyr/fov050. [ Links ]
Le, S. Y., Knudsen, F. B., & Poyton, R. O. (1985). Differentiation of brewery yeast strains by restriction endonuclease analysis of their mitochondrial DNA. Journal of the Institute of Brewing, 91, (3), 169-173. [ Links ]
Legras, J. L., & Karst F. (2003). Optimisation of interdelta for Saccharomyces cerevisiae strain characterization. FEMS Microbioly Letters, 221, 249-255. [ Links ]
López-Arboleda, W., Ramírez-Castrillón, M., Mambuscay-Menaa, L., & Osorio-Cadavid E. (2010). Diversidad de Levaduras asociadas a chichas tradicionales de Colombia. Revista Colombiana de Biotecnología, 12, (2), 176-186. [ Links ]
Mitrakul, C., Henick-Kling, T., & Egli, C. (1999). Discrimination of Dekkera/Brettanomyces yeast isolates from wine by using various DNA fingerprinting methods. Food Microbioly, 16, 3-14. [ Links ]
Muñoz, R., Gómez, A., Robles, V., Rodríguez, P., Cebollero, E., Tabera, L., Carrascosa, A. V., & Gonzalez, R. (2009). Multilocus sequence typing of oenological Saccharomyces cerevisiae strains. Food Microbiology, 26, 841-846. [ Links ]
Nardi, T., Carlot, M., De Bortoli, E., Corich, V., & Giacomini, A. (2006). A rapid method for differentiating Saccharomyces sensu stricto strains from other yeast species in an enological environment. FEMS Microbioly Letters, 264(2), 168-73. [ Links ]
Ness, F., Lavallée, F., Dubordieu, D., Aigle, M., & Dulau, L. (1993). Identification of yeast strains using the polymerase chain reaction. Journal of the Science of Food Agriculture, 62, 89-94. [ Links ]
Oda, Y., & Ouchi K. (1989). Principal-componente analysis of the cahracteristicsdesirable in baker's yeast. Applied and Environmental Microbioly, 55, 1495-1499. [ Links ]
Orberá, T. (2004). Métodos moleculares de identificación de Levaduras de Interés biotecnológico. Revista Iberoamericana de Micología, 21, 15-19. [ Links ]
Osorio-Cadavid, E., Chaves-López, C., Tofalo, R., Paparella, A., & Suzzi, G. (2008). Detection and identification of wild yeasts in Champús, a fermented Colombian maize beverage. Food Microbiology, 25 (6), 771-777. [ Links ]
Ozturk, I., & Sagdic, O. (2014). Biodiversity of yeast mycobiota in "sucuk," a traditional Turkish fermented dry sausage: phenotypic and genotypic identification, functional and technological properties. Journal of Food Sciences, 79 (11), M2315-M2322. doi: 10.1111/1750-3841.12662. [ Links ]
Praet, J., Meeus, I., Cnockert, M., Aerts, M., Smagghe, G., & Vandamme, P. (2015). Bifidobacterium commune sp. Nov. isolated from the bumble bee gut. Antonie Van Leeuwenhoek, 7 (5), 1307-1313. doi: 10.1007/s10482-015-0425-3. [ Links ]
Prakitchaiwattana, C. J., Fleet, G. H., & Heard, G, M. (2004). Application and evaluation of denaturing gradient gel electrophoresis to analyse the yeast ecology of wine grapes. FEMS Yeast Research, 4 (8), 865-77. [ Links ]
Pramateftaki, P. V., Lanaridis, P., & Typas, M. A. (2000). Molecular identification of wine yeasts at species or strains level a case study with strains from two vine-growing areas of Grece. Journal of Applied Microbioly, 89, 236-248. [ Links ]
Puig, S., Querol, A., Barrio, E., & Perez-Ortin, J. E. (2000). Mitotic recombination and genetic changes in Saccharomyces cerevisiae during wine fermentation. Applied and Environmental Microbioly, 66, 2057-2061. [ Links ]
Querol, A., Barrio, E., & Ramón, D. (1992a). A comparative study of different methods of yeast strain characterization. Systematic and Applied Microbiology, 15 (3), 439-446. [ Links ]
Querol, A., Barrio, E., Huerta, T., & Ramón, D. (1992b). Molecular monitoring of wine fermentations conducted by active dry yeast strains. Applied and Environmental Microbioly, 58, 2948-2953. [ Links ]
Querol, A., Barrio, E., Huerta, T., & Ramón, D. (1993). Utilización de técnicas moleculares para la caracterización de levaduras vínicas y el estudio del proceso de vinificación. Revista Microbiología SEM, 9, 76-82. [ Links ]
Qian, J., Cutler J. E., Cole, R. B., & Cai, Y. (2008). MALDI-TOF mass signatures for differentiation of yeast species, strain grouping and monitoring of morphogenesis markers. Analytical and Bioanalytical Chemistry, 392 (3), 439 - 449. [ Links ]
Ramirez-Castrillon, M., Camargo, S., Inostroza-Ponta, M., & Valente, P. (2014). (GTG)5 MSP-PCR Fingerprinting as a Technique for Discrimination of Wine Associated Yeasts?. Plos One, 9 (8), e105870. doi: 10.1371/journal.pone.0105870. [ Links ]
Raspor, P., Milek, D. M., Polanc, J., Moñina, S. S., & Cadez, N. (2006). Yeasts isolated from three varieties of grapes cultivated in different locations of the Dolenjska vine-growing region, Slovenia. International Journal of Food Microbiology, 109, 97-102. [ Links ]
Rodríguez, M. E., Infante, J. J., Molina, M., Domínguez, M., Rebordinos, L., & Cantoral, J.M. (2010). Genomic characterization and selection of wine yeast to conduct industrial fermentations of a white wine produced in a SW Spain winery. Journal of Applied Microbiology, 108 (4), 1292-1302. [ Links ]
Rodriguez, M. J., Fierro, J., Codon, A., Benitez, T., & Valcarcel, M.J. (2013). Selection of an autochthonous Saccharomyces strain starter for alcoholic fermentation of Sherry base wines. Society for Industrial Microbiology and Biotechnology, 40, 613-623. [ Links ]
Scannell, D. R., Zill, O. A., Rokas, A., Payen, C., Dunham, M. J., Eisen, M. B., Rine, J., Johnston M., & Hittinger, C. T. (2011). The awesome power of yeast evolutionary genetics: new genome sequences and strain resources for the Saccharomyces sensu stricto genus. G3: Genes, Genomes, Genetics,1, (1), 11-25. doi: 10.1534/g3.111.000273. [ Links ]
Schoch, C. L., Seifert, K. A., Huhndorf, S., Robert, V., Spouge, J. L., Levesque, C.A., Chen, W., & Fungal Barcoding Consortium. (2012). Nuclear ribosomal internal transcribed spacer (ITS) región as a universal DNA barcode marker for fungi. Proceedings of the National Academy of Sciences of the United States of America, 109 (16), 6241-6246. doi: 10.1073/pnas.1117018109. [ Links ]
Schuller, D., Valero, E., Dequin, S., & Casal, M. (2004). Survey of molecular methods for the typing of wine yeast strains. FEMS Microbioly Letters, 231, 19-26. [ Links ]
Stender, H., Kurtzman, C., Hyldig-Nielsen, J. J., Sørensen, D., Broomer, A., Oliveira, K., Perry-O'Keefe, H., Sage, A., Young, B., & Coull, J. 2001. Identification of Dekkera bruxellensis (Brettanomyces) from Wine by Fluorescence In Situ Hybridization Using Peptide Nucleic Acid Probes. Applied and Environmental Microbioly, 67, 938-941. [ Links ]
Suranska, H., Vranova, D., Omelkova, J., & Vadkertiova, R. (2012). Monitoring of yeast population isolated during spontaneous fermentation of Moravian wine. Chemical Papers, 66, (9), 861-868. [ Links ]
Tesfaw, A. & Assefa, F. (2014). Current Trends in Bioethanol Production by Saccharomyces cerevisiae: Substrate, Inhibitor Reduction, Growth Variables, Coculture, and Immobilization. International Scholarly Research Notices, Article ID 532852, 11p.. http://dx.doi.org/10.1155/2014/532852. [ Links ]
Tofalo, R., Perpetuani, G., Di Gianvito, P., Schirone, M., Corsetti, A., & Suzzi, G. (2014). Genetic diversity of FLO1 and FLO5 genes in wine flocculent Saccharomyces cerevisiae strains. International Journal of Food Microbiology, 191, 45-52. doi: 10.1016/j.ijfoodmicro.2014.08.028. [ Links ]
Valente, P., Boekhout, T., Fontes, M., Crestani, J., Pagnocca, F., Durae, L., Zambrano, M., Rosa, C., Brandao, L., Pimenta, R., Ribeiro, J., Marques, K., Ching-Fu, L., Sung-oui, S., Gabor, P., Dlauchy D., Fell, J, Scorzetti, G., Theelen, B., & Vainstein, M. (2012). Bandoniozyma gen.nov.a Genus of Fermentative and Non-fermentative tremellaceous Yeast Species. Plos One, 7 (10): e46060. doi: 10.1371/journal.pone.0046060. [ Links ]
Vásquez, J. A., Laguado, J. A., Lopez, J., & Gil, N. J. (2016). New sources and methods to isolate vinasse-tolerant wild yeasts efficient in ethanol production. Annals of Microbiology, 66 (1), 187-195. [ Links ]
Vezinhet, F., Blondin, B., & Hallet, J. N. (1990). Chromosomal DNA patterns and mitochondrial DNA polymorphism as tools for identification of enological strains of Saccharomyces cerevisiae. Applied Microbioly Biotecnoly, 32, 568-571. [ Links ]
Vigentini, I., Antoniani, D., Roscini, L., Comasio, A., Galafassi, S., Picozzi, C., Corte, L., Compagno, C., Dal Bello., Cardinali, G., & Foschino, R. (2014). Candida milleri species reveals intraspecific genetic and metabolic polymorphisms. Food Microbioly, 42, 72-81. doi: 10.1016/j.fm.2014.02.011. [ Links ]
Wang, H., Hu, Z., Long, F., Niu, C., Yuan, Y., & Yue, T. (2015). Characterization of Osmotolerant Yeasts and Yeast-Like Molds from Apple Orchards and Apple Juice Processing Plants in China and Investigation of Their Spoilage Potential. Journal of Food Sciences, 80 (8), M1850-M1860. doi: 10.1111/1750-3841.12946. [ Links ]
Wolters, J. F., Chiu, K., & Fiumera, H. L. (2015). Population structure of mithocondrial genomes in Saccharomyces Cerevisiae. BMC Genomics, 16, 451. doi: 10.1186/s12864-015-1664-4. [ Links ]
Yurkov, A. M., Kachalkin, A. V., Daniel, H. M., Groenewald, M., Libkind, D., de Garcia, V., Zalar, P., Gouliamova, D. E., Boekhout, T., & Begerow, D. (2015). Two yeast species Cystobasidium psychroaquaticum f.a. sp. nov. And Cystobasidium rietchieii f.a. sp. nov. Isolated from natural environments, and the transfer of Rhodotorula minuta clade members to the genus Cystobasidium. Antonie Van Leeuwenhoek, 107, (1), 173-185. doi: 10.1007/s10482-014-0315. [ Links ]