INTRODUCCIÓN
La celulosa es uno de los componentes más abundantes de la biomasa vegetal y es degradada por microorganismos (bacterias y hongos) los cuales producen un complejo enzimático que comprende por lo menos tres grandes grupos (Rabinovich et al., 2002): Endoglucanasas o endo-1-4-P-glucanasas (EC 3.2.1.4), celobiohidrolasas (EC 3.2.1.91) y P-glucosidasa (EC 3.2.1.21). Los materiales lignocelulósicos, compuestos por celulosa, hemicelulosa y lignina, son una fuente importante de materias primas para la producción de energía y biomoléculas (Ghosh & Singh 1993; Sánchez 2009), son abundantes y disponibles en los países tropicales. Un sector agroindustrial que genera grandes cantidades de biomasa lignocelulósica es la producción de aceite de palma. Con la producción de más de un millón de toneladas de aceites de palma y de palmiste, Colombia es el cuarto productor de aceite de palma en el mundo y el primer productor en América (Fedepalma, 2018). La palma de aceite está presente en cuatro zonas de Colombia: Norte, Oriental, Central y Suroccidente, que abarcan 122 municipios en 19 departamentos alrededor del país, siendo su área sembrada de 31,563 hectáreas en 2016 (Fedepalma, 2018). La palmicultura es una de las actividades agrícolas más prometedoras como eje para alcanzar el desarrollo nacional (Fedepalma, 2018). En 201 7, en Colombia se produjeron 8,6 millones de toneladas de frutos (Fedepalma, 201 8) y durante la extracción del aceite se generaron cerca de 3,5 millones de toneladas de materiales lignocelulósicos (Fedepalma, 201 8). La producción anual mundial de estos materiales alcanza 184,6 millones de toneladas (Kelly-Yong et al., 2007).
Una de las especies más estudiadas en la producción de celulasas, enzimas importantes para el aprovechamiento de biomasa lignocelulósica, son los hongos del género Trichoderma (Hypocreales, Acomycota) (Jiang et al., 2011; Juhasz et al., 2005; Liming & Xueliang 2004; Rodríguez & Piñeros 2007; Uusitalo et al., 1991) considerando su alta capacidad y conocimiento de secreción enzimática activa sobre celulosa. La identificación de especies de Trichoderma ha tenido una historia accidentada y la identificación taxonómica está en conflicto, ya que el grado de similitud morfológica es bastante alto. Rifai (1969) sugirió que más de una especie de Hypocrea produce caracteres similares de Trichoderma y la mayoría de especies, como T. harzianum se definen como agregados. Por lo tanto, parámetros adicionales más allá de los criterios morfológicos son necesarios para designar la especie y género. El análisis morfológico es altamente propenso a errores y aproximadamente el 50% de Trichoderma sp. clasificados por análisis morfológico está erróneamente realizado (Kubicek et al., 2003).
En las últimas décadas se ha dado importancia a la aplicación de técnicas moleculares para la detección e identificación de hongos. Las herramientas moleculares utilizadas para la identificación basada en la secuenciación del ADN son los marcadores ITS1- 5,8S - ITS2, una secuencia parcial de las proteínas que codifican la traducción del gen del factor de elongación (EF-1 a) y la ARN polimerasa subunidad II (RPB2) que han permitido resolver la confusión en la taxonomía de Trichoderma (Boureghda et al., 2008). Estas técnicas han sido usadas en el análisis filogenético del género Trichoderma, usando el análisis de la secuencia de la región ITS1 del ADNr (Kindermann et al., 1998).
El objetivo del presente estudio fue establecer género y especie de hongos Trichoderma sp. nativos de la región de Cumaral, Meta, Colombia, utilizando el análisis de la secuencia nucleótidica de las regiones ITS1-ITS4 y evaluar la actividad celulolítica de los complejos producidos por las cepas estudiadas usando residuos de palma de aceite como sustrato.
MATERIALES Y MÉTODOS
Cepas. Se realizaron cultivos monospóricos de cepas nativas de Trichoderma (HR-01-89, HR-03-89, HR-06-89, HR-16-89, HR-18-89, HR-04-89, HR-11-89, HR-18-89) aisladas de racimos vacíos de palma en descomposición, recolectados de la región de Cumaral-Meta (Colombia), y conservadas en glicerol a -80°C. Se utilizó como cepa de referencia a Trichoderma reesei (E. Simmons. A CECT 241 4) (ATCC 24449).
Extracción de DNA. Se aisló DNA utilizando el kit "Ultra CleanTM Tissue & Cells DNA Isolates" (MO BIO Laboratories Inc). El DNA se reveló en electroforesis de agarosa al 1% y se registró con un fotodocumentador UVP Gel-Doc-IT TM Imaging system (UVP LSC. USA). El DNA se cuantificó en un Nanodrop utilizando el programa ND-1000 V3.7.1. y luego se almacenó a -20°C previo a su utilización.
Amplificación por PCR. Se amplificaron las regiones ITS1-ITS4. Se utilizaron los oligonucleótidos iniciadores directo ITS1: 5' TCC GTA GGT GAA CCT GCG G 3' y reverso ITS4: 5'TCC TCC GCT TAT TGA TAT GC 3' (White et al., 1990). La reacción de PCR se realizó en un termociclador MJ Research PTC-100, utilizando 200 uM de dNTPs, 1.5 mM de MgCl2, 1X Buffer de reacción, 0.5 uM de oligonucleótidos, 1 U Taq polimerasa y 50 ng de DNA en un volumen final de 20 uL. Las reacciones de PCR incluyeron un ciclo inicial de denaturación a 94°C durante 5 min, seguido por 35 ciclos de denaturación a 94°C por un min, 51°C por un min y 72°C por un min, seguido por un ciclo final de extensión a 72°C durante 5 min. Los productos de la PCR se revelaron en geles de agarosa al 1% en tampón TBE 0.5X teñido con bromuro de etidio. La electroforesis se realizó a 100 voltios por 30 min en una cámara horizontal GEL XL ultra V-2 (Labnet International Inc.) y se registró con un fotodocumentador UVP Gel-Doc-IT TM Imaging system (UVP LSC. USA). Las bandas electroforéticas obtenidas se midieron con el programa VisionWorksLs determinando el tamaño en pares de bases (pb).
Purificación del DNA. Los fragmentos amplificados por PCR se purificaron con el kit UltraClean® GelSpin® DNA Extraction (MO BIO Laboratories Inc. USA) y se verificó su calidad por medio de electroforesis en geles de agarosa al 1%. Se registró en el fotodocumentador UVP Gel-Doc-IT TM Imaging system.
Secuenciación del DNA. Los productos purificados, se utilizaron para la reacción de tagDye Deoxy Terminador cycle-sequencing. La secuenciación se realizó de forma automatizada (Applied Biosystems 373A Stretch).
La calidad y ensamblaje de las secuencias obtenidas se analizaron con el programa CLC DNA Workbench 5.6.1 (CLC Bio, Denmark). Luego fueron analizadas por alineamiento con el programa BLAST (http://blast.ncbi.nlm.nih.gov) y en TrichOkey de ISTH y se asignó la especie de acuerdo a los niveles de concordancia para determinar género y especie (http://www.isth.info/tools/molkey/index.php). De los valores cuantitativos generados, solo se aceptaron las secuencias con el valor más alto para su comparación con las secuencias en estudio y se obtuvo el índice de similitud entre estas.
Alineamiento de las secuencias. Las secuencias obtenidas se alinearon con el programa BioEdit Sequence Alignment Editor 7.0 (Hall, 1999). Se obtuvieron 24 secuencias de diferentes especies de hongos previamente reportadas en GenBank de los géneros Hypocrea sp, Fusarium sp (grupo externo) y Trichoderma spp para realizar los análisis correspondientes (Tabla 2).
Tabla 1 Identificación molecular por PCR-ITS1-5,8-ITS4 de genes ribosomales rRNA en ocho cepas nativas de hongos (Trichoderma spp.) y una cepa de referencia.
| Aislamiento | Identificación morfológica | No. de nucleótidos | Género y especie identificada | Hit Genbank | Porcentaje de similaridad |
|---|---|---|---|---|---|
| HR-01-89 | Trichoderma sp | 607 | Trichoderma asperellum | DQ109538.1 | 100% |
| HR-03-89 | Trichoderma sp | 603 | Trichoderma asperellum | FJ412053.1 | 100% |
| HR-06-89 | Trichoderma sp | 593 | Trichoderma koningiopsis | DQ109538.1 | 100% |
| HR-16-89 | Trichoderma sp | 603 | Trichoderma asperellum | FJ412053.1 | 100% |
| HR-18-89 | Trichoderma sp | 642 | Trich o de rm a asperellum | KY750349.1 | 100% |
| ATCC-241489 | Trichoderma reesei/ | 640 | Trichoderma jecorina/ Hypocrea jecorina | FJ545237.1 | 100% |
| HR-04-89 | Trichoderma sp | 517 | Trichoderma koningiopsis | KY320641.1 | 100% |
| HR-11-89 | Trichoderma sp | 520 | Trichoderma koningiopsis | KP05911 6.1 | 99% |
| HR-19-89 | Trichoderma sp | 471 | Trichoderma koningiopsis | KY320641.1 | 100% |
Tabla 2 ITS1-ITS2 de especies de hongos obtenidas de GenBank para el análisis filogenético e identificación molecular de las cepas aisladas de biomasa de palma de aceite (Trichoderma sp).
| Género y especie | Código de Acceso (NCBI) | Regiones del DNA | No. de pares de bases (pb) |
|---|---|---|---|
| Fusarium oxysporum | EU545538 | ITS1-ITS2 | 496 |
| Trichoderma asperellum | GQ426036 | ITS1-ITS2 | 562 |
| Trichoderma ovalisporum | GQ426042 | ITS1-ITS2 | 591 |
| Trichoderma erinaceum | GQ426037 | ITS1-ITS2 | 591 |
| Trichoderma pleurotum | FJ418567 | ITS1-ITS2 | 593 |
| Hypocrea lixii | GQ463682 | ITS1-ITS2 | 452 |
| Hypocrea varidescens | AM498407 | ITS1-ITS2 | 548 |
| Trichoderma inhamatum | GQ426033 | ITS1-ITS2 | 602 |
| Trichoderma hamatum | GQ221830 | ITS1-ITS2 | 582 |
| Hypocrea virens | FJ654688 | ITS1-ITS2 | 531 |
| Hypocrea megalocitrina | DQ83551 1 | ITS1-ITS2 | 489 |
| Hypocrea sulphunea | DQ835510 | ITS1-ITS2 | 506 |
| Trichoderma brevicompactum | EU821800 | ITS1-ITS2 | 537 |
| Trichoderma cf. citrinoviride | DQ835514 | ITS1-ITS2 | 517 |
| Trichoderma longibrachiatum | FJ462769 | ITS1-ITS2 | 607 |
| Trichoderma citrinoviride | AM498490 | ITS1-ITS2 | 577 |
| Trichoderma cf. Minutisporum | AM498500 | ITS1-ITS2 | 561 |
| Hypocrea pezizoides | DQ835513 | ITS1-ITS2 | 491 |
| Hypocrea ochroleuca | DQ835512 | ITS1-ITS2 | 486 |
| Trichoderma gamsii | FJ790423 | ITS1-ITS2 | 528 |
| Trichoderma sp | DQ993585 | ITS1-ITS2 | 548 |
| Trichoderma koningiopsis | GQ229070 | ITS1-ITS2 | 774 |
| Trichoderma viride | GQ229073 | ITS1-ITS2 | 779 |
| Trichoderma atroviride | FJ232697 | ITS1-ITS2 | 607 |
Evaluación actividad celulolítica. Se realizó cultivo en fase líquida utilizando medio de cultivo Mandels (Mandels & Weber 1969) estéril, el cual se suplementó con 30 g/L de residuos de palma de aceite (racimos vacíos) molidos (1 mm en promedio) como fuente de carbono. Se realizaron cultivos (n=8 para cada cepa) de 20 mL en Erlenmeyer de 100 mL y se inocularon con 2 mL de suspensión de esporas (aproximadamente 1,42 x 108 esporas/mL), posteriormente se incubaron a 30°C en agitación orbital a 150 rpm, durante 5 días. Posteriormente las muestras fueron centrifugadas y al sobrenadante se le determinó la actividad enzimática de celulasas y el contenido de proteína soluble.
Medición de la actividad enzimática. La actividad celulasa total fue determinada en cada uno de los sobrenadantes (8 por cepa), mediante la cuantificación de azúcares reductores producidos por la acción enzimática sobre papel de filtro Whatman No.1, de acuerdo a lo establecido por la IUPAC (Ghose, 1987). Los ensayos fueron realizados utilizando 0,5 mL de sobrenadante, 1 mL de buffer de citrato 0.05 M a pH 4.8 e incubados a 50°C por 60 min, en presencia de 50 mg de papel Whatman No. 1. Posteriormente el contenido de azúcares reductores fue determinado usando el método de ácido 5-dinitrosalicílico (Miller, 1959). La actividad se determinó en unidades internacionales UI/mL de sobrenadante (UI en µmo[ glucosa/min). La concentración de proteína soluble se determinó mediante el método de Bradford. (1976).
Análisis estadísticos. Una vez comprobados los supuestos del análisis de varianza, los datos experimentales se analizaron mediante ANOVA y prueba de comparación de medias (Test de Tukey) con un nivel de confianza del 95%, mediante el software Statgraphics Plus 5.1 (Statistical Graphics Corp., 1994-2001).
RESULTADOS Y DISCUSIÓN
Se identificó molecularmente a nivel de género y especie ocho cepas nativas de Trichoderma, identificadas previamente por morfología hasta género, las cuales en su mayoría presentaron baja actividad celulolítica, en comparación con la cepa de referencia utilizada. Las metodologías utilizadas en este trabajo representan herramientas invaluables para la rápida caracterización de cepas de Trichoderma, tanto para su identificación como en su evaluación biológica. Los residuos de palma de aceite en Colombia representan una materia prima utilizable para la producción de azúcares fermentables y por ende de bioetanol. Resulta por esto importante, la evaluación de cepas nativas de hongos tipo Trichoderma, con novedosas y más potentes actividades biológicas que permitan la degradación de la celulosa recalcitrante, presente en los racimos de palma de aceite.
Se obtuvieron por PCR bandas electroforéticas del peso esperado, entre 593-640 pb que corresponden a las secuencias completas de las regiones genómicas ITS1-5,8S-ITS2. A partir de éstos se obtuvo la secuencia completa de cada una de las dos cadenas del DNA para las regiones ITS1 -5,8S-ITS2 para los ocho aislamientos nativos evaluados. Todas las secuencias se depositaron en la base de datos de GenBank (http://www.ncbi.nlm.nih.gov/) con los números de accesión JN704353.1, JN704351.1, JN704349.1, JN704347.1, JN704354.1, JN704352.1, JN704350.1, JN704348.1 y JN704346.1 (tabla 1).
Al analizar las secuencias nucleotídicas obtenidas, se verificó la región genómica para todos los aislamientos, obteniéndose la secuencia parcial del gen 18S rDNA, el ITS-1, el gen 5,8 rDNA, el ITS-2 y la secuencia parcial del gen 28S rDNA. Se observó para las ocho cepas la presencia de las cinco anclas que identifican hongos del género Trichoderma (tabla 1), identificándose cuatro de los hongos nativos como Trichoderma koningiopsis (HR-04-89; HR-11-89; HR-19-89; y HR-06-89) y cuatro como Trichoderma asperellum (HR-01-89; HR-03-89; HR-16-89; HR-18-89).
Por último, la secuencia de la cepa de referencia que se utilizó en este estudio (ATCC 24449) fue identificada como Trichoderma reesei/Hypocrea jecorina presentando las cinco anclas en las posiciones 78, 101, 281, 439 y 533 en la región ITS secuenciada; resultado que ratifica las bondades de esta metodología.
En estudios previos se ha descrito el aislamiento y caracterización molecular (ITS1 e ITS2 del rDNA) de cepas de Trichoderma aisladas principalmente de suelo en el Sudeste Asiático (Kubicek et al., 2003). El estudio de la producción de celulasas por hongos sobre residuos de palma se ha reportado utilizando Chaetomium globosum (Umikalson et al., 1997) Aspergillus terreus (Shahriarinour et al., 2011), Aspergillus niger (Prasertsan et al., 1997), Pycnoporus sanguineus (Teoh & Mashitah, 2010), Trichoderma harzianum (Alam et al., 2009), Trichoderma viride (Rodríguez & Piñeros, 2007) y Trichoderma reesei (Wahid et al., 2011) entre otros. Trichoderma harzianum se ha cultivado sobre los residuos de palma para ser utilizado en control biológico (Kanjanamaneesathian et al., 2003).
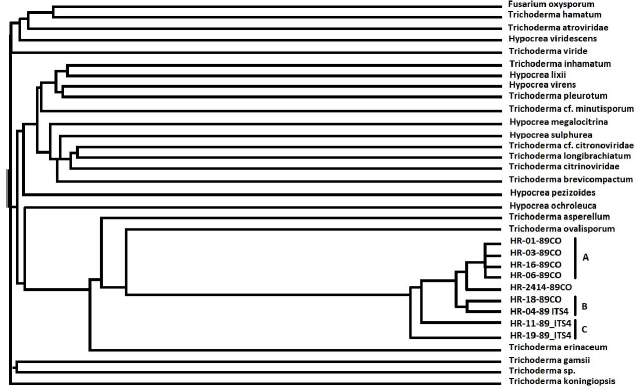
Figura 1 Cladograma basado en secuencias nucleótidicas de la región ITS1-ITS4 de cepas nativas de hongos aislados de raquis de palma africana (Trichoderma spp) y 24 especies de Trichoderma, Fusarium, e Hypocrea (GenBank; www.ncbi.nlm.nih.gov) analizadas con el algoritmo Neighbor Joining Se presentan tres ramas entre las secuencias de los hongos nativos evaluados (A, B y C) bien definidas. La rama A muestra una similitud de las secuencias del 100% entre las cuatro muestras.
Alineamiento de secuencias
Se utilizó la secuencia del ITS1-ITS4 del hongo Fusarium oxysporum como grupo externo para realizar comparaciones. Se generó un cladograma utilizando el Programa Neiborg Joining (figura 2) que evidencia que los siete aislamientos nativos aisladas de Trichoderma spp se encuentran más relacionadas entre ellas, así HR-01-89, HR-03-89, HR-16-89, HR-06-89, HR-11-89, HR-18-89, HR-04-89, HR-2414-89 y HR-18-89, formaron un grupo.
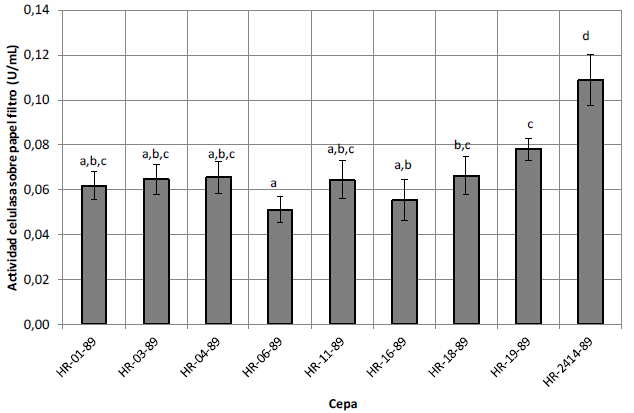
Figura 2 Actividad enzimática sobre papel filtro del complejo producido por cepas de Trichoderma spp sobre residuos de palma como sustrato. Barras blancas corresponden a proteína soluble y las negras a actividad enzimática. Las barras de error corresponden al valor de desviación estándar (n=8). Diferentes letras sobre cada columna indican diferencias significativas entre las actividades de las cepas (p <0.05).
Se observa que las cepas HR-01-89, HR-03-89, HR-16-89 y HR-06-89 conforman un solo clado, (clado A) indicando una alta similitud nucleótidica entre ellas, es posible que estas tres cepas pertenezcan a una misma colonia, debido a que se identificaron como Trichoderma koningiopsis con las posiciones idénticas para las cinco anclas observadas entre las cepas HR-03-89, HR-16-89 y HR-06-89 y ligeramente diferentes con la cepa HR-01-89 (tabla 1). La cepa de referencia ATCC 24449, Trichoderma reesei, presentó una relación más cercana a estas cuatro cepas, mostrando una alta similitud con este clado.
Las cepas HR-18-89 y HR-04-89 forman otro clado (clado B) indicando relación entre ellas y menos relación con las cepas del clado A. Por último, las cepas HR -11-89 y HR-18-89 formaron el clado C. Sin embargo, es importante anotar que la cepa HR-11-89 presentó las cinco anclas en las posiciones 74, 95, 240, 398 y 498 en la región ITS identificándose como T. koningiopsis. Por el contrario la cepa HR-18-89 solo presentó la 2da ancla en la posición 18 (tabla 1).
Determinación de la actividad celulolítica
Las cepas identificadas molecularmente se evaluaron biológicamente para determinar su actividad celulasa.
En la figura 2 se presentan los resultados de actividad enzimática de los complejos producidos por las cepas estudiadas utilizando residuos de palma como sustrato (valores promedio, n=8). Se encontraron diferencias estadísticamente significativas en la actividad celulolítica de los complejos producidos por las cepas estudiadas (ANOVA, F(8,63) =39,18, p = 0.000), (Test de Tukey, n=6, p<0,05). La prueba de Tukey, mostró que las cepas HR-01-89, HR-03-89, HR-11-89, HR-04-89 y HR-18-89 no presentan diferencias en la actividad enzimática, alcanzando un valor promedio de 0,07 UI/mL. La cepa de referencia HR-2414-89 (Trichoderma reesei) presentó mayores valores de actividad enzimática (0,11 UI/mL) comparada con las cepas nativas cultivadas sobre residuos de palma de aceite. La cepa nativa HR-18-89 (T. asperellum) presentó los mayores niveles de actividad enzimática, un 78% comparada con el valor de la cepa referencia.
Relacionado con cepas aisladas y evaluadas sobre residuos de palma, recientemente se ha estudiado una nueva cepa de Trichoderma koningii D-64 aislada de muestras de suelo de Singapur y se comparó con Trichoderma reesei Rut C30 (ATCC 56765). Se estudió la producción de celulasas sobre celulosa y salvado de trigo (además de otros compuestos), encontrando valores de actividad sobre papel filtro de 1,4 ±0,1 UI/mL y 0,5 ±0,1 UI/mL, respectivamente, actividades superiores a las obtenidas en este trabajo. También se evaluó la capacidad hidrolítica utilizando residuos de palma pretratados químicamente (tratamiento alcalino). Se encontró una mayor capacidad de hidrólisis con enzimas producidas por la cepa de Trichoderma reesei Rut C30 comparada con la cepa D-64 aislada. Sin embargo, mediante ajuste de dosis de enzimas se lograron valores similares de hidrólisis enzimática (Wang et al., 2012). En este estudio, se encontró superioridad en la capacidad hidrolítica de enzimas producidas por la cepa de Trichoderma reesei sobre las cepas nativas.
En otro trabajo publicado, se describió el aislamiento de microorganismos productores de celulasas en residuos de palma de aceite en descomposición en Malasia específicamente bacterias (Baharuddin et al., 2009) y de hongos degradadores (Abu Bakar et al., 2010). En este último trabajo se aislaron dos cepas de Trichoderma (SK1 y SK5), con actividades sobre papel filtro produciendo 0,2 y 0,4 UI/mL a los cinco días de cultivo, valores superiores a los obtenidos en este estudio (0,050,11) (figura 2). Esto puede deberse a que los residuos de palma de aceite utilizados como sustrato en la producción de las enzimas fueron materiales sin deslignificar, lo que puede haber afectado la generación de las enzimas. Se debe considerar que otras condiciones de cultivo influyen sobre la actividad celulasa de los complejos producidos por hongos Trichoderma utilizando residuos de palma como sustrato, dentro de los cuales se ha reportado el tiempo de cultivo, el tamaño del inóculo y la concentración de sulfato amonio (Kubicek et al., 2003).
CONCLUSIÓN
La identificación morfológica de hongos tipo Trichoderma spp aunada al uso de técnicas moleculares que confirmen género y especie, representa una metodología rápida, económica y que requiere poca labor, produciendo un conocimiento taxonómico de las cepas con el cual se puede proceder a realizar bioensayos que permitan reconocer las bondades de aislamientos nativos con actividades biológicas de interés para el aprovechamiento de materiales lignocelulósicos.
Se ha identificado hongos del género Trichoderma como productores de enzimas celulolíticas.
Se ha logrado establecer que todos tienen casi la misma actividad, pero La cepa nativa HR-18-89 (Trichoderma asperellum) tiene mayor actividad enzimática sobre la celulosa existente en la Palma aceitera. Esta cepa promisoria debe ser estudiada con mayor enfasis en bioensayos específicos, para revelar su verdadero potencial celulosico.














