INTRODUCCIÓN
El género Persea agrupa aproximadamente a 190 especies (Van der Werff, 2002), se estima que 90 especies se encuentran distribuidas en el neotrópico, desde el sur de los Estados Unidos de Norteamérica (Persea bor-bonia, (L.) Spreng, P. humilis Nash y P. palustris Sarg), hasta Chile (Persea lingue) (Kopp, 1966; Barrientos y López, 2001; Van der Werff, 2002). En las Antillas específicamente en las Islas Canarias, se tiene registrada a la especie Persea indica (L.) Spreng. Alrededor de 100 especies están distribuidas en el sudeste de Asia (Barrientos y López, 2001; Van der Werff, 2002). Aunque la evidencia obtenida a partir de estudios recientes indica que buena parte de las especies que se distribuyen en el sudeste de Asia corresponden a otros géneros (Rohwer, 2009).
En México existen 26 especies del género Persea Mill., las cuales tienen como principal zona de distribución el sureste del país (Campos et al., 2008), y Chiapas es el estado con mayor diversidad de especies, tanto del subgénero Persea como Eriodaphne, seguido de Oaxaca y Veracruz (Martínez et al., 2016).
Los hallazgos de aguacates primitivos desde la Sierra Madre Oriental en el estado de Nuevo León, México hasta Costa Rica en Centroamérica, apoyan la hipótesis de que se trata de un centro de origen y probablemente de todo el subgénero Persea (Sánchez, 1999; Sánchez, 2007). En los municipios de Aramberri y Zaragoza de la región sur del estado de Nuevo León y muy particularmente en las partes altas de la Sierra Madre Oriental, en donde nacen los afluentes del Río Blanco en Zaragoza, aún es posible encontrar plantas silvestres como parte de la vegetación natural, cuyas características morfológicas son contrastantes a las variedades cultivadas (Gutiérrez et al., 2009).
En la actualidad se reconocen tres razas diferentes de aguacate: La Mexicana (Persea americana var. drymifolia), la Guatemalteca (P. americana var. guatemalensis) y la Antillana (P. americana var. americana). Estás tres razas se pueden diferenciar con base a sus características morfológicas, fisiológicas y de cultivo (Barrientos et al., 2010).
Actualmente, la mayoría de las variedades comerciales de aguacate cultivadas en México, son híbridos interraciales desarrollados a partir del intercambio de materiales entre las diferentes razas (Bergh, 1 995). Las variedades más importantes de climas subtropicales, como el 'Hass', 'Bacon' y 'Fuerte', son producto de las cruzas entre las razas Mexicana y Guatemalteca, las cuales tienen diferente grado de hibridación (Newett et al., 2002). La raza Mexicana incluye variedades nativas con nombres locales, las cuales producen frutos que se consumen y comercializan localmente; también se utilizan como pie de injerto para el cultivar 'Hass', que es el más distribuido en el mundo y México es el principal exportador a nivel mundial (SAGARPA, 2007).
En las regiones americanas, en donde el aguacate se cultiva desde tiempos precolombinos, la producción proviene de fuentes distintas de árboles nativos o criollos y cultivares selectos reproducidos asexualmente, en los cuales el sabor y los valores nutritivos varían según el tipo ecológico (Mijares y López, 1998).
En Nuevo León, se cultivan varios materiales nativos producidos tanto en sistemas tradicionales de huertos comerciales como de traspatio, con características fenológicas y morfológica contrastantes (Acosta et al., 201 2; 2013; Gutiérrez et al., 2009). Los municipios del Estado donde se cultiva aguacate son, Aramberri y Zaragoza en la región sur, Sabinas Hidalgo, Bustamante, San Nicolás de los Garza y Monterrey en la región norte, y Santiago, Allende, Rayones, Montemorelos, General Terán, Linares y Hualahuises, en la región centro. Por su importancia económica y diversidad genética, destacan Aramberri, Zaragoza, Sabinas Hidalgo y Bustamante (Acosta et al., 2012, 2013).
Con base en las relaciones entre las formas cultivadas y silvestres determinadas mediante el uso de marcadores bioquímicos, moleculares y polimorfismo del ADN, a la fecha se acepta que los sistemas tradicionales de producción de aguacate criollo en la región sur del estado de Nuevo León están considerados como importantes centros de experimentación, introducción de plantas y mejoramiento empírico, así como refugios de diversidad genética única que albergan genes que aún no han sido estudiados (Gutiérrez et al., 2015). Las variedades criollas también son fuente de genes de resistencia a plagas y enfermedades (Sánchez, 1999). Además, el fruto de las variedades criollas se consume localmente y tiene mucha demanda (Rincón et al., 2011).
Adicionalmente, el aguacate criollo presenta una variada posibilidad de usos, tales como productos industrializados, entre otros: pulpas como base para productos untables, tanto frescas como refrigeradas o congeladas, mitades congeladas, y obtención de aceite tradicionalmente para fines cosméticos. En virtud de que Nuevo León forma parte del centro origen del aguacate en México y que existe una gran variabilidad genética del género Persea, se planteó este trabajo cuyo objetivo fue evaluar mole-cularmente 43 colectas de aguacate criollo de la región sur y norte del Estado.
MATERIALES Y MÉTODOS
La colecta de los materiales se realizó en huertos comerciales de aguacate criollo bajo condiciones de riego y que presentaban buenas características de adaptación a la zona, producción de frutos y buen estado fitosanitario en los municipios de Aramberri y Zaragoza (27° 49' latitud norte, 98° 26' longitud oeste y 1160 msnm) en la región sur y en los municipios de Sabinas Hidalgo (26° 29' 59'' latitud norte, 100° 9' 48'' longitud oeste y 313 msnm) y Bustamante (26° 31' 30'' latitud norte, 100° 28' 24'' longitud oeste y 425 msnm) en la región norte del estado de Nuevo León.
Material genético. Se colectaron cuatro hojas jóvenes ubicadas en las puntas de las ramas por genotipo (tres individuos por genotipo), las cuales se depositaron en bolsas plásticas con su respectivo código y trasportadas en hielo para evitar su descomposición. En la región sur se colectaron 27 materiales criollos de aguacate, provenientes de seis huertos comerciales y uno de traspatio en los municipios de Aramberri e Ignacio Zaragoza (tabla 1). En la región norte, se colectaron 16 variedades criollas de aguacate, provenientes de dos huertos comerciales, en los municipios de Sabinas Hidalgo y Bustamante, Nuevo León (tabla 1).
Tabla 1 Nombre local de las variedades de aguacate criollo seleccionadas en municipios del sur y norte de Nuevo León, México.
| Nombre local | Municipio | Nombre local | Municipio |
|---|---|---|---|
| Plátano temprano | A | Amarillo5 | A |
| Plátano grueso-1 (testigo local) | A | Leonor | A |
| María Elena | A | Anita | SH |
| Campeón | A | Pepe | SH |
| Mantequilla | A | Floreño | SH |
| Calabo | A | Blanquito | SH |
| Huevo de paloma | A | De la fosa | SH |
| Todo el año | A | Cuervo | SH |
| De agua | A | Pera | SH |
| Criollo-1 | A | Verde Pérez | SH |
| Plátano delgado | A | Sabroso | SH |
| Huevo de toro | Z | Chapeño | SH |
| Pahuita | Z | Rodríguez | SH |
| Plátano grueso-2 | Z | Pecoso | SH |
| Crema | Z | El pila | SH |
| Tamaulipas | Z | Salazareño | SH |
| Plátano grueso-3 | Z | Negro Santos | B |
| De peluquería | A | Especial | B |
| Salvador | A | ||
| Criollo boleado negro | A | ||
| Cuerno | A | ||
| Criollo boleado grande | A | ||
| Criollo boleado muy grande | A | ||
| Hule | A | ||
| Pato5 | A |
A = Aramberri; Z = Zaragoza; § Sistema de producción de traspatio; SH = Sabinas Hidalgo; B = Bustamante.
Extracciones de ADN genómico y cuantificación. Para la extracción del ADN genómico total a partir de las hojas colectadas, se utilizó el método CETAB (Doyle y Doyle, 1987), modificado por Almeyda, et al,. (2001). El ADN se cuantificó en un nanodrop (Genova Nano Spectrophotometer®) y se visualizó en geles de agarosa al 0.8 % teñidos con bromuro de etidio para verificar su calidad.
Evaluación molecular. Para la evaluación molecular del germoplasma colectado se utilizó la técnica del DNA Polimórfico Amplificado al Azar (RAPD). Inicialmente se utilizarán 40 iniciadores con diferentes porcentajes de concentración Guanina-Citocina (G-C). Con base en el polimorfismo generado se eligieron cuatro iniciadores: 3'-CGAACATGAG-5' y 3'- GTCCCTGAAT-5' (Porcentaje de G-C 50%), 3'-CTTGGGTTCG-5' (Porcentaje de G-C 60%), 3'- CGAGCCGAAC-'5 (Porcentaje de G-C 70%), los cuales se utilizaron para el análisis de todas las muestras colectadas. Las reacciones de PCR se realizaron de acuerdo a las condiciones descritas por Cazares y colaboradores (2010).
Análisis estadístico. Con el fin de determinar cuáles combinaciones RAPD's eran más informativas se calcularon algunos parámetros propuestos por Laurentini y Karlovsky (2007), tales como: Contenido de Información Polimórfica (PIC), el Indice de Marcador (MI) y el Poder de Resolución (PR).
El valor de PIC para cada combinación de iniciadores RAPD's, se calculó con la formula PIC= 2f (1- f), propuesta por Roldán-Ruiz et al., (2000). Dónde: PIC es el contenido de información polimórfica del marcador i, fi la frecuencia de los fragmentos del marcador, que estaban presentes y 1 - fi la frecuencia de los fragmentos del marcador, que estaban ausentes. El PIC fue un promedio de los fragmentos para cada primer RAPD's.
El índice del marcador (MI), se calculó con la fórmula MI = PIC x EMR (Varshney et al., 2007). Dónde: EMR es la proporción múltiple efectiva, (E) se define como el producto del número total de loci / fragmentos por iniciador (n) y la fracción de loci polimórficos / fragmentos (â), (E = n. â).
El poder de resolución (PR) de cada iniciador, se calculó con la fórmula RP = ÓI b (Prevost y Wilkinson 1999). Dónde: Ib representa la informatividad del fragmento. La I b se puede representar en una escala 0 - 1 por la siguiente fórmula: I b = 1 - [2 x (0.5 - p)j. Dónde: p es la proporción de las 43 accesiones que contiene el fragmento.
Índice de Diversidad. El índice de diversidad genética de germoplasma, se calculó sobre la base de la fórmula DI = 1 - Ó Pi 2 (Powell et al., 1 996). Donde Pi es la frecuencia del alelo i n , cada alelo individual es considerado único en sí mismo y un fragmento de amplificación.
Relaciones genéticas. Para cada iniciador RAPD's, se generó una matriz binaria de unos y ceros, para la presencia "1" y ausencia "0" de cada una de las bandas en cada accesión. Matrices de similitud por parejas fueron generadas para los datos moleculares utilizando la versión FREETREE 2.1 (Pavlicek et al., 1999). Se construyó un dendrograma utilizando el método de par-grupo no ponderado con la media aritmética (UPGMA) y el principal método de coordinar el análisis se realizó utilizando el Software Darwin 5.0.158 (Perrier & Jacquemoud-Collet, 2006). Un remuestreo bootstrap (10,000), se realizó para determinar la robustez del dendrograma. A partir de estas matrices, se determinaron los límites de confianza para cada comparación por pares (Felsenstein, 1985).
RESULTADOS Y DISCUSIÓN
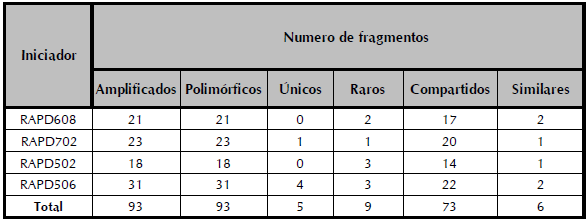
Un total de cuatro diferentes iniciadores RAPD's se usaron para el análisis de todas las muestras colectadas en este estudio. Cada uno de los iniciadores RAPD's generó información de los 43 genotipos de aguacate (tabla 2). El número de productos amplificados por ensayo varió de 18 a 31. En total las cuatro combinaciones de oligonucleótidos selectivos amplificaron 93 productos, de los cuales el 100 % se consideran polimórficos (tabla 2).
En el análisis de las frecuencias y distribución de los fragmentos polimórficos, se observó un total de cinco fragmentos específicos, con un promedio de 1.25 fragmentos por iniciador (tabla 2). Los genotipos que presentaron fragmentos únicos fueron Huevo de toro, Criollo Uno, Salazareño y Especial (tabla 3).
Tabla 3 Combinación RAPD y genotipo con fragmentos únicos.
| Iniciador | Genotipo | Fragmentos únicos |
|---|---|---|
| RAPD702 | Huevo de toro | 1 |
| RAPD506 | Criollo Uno | 1 |
| Salazareño | 2 | |
| Especial (Bustamante) | 1 |
Así mismo, se evaluó el número de fragmentos raros (presentes hasta en el 10 % de las accesiones) por iniciador (tabla 2). Se observó un total de 9 fragmentos raros con un promedio de 2.25 por iniciador RAPD's.
El mayor número de fragmentos raros, se observó en los iniciadores RAPD502 y RAPD506 con tres, mientras que el iniciador RAPD702 solo presentó un fragmento raro (tabla 2).
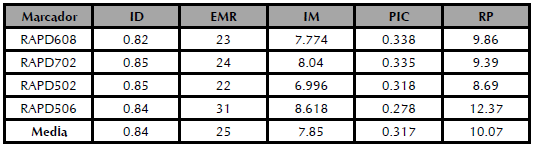
Se observaron un total de 73 fragmentos compartidos (fragmentos presentes hasta en un 70 % de las accesiones para un locus en particular), con un promedio de 18 fragmentos compartidos por iniciador (tabla 2). Este tipo de fragmentos son los más informativos ya que presentan un PIC en promedio de 0.317 por iniciador RAPD's (tabla 4). El número de fragmentos similares (fragmentos presentes en más del 70 % de las accesiones para un locus en particular), fue 6 con un promedio de 1.5 por iniciador (tabla 2).
Para determinar cuáles iniciadores RAPDs son más informativos, se calculó el contenido de información poli-mórfica (PIC) para los cuatro iniciadores, la que varió de 0.330 a 0.278, con un promedio de PIC 0.317. El índice de marcador (IM) presentó valores de 6.996 a 8.618 con un promedio de 7.85, mientras que el poder de resolución (RP) vario de 8.69 a 12.37 con un promedio de 10.07. Por su parte el índice de diversidad (ID) promedio fue del 84 % (tabla 4).
Relaciones genéticas. En el dendrograma generado, se formaron dos grandes grupos o nodos, los cuales se agruparon por su origen geográfico, es decir en un nodo están agrupados los materiales colectados en la región sur y en otro nodo, se agruparon los materiales de la región norte, dichas regiones presentan condiciones agroecológicas diferentes. Adicionalmente en el nodo donde se agrupan los materiales de la región norte se divide en dos subgrupos, en uno de ellos, se agrupan los materiales colectados en el municipio de SabinasHidalgo y en el otro, los colectados en el municipio de Bustamante (figura 1).

Figura 1 Relaciones genéticas obtenidas mediante la técnica de RAPD entre materiales de aguacate criollo de Nuevo León.
Estos resultados son coincidentes con lo reportado por Cañas et al. (2015), quienes al realizar la caracterización molecular y morfológica de 111 cultivares criollos y comerciales de aguacate (Persea americana Mill) en el departamento de Antioquia, Colombia, encontraron altos niveles de heterocigocidad, y la distancia genética de Nei, produjo tres agrupaciones que incluyeron individuos que se encontraban con gran cercanía geográfica y similares condiciones climáticas. Un análisis de componentes principales (ACP) basado en los caracteres morfológicos de los materiales en estudio, produjo tres agrupaciones concordantes con lo encontrado en el dengrograma generado con el algoritmo UPGMA (Unweighted Pair Group Method with Arithmetic Mean).
Cuiris et al., en el 2009, también encontraron alta variación genética entre 77 accesiones de aguacate de la raza mexicana colectados en diez estados de México (Chiapas, Guanajuato, México, Michoacán, Morelos, Nuevo León, Oaxaca, Puebla, Tabasco, Veracruz), y mantenidos en un banco de germoplasma del Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias de México, y señalan que esos altos valores pueden tener su explicación en el constante movimiento del material de aguacate para usos comerciales en todo el país, lo anterior aunado a la diversificación del suelo y las condiciones climáticas en esas regiones han favorecido la variación genética dentro de esta especie.
Un gran número de marcadores de ADN han sido desarrollados para evaluar la diversidad genética en plantas, tanto en especies silvestres como en plantas cultivadas (Bensch y Akesson 2005; Tatikonda et al., 2009). En este trabajo se utilizaron marcadores tipo RAPD para evaluar la diversidad genética de aguacate nativo en el estado de Nuevo León, México. Estos marcadores revelaron robustos perfiles de amplificación y fueron 100% polimórficos, lo que permitió la diferenciación entre genotipos.
El nivel de diversidad genética detectado fue alto (84%), lo que coincide con lo observado por Gutierrez-Diez et al. (2009), e incluso fue superior a lo observado por Ashworth and Clegg en el 2003 quienes reportaron un promedio de diversidad genética del 60.7 % cuando analizaron 35 cultivares de aguacate y dos parientes silvestres por medio de marcadores microsatélites y por polimorfismos de longitud de fragmentos de restricción (RFLP). En el análisis de las frecuencias y distribución de los fragmentos polimórficos, se observaron fragmentos específicos o únicos tipo RAPD's, presente en un solo individuo.
Según Foster et al. (2010), la presencia de este tipo de fragmentos en el germoplasma, está relacionado con pérdida de diversidad genética debido posiblemente a la selección. Sin embargo, Agrama y Tuinstra (2003) mencionan que los alelos específicos y raros son de particular interés ya que pueden estar ligados a un genotipo en particular y servir en el diagnóstico para diferenciar un genotipo o una región específica del genoma. En este estudio los genotipos con el mayor número de fragmentos únicos y/o raros fueron el Huevo de toro y Amarillo. Los resultados obtenidos en este trabajo, soportan la eficiencia de los marcadores RAPD's en la caracterización molecular del germoplasma de aguacate criollo de Nuevo León.
López et al. (2007), mencionan que el 42% de los genes secuenciados del aguacate criollo están relacionados con metabolismo, 20% son de función desconocida, 14% genes de maduración de fruto, 8% síntesis de Ac. Grasos, 6% de respuesta a patógenos, interesantemente un 6% son genes no reportados y 4% genes involucrados en senescencia. El entendimiento de lo que sucede a nivel molecular en el proceso de maduración de aguacate tiene una gran importancia para diferentes aspectos del cultivo, principalmente para el manejo postcosecha y para programas de mejoramiento genético.
Lo anterior es de particular interés para el aguacate nativo del Estado de Nuevo León, cuyo problema para su comercialización es la corta vida de anaquel que presenta, por lo tanto, encontrar gran variación genética como se detectó en este trabajo, incrementa la posibilidad de generar nuevos materiales cuya vida de anaquel sea más prolongada, lo cual incrementaría su valor comercial y por otro lado, se podría aprovechar la característica de los aguacates nativos colectados para generar variedades con altos contenidos de aceites esenciales superior a lo registrado en variedades comerciales como el Hass, característica observada en algunas de las variedades criollas colectadas.
CONCLUSIONES
Los marcadores RAPD resultaron altamente informativos y permitieron diferenciar el total de los materiales de aguacate evaluados, los cuales se agruparon por su origen geográfico. La diversidad genética detectada fue elevada (84%), lo que permite inferir que hay variabilidad suficiente en el germoplasma de aguacate de Nuevo León para la generación de nuevas variedades de aguacate con mejores características de interés comercial.