INTRODUCCIÓN
El uso de la biotecnología moderna ha hecho posible modificar la composición genética de los organismos, utilizando técnicas de biología molecular, las cuales permiten aislar, alterar, manipular y transferir material genético de un organismo a otro para brindarles características nuevas como resistencia a insectos, tolerancia a herbicidas (Instituto Colombiano Agropecuario, 2015; Hilder & Boulter, 1999), mayor contenido de nutrientes (proteínas, ácidos grasos, vitaminas o minerales) (Beyer et al., 2002) así como resistencia al estrés abiótico (menor contenido de agua, pH ácidos o básicos o alta concentración de sales) (Wang et al., 2003). Dichos organismos son denominados OGM, o cultivos GM en el caso de especies cultivadas.
El desarrollo de los cultivos GM se ha enfocado en el aumento de la productividad y del valor nutricional de las especies de interés. A nivel mundial la adopción de los cultivos GM ha pasado de 1.7 millones de hectáreas cultivadas en 1996 a 179.7 millones de hectáreas en 2015, con cerca de 18 millones de agricultores (James, 2016). Colombia inicio con los cultivos GM en el año 2000, con la autorización para siembra de clavel de flor azul para la producción de flor cortada en una extensión de 12 hectáreas, (Instituto Colombiano Agropecuario, 2000). A 2016, el país cuenta con más de 160 autorizaciones para la introducción de diferentes tipos de cultivos GM y cerca de 100.000 hectáreas sembradas con algodón (Gossypium hirsutum), maíz (Zea mays), soya (Glycine max) y arroz (Oryza sativa), para diferentes usos.
Con el avance de este tipo de cultivos, se han desarrollado técnicas de análisis que permiten detectar, identificar y cuantificar diferentes tipos de modificaciones presentes en el cultivo, diferenciándose en factores como su sensibilidad y capacidad de detección. Las técnicas convencionales son reacción en cadena de la polimerasa (PCR por sus siglas en inglés) y ensayo de inmunoabsorción enzimática (ELISA por sus siglas en inglés) para ADN y proteínas, respectivamente; siendo el primero, el más empleado. Sin embargo, el desarrollo de nuevos cultivos GM con un mayor número de modificaciones, y su impacto en el comercio internacional, ha impulsado la implementación de técnicas más avanzadas para su detección y cuantificación. Así el propósito de este documento es presentar una revisión de las principales técnicas de análisis disponibles, especialmente aquellas basadas en ADN, frente a las capacidades técnicas nacionales para su detección, con base en el estado actual de los cultivos GM en Colombia y el mundo.
LOS CULTIVOS GENÉTICAMENTE MODIFICADOS EN EL MUNDO
En 2014, la superficie total sembrada con OGM fue de 181.5 millones de hectáreas, que corresponde al 13% de la superficie cultivable total en el mundo. El 82% del total de área cultivada con soya, el 68% de algodón, el 30% de maíz y el 25% de canola a nivel mundial, correspondían a variedades genéticamente modificadas (James, 2014). Se habían aprobado 357 eventos de modificación genética, de los cuales Japón, el país número uno en el ranking mundial había aprobado 201, Estados unidos 171 y la Unión Europea 67 (Inghelbrecht et al, 2014; Wong & Chan, 2016).
En 2015, el área total cultivada a nivel mundial con OGM fue de 179.7 millones de hectáreas, 1% menos que el año anterior (James, 2014), concentrándose en su mayor parte en el continente americano (87.4%), con Estados Unidos, Brasil y Argentina a la cabeza. Europa tuvo el 1.4% de participación con Portugal, España, República Checa y Eslovenia, quienes han aprobado el maíz MON810. Por su parte, el continente asiático contribuyó con el 10.9%, siendo india el país con mayor porcentaje de participación (57%) (James, 2015) (tabla 1).
Basados en el área destinada para este tipo de cultivos respecto al total disponible por país, Paraguay ocupa el primer lugar, con un 78.2% de ocupación de cultivos GM vs el área disponible para cultivo, seguido de Argentina, Uruguay, Brasil y Estados Unidos (Figura 1), lo que demuestra cómo algunos países han hecho una apuesta muy fuerte por el uso de cultivos GM, especialmente de soya, algodón y maíz. Se estima que del 70% al 90% de la producción mundial de OGM se destina a la elaboración de alimentos para animales (Lucht, 2015).
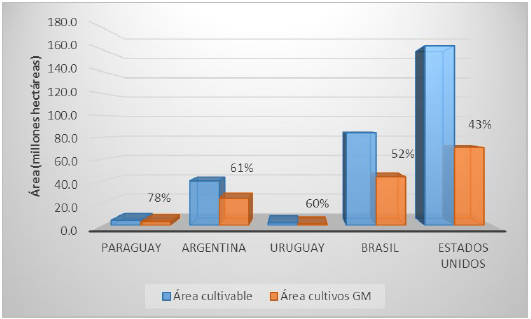
Figura 1 Porcentaje ocupación cultivos GM, calculado como la fracción de área con cultivos CM vs el área cultivable por país. Elaborado a partir de Banco Mundial (2017), Ministerio de Agricultura (2017), Ministerio de Agricultura Pecuária Abastecimento.
En China, el principal importador y consumidor de productos GM, los cultivos GM son regulados por la legislación dada para los productos convencionales, (Wong & Chan, 2016). En Estados Unidos, la Administración de Medicamentos y Alimentos (FDA, por sus siglas en inglés), considera que los alimentos GM no difieren significativamente de los alimentos no modificados genéticamente; sin una evidencia contraria, los alimentos GM son seguros y no requieren etiquetado o evaluación de seguridad particular, por lo anterior, el etiquetado para alimentos derivados de estos, es voluntario (Wong & Chan, 2016).
En la Unión Europea son poco comunes los cultivos GM, actualmente, solo se ha aprobado el cultivo de una especie genéticamente modificada, el maíz MON810 cultivado principalmente en España (Lucht, 2015). Adicionalmente, el parlamento europeo y el Consejo de la Unión Europea con la directiva 2015/412 del 11 de marzo de 2015, abrieron la posibilidad para que los países miembros restrinjan o prohiban el cultivo de OGM en su territorio. No obstante, dicha legislación no evita la presencia de ingredientes procedentes de OGM en los alimentos comercializados, los cuales pueden estar presentes en alimentos siempre y cuando su contenido sea inferior al 0.9 % en masa, ni implica la necesidad de una etiqueta indicando el uso de OGM en productos tales como huevos, leche y carne provenientes de animales a los que se les ha suministrado alimentos con OGM (Inghelbrecht et al., 2014; Parlamento Europeo y Consejo de las Comunidades Europeas, 2008).
MÉTODOS DE DETECCIÓN DE OGM
El monitoreo y la verificación de la cantidad y presencia, así como la aplicación de valores límite y requisitos legales de etiquetado y comercialización de los OGM en diferentes países, ha generado un incremento en la demanda de métodos confiables, rápidos y económicos; capaces de detectarlos, identificarlos y cuantificar-los (Kamle & Ali, 2013; Popping, 2003).
Una modificación genética consiste en la incorporación de un constructo o elemento transgénico en el genoma del huésped. El constructo está constituido por un promotor que favorece la expresión del gen insertado, un transgen que confiere la característica nueva al organismo huésped, un gen marcador que favorece la selección de las especies transformadas y un terminador que facilita la detención de la transcripción (figura 2). Si la célula expresa la información codificada en el constructor, se producirá la proteína responsable de la característica o modificación de interés. De tal forma, los métodos de detección de OGM están dirigidos básicamente a dos tipos de biomoléculas: ADN y proteínas recombinantes. El primero tiene la ventaja de ser estable, abundante y fácil de amplificar (Holst-Jensen et al., 2012), por lo que la detección de secuencias específicas de ADN, especialmente utilizando la PCR es la más empleada (Querci et al., 2008). La complejidad de la estrategia empleada para la identificación de los OGM varía de acuerdo con el tipo de modificación a estudiar, pasando del uso de métodos simples para el estudio de modificaciones individuales al uso de combinaciones de métodos para el análisis de eventos múltiples (Holst-Jensen, 2009).
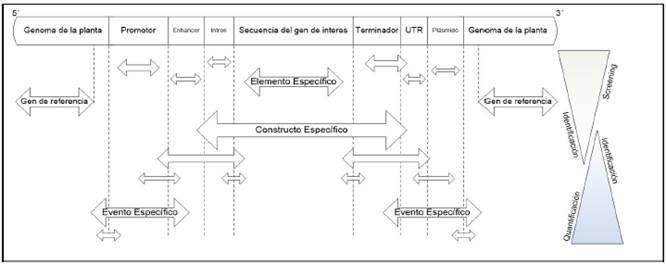
Figura 2 Niveles de especificidad para la detección e identificación de OGM. modificado a partir de Lusser etal., (2011).
Independientemente de la estrategia y los métodos usados para la cuantificación de los OGM, los resultados cuantitativos deben estar relacionados con sistemas de medición de referencia globalmente aceptados; sin embargo, en muchos casos, sus principales componentes y características metrológicas tales como unidades de medición y trazabilidad metrológica aún no han sido implementados (Emons, 2010), por lo tanto no es posible garantizar la comparabilidad de las mediciones en el tiempo y el espacio respecto a, por ejemplo, especificaciones legales y/o de producto (Koeber et al., 2010).
Validación de métodos
Para asegurar la calidad del ensayo se deben evaluar parámetros como la selectividad o especificidad, el intervalo lineal, exactitud del método, en términos de su precisión y veracidad, límites de detección y cuantificación, tanto en simplex como multiplex, empleando materiales de referencia como controles internos, de modo que se garantice que el método así desarrollado es adecuado para el fin previsto (Broeders et al., 2014; Scholtens et al., 2010). La unión europea dispone de una base de datos para métodos de referencia para análisis de OGM (GMOMETHODS, http://gmocrl.jrc.ec.europa.eu/gmomethods/) que proporciona información detallada de los parámetros de desempeño y los criterios de aceptación empleados en la validación de las metodologías de detección de un amplio número de OGM, a nivel de evento, constructo o elemento específico, tanto cuantitativos como cualitativos.
Métodos de detección basados en proteínas
Los métodos de detección basados en proteínas comprenden las diferentes tecnologías de inmunoensayos con anticuerpos mono o policlonales, varían según la especificidad del sistema de detección, la aplicación particular y el costo; y poseen límites de detección entre el 0.25% - 1% (FAO, 2007). Entre los ensayos disponibles se encuentran: ELISA, tiras de flujo lateral y Western Blot. Los dos primeros son los más utilizados para la detección de proteínas por su bajo costo, facilidad y los escasos requerimientos para el manejo de la muestra (Brett et al., 1999; Vela, 2013); sin embargo, la exactitud de éstos métodos en términos de su veracidad y precisión (JCGM, 2012) puede ser afectada por efectos de matriz y por la heterogeneidad de la concentración de la proteína a lo largo de la planta, los resultados pueden depender del origen de la muestra (Ahmed, 2002; Querci et al., 2008). Además, existe la probabilidad de falsos negativos, especialmente en alimentos procesados, puesto que el método solamente detecta proteínas y estas pueden degradarse en el procesamiento de la muestra (Vijayakumar et al., 2009), limitando el uso de estos métodos.
Métodos basados en ADN
La PCR es uno de los métodos más usado para la detección de OGM en virtud de su especificidad, sensibilidad, y aplicabilidad a un amplio intervalo de materiales (Lusser, Parisi, Plan, & Rodríguez-cerezo, 2011); de acuerdo con la secuencia de interés los ensayos basados en PCR se pueden clasificar en diferentes niveles de especificidad (Manzanares-Palenzuela et al., 2015; Miraglia et al., 2004) (figura 2): Tamizaje, elemento-especifico, constructo-especifico y evento-específico. En el tamizaje se busca la detección de elementos reguladores como promotores o terminadores, el elemento-específico reconoce la secuencia de genes específicos especialmente los transgenes, responsables del nuevo fenotipo en el organismo huésped. En el constructo-específico se busca identificar regiones constituidas por la unión de un elemento regulador y un transgen. El evento-especifico reconoce una secuencia correspondiente a la unión del genoma del huésped con el elemento insertado, este último, constituye la base para la asignación de un identificador único para cada OGM (Taverniers et al., 2005).
La primera versión de la PCR conocida como punto final, utiliza electroforesis como método de detección, es usada en la determinación de la presencia o ausencia de elementos específicos o eventos de transformación genética; sin embargo, carece de la capacidad para proporcionar información cuantitativa confiable y precisa, debido a que la variación de la eficiencia aun entre reacciones de una misma amplificación impide que se establezca una correlación directa entre el producto final de amplificación y la cantidad inicial de ADN en la muestra (Anklam et al., 2002).
En la segunda versión denominada PCR en tiempo real (qPCR), la cuantificación en la fase logarítmica de la reacción se da a través de una señal fluorescente, ciclo a ciclo (Higuchi et al., 1993; Aguilera et al.,2014), por interpolación en una curva de calibración preparada con un patrón de referencia, preferiblemente certificado (MRC). En el caso particular de la cuantificación de OGM, el resultado puede ser expresado en términos del porcentaje del número de copias del transgen respecto al número de copias de un endogen, estable y altamente conservado entre las especies de un género en particular (Official Journal of the European Union, 2004). Se han desarrollado métodos en simplex (detección de una única secuencia por reacción) y multiplex (dos o más secuencias por reacción), dependiendo de los sistemas de detección y adquisición de datos disponibles. Los sistemas en multiplex ofrecen la posibilidad de aumentar el rendimiento en el procesamiento y análisis de las muestras, al tener la capacidad de evaluar diferentes eventos de transformación genética en una misma reacción (Park et al., 2015; Zhong et al., 2011); sin embargo, es necesario optimizar adecuadamente el método, para evitar la formación de productos de amplificación entre las parejas de primers empleados; lo cual constituye una de las principales limitantes respecto al número de targets que pueden ser evaluados empleando ésta metodología.
Nuevos métodos para detección de OGM
El aumento en el desarrollo y complejidad de nuevos OGM, implica un proceso más laborioso y costoso en cuanto a su detección (Novak et al., 2009). Los OGM que portan una sola modificación pueden ser identificados de forma sencilla por PCR convencional o tiempo real, sin embargo, la detección de OGM con modificaciones múltiples o apiladas, requiere del diseño de nuevas metodologías de alto rendimiento para su detección, algunas aplicables no solo en laboratorio sino en campo (puertos, cultivos, etc.) (Randhawa et al., 2013), razón por la cual se han desarrollado nuevos métodos que se han ido posicionando en el sector de análisis para OGM, entre los más importantes se encuentran amplificación isotérmica mediada por bucle (LAMP por sus siglas en inglés) (Notomi et al., 2000) y, PCR digital (dPCR) (Vogelstein & Kinzler, 1999), microarreglos (Doménech-Sánchez & Vilab, 2004) y secuenciación de nueva generación (NGS por sus siglas en inglés) (Pettersson et al., 2009).
Amplificación isotérmica mediada por bucle (LAMP)
Consiste en la amplificación del fragmento de interés a temperatura constante (60-65°C), se emplean 4 primers (2 internos y 2 externos) que deben ser diseñados para reconocer seis regiones diferentes en el ADN molde (los primers internos reconocen 2 regiones y los externos 1), la reacción inicia por uno de los primers internos quien porta un fragmento con una secuencia complementaria y un fragmento con una secuencia igual a la hebra molde respectivamente, en paralelo los primers externos inician el desplazamiento del fragmento amplificado por los primers internos (gracias a la Taq polime-rasa empleada quien tiene actividad polimerasa pero no 5'-3' exonucleasa) (Notomi et al., 2000). Posteriormente los primers internos generan un bucle en uno de los extremos del producto de amplificación, lo que permite generar una nueva hebra complementaria al fragmento previamente amplificado, pero unido a este a través del bucle. El ciclo se repite isotérmicamente generando estructuras en coliflor de diferentes tamaños.
La técnica presenta entre sus ventajas, alta especificidad al tener 4 primers por fragmento, tolerancia a inhibidores como polisacáridos (Zahradnik et al., 2014) y no requerimiento de equipos especializados, por lo que un baño termostatado o un bloque de calentamiento pueden servir para desarrollar la reacción, inclusive en campo (Fraiture et al., 2015). Su principal desventaja reside en el diseño de los 4 primers, que son la clave de su alta especificidad, así como la dificultad de realizar el ensayo en modo multiplex. Se han desarrollado ensayos por LAMP para algodón (Randhawa et al., 2013), maíz (Zahradnik et al., 2014), arroz (Chen et al., 2012), entre otros.
Microarreglos
Los microarreglos son un conjunto de sondas dispuestas por puntos, enlazadas a un soporte sólido en una disposición regular fija, donde cada punto contiene varias copias de la misma sonda, llegando a obtener entre 20000 y 60000 sondas por soporte, dependiendo el tipo de tecnología empleada en su síntesis (Doménech-Sánchez & Vilab, 2004). El microarreglo se híbrida con el ADN aislado de la muestra de interés, el cual previamente ha sido marcado. Los productos no hibridados son lavados, para permitir el análisis del microarreglo, donde se busca evaluar el grado de intensidad de fluorescencia para cada uno de los puntos sobre el soporte, indicando así la presencia de los eventos de transformación positivos en la muestra de interés.
La técnica de microarreglos presenta como ventaja principal el alto rendimiento, en cuanto al número de elementos específicos que pueden ser detectados en paralelo para muestras complejas de ADN en un solo ensayo, con una sensibilidad similar a qPCR (Turkec et al., 2016). Se ha reportado la detección de promotores terminadores y elementos transgénicos para la evaluación del contenido de OGM en maíz, soya, algodón, canola, entre otros (Miraglia et al., 2004; Shao et al., 2014;Turkec et al., 2016).
dPCR
Esta tercera versión de la PCR, se basa en diluir y dividir la muestra en pequeñas porciones denominadas particiones, a tal punto que cada reacción se realiza sobre una única molécula de ADN contenida en una partición. Posterior al proceso de amplificación, se cuentan las particiones positivas respecto del total de particiones y se establece su proporción de acuerdo con la distribución de Poisson (Pohl & Shih, 2004).
Existen dos plataformas disponibles: en chips, donde las particiones se dan en micro-cámaras dispuestas sobre un soporte sólido (chip) (Corbisier et al., 2015) y en gotas, donde las particiones están compuestas por microgotas, formadas a partir de una emulsión agua -aceite (Pinheiro et al., 2012). Un factor importante es que dado que la muestra se divide en un gran número de particiones (1.000 - 10.000.000) dependiendo el sistema de partición, la eficiencia de la reacción de amplificación se ve menos afectada por la presencia de inhibidores, lo cual tiene un impacto importante en la precisión del ensayo y en la incertidumbre, especialmente a bajas concentraciones (Milavec et al., 2013).
A diferencia de qPCR, la cuantificación es absoluta, por lo que no requiere un estándar o material de referencia (MR); esto solventa dos de los problemas más frecuentes en qPCR: a) disponibilidad de materiales de referencia conmutables (Vesper et al., 2007) con las muestras de estudio, lo cual implica igualdad de secuencias tanto de transgén como endogen, así como similitud en las matrices de estudio, y b) trazabilidad del resultado de medición, ya que si no se dispone de un MRC, no es posible establecer la cadena de trazabilidad metrológica del resultado de medición y por tanto su comparabilidad con otras mediciones en tiempo o lugares diferentes (Kamle & Ali, 2013).
Basado en sus características, la dPCR se ha postulado como un método potencialmente primario (Milton & Quinn, 2001) para la cuantificación de ácidos nucleicos (Bhat et al., 2010; Burke et al., 2013), ya que permite establecer una relación directa al sistema internacional de unidades (SI), garantizando así la trazabilidad metro-lógica de los resultados de medición asociados. Por lo tanto una de las principales aplicaciones está asociada con la asignación de valor en el proceso de producción de materiales de referencia para diversos usos, entre los que se encuentran la detección y cuantificación de virus (Bhat & Emslie, 2016; Haynes et al., 2013), bacterias (üong et al., 2014), OGM (Demeke et al., 2014; Ganceberg et al., 2007; Pérez & Acatzi, 2014); además como ítem de comparación en pruebas interlaboratorio (Burns et al., 2010; (Corbisier et al., 2010).
Secuenciación
La caracterización molecular de los eventos de modificación genética requiere conocer el número de copias y la localización de los fragmentos insertados, así como la secuencia de las regiones flanqueantes, importantes en la detección evento-específico. Frente a esto, las técnicas de NGS han abierto un nuevo camino en la identificación de OGM, ofreciendo la posibilidad de secuenciar de forma simultanea diferentes tipos de muestras (Pettersson et al., 2009). Existen dos alternativas: secuenciación parcial o secuenciación total. La primera es útil cuando se conoce a priori la secuencia de los fragmentos de interés especialmente de genomas complejos, como son las plantas. En este caso se puede iniciar el proceso a partir de una librería de productos de PCR (amplicones) o una librería genómica (Rohland & Reich, 2011). La librería de ADN es similar al tamizaje que se hace por qPCR, sin embargo, ofrece como valor adicional la secuencia de los productos o dianas de amplificación, generando un mayor grado de confianza sobre la identificación del OGM. En el segundo caso, el proceso implica la selección de las secuencias de interés a partir de las librerías genómicas, en este caso, la eficiencia del proceso de hibridación es el paso crucial. Posteriormente los fragmentos que contienen la secuencia de interés pueden ser secuenciados.
La segunda alternativa permite en principio caracterizar una muestra sin un conocimiento previo de su secuencia. En este caso la librería entera es secuenciada, luego por análisis bioinformáticos y con base en la secuencia de genomas de referencia e información de otros OGM, es posible establecer la identidad de la muestra de interés. Cuando no se dispone de genomas de referencia, es necesario ensamblar los fragmentos de novo, comparando todos los fragmentos entre si hasta generar la secuencia de interés por solapamiento de los fragmentos secuenciados (Kovalic et al., 2012), sin embargo, es una tarea difícil debido al tamaño de los genomas de las plantas en estudio, a su ploidia, heterocigocidad y a la presencia de diferentes tipos de modificaciones genéticas. Empleando esta técnica, se han caracterizado OGM como el arroz LLRICE 62 (Yang et al., 2013), maíz, soya (Song et al., 2014) y algodón (Liang et al., 2014).
Entre las desventajas de la técnica, se encuentra que los ensayos aún son costosos y laboriosos, requiriendo equipos especializados y personal altamente competente no solo a nivel técnico sino bioinformático. A pesar de esto, la NGS se perfila como una de las más poderosas técnicas para la caracterización de OGM (Liang et al., 2014)
MATERIALES DE REFERENCIA PARA OGM
Los materiales de referencia constituyen un eslabón clave en el establecimiento de la cadena de trazabilidad metrológica en las mediciones químicas y biológicas, y su uso contribuye a garantizar confiabilidad y comparabilidad en dichas mediciones (Panteghini, 2007). Son elementos indispensables en el proceso de validación de cualquier técnica analítica, así como en la evaluación del desempeño tanto de los métodos como de los laboratorios. El Vocabulario Internacional de Metrología los define como "materiales suficientemente homogéneos y estables, acompañados por la documentación emitida por un organismo autorizado, que proporciona uno o varios valores de propiedades especificadas, con incertidumbres y trazabilidad asociadas" (JCGM, 2012); lo que se traduce en que son materiales sólidos, líquidos o gaseosos, que cuentan con estudios de homogeneidad y estabilidad para las propiedades objeto de análisis, y para las vigencias o periodos establecidos en la validez del material; con una declaración del valor asignado y la incertidumbre de la(s) propiedad(es) certificadas, trazables al SI u otro sistema consensuado a nivel internacional (ejemplo, las unidades internacionales (IU por sus siglas en inglés) de la Organización Mundial de la Salud), producidas por una organización técnicamente competente; durante un proceso de certificación del material.
En el caso de los cultivos GM sirven como control positivo, calibrantes para el análisis cuantitativo y para el aseguramiento de la calidad de la medición. Estos deben ser independientes del método analítico y estar enfocados en el material origen o en los ingredientes base más que en el alimento terminado; por consiguiente, cada cultivo GM requiere un MR específico. Según la directiva EU 619/2011 (Oficial Journal of the European Union, 2011), para que un organismo genéticamente modificado pueda ser aprobado en la Comunidad Europea, el productor de la tecnología debe proporcionar controles tanto positivos como negativos para que los laboratorios de la EURL-GMFF (por sus siglas en ingles European Reference Laboratory for GM Food & Feed) puedan implementar y validar los métodos de medición, y producir y certificar los materiales de referencia correspondientes, para diferentes concentraciones. Además, establece que para alimentos que sean o contengan OGM deben estar disponibles los correspondientes materiales de referencia (Caprioara-Buda et al., 2012). Sin embargo, no todos los OGM que se comercializan en el mundo están autorizados en la Unión Europea, en consecuencia, no todos cuentan con materiales de referencia que permitan su análisis. Frente a esto, se han postulado algunas opciones como caracterizar materiales provenientes de productos modificados genéticamente, así como desarrollar plásmidos con las secuencias de interés para ser usados como MR; sin embargo, éstos carecen de trazabilidad metro-lógica y en consecuencia las mediciones realizadas para un mismo OGM, por diferentes laboratorios con dichos materiales, no son comparables (Charles et al., 2007; Miraglia et al., 2004; Panteghini, 2007). Algunos elementos críticos en la selección del material para el desarrollo de un MR están relacionados con la ploidia y la cigocidad, el grado de conmutabilidad entre el método de asignación de valor en el proceso de certificación y el método de rutina empleado por los laboratorios; los cuales deben ser previstos antes de iniciar un proceso de producción de un MR.
A nivel mundial hay dos productores de materiales de referencia para OGM, el primero es el Centro de Investigación Conjunto de la Unión Europea (JRC por sus siglas en ingles) que produce materiales de referencia certificados necesarios en la validación de métodos cuantitativos y en la diseminación de la trazabilidad metrológica. El segundo, es la Asociación Americana de Químicos del Petróleo (AOCS por sus siglas en inglés) que produce materiales de referencia no certificados cuyo uso se limita al aseguramiento de la precisión de las mediciones, es decir, no constituyen un eslabón en la cadena de trazabilidad metrológica.
Los materiales de referencia disponibles en la actualidad, basados en ADN, han sido producidos a partir de granos y semillas (como matriz completa), o ADN ge-nómico y plasmídico (Ahmed, 2002); los granos y semillas resultan ser los mejores materiales debido a que representan mejor las muestras de interés en cuanto a características como el efecto matriz, el cual tiene implicaciones importantes en el procesamiento de la muestra, especialmente en la extracción de ADN. Por su parte, el ADN genómico resulta ser un poco más fiel en términos del efecto matriz que genera la presencia del genoma completo sobre las secuencias de interés durante el proceso de amplificación por PCR. El ADN plas-mídico por otro lado presenta la ventaja de prepararse más fácilmente en grandes cantidades. En particular, el ADN genómico y el plasmídico han sido empleados como MR en la cuantificación de eventos de modificación genética basada en la razón del número de copias entre dos genes previamente seleccionados (Caprioara-Buda et al., 2012; Wang et al., 2013).
En cuanto al proceso de certificación, hasta 2004, los MR en matriz se certificaban en fracción en masa, posteriormente, por recomendación expresa de la Comunidad Europea, este tipo de materiales se comenzaron a certificar en términos del porcentaje de número de copias del transgen, respecto al número de copias de un endogen taxón específico de un genoma haploide (Official Journal of the European Union, 2004). El proceso de asignación de valor inicialmente se hizo por conceso entre un conjunto de laboratorios con competencia técnica demostrada para la medición en cuestión, por qPCR. Sin embargo, con el desarrollo de la dPCR, y su potencial como método primario, los procesos de certificación de materiales de referencia se están haciendo por esta técnica, donde el productor estima el valor asignado a partir de la combinación de los resultados obtenidos por diferentes plataformas de dPCR (chips y gotas), lo que contribuye a aumentar el número de materiales de referencia disponibles (Catalogo materiales de referencia JRC https://crm.jrc.ec.europa.eu/).
ESTADO ACTUAL EN COLOMBIA
En el año 2000 se autorizó el primer OGM en Colombia, el clavel de flor azul, para la producción de flor cortada (Instituto Colombiano Agropecuario, 2000). A 2016, se habían aprobado un total de 162 autorizaciones para la introducción de especies como algodón (Gossypium hirsutum), maíz (Zea mays), soya (Glycine max) y arroz (Oryza sativa), destinadas a siembra controlada o comercial en diferentes regiones geográficas, alimento o materia prima para la elaboración de alimentos para consumo humano o animal. De las 162 autorizaciones el 59% han sido para maíz, el 19% para algodón y el 13% para soya; el 9% restante se encuentra representado por cultivos como arroz (2 eventos), remolacha (2), trigo (1), claveles (3), rosas (2) y vacunas para animales (5) (figura 3); cifras que coinciden con la disponibilidad y proporción de eventos de modificación genética por cultivo a nivel mundial reportada en la base de datos de OCDE (Organización para la Cooperación y el Desarrollo Económicos, 2017), según la cual, para el maíz hay un total de 90 eventos de modificación genética, entre simples y combinados o Stacks, para algodón 37 y para soya 31.
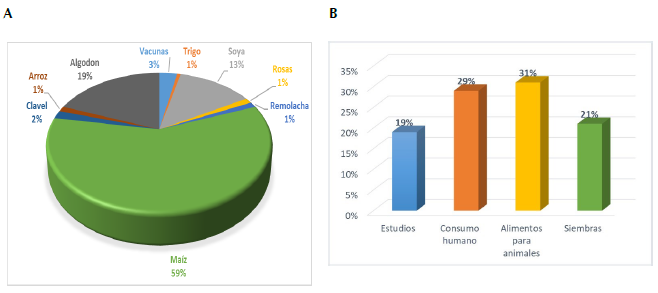
Figura 3 Relación de las autorizaciones por especie (A) y propósito (B), en porcentaje, respecto al total de autorizaciones en Colombia para 2016 (Elaborado a partir de BCH, 2016).
Las características de los cultivos GM que se han autorizado en el país son en su mayoría resistencia a insectos, en particular de los órdenes Coleóptera y Lepidóptera; tolerancia a herbicidas como el glifosato y el glufosinato de amonio; cambios en la calidad y contenido de metabolitos, como proteínas, aminoácidos (Maíz Mavera™ evento REN-00 038-3) o ácidos grasos insaturados (Soya MON-87769-7); y tolerancia al estrés abiótico, por ejemplo, tolerancia a la sequía (maíz MON 87460-4).
Entre los eventos aprobados se encuentran tanto de tipo individual como apilados; de los 38 eventos autorizados para maíz 18 son simples y 20 son apilados, en el caso de soya hay 9 eventos simples y 2 apilados y para algodón se reportan 5 eventos simples y 5 apilados; razón por la cual varios de éstos cultivos presentan hasta 4 características diferentes, un ejemplo es el maíz Herculex XTRA™ Roundup Ready™ 2 DAS-59122-7 x DAS-01507-1 x MON-00603-6, que reúne tanto resistencia a lepidópteros y coleópteros, como tolerancia al glifosato y glifosinato de amonio. En el caso de arroz, trigo y remolacha solo hay eventos simples.
Según el ranking mundial 2015 de países con mayor extensión sembrada con cultivos GM, Colombia ocupa el puesto 18 con 100.000 hectáreas, distribuidas así; 85.251 ha de maíz, 15.868 ha de algodón y 12 ha de clavel (James, 2015), distribuidos en 24 de los 32 departamentos del país. (http://www.agrobio.org/ Revisado 2017-05-21). Algunas de las especies que cuentan con autorización para siembra, consumo animal y/o humano, como la soya, el algodón, la remolacha azucarera, el arroz y el trigo, actualmente no se cultivan en el país, dado que las autorizaciones, especialmente de cultivos para consumo humano, responden a las dinámicas mundiales para la comercialización de granos más que al interés nacional (Castaño-Hernández, 2015).
Regulación OGM en Colombia
En Colombia, los OGM están regulados por la Ley 740 de 2002, por la cual se aprueba el Protocolo de Cartagena sobre Seguridad de la Biotecnología del Convenio sobre la Diversidad Biológica.
Por el decreto 4525 de 2005, el cual dicta el marco regulatorio de los Organismos Vivos Modificados (OVM) en el movimiento transfronterizo, el tránsito, la manipulación y la utilización de los OVM que puedan tener efectos adversos para el medio ambiente y la diversidad biológica, teniendo en cuenta los riesgos para la salud humana, la productividad y la producción agropecuaria. Adicionalmente, establece las autoridades competentes encargadas de autorizar el uso de OVM en el sector Agropecuario, Ambiental y en Salud humana, el tratamiento diferencial en medio confinado, la evaluación y gestión del riesgo, los Comités Técnicos Nacionales de Bioseguridad (CTN) de acuerdo con el uso de los OGM, el control y seguimiento en materia de bioseguridad, el acceso a la información, la posibilidad de tener disposiciones adicionales en términos de etiquetado o rotulado, la educación a usuarios y consumidores de esta tecnología y la participación del público en las solicitudes y en el proceso de adopción de decisiones (Ministerio de Agricultura y Desarrollo Rural, 2005).
La resolución 4254 de 2011, contempla las disposiciones relacionadas con el rotulado o etiquetado de alimentos derivados de OGM para consumo humano y la identificación de materias primas que los contengan (Ministerio de la Protección Social, 2011), además establece la obligatoriedad de declarar un alimento derivado de OGM cuando: a) no exista una equivalencia entre el alimento derivado de OGM y su contraparte convencional, b) producto de la modificación, se requiera condiciones de almacenamiento, preparación o cocción diferentes al del alimento convencional, c) por presencia de un alérgeno introducido por la modificación y, d) por cambio en las propiedades organolépticas respecto a su contraparte convencional.
La resolución 3168 de 2015 del Instituto Colombiano Agropecuario (ICA), la cual reglamenta y controla la producción, importación y exportación de semillas producto del mejoramiento genético para la comercialización y siembra en el país, así como el registro de las unidades de evaluación agronómica y/o unidades de investigación en fitomejoramiento; define dentro de las condiciones finales para la certificación de la semilla (como básica, registrada o certificada) límites de concentración de contenido de OGM (con las tecnologías aprobadas en el país), entre 0.5%/kg - 1%/kg para maíz, 0 %/kg - 1.5 %/kg para algodón y 0%/kg - 2%/kg para soya (Instituto Colombiano Agropecuario, 2015).
Capacidad de detección de OGM en Colombia
Después de la firma del protocolo de Cartagena, con el propósito principal de fortalecer la capacidad de detección de OGM en Colombia, gracias al apoyo del Banco Mundial a través del proyecto Global Environment Faility (GEF), se estableció el convenio para la creación del Laboratorio Central Interinstitucional de Detección y Monitoreo de OGM, el cual estaba conformado por el Instituto Nacional de Vigilancia de Medicamentos y Alimentos (INVIMA), el Instituto Nacional Agropecuario (ICA) y el Instituto de Investigación de Recursos Biológicos Alexander von Humboldt (IAvH), representando los tres sectores de interés en el país, a través de un convenio de cooperación científica vigente hasta 2015.
En la actualidad, la infraestructura para la detección y cuantificación de OGM en Colombia está centrada en 4 instituciones que difieren en objeto pero que unidas, abarcan los requerimientos para el aseguramiento de la calidad de las mediciones que se realizan en este campo.
El ICA cuenta con el laboratorio de detección y monito-reo de OGM localizado en Tibaitatá, éste ofrece el servicio de detección y está en capacidad para apoyar el esquema de certificación de semillas, la inspección en puertos y aeropuertos y el control de liberación intencional y no intencional en campo. Allí se han estandarizado una serie de métodos de detección de proteínas transgénicas y qPCR aplicables a tamizaje/screening o detección evento-específica, también se han implementado métodos de detección cuantitativa. Teniendo en cuenta la creciente necesidad de verificar los envíos de semilla y grano en el comercio internacional, se encuentran desarrollando estrategias de detección de alto rendimiento y bajo costo basados en PCR.
El IAvH ha desarrollado investigaciones para la realización de análisis técnicos asociados a la evaluación del riesgo desde el punto de vista ambiental, y del impacto de esta tecnología en parientes silvestres colombianos (Villafañe, 2010).
El Laboratorio de OGM del INVIMA, es el primer laboratorio acreditado con alcance flexible del país, para la detección cualitativa y cuantitativa de OGM por el método de q-PCR / Sondas de hibridación. Bajo esta acreditación, se cuenta con un total de 12 métodos de detección en formato Simplex, Multiplex y MultiTarget para la detección cualitativa de 8 elementos y 14 eventos específicos, y la cuantificación de 9 eventos. Gran parte de estos métodos se han desarrollado en respuesta a las necesidades de detección de OGM en alimentos (Instituto Nacional de Vigilancia de Medicamentos y Alimentos, 2017).
En 2011, se crea el Instituto Nacional de Metrología, cuyas funciones incluyen el apoyo a las actividades de control metrológico y la diseminación de mediciones trazables al SI, por lo cual en 2015 creó el Grupo de Metrología en Bioanálisis, el cual ha iniciado el proceso de desarrollo de la competencia técnica para la cuantificación de OGM; validando técnicas de detección por dPCR en formato simplex y multiplex, en el marco de comparaciones clave (como la CCQM-K86c) con otros institutos de metrología, lo cual le permitirá soportar el desarrollo de materiales de referencia y métodos de medición para OGM en el país.
Perspectivas de los cultivos GM en Colombia.
De acuerdo con la Organización de las Naciones Unidas para la Alimentación y la Agricultura (FAO), Colombia junto con Brasil, República Democrática del Congo, Angola, Sudán, Argentina y Bolivia, cuentan con las condiciones para incrementar la producción de alimentos de manera sostenible en los próximos años (FAO, 2003). En esa misma línea, el plan nacional de desarrollo 2014-2018, a través de los ejes de transformación del campo y crecimiento verde, ha considerado el sector agropecuario como uno de los sectores encargados de definir el rumbo de la economía en los próximos años (Departamento Nacional de Planeación, 2015).
Para 2016, en Colombia, el área total cultivada fue de 5.000.000 de hectáreas, predominando los cultivos agroindustriales (café, cacao, tabaco, caucho, algodón, soya, caña panelera) con 32.4% y los cereales (arroz, maíz amarillo, maíz blanco y otros) con 19.8% (DANE, 2016); sin embargo, esto solo constituye una pequeña fracción del total disponible, considerando que de 114.000.000 de hectáreas que constituyen el territorio Colombiano, 26.500.000 tienen vocación productiva, y de éstas, 11.000.000 poseen vocación agrícola (Pinilla, 2017), lo cual indica que su grado de aprovechamiento es menor del 50%.
Por otro lado, se han generado diferentes estrategias encaminadas a incrementar la productividad y competitividad del sector agrícola colombiano, así como a garantizar disponibilidad de alimentos para una población en aumento; entre las cuales puede citarse el Programa de Transformación Productiva (PTP),creado por el Ministerio de Comercio, Industria y Turismo, que busca fortalecer el sector agroindustria para mejorar la productividad, calidad, comercialización, y exportación de productos tales como cacao, café, aguacate Hass, lácteos, palma, frutas, alimentos procesados, acuícola y carne bovina, a mercados internacionales (L. Pérez, 2018); y el plan Colombia Siembra, orientado a aumentar en 1.000.000 las hectáreas sembradas en el país para 2018 especialmente con cultivos como maíz amarillo y blanco, soya, palma de aceite, cacao, forestales comerciales y algodón (Ministerio de Agricultura y Desarrollo Rural, 2016).
Aunque, ninguna de las estrategias anteriormente mencionadas plantea el uso de cultivos GM, la realidad es que éstos han ido ganando cada vez más fuerza. En 2012, el cultivo de maíz GM representaba aproximadamente el 12% del total de superficie sembrada con este grano (Fenalce, 2014), para 2015, esta cifra ascendió al 20% (DANE, 2016; James, 2016), indicando así que efectivamente pueden constituir una alternativa para el país con miras a desarrollar sus objetivos de aumentar su productividad y competitividad por un lado y garantizar la disponibilidad de alimentos por el otro.
A la fecha, varias universidades, centros de investigación y corporaciones están trabajando en el desarrollo de variedades genéticamente modificadas de yuca (Manihot esculenta): modificación de las características de producción de almidón, enriquecimiento en provitamina A, control de floración, producción de etanol; arroz (Oryza sativa): resistencia al virus de la hoja blanca; café (Coffea): resistencia a la broca; caña de azúcar (Saccharum officinarum): resistencia al virus del síndrome de la hoja amarilla; y papa (Solanum tuberosum): resistencia a tecia solanivora (James, 2016); por lo cual, cabe esperar que en algunos años se estén comercializando uno o varios de estos desarrollos. Adicionalmente, y de acuerdo con la tendencia mundial en la producción de cultivos GM, se espera que el comercio de estos aumente en los próximos años.
CONCLUSIONES
Los cultivos GM han mostrado ser una alternativa para aumentar la productividad y competitividad del campo y proporcionar alimentos con alto valor nutricional; frente a su evaluación, existen diferentes alternativas basadas tanto en análisis de proteínas como ADN, siendo este último el más empleado, a través de la técnica de PCR, tanto punto final como tiempo real, en modo simplex o múltiplex. Sin embargo, el aumento en el número tanto de especies modificadas como del tipo y número de modificaciones genéticas plantea nuevos desafíos en términos de las capacidades de detección de OGM, las estrategias a adoptar dependerán de las necesidades particulares, así como de la infraestructura y la información disponible: LAMP para la detección de OGM individuales in situ a costos más bajos, microarreglos para la detección simultanea de un gran número de targets GM, dPCR para la cuantificación absoluta, y NGS en aquellos casos en los que la secuencia de interés no se conoce o se conoce parcialmente, además de requerir su confirmación.
En Colombia, existen diferentes estrategias encaminadas a aumentar la productividad y competitividad nacional apalancado por el sector agrícola, y aunque ninguna menciona abiertamente el uso de cultivos GM, los procesos de investigación en cultivos de interés nacional, así como la oferta mundial, suponen un crecimiento de la extensión de los cultivos GM. Aunque la infraestructura disponible en el país para la detección y cuantificación de los cultivos GM autorizados es suficiente para garantizar la calidad de los resultados obtenidos, el desarrollo de nuevos cultivos GM, su complejidad y la variedad de modificaciones, así como el aumento en el comercio internacional; implican el fortalecimiento de las capacidades nacionales en cuanto al desarrollo e implementación de nuevos métodos de medición y herramientas para el aseguramiento de la calidad de los resultados, que permitan aumentar el rendimiento y reducir los costos asociados a la evaluación de los cultivos GM, garantizando el cumplimiento del marco regulatorio para OGM a nivel nacional y las condiciones del mercado internacional, brindando la confianza requerida al consumidor final.














