INTRODUCCIÓN
El Cáncer de Cuello Uterino (CCU) es el cuarto tipo de cáncer más frecuente en la población femenina a nivel mundial, con cerca de 528.000 nuevos casos y 266.000 muertes reportadas en el 2012. En Colombia ocupa el segundo tipo de cancer más frecuente en mujeres con una incidencia de 12.6%, seguido del cáncer de mama y por lo tanto convirtiéndolo en un problema de salud pública (GLOBOCAN, 2012).
El principal factor de riesgo para el desarrollo del CCU es la infección con Virus de Papiloma Humano (VPH), específicamente en aproximadamente el 10% de las mujeres donde el virus persiste en el tejido, lo que parece favorecerse por diversos factores como la inhibición funcional de células natural killer (NK), considerada como la primera línea de defensa inmunológica asociada con la lisis de células infectadas por virus y/o células tumorales. Asi, el VPH utiliza está estrategia para evadir la respuesta inmune innata, la cual a pesar de ser una de las principales barreras iníciales del sistema inmune, ha sido poco estudiada en la patología cervical (Textor et al., 2008).
Las celulas NK son pertenecientes a la inmunidad innata porque carecen de receptores específicos de antígenos en la superficie celular y participan en el control temprano de células estresadas. Su actividad citotóxica se realiza a través del reconocimiento y movilización de sus gránulos secretorios hacia el sitio de contacto con las células blanco, principalmente gránulos de granzima B y la perforina que junto con una tercera proteína de alto peso molecular denominada serglicina forman un complejo liberado en la zona de contacto entre las dos células y conduce a la formación de una vacuola de ph ácido que induce la activación de las perforinas ejerciendo los efectos desestabilizantes sobre la membrana de la vacuola endocítica y permitiendo que la granzima B acceda al citosol de la célula blanco activando al sistema de caspasas e induciendo la muerte por apoptosis (Lieberman, 2003; Barry & Bleackley, 2002; Vivier et al., 2011).
Con respecto a los receptores que posee la celulas NK para ejercer su función citotóxica, éstos se han clasificado en activadores, inhibidores, qumiotacticos y de adhesión. Dentro de los receptores activadores se destaca el NKG2D, el cual es un glicoproteina transmembrana de superficie tipo II que se expresa en forma dimerica junto con una molécula coadaptadora DAP10 en células NK, LT CD8+ TCR αβ y LT CD4+ TCR αβ y posee la capacidad unica de reconocer diversos ligandos sobreexpresados en las células tumorales (NKG2DLs) (Obeidy & Sharland, 2009; Huergo-Zapico et al., 2014).
Los ligandos del receptor NKG2D son moleculas similares a MHC-I que se dividen en dos familias: mic (a y b) y ulbp (1-6) que junto con la posible ausencia del MHC-I son indicadores de infección y/o transformación del DNA propia de un microambiente tumoral generando señales de activación e inducción de citotoxicidad (Textor et al., 2008).
En estudios con líneas celulares de cáncer cervical, se ha observado una disminución en la expresión de receptores activadores de NK como NKG2D, NKp30, NKp46 entre otros y de algunos ligandos como cd155, mica, micb - ulbp-2 (Choy & Phipps, 2010). En el 2002, Pende y colaboradores evaluaron la expresión de mica en un panel de líneas celulares que incluía: tres carcinomas de colón, dos carcinomas de ovario, un carcinoma hepático y la línea celular HeLa de cáncer cervical; como resultado encontraron que todos los tumores estudiados expresaban el ligando mica (Pende et al., 2002).
Recientemente Del Toro y colaboradores en el 2011 (Del Toro et al., 2011) describieron que mica y micb exhibían un patrón de expresión diferencial entre las líneas celulares infectadas con VPH, SiHa y HeLa, y las lineas celulares no infectadas con VPH, C33-A y HaCaT (línea celular de queratinocitos inmortalizados). Observaron que la expresión celular de mica en lineas infectadas era mayor que la expresión de micb y bajos niveles de expresión de mica en las líneas celulares no infectadas, por lo tanto se pudo evidenciar que por mayor grado de homología que compartan estos ligandos, al parecer hay una regulación diferencial de su expresión en la superficie celular en el cáncer de cuello uterino y proponen que una sobreexpresión sostenida de mica puede promover una baja modulación del receptor NKG2D en células NK de pacientes con cáncer de cuello uterino.
De igual forma se describen otros mecanismos de regulación de estos ligandos como el clivaje por proteasas de los ligandos de NKG2D de la superficie celular para evitar el reconocimiento por parte de la célula NK, tales como metaloproteinasas de matriz y desintegrinas metaloproteinasas; particularmente mmp14, adam 10 y adam17, que se han relacionado con el clivaje de mica y micb de la superficie de células tumorales (Doubrovina et al., 2003; Jiménez et al., 2012). Los ligandos liberados se pueden detectar en el suero de pacientes con diferentes tipos de cáncer donde se han encontrado grandes cantidades de mica y micb solubles comparado con el suero de pacientes sanos (Salih et al., 2002; Boutet et al., 2009).
Por lo anterior nosotros evaluamos por primera vez la expresión génica de todos los ligandos descritos (mica, micb y ulbp 1-6) para el receptor activador NKG2D en humano, además de las metaloproteinasas adam10, adam17 y mmp14 involucradas; en las lineas celulares de referencia para el cancer cervical como HeLa positiva para el tipo viral VPH 18 y C33A negativa para VPH.
METODOLOGÍA
Se utilizaron lineas celulares de referencia para cancer cervical: HeLa (positiva para VPH 18) y C33 A (negativa para VPH), las cuales fueron mantenidas en cultivo de acuerdo a las recomendaciones de la ATCC.
Extracción de RNA
A partir de las líneas celulares se realizó la extracción del RNA bajo las indicaciones y recomendaciones del fabricante del Kit RNeasy Quiagen®. Una vez obtenido el RNA fue cuantificado por espectrofotometría utilizando NanoDrop®ND-100 y de acuerdo a su calidad fue tratado bajo las indicaciones y recomendaciones del fabricante del Kit de DNasa I de Invitrogen®.
Síntesis de cDNA
Posteriormente, se realizó la retrotranscripción de la muestra de RNA para obtener cDNA bajo las indicaciones y recomendaciones del fabricante del Kit SuperScript® III First-Strand Synthesis System (Invitrogen), donde se adiciono al RNA previamente tratado con DNAasa 1μl de dNTP (10mM), se incubo a 65°C por 5 minutos e inmediatamente se coloco sobre hielo por un minuto. Luego se adiciono 2μl de RT buffer 10X, 4μl cloruro de magnesio (25mM), 2μl de DTT (0,1M), 1μl RNAaseOut (40U/μl) y 1μl de SperScript III RT (200U/μl); se incubo a 50°C por 50 minutos seguido de una temperatura a 85°C por cinco minutos e inmediatamente se coloco en hielo durante un minuto. Finalmente se adiciono 1μl de RNAase H (2U/μl) seguido de una incubación de 20 minutos a 37°C.
El cDNA se cuantifico espectrofotométricamente por NanoDrop®ND-100 y se realizaron alícuotas de trabajo de 125ng/μl las cuales se almacenaron a -70°C para su uso posterior.
Diseño de primers
En la tabla 1 se resume las secuencias de los primers para el cual se utilizó el software Primer Blast y Primer 3 IDT y corroborados mediante alineamiento con BLAST para comprobar que los iniciadores diseñados correspondían a los genes de interés, además de las condiciones de estandarización para los ensayos de RT-PCR de cada uno de los genes del estudio.
Tabla 1 Secuencias y condiciones de estandarización de primers de NKG2DL - ulbps 1-6, mic-a, mic-b; metaloproteinasas (adam 17, adam 10 y mmp14) y gen house keeping de β-actina.
| Gen | Secuencia primer (5’-3’) | Temperatura (°C) | Tamaño del amplímero (pb) |
|---|---|---|---|
| ulbp- 1 | F: GCAGAGGATCTTGGCAGTTC R: ATGAGAAGGCTCCAGGGACT | 62°C | 298 pb |
| ulbp- 2 | F: CAGAGCAACTGCGTGACATT R: CATGCCCATCAAGAAGTCCT | 62°C | 299 pb |
| ulbp- 3 | F: TGGAACTGGCTGACACTGAG R: GCCTCTTCTTCCTGTGCATC | 62°C | 306 pb |
| ulbp- 4 | F: TGGAACTGGCTGACACTGAG R: GCCTCTTCTTCCTGTGCATC | 62°C | 93 pb |
| ulbp- 5 | F: CCATGTCCTCAGGCACAGC R: CAGGGAACCATCAAGATATGG | 64°C | 375 pb |
| ulbp-6 | F: CGCCATCCCAGCTTTGCTTC R: TTTTCATCCACCTGGCCTTGA | 60°C | 165 pb |
| mic-a | F: GGGAACGGAAAGGACCTCAG R: GGGGCATTGTCCATTCCTCA | 60°C | 190 pb |
| mic-b | F: GATAAGCGGTCGCTGAGCGGG R: AGCCCCATGGCCCCTACGTC | 62°C | 120 pb |
| adam17 | F: TTGTGTGGTTTGGCCCTTCT R: CTGCTTTTGCACCACAGGTC | 60°C | 116 pb |
| adam10 | F: CGAACTCTGCCATTTCACTC R: ACTTCCTCTACACCAGTCAT | 62°C | 140 pb |
| mmp14 | F: CGAACTCTGCCATTTCACTC R: ACTTCCTCTACACCAGTCAT | 60°C | 124 pb |
| β-actina | F: ATTGCCGACAGGATGCAGA R:GAGTACTTGCGCTCAGGAGGA | 60°C | 89 pb |
PCR en tiempo real (RT-PCR)
Los ensayos de qRT-PCR para las líneas celulares de cáncer de cérvix se realizaron bajo las siguientes condiciones:
En placas de real time del equipo Light Cycler® 480 System, por cada gen se preparó la mix 1X para cada línea celular por triplicado con 0.2 μM de los iniciadores tanto forward como reverse correspondiente, 5.0 μl de solución Master Syber Green, 2 μl de cDNA (250 ng /μl) y se completó con agua desionizada libre de RNAasa hasta un volumen final de 10 μl. En el equipo Light Cycler® 480 System se procesaron las reacciones bajo el programa establecido así: Un ciclo de denaturación a 95°C por 4 minutos; treinta cinco (35) ciclos de amplificación cada uno de 95°C por 10 segundos; la temperatura descrita para cada tipo de gen en la tabla 1 (60°C, 62°C o 64°C) por 20 segundos y 72°C por 1 segundo y finalmente un paso de enfriamiento a 4°C por 10 segundos. Como control negativo fue utilizado agua desionizada libre de RNAasa en las mismas condiciones mencionadas.
Para la optimización de la qRT-PCR se obtuvieron las eficiencias para cada uno de los genes en estudio, realizando curvas estandar utilizando diluciones seriadas de DNA de la linea celular HeLa y aplicando la formula E= 10 (-1/pendiente), dicha eficiencia debia estar entre 1.7 - 2.0 siendo indicativo de adecuadas condiciones en la corrida de las reacciones (Yun et al., 2006).
El análisis de la expresión se realizó utilizando el método de cuantificación relativa donde cada muestra fue normalizada con la expresión del gen de referencia β-actina. La expresión relativa de los genes de interés fue calculada y fundamentada en el método 2-ΔCT teniendo en cuenta las eficiencias obtenidas para cada set de iniciadores como factor de corrección, por lo que el cálculo se realizó basado en la siguiente fórmula: (E actina) * CP muestra/ (E gen de interés) *CP muestra (Pffafl, 2001).
Análisis estadístico
El análisis estadístico de los datos obtenidos de tiempo real fue realizado utilizando el software Graph PadPrism 6, en el cual se realizaron gráficos de dispersión con media y desviación estándar DS. Para la significancia estadística, se realizó la prueba t-test. Un p valor < 0,05 fue considerado como significante.
RESULTADOS
La expresión relativa de los genes de interés ulbp (1-6), mica, micb y metaloproteinasas (mmp14, adam10 y adam17), se realizó a través qRT-PCR por triplicado en las lineas celulares: HeLa (positiva para VPH tipo 18) y C33- A (negativo para VPH). Se obtuvieron valores óptimos de eficiencia para todos los primers de los genes utilizados incluyendo nuestro gen constitutivo de β- actina.
Expresión de los ligandos de NKG2D
Como se observa en las figura 1, la expresión genica relativa de los ligandos ulbp-1 y ulbp-2 en las líneas celulares presento una diferencia significativa con un valor p de 0.01 y 0.04 respectivamente, siendo la línea C33 la que obtuvo un mayor nivel de expresión de estos dos ligandos respecto a HeLa donde los niveles de expresión génica son prácticamente nulos. Para el ligando ulbp-3 su expresión presento también diferencias significativas con un valor p de 0.03 siendo igualmente la línea C-33A la que mayores niveles presentó, mientras que la línea celular HeLa presentó menor expresión de éste ligando. Para el ligando de ulbp-4, no se observó expresión en ninguna de líneas celulares evaluadas. La expresión génica de los ligandos ulbp-5 y ulbp-6 fue ligeramente mayor en la línea celular C-33A, con respecto a la HeLa, pero éstas diferencias no fueron estadísticamente significativas.
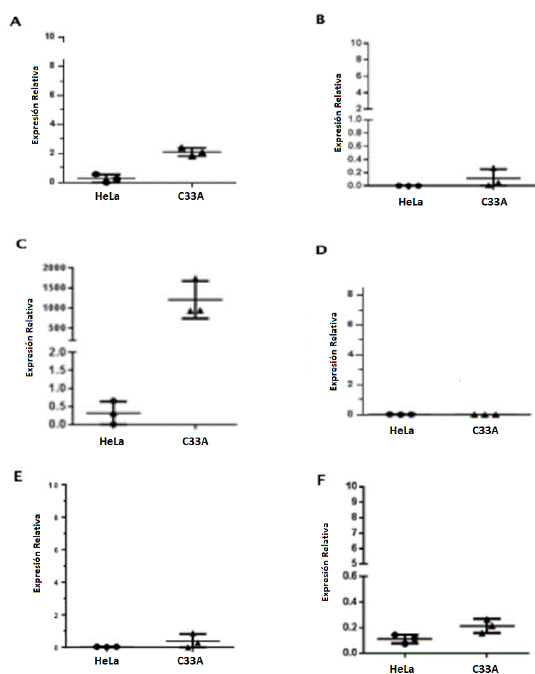
Figura 1 Expresión del ligandos ulbp (1-6) de NKG2D en líneas celulares de CCU: HeLa: Positivo para VPH -18 y C33A: Negativo para VPH. A) ulbp-1 p=0.01, B) ulbp-2 p=0.04, C) ulbp-3 p=0.03, D) ulbp-4 p= 0.81 E) ulbp-5 p= 0.32 F) ulbp-6 p=0.14. Se considera valores estadísticamente significativos p = <0.05.
En la figura 2, se observa que la expresión génica de mica presenta diferencias estadísticamente significativas con un valor de p de 0,02 siendo la línea celular C-33A la de mayor nivel de expresión, en contraste la línea celular HeLa dicha expresión fue nula. En relación al ligando micb su expresión fue baja en las dos líneas celulares evaluadas, sin embargo, se observa un leve incremento en la línea celular C-33A.
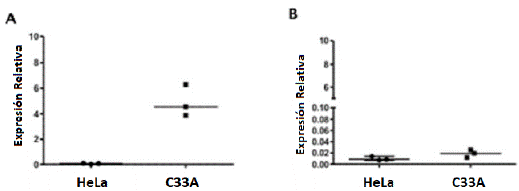
Figura 2 Expresión del ligandos mica y micb de NKG2D en líneas celulares de CCU: HeLa: Positivo para VPH -18 y C33: Negativo para VPH. A) mica p=0.02 B) micb p=0.19. Se considera valores estadísticamente significativos p = <0.05.
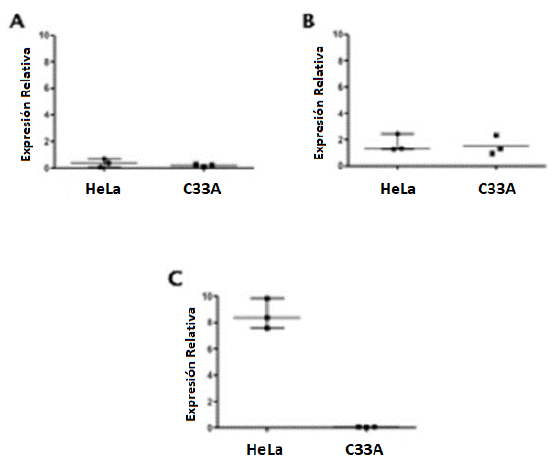
Finalmente, para las desintegrinas y metaloproteinasas, observamos en la figura 3, la expresión relativa para adam10 y adam17 fue similar en las dos líneas celulares evaluadas, sin embargo, la mmp14, mostró diferencias estadísticamente significativas con un p=0.006, con un incremento en su expresión en la línea celular HeLa positiva para VHP18 y una expresión nula en la línea celular C-33A negativa para VPH.
DISCUSIÓN
La mayoría de ulbp no se expresan en tejido normal a diferencia de los ligandos mica y micb los cuales son expresados en células gastrointestinales por acción de flora bacteriana. Para el caso especifico de la patología cervical, el VPH ocasiona la transformación del DNA en la célula blanco induciendo la disminución de la expresión de moléculas MHC-I y la expresión de ligandos ulbp y mic que a su vez activa el receptor NKG2D de las células NK promoviendo la lisis celular; es así como se ha observado la expresión de todos estos los ligandos mencionados en líneas de cáncer de cérvix tanto positivas (HeLa) y negativas (C33) para VPH pero con patrones totalmente diferentes.
Tabla 2 Eficiencias de ULBPs (1 al 6), mica, micb, adam10, Adam 17 y mmp14 utilizando DNA de línea celular HeLa.
| Gen | Eficiencia Obtenida |
|---|---|
| ulbp- 1 | 1.797 |
| ulbp- 2 | 1.989 |
| ulbp- 3 | 2.013 |
| ulbp- 4 | 1.915 |
| ulbp- 5 | 1.769 |
| ulbp-6 | 1.816 |
| mic-a | 1.781 |
| mic-b | 1.889 |
| adam17 | 1.762 |
| adam10 | 1.891 |
| mmp14 | 1.848 |
| β-actina | 2.301 |
Eficiencia recomendada: 1.70 - 2.10 (Yun et al., 2006)
La expresión de ulbp-1; ulbp-2 ulbp-3 y mica presentaron diferencias estadísticamente significativa (p=0.01; p=0.04; p=0.003; p= 0.02 respectivamente) entre las dos líneas celulares (HeLa y C33) pero destacándose su expresión en la línea celular C33 la cual no contiene genoma de VPH, contrario a lo presentado en el estudio realizado por Jiménez y colaboradores en el 2012 (Jiménez et al., 2012) y Textor y colaboradores en el 2008 (Textor et al., 2008) en el que se destaca la expresión genica del ligando ulbp-2 en SiHa (línea celular asociada con VPH tipo 16) y los reportados por Del Toro y colaboradores en el 2011 (Del Toro et al., 2011) donde reportaban una baja expresión de mica en la línea negativa para VPH. En cuanto a micb no se observaron diferencias de la expresión del gen en las líneas celulares empleadas las cuales fueron menores a las reportadas Del Toro y colaboradores (Del Toro et al., 2011), quienes encontraron una mayor expresión en la línea C-33 que en HeLa. Sin embargo, estudios recientes de McCarthy y colaboradores en el 2018 (McCarthy et al., 2018), demuestran como en líneas celulares tumorales de cáncer cervical (HeLa), fribrosarcoma (HT1080) y cáncer de seno (MCF7) al ser tratadas con glucosa en distintas concentraciones (2,5 - 25mM) incrementan la expresión de mica y micb, esto se relaciona con el estudio realizado por Jung y colaboradores en el 2017 (Jung et al., 2017), donde en líneas celulares positivas y negativas para VPH en cáncer de cabeza y cuello, demuestran una regulación diferencial en el metabolismo anaeróbico de la glucosa y la fosforilación oxidativa mitocondrial, estos autores observan que las células positivas para VPH y con deprivación de glucosa durante 48 horas, sobreviven en un 90% ó mas, mientras que las células negativas para el virus únicamente sobreviven un 40 a 60%, por otra parte, alta producción de lactato y bajo consumo de oxígeno es característico de las células VPH negativas contrario a las VPH positivas. Estos hallazgos pueden explicar como por las diferencias metobólicas entre las líneas celulares positivas y negativas para el virus, pueden influenciar la expresión de mica y micb, encontrándose en menor proporción en células HeLa que en C33.
Los ligandos ulbp-4, ulbp-5 y ulbp-6 no presentaron diferencias estadísticamente significativas (p= 0.92, p=0.72 y p=0.64 respectivamente), contrario al estudio de Eagle y colaboradores en el 2009 (Eagle et al., 2009) donde para la expresion del gen ulbp-6, esté se caracteriza por ser restringido para líneas celulares positivas para VPH y derivadas de cáncer de cérvix (SiHa, CaSki, HeLa) al compararlo con 10 tipos diferentes de líneas celulares tumorales y por lo tanto es considerado como un indicador de la respuesta inmune frente al VPH.
Diversos estudios describen el perfil de expresión de los ligandos del receptor NKG2D en otras patologías malignas como el cáncer de seno y cáncer de ovario presenta un comportamiento totalmente diferente como por ejemplo incrementos estadísticamente significativos en la expresión de mica, micb, ulbp-4 y ulbp-2 siendo este ultimo de mal pronóstico para cáncer de ovario (Huergo-Zapico et al., 2014; de Krujif et al., 2012).
Es importante aclarar que en la expresión de las proteínas ulbp se han descrito diferentes mecanismos que intervienen en su modulación como el clivaje de algunas isoformas solubles de ulbp2- ulbp-4 que se unen al receptor NKG2D, realizando una alteración en la expresión del receptor NKG2D favoreciendo una disfunción de las NK. Otro mecanismo es el contacto persistente entre células NK y células que expresan NKLs el cual modulan la expresión de ligandos como el ulbp-3 o del receptor NKG2D, como por ejemplo, al realizar cocultivos entre células de cérvix SiHa (VPH 16) o HeLa (VPH 18) con NK se disminuye la actividad citotóxica, contrario a lo ocurrido con línea celular C33 (VPH negativo) donde se incrementa la actividad citotóxica.
De igual forma, se han descrito mecanismos de regulación de estos ligandos que involucran procesos transcripcionales y postranscripcionales, como por ejemplo la capacidad de las oncoproteinas E6 y E7 de VPH-AR de interferir en la unión de ciertas inmunoproteinas como factores de transcripción (IRF1, IRF3) implicados en la respuesta inmune innata, los cuales podrían realizar la retención intracelular de esos ligandos evitando su expresión a la superficie tal como sucede en infecciones con citomegalovirus (CMV) y herpes virus (Jiménez et al., 2012). Por otro lado, algunos procesos de metilación del DNA se correlacionan con la ausencia de transcripción de ligandos del receptor NKG2D, constituyendo un mecanismo de evasión de las células tumorales principalmente en leucemia mieloide aguda (LMA) (Raneros et al., 2015).
Teniendo en cuanta lo anterior podría sugerirse que la presencia o no del virus (VPH) interviene de forma directa en la expresión de los diferentes ligandos tanto ulbp y mica y micb en líneas celulares de cérvix.
Otra característica descrita para los ligandos de NKG2D, son los polimorfismos presentes principalmente en mica y micb, para el caso de mica se han descrito 68 alelos y para micb 25, los cuales se distribuyen de manera diferente entre las distintas poblaciones como caucásicos, japoneses, amerindios entre otros. Algunos de esos polimorfismos se han asociado a distintas patologías como por ejemplo para artritis reumatoide el polimorfismo MICB*004, enfermedad celiaca MICA-A5.1 y MICB*0106, artritis psoriatica MICA-A9 y enfermedad de Behcet´s MICA*009 entre otras (Mendoza, 2007). En cuanto a estudios de relación de polimorfismos de mica ó micb asociados a cáncer cervical, Chen y colaboradores en el 2005, no encontraron ninguna asociación en población de Taiwan (Chen et al., 2005) y más recientemente Chen y colaboradores pero en el año 2013 (Chen et al., 2013), en un estudio de GWAS describe asociación a mayor riesgo de desarrollar cáncer cervical en personas que presentaran el alelo A5.1 del gen mica.
Se ha determinado que uno de los posibles mecanismos del tumor para evadir el sistema inmune es clivar los ligandos como mica y micb para evitar ser reconocido por el receptor NKG2D; algunas de las proteínas que al parecer están involucradas en este proceso hacen parte de las metaloproteinasas de matriz y desintegrinas, como es el caso de adam10, adam17 y mmp14. Además, la unión del NKG2D a los ligandos solubles promueve su internalización y posterior degradación, como consecuencia se obtiene una disminución de la expresión del receptor y por lo tanto una menor actividad citotóxica de las células NK (Chen et al., 2005; Chen et al., 2013).
En una línea celular de carcinoma pancreático se encontró que solo la baja regulación de adam10 y adam17 inhibía el clivaje de MICA mientras que ADAM17 se identificó como el principal responsable del clivaje de esta proteína en otras líneas celulares como es el caso de la línea MDA-MB-231 (cáncer de seno). En el clivaje de micb tanto adam10 como adam17 son igualmente efectivas en la línea PancTu-1 (cáncer de páncreas), mientras que adam17 fue más efectiva en otras líneas como la de cáncer de próstata PC-3. De lo anterior se podría llegar a concluir que el papel que juegan estas proteínas en el clivaje es mas una cuestión selectiva por parte de la célula tumoral que una especificidad de sustrato (Chitadze et al., 2013; Waldhauer et al., 2008).
Para el caso de adam17, no se encontraron diferencias estadísticamente significativas entre las líneas C-33A y HeLa; además se observo una expresión baja en las dos líneas celulares empleadas. Estos resultados son contrarios a lo reportado por Xu y colaboradores en el 2013, quienes encontraron altos niveles de la proteína en tejidos con CCU en comparación con tejidos adyacentes no cancerígenos y una alta expresión de ésta se ha asociado con un mal pronóstico en pacientes con CCU (Xu et al., 2013), por lo que podría sugerirse que podría llegar a ser un buen marcador de pronóstico, por lo tanto, era de esperarse que un modelo de CCU en líneas celulares se encontrará una alta expresión de adam17.
Según nuestros resultados la expresion del gen mmp14 muestra diferencias de expresión entre las líneas celulares, siendo mayor expresión en la línea celular HeLa. Sheu y colaboradores en el 2003 (Sheu et al., 2003), encontraron que queratinocitos primarios humanos que expresaban las oncoproteínas virales exhibían una sobre-expresión de mmp9 asociado, probablemente, a que las proteínas E6 y E7 mediaban la baja regulación de inhibidores de mmp como RECK y TIMP-2, sin embargo, las otras metaloproteinasas no presentaron cambios.
CONCLUSIONES
En los análisis de expresión génica de ulbps y mic-a en líneas celulares de cáncer de cérvix que fueron estadísticamente significativos, similar al predomino de la expresión de la metaloproteinasa mmp14 en la línea celular positiva para VPH, por lo que se sugiere una posible estrecha relación entre la patología cervical asociada a la infección por el VPH y la respuesta inmune innata a expensas del receptor NKG2D de las celulas NK.