INTRODUCCIÓN
La diversidad bacteriana es un tema de trascendencia, puesto que cumple funciones relacionadas con la nutrición al intervenir directamente en los ciclos biogeoquímicos, la producción de sideróforos (Camelo et al., 2011), la promoción del crecimiento de las plantas mediante la producción de sustancias orgánicas (Beltrán 2014; Ortiz et al., 2016) y el control biológico sobre organismos patógenos (Compant, et al., 2005).
Investigaciones realizadas, sugieren que el uso de abonos orgánicos compostados producen cambios en la diversidad bacteriana asociada a la rizósfera de diferentes cultivos, reflejados en la abundancia y estructura (Julca 2006; Fujino et al., 2008).
Es de resaltar que el estudio de la relación entre las alteraciones que se dan al interior de las comunidades bacterianas, el manejo de los cultivos y las características de los ambientes particulares como suelo y agua entre otros, se han facilitado con el estudio de los metagenomas (Cortés et al., 2014), los cuales parten de la extracción del ADN de las muestras, el clonado de fragmentos de ADN, la amplificación de secuencias del gen ADNr 16S con cebadores universales, y finalizan con la secuenciación y asignación taxonómica para su respectivo análisis mediante indicadores de diversidad alpha, beta y gama (Montenegro, 2012).
En Colombia no existen estudios metagenómicos relacionados con la rizósfera de morera (Morus indica), planta que es usada como único alimento para la cría del gusano de seda Bombix mori, adicionalmente tiene la capacidad de crecer bien en zonas templadas y tropicales, es muy extractora de nutrientes, de ahí que su producción dependa de la disponibilidad en el suelo o del suministro (Pentón et al., 2016), sin embargo, la aplicación de fuentes orgánicas compostadas no siempre han causado beneficio a este cultivo especialmente cuando se usa compost de mala calidad, (Bello et al., 2011).
En Colombia, y para el caso específico del Departamento del Cauca no existen estudios que evalúen tal efecto sobre el rendimiento de la morera, ni tampoco sobre la diversidad de las bacterias rizosféricas, razón por la cual se planteó como objetivo para esta investigación, indagar el efecto que causa el uso de fuentes orgánicas compostadas a partir de residuos de sericultura sobre la diversidad genética bacteriana asociada a la rizosfera de morera.
MATERIALES Y MÉTODOS
Localización y caracterización de la zona de estudio
Esta investigación se realizó en la unidad productiva denominada Parcela 17, ubicada en la vereda Clarete Alto, municipio de Popayán (Dpto. del Cauca), a 2056 msnm, con coordenadas 2°29.38¨´N - 76°30.26¨ W, 18°C promedio anual de temperatura, precipitación entre 1850 y 2300 mm y la humedad relativa entre 68 y 82 % (Paz y Ospina, 2014), propiedad de Carlos Gurrute, Sericultor con más de diez años de antigüedad.
Diseño experimental y descripción de parcelas experimentales
El suelo experimental derivado de cenizas volcánicas se caracterizó por ser franco arenoso; con pH de 5.0, contenido de Materia orgánica de 5%, disponibilidad baja de nitrógeno y fosforo, y alta de potasio. En cuanto al manejo, durante el último año solo se le había aplicado compost elaborado con residuos de cosecha y cría de gusano en dosis de 0,5 Kg.m-2 por corte (cada 120 días).
Se empleó un compost elaborado por los mismos investigadores con residuos provenientes de la cría del gusano de seda Bombix mori, en La Parcela 17 (lugar de investigación), mediante el método de pila abierta bajo cobertura y condiciones controladas, monitoreado por 170 días y evaluado en sus propiedades físicas, químicas y biológicas, calificándose según la norma ICONTEC 5167 apto para su aplicación en el suelo, cuya diversidad bacteriana identificada mediante ADN metagenómico, a partir de fragmentos de 200 a 300 pb de la región hipervariable V4 del gen 16S y secuenciación por el método ILLUMINA Miseq, se determinó que estaba dominada por los filos Proteobacteria, Firmicutes y Actinobacteria, y los géneros Agromyces, Pseudomonas, Candidatus solibacter, Candidatus korinebacter.
Para el desarrollo del ensayo, se podaron las plantas de morera y se establecieron 12 parcelas experimentales, utilizando un diseño en bloques completos al azar, con 4 tratamientos conformados por un control y 3 bloques. Cada una de ellas compuesta por 16 plantas, para un total de 192 plantas de morera de 7 años de edad sembradas a distancia de 0,4 m entre plantas y 0,8 m entre surcos y dispuestas en un área experimental efectiva de 5,5 m2.
Los tratamientos dispuestos para el experimento fueron los siguientes:
T0 (Control) Suelo cultivado con morera sin ninguna aplicación.
T1 Suelo cultivado + aplicación de compost en dosis de 0,25 Kg.m-2 por corte (50% menos de la dosis aplicada normalmente por sericultores).
T2 Suelo cultivado + aplicación de compost en dosis 0,5 Kg.m-2 por corte (dosis aplicada normalmente por sericultores).
T3 Suelo cultivado + aplicación de compost en dosis de 1,0 Kg.m-2 por corte (100% más de la dosis aplicada normalmente por sericultores).
El compost fue aplicado alrededor del tallo de cada planta en un área de 40x40 cm, en las dosis que indica cada tratamiento, e incorporado a profundidad entre 0 y 5cm. En cuanto a los muestreos al suelo se realizaron cuatro; antes de la aplicación del compost y a los 5, 10 y 90 días después de aplicado. Para cada muestreo se tuvo en cuenta las cuatro plantas internas de cada parcela con el fin de eliminar el efecto de borde, tomándose 5 g de suelo rizosférico en cada planta a profundidad ya mencionada. De cada parcela se conformó una muestra de 20 g y se transportaron en nevera portátil a 2°C y en lapso de tiempo menor a 24 horas fueron preparadas para la extracción del ADN en SEQMATIC y así evitar cambios en composición microbiana de la muestra (Neher et al., 2013).
Extracción y secuenciación del ADN
La extracción de ADN se realizó usando el kit comercial PowerSoil™ ADN Isolation (Mo Bio Laboratories Inc., California, USA) a partir 48 muestras de suelo, de cada muestra se tomó 0.25 g de diferentes partes. El procedimiento para aislar ADN microbiano genómico se realizó según instrucciones recomendadas por MoBio Laboratories para suelos (Storey et al., 2014; De Gannes et al., 2013). Para observar la integridad del ADN extraído de todas las muestras y cuantificarlo una alícuota de 2μl de cada muestra se sometió a electroforesis (80 voltios) en gel de agarosa al 1 % en tampón BE 0.5 (Montenegro, 2012), durante 30 minutos. Por último para determinar la concentración y pureza del ADN extraído en cada muestra, se utilizó el espectrofotómetro ND Colibrí de BERTHLOD.
El ADN total extraído del suelo fue enviado a SEQMATIC con sede en Fremont California en Estados Unidos, donde se amplificó fragmentos de 200 a 300 pb de la región hipervariable V4 del gen 16S, por presentar alta resolución para distinguir géneros (Mureau et al., 2014; Decelle et al., 2014), sobre los cuales se prepararon bibliotecas utilizando como cebadores el 515F (GTGCCAGCMGCCGCGGTAA) y 806R (GGACTACHVGGGTWTCTAAT), generadas con protocolo PCR, cuya configuración de las reacciones fue: 95°C por 10 min, 95°C por 30 segundos, 60°C por 30 segundos, 72°C por 90 segundos y 72°C por 5 minutos. El producto de la PCR en cada muestra se limpió con perlas AMPure XP y etiquetado con códigos de barras y secuenciado por el método ILLUMINA Miseq.
La información generada por SEQMATIC se analizó a través del programa Galaxy Project versión 17.01, para mejorar la calidad de las secuencias, eliminando de cada muestra secuencias de longitud menores de 50pb y mayores de 400pb. Posteriormente se reagruparon las fracciones restantes de 20 pares de nucleótidos y con el mismo programa fueron solapadas las secuencias F y R. Posteriormente se seleccionaron aquellas muestras con más de 9000 secuencias solapadas para la nueva asignación taxonómica de OTUS mediante el programa QIIME versión libre.
Variables evaluadas
Para el análisis de las secuencias se cuantificó el número de OTUS, la abundancia en función de la frecuencia relativa de cada unidad taxonómica asignada, las diversidades alpha y beta, y la variación en el número de unidades taxonómicas identificadas por efecto de los tratamientos.
La diversidad alpha representa la riqueza de una comunidad estimada a nivel local (García et al., 2016) siendo definida dentro de cada parcela experimental, y medida a partir de los índices de Simpson, inverso de Simpson y Shannon. La diversidad beta representa el grado de similitud o disimilitud entre las comunidades obtenidas en un área mayor (García et al., 2016), esta se obtuvo a partir la comparación de muestras por tratamientos mediante los índices de Jaccard y Sorensen. Finalmente, se estimó la variación en el número de unidades taxonómicas identificadas por efecto de los tratamientos, representada en la dinámica de los grupos que surgen y que disminuyen por efecto de la aplicación de compost a través del tiempo.
Los datos fueron analizados mediante el programa libre Galaxy Project versión 17.01, suministrado por la Universidad Johns Hopkins. Posteriormente fueron seleccionadas las muestras con más de 9000 secuencias solapadas para la nueva asignación unidades taxonómicas operativas (UTOS) en ingles OTUS utilizando el programa QIIME versión libre (Caporaso et al., 2010)
RESULTADOS Y DISCUSIÓN
En cuanto a las muestras de suelo el resultado de 2649264 de secuencias solapadas provenientes de 48 muestras permitió la clasificación de 253469 OTUS unidades taxonómicas operativas), cuya distribución en relación con el número de secuencias, indicó que en aquellas muestras donde se obtuvo menos de 150000 secuencias, se encontró mayor concentración de OTUS o de niveles de interés taxonómico expresando de mayor diversidad en el rango de secuencias mencionado. El análisis de la diversidad se presenta a continuación.
Índices de diversidad Alpha y Beta
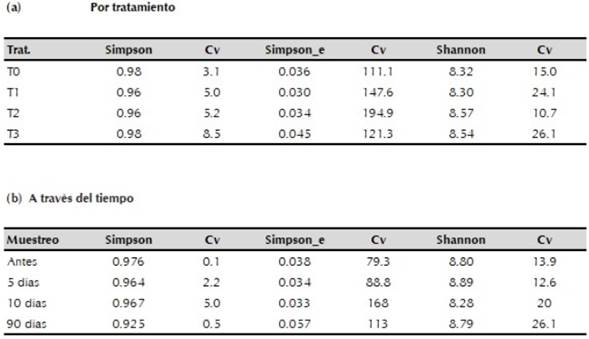
Para la diversidad Alpha, el índice de Simpson generó valores cercanos a uno (tabla 1a) en todos los tratamientos, sin diferencias significativas entre ellos (α=0.05), manifestando que algunos grupos taxonómicos dominaban sobre otros existentes, lo que se expresó en la mayor frecuencia relativa. El índice inverso de Simpson, osciló con valores muy bajos entre 0.030 y 0.045 indicando que las muestras evaluadas tenían grupos taxonómicos compuestos por alto número de individuos, y el índice de Shannon que oscilo entre 8.3 y 8.57, reflejó mayor riqueza en los tratamientos T2 (dosis normal) y T3 (doble dosis) lo cual se pudo comprobar al identificarse mayor número de grupos taxonómicos a nivel de familia y género (α=0.05).
Para los resultados obtenidos a través del tiempo según el momento del muestreo (tabla 1b) el índice de Simpson también indicó dominancia de algunos grupos taxonómicos, pero sin diferencias significativas entre los tres primeros muestreos (α=0.05). De donde se puede infir que a los 90 días (periodo bajo influencias de lluvias) la dominancia presentada en los primeros muestreos había disminuido en favor de una mayor riqueza y equidad, causada por los cambios de pH y humedad ocurridos en el suelo, comprobándose esto al observarse mayor número de grupos taxonómicos a nivel de familia y género en este muestreo. El inverso de Simpson detectó disminución en el número de individuos por grupo a los 90 días, mientras que Shannon registró menor riqueza en el muestreo tomado a los 10 días, comprobándose posteriormente que en este periodo ocurrió una disminución de grupos taxonómicos a nivel de familia y género, como se detallará más adelante.
Los índices anteriores sugirieron un reacomodamiento en el número de individuos por grupo en pro de la estabilidad natural de la comunidad bacteriana con el paso del tiempo, luego del cambio causado en la rizosfera por efecto de la poda de las plantas de morera y el manejo del suelo, tal como lo registran Culman et al. (2006), puesto que encontraron con el paso del tiempo recuperación de las estructuras bacterianas de la comunidad del suelo en un sistema de cultivo de arroz-trigo, luego de ser afectadas con perturbaciones por manejo, solarización y temperatura.
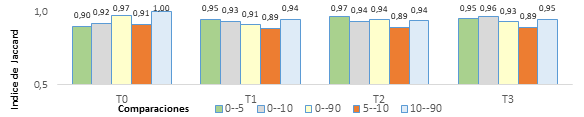
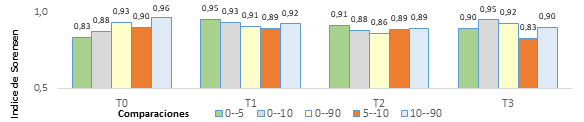
Con relación a la diversidad beta medida en función de Jaccard y Sorensen (figuras 1 y 2), los valores sugirieron mayor variación solo al comparar el tratamiento T0 contra los demás, mostrando rangos que se incrementaron a través del tiempo y expresanron ganancia en la similitud hacia el último muestreo (90 días) y recuperación frente a las condiciones iniciales. En contraste en el caso de los tratamientos donde se aplicó compost (T1, T2 y T3) la tendencia de estos índices, a través del tiempo, fue decreciente con el aumento de la dosis, indicando que las poblaciones fueron más disimiles en T3 y al final del periodo.
La tendencia anterior coincidió con el aumento de las unidades taxonómicas identificadas ocurridas a los 5, 10 y 90 días, especialmente en los niveles de familia y género, debido a los beneficios que el compost aportó (Carbono, nutrientes y energía). En consecuencia entre mayor disponibilidad de material orgánico descompuesto existió en la rizosfera, mayor fue la posibilidad de que se sustentara una población microbiana más grande y diversa en el suelo (Guerrero, 2012).
Al comparar la diversidad beta entre el muestreo tomado a los 5 días contra el tomado a los 10 días en términos generales, Jaccard y Sorensen detectan una disminución en la similitud, mostrando que algún factor influyó sobre las poblaciones alterando su abundancia. En contraste se observó una recuperación de similitud en el periodo comprendido entre los 10 y 90 días en todos los tratamientos.
Esta tendencia se asoció con la actividad radical, especialmente en el periodo comprendido entre el día 20 y 25 después de poda (muestreo a 10 días), cuando la raíces están en proceso de degradación, lo que pudo generar cambios en la excreción y secreción exudados al suelo y cambios en la composición de las poblaciones bacterianas según su hábito y metabolismo (Marshner, 2012).
Composición y abundancia bacteriana por efecto del compost aplicado
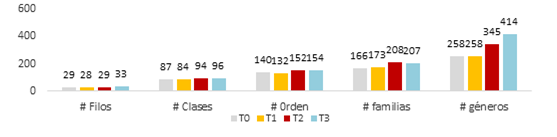
En la figura 3 se observa el incremento en el número de unidades identificadas en la medida en que aumenta la especificidad bacteriana. De igual manera, al comparar el número de OTUS por tratamiento, se observa que en todas las categorías taxonómicas se incrementó el número con respecto a T0, especialmente en T2 (dosis normal) y T3 (doble dosis) con diferencia del 30% entre estos dos, en favor del tratamiento T3.
Lo anterior permitió inferir que los aportes de C y principalmente de N y P realizados por las dosis mayores de compost favorecieron la diversidad bacteriana del suelo incrementándola en función de su riqueza, factor importante para la estabilidad del ecosistema (Conforto et al., 2012; Zhong et al., 2010) especialmente en los niveles; orden, familia y género.
Grupos bacterianos dominantes
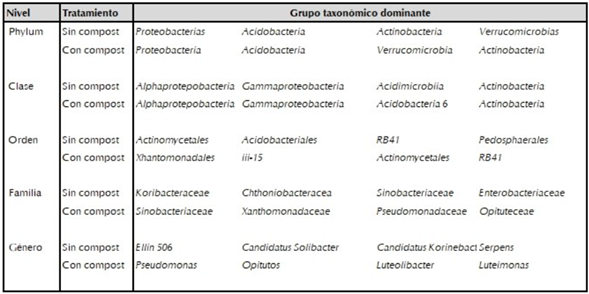
En las muestras de suelo con influencia de compost se encontraron entre los grupos taxonómicos dominantes a los filos: Proteobacteria con frecuencia relativa entre 58 y 80%, Acidobacteria entre 7 y 17% y Verrucomicrobia entre 4 y 11%; las clases Alphaproteobacteria (entre 28 y 61%), Gammaproteobacteria (9 y 18%) y Acidobacteria 6 (4 y 8%); dentro de los órdenes Xhanthomonadales (11 y 15%), iii-15 (10 y 11%) y Actinimycetales (6 y 10%); entre las familias Xanthomonadaceae (6 y 11%), Sinobactereaceae (7 y 10%) y dentro de los géneros Pseudomonas (2 y 11%), Opitutus (4 y 7%), Luteimonas (3 y 6%), Luteolibacter (3 y 5%) y Nitrospira (3 y 4%). Se infirió que es posible que los Nitratos provenientes del compost, hayan causado el dominio de Pseudomonas y Opitutus por ser reductoras de nitratos, y el nitrógeno orgánico mineralizado haya permitido el crecimiento de Luteolibacter y Nitrospira.
Al comparar los grupos dominantes existentes en las muestras sin aplicación y con aplicación de compost se observa en la tabla 2 que se conserva una estructura muy parecida a nivel de phylum y clase, sin embargo, la composición bacteriana y su distribución cambian desde el nivel de orden posiblemente por efecto de la aplicación de compost, hasta el punto de que ningún género coincide, posiblemente debido a la presencia de poblaciones zimógenas (Gómez, Paulini y Hernández, 2010)
Tabla 2 Comparación de grupos taxonómicos más abundantes en ecosistemas cultivados sin y con aplicación de compost.
También fue posible cuantificar que la dominancia existente de algunos grupos taxonómicos disminuyó en la medida en que aparecieron nuevos grupos. Así como también se determinó que en aquellas parcelas donde se aplicó la mayor dosis de compost se dieron los mayores cambios incrementándose la diversidad bacteriana en función de la riqueza, esto pudo ocurrir por la mayor disponibilidad de N y P presente en el suelo. Hu et al. (2011), y Guerrero (2012) afirman que estos nutrientes son clave en el crecimiento de tal diversidad, especialmente el fósforo. Autores como Julca (2006) reporta incrementos de la diversidad bacteriana en suelos cultivables por influencia de las aplicaciones de materia orgánica, y otros como Conforto et al. (2012), encontraron que las comunidades microbianas en suelos con fertilización orgánica presentaron cambios en la biodiversidad, sugiriendo que esto se debido a la mayor utilización de sustratos carbonados y la mayor disponibilidad de nutrientes en el suelo.
Variación en el número de unidades taxonómicas identificadas
A pesar de la diversidad bacteriana existente en los suelos T0 (suelo sin aplicación de compost); 29 filos, 87 clases, 140 ordenes, 166 familias y 258 géneros, se encontró en este entorno menor cantidad de unidades taxonómicas con respecto a T1, T2 y T3, lo que puedo estar relacionado principalmente con el incremento de material orgánico registrado después de la aplicación del compost, lo que generó mayor disponibilidad en el suelo de C, N y P, elementos que influyen directamente en el crecimiento de la diversidad bacteriana (Hu et al., 2011). Aún, si se considera que estos elementos son deficientes en suelos de Clarete Alto. Sin embargo, los cambios de pH ocurridos en el suelo, variando entre 0.7 y 1.4 unidades por efecto de tal adición también influyeron en el aumento de unidades Taxonómicas. Tal es el caso de Fernández y Baath (2010) quienes encontraron cambios en la comunidad bacteriana del suelo hasta en un 50%, cuando el pH tuvo variaciones de 1.7 unidades.
Al comparar y detallar las unidades taxonómicas que aumentaron y disminuyeron en población en cada tratamiento frente a T0 (sin compost), se observó que a nivel de phylum los cambios no fueron significativos para la estructura de la comunidad. A nivel de clases solo dominaron las Gammaproteobacteria con frecuencia relativa importante (16.7 %), al aplicarse la dosis mayor de compost, generando posibles cambios en la estructura bacteriana. Algunas de estas bacterias se ha definido que participan en el ciclo del carbono, otras oxidantes del ácido sulfhídrico y del metano (Shapovalova et al., 2008). Las pocas clases que mostraron sensibilidad a la aplicación de compost registraron frecuencias inferiores al 1% (Fusarium y Spirochaetes), sin causar cambios significativos en la estructura.
Con relación a los órdenes, con la aplicación de las mayores dosis de compost (T2 y T3) surgieron 24 órdenes, resaltándose a Entomoplasmatales (4.4%), Solilubrobacteriales (1.7%), Rhodospirillales (1.5%) con las mayores frecuencias. Las entomoplasmatales, viven dentro de los intestinos o hemolinfa de insectos, no son degradadores de M.O en el suelo (Davis et al., 2017). Las Rhodospirillales incluyen bacterias fijadoras de nitrógeno, otras oxidan el CO con gas de hidrógeno, y pueden usar sulfuro en bajas concentraciones para la reducción de CO2 (Amoozegar et al., 2013). Sobre Solilubrobacteriales no se encontró información disponible de carácter fisiológico. Solo las Desulfuromonadales que con frecuencia mayor al 1% mostraron sensibilidad al compost, al parecer debido al cambio de pH (Holmes et al., 2016).
De las 77 familias surgidas luego de la aplicación de compost solo dos presentaron frecuencia mayor a 1%; Chthoniobacteraceae con 4.5% y Rhodospirillaceae con 2.8%, considerándose familias importantes dentro de la nueva estructura. Las Chthoniobacteraceae metabolizan el carbono orgánico proveniente de residuos vegetales y el aumento de este en el suelo favorece su presencia (Victoria et al., 2012). Las Rhodospirillaceae algunas son heterótrofas aerobias facultativas y otras estrictamente aeróbicas son diazotróficas que habitan la rizósfera y promueven el crecimiento de las plantas. En cuanto a las 40 familias que perdieron visibilidad luego del abonamiento, ninguna registró frecuencias por encima de 1%.
Con relación a los 70 géneros que surgieron luego de la aplicación de compost y los 64 que disminuyeron hasta perder visibilidad, la variación más significativa ocurrió en T3 (mayor dosis de compost), sin embargo ninguno alcanzó frecuencia mayor a 1%, como para significar cambios considerables en la estructura bacteriana.
La disminución de los grupos taxonómicos analizados por tratamientos sugiere que la supresividad en el suelo pudo haberse manifestado impidiendo que algunas bacterias se mantuvieran en la rizósfera e interfiriendo en el restablecimiento de otras. Entre los factores que pudieron inducirla se puede mencionar al cambio de pH por la adición de compost, la mayor disponibilidad de nutrientes (De Luca y Pérez 2014) y la supresividad implícita del compost utilizado (Doussoulin y Moya, 2011) inducida posiblemente por la presencia Pseudomonas y Xanthomonas, las cuales fueron encontradas en el compost (Robledo et al., 2010).
CONCLUSIONES
Se evidenció un efecto sobre la diversidad bacteriana de suelos con aplicación de 1 Kg.m2 de compost, mostrando cambios positivos en la composición, riqueza y abundancia en los niveles taxonómicos orden, familia y género, como consecuencia de la variación presentada por el pH y la humedad del suelo.