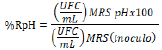INTRODUCCIÓN
Actualmente, la producción animal a nivel mundial enfrenta un sin número de desafíos, los aspectos de bienestar animal tienden a predominar, pero la nutrición y la salud también resultan ser problemáticas. Es por ello que en los sistemas de producción animal intensiva se introducen nuevos productos y tecnologías que permiten la obtención de alimentos más seguros y que al mismo tiempo contribuyan a producciones con una sostenibilidad económica adecuada (Pedroso et al., 2012).
Los recursos forrajeros naturales de la región Caribe se consideran insuficientes para la sostenibilidad de los sistemas de producción con rumiantes, particularmente por su disponibilidad biestacional (Rangel-Ch, 2015). Como el Departamento de Sucre basa su economía principalmente en la ganadería no es ajeno a esta problemática (Lombana et al., 2012) y se proponen diferentes alternativas para cubrir las demandas nutricionales de los animales, una de ellas es la práctica de suplementación con probióticos.
Por otro lado, la especie bovina, está expuesta a diferentes enfermedades durante su ciclo de producción y una de ellas es el síndrome diarreico de origen infeccioso que se presenta frecuentemente durante los primeros diez días de edad. Entre los agentes infecciosos más importantes que intervienen en la enfermedad están Escherichia coli y Salmonella (Zurita et al., 1990). Para contrarrestar los efectos de dichas enfermedades se utilizan los antibióticos de amplio espectro; pero estos compuestos han originado graves problemas de resistencia microbiana y efectos residuales colocando en riesgo la inocuidad alimentaria (Mathur & Singh, 2005).
Debido a esto se han introducido los probióticos como una alternativa natural al uso de los antibióticos, pues no generan efectos colaterales, al contrario, benefician la salud del animal, mejorando el equilibrio de su microbiota intestinal (Gutiérrez et al., 2013).
Una microflora intestinal equilibrada y estable es de vital importancia para las funciones optimizadas del sistema digestivo y por lo tanto para la prevención de enfermedades (Salminen et al.,1998).
La mayoría de microorganismos que se utilizan como probióticos en los animales pertenecen generalmente a las especies Lactobacillus. Enterococcus y Bacillus, aunque también se utilizan levaduras (Saccharomyces cerevisiae) y hongos (Aspergillus oryzae) (Castro y Jimeno, 2001 ).
Los probióticos usados en bovinos reducen o eliminan los patógenos en el tracto gastrointestinal, así como residuos de antibióticos y otras sustancias análogas en productos finales, mejorando el índice de conversión y reduciendo la incidencia de diarreas (Matin & Nisbet, 2005).
Los microorganismos utilizados como probióticos en rumiantes provienen de otras regiones geográficas, tienen un alto costo y son de dudosa calidad e incluso de otras especies animales, sin contar con especies nativas autóctonas. Por lo anterior, es necesario el aislamiento y caracterización molecular de microorganismos con potencial probiótico de heces de terneros de levante de ganado cebú en el Departamento de Sucre, Colombia, con la finalidad de seleccionar las mejores cepas para su posterior aplicación como aditivos microbianos en la alimentación bovina.
MATERIALES Y MÉTODOS
Localización del estudio
Este estudio se realizó en el Departamento de Sucre, Colombia, en dos fincas perteneciente la subregión Montes de María (Municipio de Ovejas), en donde se seleccionaron terneros de levante con edades de entre 1 y 2 años, machos y hembras, de raza Cebú.
Aislamiento de los microorganismos
A partir de un pool de heces de al menos tres terneros sanos por finca, alimentados sin concentrado y sin ser tratadas con antibióticos por lo menos tres meses antes, se aislaron en el laboratorio de Biotecnología GRUBIO-DEQ de la Universidad de Córdoba, bacterias del género Bacillus spp., Lactobacillus spp., y levaduras del género Candida, utilizando medios selectivos, Agar de Man Rugosa Sharpe (MRS) (Lactobacillus spp.), Nutritivo modificado con CMC y Almidón al 0.1% (Bacillus spp.) y Agar Sabouraud Dextrose (SDA) para Levaduras; para su crecimiento e identificación (Rondón et al., 2008).
En cada cepa aislada se realizaron pruebas preliminares: Tinción de Gram, tinción de endosporas (método de Wirtz), prueba de la catalasa, prueba de oxidasa.
Evaluación de las características probióticas "in vitro"
Las cepas preseleccionadas se sembraron en tubos de cultivo con 10 mL de caldo MRS, nutritivo modificado o SDA según su aislamiento. Posteriormente se incubaron por 24 h a 37 °C. A partir de estos cultivos con 1 mL de cada cultivo se realizaron diluciones seriadas; de la dilución 105 y 106 se tomó 0,1ml y se sembró en los medios MRS para conteo de células viables. Conocida la concentración de los inóculos de cada microorganismo, se le realizó a cada cepa las pruebas probióticas.
Tolerancia a cambios de pH
La sobrevivencia y resistencia a pH 3, 4, 5.6 y 7 (ajustado con HCI 5%), el porcentaje de resistencia a pH fue calculado por la siguiente ecuación (Kociubinski et al.,1999):
La tolerancia a la acidez se estimó al comparar el conteo de bacterias viables en agar MRS para la h0 y las células sobrevivientes después de la incubación a 37°C en condiciones de anaerobiosis durante 24 horas pH ácido por 3 h.
Tolerancia a sales biliares
El ensayo se realizó a diferentes concentraciones de sales 0.05, 0.1, 0.15 y 0.3 % (p/v) ajustado el pH =7 con HCI 5 %, e incubación a 37 °C durante 24 horas; al cabo de este tiempo la sobrevivencia y resistencia a sales biliares se comprobó mediante la determinación del número de células viables (Avila et al., 2010).
Tolerancia a cambios de temperatura
Las diferentes cepas fueron sometidas a una temperatura de 40 °C, durante 24 horas; la sobrevivencia y resistencia a esta temperatura se comprobó mediante la determinación del número de células viables (UFC) (Rubio et al., 2008).
Tolerancia a altas concentraciones de Cloruro de sodio (NaCl)
La prueba se realizó utilizando los caldos MRS, Malta, Nutritivo y diferentes concentraciones de NaCl: 2, 4, 6, 8 y 10 % (p/v), con incubación a 37 °C durante 24 horas; finalizado el tiempo se determinó el crecimiento a altas concentraciones de NaCl mediante la medición de la densidad óptica (DO) a 600 nm a 0 y 24 horas (Rondón et al., 2008).
Fermentación de la glucosa
Se utilizaron los caldos MRS, Malta, Nutritivo, que contenían 0.2 % (v/v) de una solución de purpura de bromocresol (0.5 %) y campanas de Durham; luego de la inoculación las muestras se incubaron a 37 °C durante 48 horas. Se consideraron positivos los tubos con burbujas de gas en los viales (Rondón et al., 2008).
Prueba de antagonismo
Esta prueba se realizó contra Salmonella spp., y E. coli pertenecientes al banco de cepas del laboratorio GRU-BIODEQ, las cuales se sembraron en forma masiva en agar Mueller Hinton; en la superficie de éstos se colocaron discos impregnados con los microorganismos de ensayo; se introdujeron en la nevera durante 30 minutos (T = 15 °C) y después se incubaron a 37 °C durante 48 horas. La acción antagónica se evidenció por la presencia de halos de inhibición y crecimiento alrededor de los discos (Mejía et al., 2007).
Capacidad de crecimiento
Las cepas que resistieron la presencia de sales biliares y acidez se cultivaron en 30 mL de caldo MRS, Nutritivo modificado, SDA y se incubaran a 37 °C durante 24 y 48 horas; el crecimiento se comprobó mediante la determinación del número de células viables (UFC) y se determinó su porcentaje (Laurencio et al., 2002).
Identificación molecular y análisis filogenético
Para la identificación molecular se utilizó una de las cepas que superó las pruebas probióticas, de la siguiente forma:
Extracción del DNA genómico
Para la extracción del DNA genómico del microorganismo con características probióticas se utilizó el kit comercial QIA amp® DNA Stool Mini Kit (QIAGEN Corporation). Siguiendo las instrucciones del fabricante.
Amplificación por PCR para la detección del DNA genómico
Se realizó la amplificación por PCR de la región genética de 1465 pb que codifica para la subunidad 16S del RNA ribosomal de procariotas. Utilizando los cebadores (Primers) 27F, 518F, 800R, y 1492R para la identificación molecular de la cepa aislada con características probióticas, la cual fue caracterizada previamente.
Electroforesis
Los productos amplificados por PCR se separaron mediante electroforesis en gel de agarosa al 2 %, previa tinción con GelStar™ Nuclein Acid Gel Stain 10,0000X, según las especificaciones del fabricante. El gel se corrió utilizando el buffer Tris- Acido Borico- EDTA (TAE) al 0.5X durante 50 minutos, a 80 voltios, y se visualizó en un transiluminador de luz ultravioleta. Los productos se analizaron en base al tamaño del segmento y se compararon con los segmentos obtenidos de los controles positivos de cada cepa.
Purificación del fragmento de PCR amplificado
Para la purificación se utilizó MinElute Gel Extraction Kit (QIAGEN), según indicaciones del fabricante.
Secuenciación del DNA
Los productos de PCR una vez purificados se enviaron a la empresa (Macrogen) para su secuenciación.
Análisis bioinformático
El análisis taxonómico de la secuencia problema se realizó mediante la comparación contra las bases de datos National Center for Biotechnology Information (NCBI), Greengenes (Laurence Berkeley National Laboratory) y Ribosomal Database Project (RDP). El alineamiento y generación de un árbol de distancias se hizo utilizando las secuencias con mayor similitud a la secuencia problema.
Diseño y análisis estadístico
Los ensayos se distribuyeron en un diseño completamente al azar (DCA) y los resultados, obtenidos por triplicado, se sistematizaron y analizaron en el software Statgraphics Centurion XVI. Se comprobó la homogeneidad de varianza por el método de Bartlett y el test de normalidad de Shapiro-Wilk, luego se realizó análisis de varianza (ANOVA) dentro de cada concentración y para cada cepa aislada, con el fin de comparar los tratamientos. De igual forma se evaluó la diferencia de los valores de las medias de los grupos en términos de varianza intragrupal utilizando el método de diferencia honestamente significativa (HSD) de Tukey a un 95 % de confianza, además de métodos gráficos de representación de resultados.
RESULTADOS Y DISCUSIÓN
Se aislaron y caracterizaron morfológicamente 10 colonias nativas presentes en muestras de heces de terneros de levante de la raza cebú, correspondientes a cuatro cepas de bacterias ácido lácticas (BAL) (agar MRS), tres bacilos Gram positivos (agar nutritivo) y tres levaduras (Sabouraud Dextrose agar), determinadas a través de las distintas pruebas preliminares. Posteriormente se realizaron todas las pruebas probióticas para cada una de las cepas, como se describe a continuación:
Tolerancia a cambios de pH
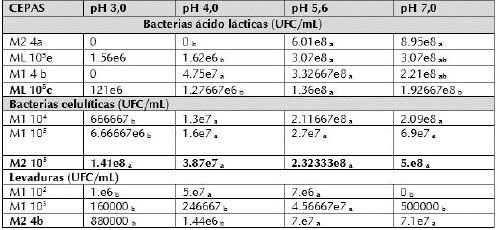
Los análisis para las cepas BAL muestran los siguientes resultados (tabla 1). La cepa M1 4b, aislada en agar MRS, presentó un crecimiento superior para los valores de pH 4, mientras que la cepa M2 4a lo estuvo para un pH de 7; las demás cepas se comportan similar al pH 5,6. Esto sugiere que el máximo crecimiento de estas cepas se encuentra enmarcado en estos niveles de pH. Algunos microorganismos acido lácticos evaluados no soportaron pH 3.0 lo que implica que no son lo suficientemente aptos para colonizar el intestino ya que primero deben superar el ambiente ácido del estómago. Delgado (2005) reportó datos similares a lo mostrado en esta investigación donde algunos aislados evaluados no crecieron a pH 3. En cuanto a las bacterias celulolíticas no hubo diferencias significativas en cuanto a los diferentes niveles de pH, sólo la cepa M2 105 presentó diferencia frente a las demás cepas. Respecto a las levaduras la cepa M2 4b presentó un mejor comportamiento frente a las variaciones de pH siendo superior en valores de pH 4 y 7; lo cual indica la supervivencia en condiciones extremas de pH.
Tolerancia a sales biliares
De las cepas seleccionadas como microorganismos con potencial pro biótico respecto a las BAL sólo la M2 4a no presentó tolerancia ante altas concentraciones, las otras cepas tuvieron igual comportamiento. En cuanto a bacterias celulolíticas la cepa M2 105 tuvo mayor resistencia ante las diferentes concentraciones de sales biliares y para las levaduras la cepa M2 4b mostró igual resistencia para las diferentes concentraciones analizadas (tabla 2).
Tabla 2 Población viable de bacterias y levaduras en medios con cuatro concentraciones de sales biliares.

Nota: No existen diferencias estadísticamente significativas entre aquellos niveles que compartan una misma columna de letras. M: muestras y alfanumérico: código.
Todas las cepas evaluadas fueron capaces de resistir y crecer en presencia de las sales biliares. Para que una cepa probiótica produzca una buena actividad metabólica es necesario que sea capaz de colonizar el intestino delgado, por lo que la tolerancia a bilis es un criterio esencial para la selección de bacterias lácticas (Maré et al., 2006).
Tolerancia a cambios de temperatura (Crecimiento a 40 °C)
Todas las cepas presentaron crecimiento a temperatura de 40 °C, siendo las cepas M2 105 y M1104 las que mostraron los mayores valores en población, el resto también creció, pero con valores menores.
En su mayoría las cepas presentaron crecimiento a 40 ° C, siendo las cepas M2 105 y M1104 las que mejor concentración tuvieron a esta temperatura (tabla 3).
Estos resultados eran de esperarse, ya que estos microorganismos fueron aislados del rumen donde normalmente la temperatura es de 40 oC debido a la enorme cantidad de procesos metabólicos que se producen, la temperatura aquí suele ser 1 o 2 grados por encima de la temperatura corporal del animal (38 a 42 oC) (Van y Regueiro, 2008).
Tolerancia a las altas concentraciones de Cloruro de Sodio
En la tabla 4 se presentan los resultados de los microorganismos seleccionados enfrentados a concentraciones altas de cloruro de sodio. Se observó un efecto significativo en el crecimiento de los diferentes microorganismos a las concentraciones de 2 y 4% de NaCl, sin embargo, a la concentración de 8 y 10% hubo una disminución (tabla 4). Las cepas que mostraron mayor resistencia a altas concentraciones de sal fueron los aislados de bacterias BAL seguido por la bacteria celulolítica y la levadura.
Tabla 4 Evaluación del crecimiento de bacterias y levaduras en medios con cinco porcentajes de NaCI (Absorbancia a 600 nm). Densidad óptica (DO).
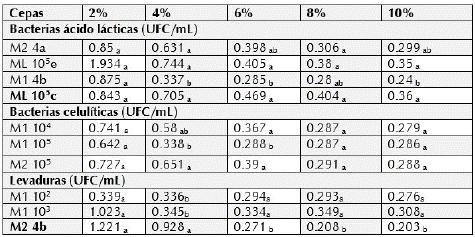
Nota: No existen diferencias estadísticamente significativas entre aquellos niveles que compartan una misma columna de letras. M: muestras y alfanumerico: código.
De acuerdo a los resultados obtenidos en esta prueba, los microorganismos seleccionados en este estudio tienen capacidad para adaptarse fácilmente a condiciones de salinidad relativamente altas. Esta estrategia es fundamental para que los microorganismos soporten un estrés osmótico debido a la presencia de una alta concentración de sal, como ejemplo, la mayoría de las levaduras, independientemente de su hábitat normal, son capaces de crecer en las concentraciones de NaCl que se encuentran en el agua de mar (Van Uden, 1968).
Prueba de Antagonismo
La prueba de antagonismo frente a patógenos, Los microorganismos aislados presentaron poca actividad antagónica frente a E. coli y Salmonella, con excepción de M1 105 y ML 105e (tabla 5).
Tabla 5 Medias para halos de inhibición (mm) por cepas acido lácticas, nutritivo y levaduras frente a Salmonella spp., y E. coli.
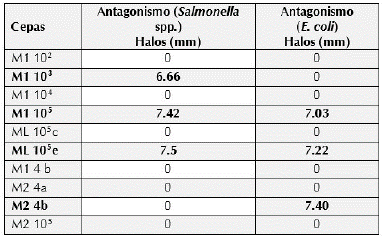
La capacidad antagónica frente a enterobacterias es uno de los criterios para la selección de cepas probióticas en nutrición animal (Pérez, 2008). Lara y burgos (2012) reportaron que Lactobacillus spp., produjo halos de inhibición de 1 mm frente a Salmonella spp., y E. coli, lo cual puede deberse a la producción de sustancias inhibitorias como bacteriocinas y ácido láctico (Domitille et al., 2005).
IDENTIFICACIÓN DE LOS MICROORGANISMOS CON POTENCIAL PROBIÓTICO
Identificación molecular y análisis filogenético
Debido a que la cepa M1 105 fue uno de los microorganismos que superó las pruebas probióticas, se utilizó éste para realizarle análisis molecular. Se determinó mediante la herramienta Ribosomal Database Project (RDP), que se trata de una secuencia de un microorganismo perteneciente al género Bacillus con un 100 % de confidencia.
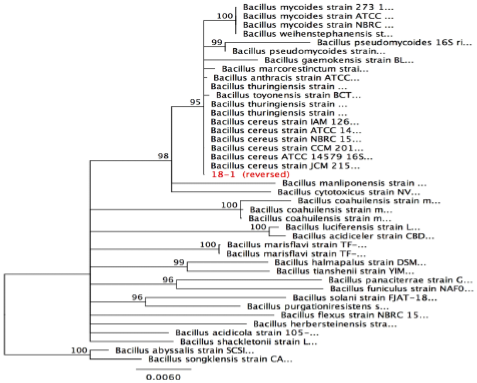
La base de datos de secuencias 16S RDP, usando el algoritmo Seqmatch RDP contra aislamientos cultivados, indica que la secuencia problema ensamblada es muy similar en la mayoría de su longitud con las cepas tipo identificadas como B. toyonensis, B. thuringiensis, B. cereus, o Bacillus spp., la búsqueda en la base de datos Greengenes indica que la secuencia estudiada es muy similar a secuencias de B. cereus, B. thuringiensis, Bacillus spp., los resultados del análisis taxonómico de la secuencia problema ensamblada de 1202 pb contra la base de datos refseq del NCBI indican que tiene un 100 % de identidad en el 99 % de su longitud, con secuencias del gen ribosomal 16s pertenecientes a B. toyonensis (figura 1).
En la figura anterior se observa la relación entre la cepa M1 105 (código en el árbol: 18-1 (reversed)) y B. toyonensis, B. thuringiensis, y B. cereus. En la base de cada clado se muestra el soporte de rama (bootstrap), expresado como el porcentaje de veces en que el análisis produjo la misma asociación entre las secuencias. Este árbol fue construido a partir de las 40 secuencias tipo de microorganismos cultivables más cercanas (Neighbor joining y Bootstrap 100), disponibles en la base de datos de Refseq NCBI, muestra que la secuencia problema se agrupa con las especies B. toyonensis, B. thuringiensis, y B. cereus. De acuerdo con los resultados de búsquedas en las distintas bases de datos, el aislamiento M1 105 pertenece al grupo de B. cereus este es un grupo de bacterias Gram-positivos, formadores de esporas, el cual constituye una rama independiente homogénea dentro de B. genus, compuesto entre otras especies por B. thuringiensis, B. anthracis y B. toyonensis (Sacchi et al., 2002; Jiménez et al., 2013). Estas especies tienen alto grado de similitud a nivel del gen ribosomal 16S, por lo tanto, para poder discriminar entre ellas, es necesario usar regiones específicas de otros genes menos conservados.
Entre las diferentes cepas probióticas formadoras de esporas, B. cereus var. Toyoi (cepa BCT-7112T) ha estado en uso desde 1975 cuando fue aprobada oficialmente por el Ministerio Japonés de Agricultura y Silvicultura como la preparación comercial TOYOCERIN®. Las esporas de BCT-7112T se han utilizado en la nutrición animal para porcinos, aves, ganado, conejos y acuicultura durante más de treinta años en una amplia gama de países alrededor del mundo. En la Comunidad Europea, TOYOCERIN® fue autorizado por primera vez por la Comisión Europea en 1994 para su utilización en la especie porcina, siendo el primer microorganismo autorizado por la Unión Europea para ser utilizado como aditivo en la alimentación, y posteriormente se autorizó también para su uso en aves de corral, ganado y conejos (Jiménez et al., 2013).
Los estudios de identificación inicial asignaron esta cepa dentro de la especie B. cereus (Kozasa et al., 1977). Sin embargo, dado a que una serie de caracteres fenotípicos pueden diferenciar BCT-7112T del fenotipo principal de la especie, se consideró que era una variante y se denominó B. cereus var. Toyoi (Kozasa et al., 1977). Durante muchos años esta cepa ha sido objeto de numerosos estudios para demostrar su naturaleza no toxigénica en la producción industrial (Trapecar et al., 2011; Williams et al., 2009), así como estudios de identificación molecular para diferenciar BCT-7112T de cepas de intoxicación alimentaria (Klein, 2011; Mietke et al., 2010; Pecoraro y Bucher, 2002).
Sin embargo, esta cepa es resistente a cloranfenicol y tetraciclina (genes tetM y catQ), un rasgo que generalmente se considera desaconsejable para su introducción en la cadena alimentaria si se asocia con elementos genéticos móviles (MGEs). Por lo tanto, Glenwright et al. (2017), realizaron una investigación para identificar los genes responsables de estos fenotipos de resistencia y establecer si eran intrínsecos o adquisiciones recientes asociados con la transferencia horizontal de genes. En conclusión, esta investigación mostró claramente que los genes tetM y catQ de B. toyonensis BCT-7112 T son intrínsecos no sólo a esta especie, sino también al grupo B. cereus (sensu lato). La naturaleza intrínseca probada y no transferibilidad de estos genes de resistencia a antibióticos en B. toyonensis BCT-7112 T corroboran recomendar el uso seguro de esta cepa como aditivo en la alimentación animal.
Una investigación realizada por Jiménez y colaboradores (2013) demostró que a pesar de que la cepa BCT-7112T fue, inicialmente, clasificada como B. cereus, mostró diferencias genómicas significativas de las cepas tipo del grupo B. cereus que fueron lo suficientemente grandes (valores ANI por debajo del 92 %) y permitieron considerarla como una especie diferente dentro este grupo. El estudio taxonómico polifásico proporcionó suficientes parámetros discriminatorios para clasificar BCT-7112T como una nueva especie dentro del género Bacillus para la cual el nombre B. toyonensis spp., nov. fue propuesto, con la designación de cepa tipo BCT-7112T (= CECT 876T; = NCIMB 14858T), (Oren y Garrity, 2014).
Recientemente Janssens y colaboradores (2017) publicaron una investigación donde presentaron el proyecto del genoma de otra cepa de B. toyonensis (B. toyonensis VUDES13), la cual fue aislada del intestino medio del colémbolo que vive en el suelo Folsomia candida. Comparando VUDES13 con BCT-7112T (cepa tipo), ésta exhibió "hits" para tres grupos de genes biosintéticos adicionales por antiSMASH, así como 13 -lactamasas adicionales. Y además no observaron genes de virulencia relacionados con otros miembros del clado de B. cereus (B. anthracis, B. cereus y B. thuringiensis).
Esta investigación representa el primer reporte molecular de B. toyonensis en estiércol de terneros de levante cebú en el Departamento de Sucre, el cual ha sido ampliamente utilizado como probiótico en la nutrición animal. Los resultados obtenidos en las pruebas in vitro realizadas demostraron que esta cepa posee características para ser utilizada potencialmente como probiótico.
CONCLUSIONES
Esta investigación representó el primer reporte molecular de Bacillus toyonensis en estiércol de terneros de levante cebú en el departamento de Sucre, los resultados obtenidos en las pruebas in vitro realizadas demostraron que esta cepa posee características para ser utilizada potencialmente como probiótico. Con esta investigación se pretende crear el primer banco de cepas probióticas nativas, aisladas de ganado cebú en el departamento de Sucre que serán usadas para futuros estudios y para la elaboración de biopreparados con acción probiótica como alternativa a la utilización de antibióticos en la nutrición animal.