INTRODUCCIÓN
La carne de pollo y el huevo se encuentran entre los alimentos de origen animal de alto consumo a nivel mundial (Mottet y Tempio, 2017). La demanda de alimentos derivados de animales está aumentando debido al crecimiento de la población, el aumento de los ingresos y la urbanización (OCDE/FAO, 2014). De esta manera, la carne de pollo ha mostrado la tendencia más rápida en crecimiento en los últimos años. Se estima que se produzcan alrededor de 150 millones de toneladas de carne de pollo en 2020 (OCDE / FAO, 2018). Pese al progreso observado en la explotación avícola, este sector se enfrenta a amenazas significativas como enfermedades infecciosas ocasionadas por microorganismos patógenos como Salmonella ssp, Campylobacter spp y Escherichia coli, entre otros (Davies y Wales, 2010). Además, la naturaleza ubicua de estos microorganismos puede crear una fuente constante de contaminación del producto alimenticio, debido a que las aves de corral son uno de los principales reservorios de estos patógenos (Davies y Wales, 2010).
La caracterización del microbioma de aves de corral se centra actualmente en el estudio del tracto gastrointestinal, dado que es el área de mayor abundancia y diversidad bacteriana (O'Hara y Shanahan, 2006), mayor relevancia para la salud del huésped, dado que el mismo desempeña un papel esencial en la digestión y absorción de nutrientes, el desarrollo del sistema inmune y la exclusión de patógenos (Pan y Yu, 2014; Yeoman et al., 2012). Por otra parte, se ha señalado que la dieta (Hammons et al., 2010; Jia et al., 2009) y los aditivos alimentarios (Amerah et al., 2011; Danzeisen et al., 2011) pueden afectar la microbiota gastrointestinal de las aves de corral con respecto a la diversidad y composición (figura 1). En consecuencia, la microbiota intestinal se ve influenciada por factores genéticos y factores externos como la dieta y el ambiente (Apajalahti et al., 2004; Smulikowska, 2006; Wang et al., 2016; Ballou et al., 2016; Wang et al., 2018). No obstante, a pesar de la importancia de la microbiota intestinal, y el papel que tiene en la salud y bienestar de las aves de corral (Awad et al., 2016; Stanley et al., 2013), se ha estudiado de manera limitada la composición y diversidad bacteriana dentro del tracto gastrointestinal; adicionalmente el rol de los microorganismos en el desarrollo de un tracto gastrointestinal saludable aun es desconocido. Se conoce poco sobre la diversidad de este ecosistema, su desarrollo y la respuesta a las infecciones microbianas, principalmente bacterianas, su impacto en el huésped y las interacciones entre la microbiota y el huésped.

Figura 1 Asociación entre microbioma intestinal, huésped, dieta y microbioma de la cama. (Adaptado de Pan y Yu (2014), Wang et al. (2016), Ballou et al. (2016).
Los primeros estudios de las comunidades microbianas se limitaban a métodos convencionales usando medios de cultivo selectivo para el crecimiento bacteriano. (Barnes et al., 1972). Sin embargo, estas técnicas recuperan una fracción minoritaria de los microorganismos. Es decir, para los taxones bacterianos que habitan en el tracto gastrointestinal de las aves de corral, menos del 20% es recuperado a través de este método (Gaskins et al., 2002). Por esta razón, se han utilizado nuevos enfoques de estudio independientes del uso de medios de cultivo, estos hacen uso de herramientas moleculares con el objeto de superar los sesgos presentados y obtener información detallada sobre la diversidad, estructura y función del microbioma.
Entre las técnicas introducidas, se incluye el enfoque molecular de electroforesis en gel de gradiente desnaturalizante (DGGE) (Gaskins et al., 2002), electroforesis en gel con gradiente de temperatura temporal (TTGE) (Zhu et al., 2002) y la técnica del polimorfismo de longitud de fragmento (T-RFLP) (Gong et al., 2008; Torok et al., 2011). De igual manera, la secuenciación de Sanger también ha sido utilizada para estudiar la diversidad de la microbiota intestinal del pollo parrillero (Lu et al., 2003). Aunque estos enfoques son más robustos que los métodos dependientes de cultivo, aún son incapaces de representar con precisión la diversidad del microbioma de las aves, debido a su baja cobertura, rendimiento y sumado a los altos costos que implica su utilización (Choi et al., 2015). En los últimos años, se han introducido técnicas de biología molecular que permiten acceder a la diversidad microbiana de un ecosistema determinado. Estas hacen uso de la secuenciación de próxima generación (NGS), la cual proporciona un análisis a gran escala de la biodiversidad y abundancia de microorganismos (Josefsen et al., 2015). A partir de esta tecnología, es posible desarrollar estudios metagenómicos, facilitando un análisis completo y complejo de las comunidades microbianas (Simon y Daniel, 2011), además, es posible acceder a información en un único análisis sobre la presencia, proporción, tipo, virulencia, predicción de roles nutricionales y resistencia a antibióticos de un determinado grupo de microorganismos (Josefsen et al., 2015). Por otra parte, este nuevo enfoque metagenómico ha permitido el desarrollo de análisis genómicos funcionales completos para delinear los requisitos genéticos requeridos para la colonización, infección y supervivencia de microorganismos patógenos en condiciones ambientales a las que éste se encuentra expuesto. El desarrollo y la aplicación de tales herramientas novedosas de la metagenómica funcional pueden revelar vulnerabilidades previamente desconocidas, las cuales pueden explorarse para desarrollar nuevas intervenciones y romper la cadena de transmisión de microorganismos patógenos en la industria avícola. El propósito de esta revisión fue estudiar las metodologías utilizadas para análisis metagenómicos, con el fin obtener la taxonomía bacteriana e inferencias funcionales de la microbiota gastrointestinal de aves de corral. Se describe y se analiza el procesamiento de muestras, el tipo de enfoque metagenómico, la tecnología de secuenciación y las herramientas bioinformáticas utilizadas para el análisis de la información.
Recolección y extracción de muestras de ADN
El procesamiento de muestras es el primer paso en los proyectos metagenómicos. El ADN extraído debe ser representativo de todas las células presentes en la muestra y debe ser idóneo para la generación de las bibliotecas genómicas. Actualmente están disponibles diferentes métodos de extracción de ADN para cada tipo de muestra (e.g. muestras fecales, suelo) (Wise y Siragusa, 2007; Hang et al., 2014; Josefsen et al., 2015). Sin embargo, la selección del método óptimo no está determinada (Hamady y Knight, 2009). Existen diferentes kits de extracción de ADN, la mayoría de estos utilizan lisis celular, eliminación de macromoléculas diferentes a ácidos nucleicos y recolección del ADN. En general, la lisis es la estrategia más aplicada y de mayor relevancia, puesto que la intensidad de la lisis puede dar lugar a un sesgo hacia un grupo taxonómico en particular (Josefsen et al., 2015). Por tal razón, es necesario utilizar una combinación de medios químicos, físicos y mecánicos para lisar las células de manera eficiente. Trabajos previos recomiendan el uso combinado de los tres, específicamente cuando se analizan comunidades microbianas complejas (Yu y Morrison, 2004; Josefsen et al., 2015; Burbach et al., 2016; Wang et al., 2016).
Para la extracción eficiente de ADN de muestras particulares como como las heces o el intestino de las aves se requiere de gran cuidado ya que son una clase de muestras divergentes y de variabilidad en composición (Gill, 2014). Lo anterior está dado por las diferencias encontradas en la fisiología del tracto digestivo de las aves y la forma de procesamiento del alimento (Wise y Siragusa, 2007). Esto hace que la extracción de ADN del microbioma de las aves sea un desafío, por ejemplo, debido al alto contenido de e.g. ácido úrico y sustancias húmicas encontradas en las muestras (Eriksson et al., 2017). De acuerdo con la literatura el protocolo más común de extracción de ADN en muestras de heces y ciego intestinal ha sido el planteado por Yu y Morrison (2004), al igual que el Kit de extracción QIAmpH DNA Stool Mini Kit (tabla 1).
Por otra parte, estudios recientes han sugerido cambios en la microbiota gastrointestinal en pollos recién nacidos asociados a factores contaminantes fecales o ambientales, adheridos a la cáscara de los huevos (Donaldson et al., 2017). El estudio realizado por Lee et al. (2019), ha indicado que más del 50% de la diversidad de la microbiota de la clara de huevo y del intestino del embrión tiene su origen a partir de la microbiota de la cáscara de huevo. Del mismo modo, sugieren que la microbiota de la cloaca y del oviducto materno posiblemente determina la microbiota de la cáscara de huevos.
De esta manera, se han incluido técnicas para recuperar el ADN en muestras de cáscara de huevo, como el aislamiento de ADN basado en la resina chelex (Martín-Platero et al., 2010). Este protocolo ha sido ampliamente utilizado para muestras de diferentes orígenes por su bajo costo y simplicidad. Para realizar el aislamiento de muestras de cáscaras de huevos, el procedimiento indica que los huevos deben limpiarse con un hisopo estéril, luego éste debe ser almacenado en una solución fosfato en un tubo de microcentrífuga hasta la extracción del ADN. Para la obtención del ADN, las células bacterianas se recolectan por varias etapas de centrifugación. Posteriormente, se procede a realizar la extracción de ADN a partir del sedimento recolectado (Martín-Platero et al., 2010. No obstante la sencillez del protocolo mencionado, el método de frotis de la cáscara de huevos presenta diferencias en los perfiles de las comunidades bacterianas y una menor eficiencia en la extracción comparado con el método de trituración de la cáscara de huevo (Grizard et al., 2014). En este último, se utiliza nitrógeno líquido para la trituración de las cáscaras de huevo, el polvo obtenido se procesa utilizando el Kit NucleoSpin Soil Kit (Grizard et al., 2014).
De acuerdo con lo mencionado anteriormente es de interés optimizar un protocolo de extracción de ADN de alto rendimiento para muestras aviares, con el fin de obtener una representación correcta del microbioma estudiado.
Estudios de Microbioma
Los estudios metagenómicos para el análisis de microbioma se pueden clasificar en dos categorías; estudios de amplicón dirigidos, los cuales se centran en uno o varios genes marcadores para revelar la composición y diversidad de la microbiota (Torok et al., 2011; Thomas et al., 2012; Neelakanta et al., 2013; Bhogoju et al., 2018) y el enfoque metagenómico completo, también denominado megenómica shotgun, debido a la aleatoriedad con que se obtienen las secuencias genómicas (Thomas et al., 2012; Sharpton, 2014). La figura 2 muestra una descripción general de ambos tipos de estudio y su combinación.

Figura 2 Análisis metagenómico del tracto gastrointestinal de aves. Adaptado de Morgan et al. (2013), Choi et al. (2015).
La metagenómica shotgun proporciona datos más completos sobre el potencial funcional presente en las comunidades microbianas. Para este último enfoque, la naturaleza aleatoria de la secuenciación garantiza una cobertura adecuada para evaluar la estructura de la comunidad y abre nuevas vías para descubrir novedades estructurales y funcionales (Kuczynski et al., 2012).
Metagenómica Dirigida 16S RNA
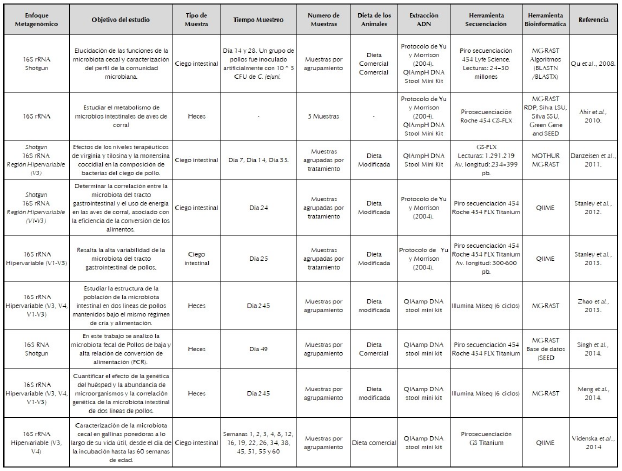
Las subunidades de proteína ribosómica, los factores de elongación, y las subunidades de ARN polimerasa han sido utilizados como genes marcadores para estudiar la composición y la diversidad microbiana, sin embargo, estos representan poblaciones diferentes (Ríos y Ríos, 2011). En consecuencia, la mayoría de estudios de comunidades microbianas incluyen la secuenciación de amplicones dirigidos filogénicamente informativos, como el gen de la subunidad ribosomal 16 rRNA para bacterias y Archaea, el gen 18S rRNA de especies eucarióticas y las regiones nucleares de espaciador transcrito ribosomal (ITS) para hongos (Meyer et al., 2010). Una característica diferenciadora de los genes rARN es su presencia en todos los organismos, mientras que otros marcadores usados tienen una distribución taxonómica limitada (Kuczynski et al., 2012). Además, el gen ribosómico 16S rRNA consta de nueve regiones hipervariables flanqueadas por regiones conservadas, las regiones hipervariables son específicas de cada género y especie, característica utilizada en la identificación de microorganismos (Baker et al., 2003). En consecuencia, el gen 16S rRNA es un marcador molecular óptimo para la identificación a nivel de especie en poblaciones microbianas. Por ejemplo, para estudios microbianos del intestino de aves productoras de carne y huevos se han cubierto las regiones V1-V3, V3-V4, V4-V5, V6-V7, V1, V2, V3, V4, V8, V9 del gen 16S rRNA. (Danzeisen et al., 2011; Stanley et al., 2012; Stanley et al., 2013; Zhao et al., 2013;Meng et al., 2014; Videnska et al., 2014; Sergeant et al., 2014; Ma et al., 2015;Borda-Molina et al., 2016; Hou et al., 2016;Wang et al., 2016; Bortoluzzi et al., 2017; Pineda-Quiroga et al., 2017; Bhogoju et al., 2018; Lee et al., 2019), ver tabla 1.
Por otra parte, otra ventaja que proporciona el gen 16S rRNA sobre otros genes marcadores potenciales, es la disponibilidad de varias bases de datos de secuencias de referencia y taxonomía, como greengenes (DeSantis et al., 2006), SILVA (Pruesse et al., 2007) y Ribosomal Database Project1 (Claesson et al., 2010). El gen 16S rRNA ha sido utilizado para análisis filogenéticos de la microbiota cecal en aves de corral (Lan et al., 2002). Sin embargo, el primer estudio de metagenómica dirigida que caracterizó la microbiota gastrointestinal de los pollos se publicó en el año 2008 (Qu et al., 2008). En este estudio, se analizó la microbiota cecal de aves comerciales usando el gen 16S rRNA y metagenómica shotgun, utilizando piro secuenciación; los resultados obtenidos revelan una visión más completa de la comunidad microbiana del ciego del pollo. Se encontró que los elementos móviles de ADN son un componente funcional de los microbiomas cecales, lo que contribuye a la transferencia horizontal de genes y la evolución funcional de los microbiomas. Además, esto sugiere que la evolución de metavirulomas específicos del huésped es un factor que contribuye a la resistencia de la enfermedad a patógenos zoonóticos (tabla 1).
Como resultado, los perfiles de secuenciación del gen ribosomal 16S rRNA ha permitido determinar la relación entre la diversidad microbiana y las condiciones ambientales a las que se encuentra expuesta. Además, las modulaciones de la comunidad microbiana en respuesta a los promotores de crecimiento, antibióticos, indicando que los cambios en la estructura de las comunidades microbianas cecales o fecales que han sido inducidas por antibióticos son un factor determinante en la abundancia de microorganismos presentes en el huésped y los retos a futuro para reemplazar los efectos generados por los promotores de crecimiento y antibióticos utilizando enfoques alternativos (Danzeisen et al., 2011; Xiong et al., 2018)
En el caso de la microbiota del tracto gastrointestinal de aves, se ha generado una visión sobre las interacciones huésped-microorganismos, además contempla posibles hipótesis sobre mecanismos de la enfermedad (Ding et al., 2016; Sergeant et al., 2014; Stanley et al., 2013; Wang et al., 2016).
Adicionalmente, estudios metagenómicos utilizando el gen 16s RNA han asociado el genotipo del hospedador y la dieta como factores influyentes en la sucesión de la micro-biota intestinal de las aves de corral. (Zhao et al., 2013; Meng et al., 2014; Borda-Molina et al., 2016; Varmuzova et al., 2016; Ding et al., 2016; Yan et al., 2017; Bortoluzzi et al., 2017; Pineda-Quiroga et al., 2017; Huang et al., 2018).
Metagenómica Shotgun
La metagenómica dirigida es un enfoque que hace posible estudiar la diversidad microbiana a gran escala, no obstante la secuenciación de amplicones tiene sus limitaciones; en primer lugar, puede producir estimaciones de diversidad ampliamente diferentes (Jumpstart Consortium Human Microbiome Project Data Generation Working Group, 2012), en segundo lugar, la secuenciación de amplicones generalmente solo proporciona información sobre la composición taxonómica de la comunidad microbiana (Liu et al., 2008). En consecuencia, es imposible resolver directamente las funciones biológicas asociadas a los taxones utilizando este enfoque, además, la identificación de bacterias altamente divergentes son difíciles de caracterizar mediante este enfoque (Sharpton, 2014).
En consecuencia, el enfoque metagenómico "shotgun" es un método alternativo para estudiar ecosistemas complejos, en el cual se reducen las limitaciones anteriormente mencionadas. En este procedimiento, el ADN extraído se corta en pequeños fragmentos que se secuencian de forma independiente, en lugar de apuntar a un locus genómico específico para la amplificación, dando como resultado una vista completa de todos los microorganismos y genes presentes en la microbiota (Sharpton, 2014). De esta forma, la metagenómica "shotgun" describe el conjunto de genomas y los genes correspondientes de un ecosistema dado, facilitando la caracterización de la funcionalidad bacteriana potencial en ambientes específicos (Marchesi y Ravel, 2015). Asimismo, la combinación del uso de marcadores filogenéticos 16S rRNA, enfoques metagenómicos shotgun, técnicas moleculares de secuenciacion masiva y las herramientas para análisis de datos, han permitido obtener información sobre el microbioma gastrointestinal de aves de corral, en cuanto a taxonomía y función de microorganismos (Borda-Molina et al., 2016; Bortoluzzi et al., 2017; Danzeisen et al., 2011; Ding et al., 2016; Meng et al., 2014; Pineda-Quiroga et al., 2017; Sergeant et al., 2014; Stanley et al., 2013; Wang et al., 2016). No obstante, hasta el momento solo unos pocos estudios se han enfocado en estudiar las funciones de los microorganismos que se encuentran presentes en el tracto gastrointestinal de las aves (Yeoman et al., 2012). Hasta la fecha se han realizado ocho estudios metagenómicos shotgun en pollos, enfocados en el ciego intestinal. El primero, realizado por Qu et al. (2008), se determinó la distribución de 200.000 genes presentes en los pollos sanos y en pollos infectados experimentalmente con Campylobacter jenuini. En el segundo, Danzeisen et al. (2011), analizaron las diferencias en los genes entre los pollos de control y los alimentados con niveles sub-terapéuticos de antibióticos para mejorar el crecimiento. En el tercero, Singh et al. (2014), determinaron la variación de la microbiota intestinal y la eficiencia de la conversión de los alimentos representado en ganancia de peso, encontrando que la microbiota cecal presentó una mayor diversidad en muestras con alta conversión de alimento comparada con la microbiota encontrada en aves con baja conversión de alimento. Por otra parte, las diferencias encontradas en el análisis funcional de los genes asociados con el metabolismo de carbohidratos y aminoácidos o asociados a la virulencia y motilidad flagelar, estaban en mayor proporción en aves con baja conversión de alimento. En este sentido, esta información facilita estrategias para mejorar la eficiencia de la alimentación y la formulación para pollos de engorde.
En el cuarto estudio Sergeant et al. (2014), se centraron en las funciones de ciego intestinal encontrando que el microbioma cecal codifica varias fermentaciones o rutas que conducen a la producción de ácidos grasos de cadena corta con características novedosas. En el quinto, Ma et al. (2015), investigaron genes de resistencia a los antibióticos (ARG) y su coexistencia con elementos genéticos móviles asociados, detectando altos niveles de genes de resistencia a múltiples medicamentos ARGs (7762 x/Gb) como tetraciclina, eritromicina y a los aminoglucósidos en las heces de pollos adultos. Estos resultados proporcionan conocimiento con respecto a los genes asociados ARG y la posible evaluación del riesgo ambiental y público. En otro estudio, realizado en la investigación de genes de resistencia a los antibióticos, Tong et al. (2017), encontraron que la especie Escherichia coli fue representativa en las heces de las aves. Por otra parte el análisis de Blast y la comparación con la base de datos de genes de resistencia a antibióticos revelo que las heces de pollo contenían gran abundancia de genes de resistencia a antibióticos ARG, entre los cuales la vancomicina (van) fue más abundante (36% de todas las lecturas). Finalmente, el enfoque Metagenómico shotgun ha permitido comparar el microbioma intestinal de dos líneas de pollos (Magras y grasa), observando diferencias estructurales significativas tanto en los metagenomas filogenéticos como en los funcionales entre las dos líneas de pollos (Hou et al., 2016; Ding et al., 2016).
En este sentido, los proyectos metagenómicos del tracto gastrointestinal de pollos se han centrado en las funciones de la microbiota cecal (Sergeant et al., 2014), el papel fundamental de la microbiota de aves en los parámetros de rendimiento y cambios en la dieta (Yan et al., 2017), las comparaciones entre líneas gordas y magras (Singh et al., 2014; Ding et al., 2016; Hou et al., 2016), los mecanismos de respuesta a microorganismos patógenos (Qu et al., 2008), representación del viruloma (Qu et al., 2008) y de los genes de resistencia a antibióticos (Ma et al., 2015; Tong et al., 2017), ver tabla 1.
Del mismo modo, estas investigaciones descubrieron una gran cantidad de información relacionada con la prevalencia de elementos móviles y genes involucrados en la nutrición, la virulencia y la resistencia a los antibióticos. Por otra parte, generaron una visión detallada de la diversidad y distribución de todos los tipos de microorganismos, incluidas las bacterias, las arqueas, los virus y los microorganismos eucarióticos.
Tecnologías de Secuenciación de microbiomas de aves de corral
Se han utilizado varias tecnologías de secuenciación para realizar estudios de microbioma a partir de perfiles taxonómicos basados en 16s RNA y shotgun (Danzeisen et al., 2011). Dentro de estas tecnologías se encuentra incluida la secuenciación capilar (Sanger), este método pertenece a la primera generación de técnicas de secuenciación. Lu et al. (2003), utilizaron esta técnica para estudiar la sucesión de la microbiota intestinal de pollo, encontrando que la composición y la diversidad de la microbiota varía con diferentes dietas, aditivos para piensos y probióticos (Ballou et al., 2016). Pese a los resultados encontrados, el método convencional de secuenciación de Sanger ha limitado los estudios metagenómicos de microbiota gastrointestinal debido a los altos costos de secuenciación, insuficiencia en las secuencias y su bajo rendimiento (Choi et al., 201 5). En consecuencia, hubo la necesidad abordar nuevos métodos de alto rendimiento, capaces de generar cientos de miles de secuencias de manera más rápida y económica. Estas técnicas son denominadas tecnologías de secuenciación de próxima generación (Metzker, 2010), las cuales constituyen diversas estrategias que se basan en una combinación de plantillas, secuenciación e imágenes, alineación del genoma y métodos de ensamblaje (Diaz-Sanchez et al., 2013). Actualmente, existen diferentes plataformas disponibles para secuenciación; dentro de éstas, se incluyen plataformas de segunda generación HT-NGS, las cuales pueden generar alrededor de quinientos millones de bases de secuencia en bruto (Roche) a miles de millones de bases en una sola ejecución (Illumina, SOLiD). Estos nuevos métodos tienen algunas de las siguientes características en la forma de realizar la secuenciación:
(i) Secuenciación por síntesis:
-PCR en emulsión (Roche SoliD)
-PCR en puente (Ilumina)
(ii) Secuenciación por semiconducción:
-Ion Torrent
En la tabla 2 se señalan características distintivas de cada tecnología, como longitud de lectura, tiempo de corrida, tasa de error, tamaño de inserción y costo.
Tabla 2 Comparación de plataformas de secuenciación de próxima generación. (Segunda y tercera generación)

La descripción de cada una de las plataformas mencionadas permite evidenciar los rápidos avances tecnológicos en las metodologías de secuenciación, sus aplicaciones y las herramientas optimizadas resaltan la importancia de la investigación del genoma. Por otra parte, se hace evidente que en los últimos años los estudios sobre la evolución experimental microbiana han utilizado la secuenciación como una herramienta fundamental para investigar el rol ecológico de los microorganismos en diferentes ambientes (Kuczynski et al., 2012).
Por otra parte, la aplicación de las técnicas de secuenciación de próxima generación son herramientas útiles y valiosas para comprender la evolución de microorganismos patógenos y sus cambios mutacionales dentro y en el huésped, además, son idóneas para identificar los microorganismos patógenos que causan infecciones o epidemias en las poblaciones huéspedes (Schmid et al., 2000).
Lo anterior evidencia como la secuenciación de próxima generación revolucionó y cambió las perspectivas en la caracterización de las comunidades microbianas. Los estudios metagenómicos realizados en microbioma de aves de corral demuestran que las tecnologías de secuenciación seleccionadas para este tipo investigaciones han sido las tecnologías: Illumina MiSeq, HiSeq, Pirosecuenciación e Ion Torrent (tabla 1).
De este modo, las tecnologías de secuenciación de próxima generación en investigaciones del microbioma de aves de corral muestran una nueva orientación e integración de los antiguos estudios de laboratorio, resaltando su veracidad en el seguimiento y detección de los patrones de evolución encontrados en microorganismos patógenos en diferentes entornos (Danzeisen et al., 2011). Además, estas tecnologías soportan el desarrollo de estrategias para el control de patógenos, puesto que permite identificar genes de resistencia, diversidad microbiana a gran a escala, generando como resultado un análisis completo del microbioma intestinal (Qu et al., 2008; Ma et al., 2015; Tong et al., 201 7; Lee et al., 201 9).
Herramientas de análisis de datos bioinformáticos
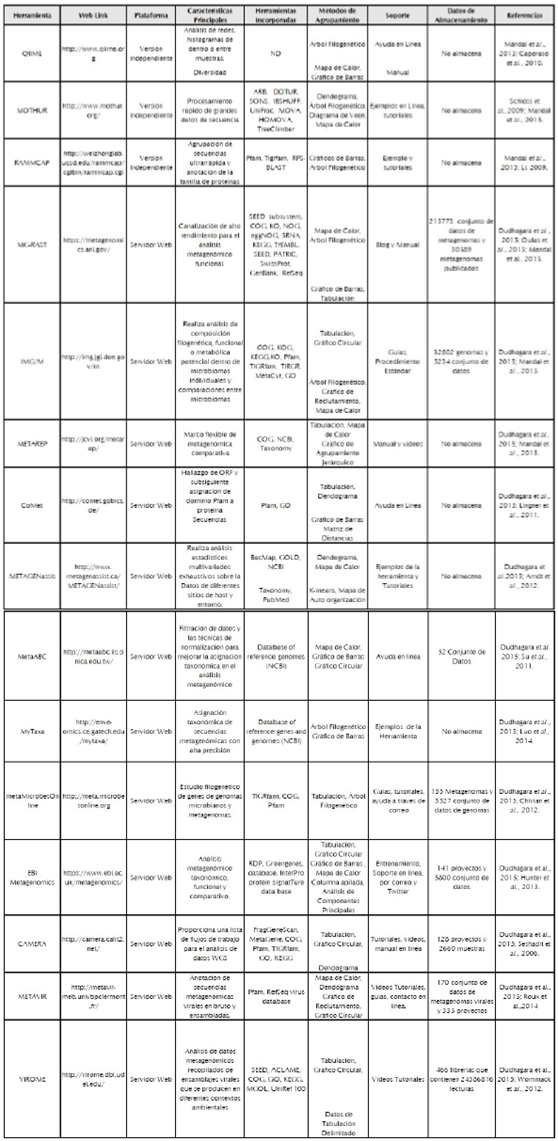
Después de obtener las secuencias de ADN, éstas deben ser analizadas e interpretadas. Es primordial analizar los datos obtenidos para determinar las identidades y los roles de las especies microbianas presentes en las muestras analizadas (Ounit et al., 2015). La gran cantidad de datos producidos en los proyectos metagenómicos requiere de herramientas bioinformáticas para su análisis dado que la manipulación directa de datos, como la alineación manual de secuencias de ADN es dispendiosa y demanda mucho tiempo (Kuczynski et al., 2012). Por ejemplo, el ensamblaje de los datos de las secuencias y la identificación de unidades taxonómicas operativas (OTU), es uno de los principales retos técnicos para las herramientas de análisis metagenómico (Behnam y Smith, 2014). Por otra parte, además de la alta complejidad de los datos metagenómicos, es crítico y difícil analizar los mismos utilizando herramientas independientes. Existen diferentes herramientas para clasificar lecturas metagenómicas contra conjuntos de datos de referencia (Mandal et al., 2015). Aquí revisamos algunas herramientas software, útiles para el análisis de los datos de secuenciación del gen del marcador 16S rRNA y los datos de metagenómicos shotgun (tabla 3).
En esta revisión se describen diferentes enfoques bioinformáticos con el fin de soportar la información principal para la selección y aplicación idónea de los mismos en los análisis de datos metagenómicos. Para ello, se han usado diferentes softwares para la eliminación de secuencias de baja calidad y verificación de secuencias quiméricas (Haas et al., 2011), eliminación de errores generados en la secuenciación, producción de un grupo de secuencias casi idénticas denominadas unidades taxonómicas operativas y el análisis estadístico para determinar las diferencias significativas comparadas con la secuencia de referencia (Caporaso et al., 2010), ver figura 3.
De acuerdo con Medinger et al. (2010), las unidades taxonómicas operativas (OTU) están definidas por dos enfoques:
i) La agrupación no supervisada en la similitud de secuencia, ii) la agrupación supervisada que asigna directamente secuencias a grupos taxonómicos.
De esta manera, las unidades taxonómicas operativas (OTU) representan la información taxonómica y proporcionan información sobre la diversidad de la población. Posteriormente a la asignación taxonómica, se procede a realizar el análisis estadístico para establecer diferencias significativas comparadas con la secuencia de referencia (Videnska et al., 2014).
El análisis estadístico contiene el estudio de la biodiversidad direccionada a dos indicadores relevantes: diversidad alfa y beta (Choi et al., 2015). La riqueza y uniformidad de las especies individuales en una muestra es denominada diversidad alfa (Hughes, et al., 2001), mientras que la diversidad beta permite comparar múltiples comunidades con el fin de identificar OTU o taxones compartidos entre las mismas y relacionarnos con la metadata (Colwell, 2009). Así mismo, tanto la riqueza como la uniformidad de las especies son indicadores importantes de la salud de los animales. Por ejemplo, en los estudios realizados por Danzeisen et al. (2011), Borda-Molina et al. (2016), Pineda-Quiroga et al. (2017) y Bortulozi et al. (2017), se identificó que el número de microorganismos en el intestino del huésped disminuye durante el tratamiento con antibióticos o variaciones en los nutrientes de la dieta suministrada. Así por ejemplo, herramientas bioinformáticas tales como MOTHUR y QIIME, las cuales permiten el cálculo de varios índices, incluidos Chao, son estimadores de cobertura basados en abundancia. Adicionalmente, los índices Shannon y Simpson calculan la abundancia o distribución de las unidades taxonómicas dentro de una población particular (Caporaso et al., 2010; Schloss et al., 2009).
Por otra parte, algunos estudios sugieren que la micro-biota intestinal con una alta diversidad alfa está fuertemente correlacionada con funciones genéticas específicas, concluyendo que el huésped que presenta una alta diversidad en el tracto gastrointestinal puede ser más estable o más saludable que aquellos que tienen una baja diversidad (Caporaso et al., 2010). De esta manera, los resultados proporcionados por MOTHUR y QIIME, representados en tablas o como curvas de rarefacción, permiten la comparación de las comunidades microbianas y soportan la emisión de conclusiones basadas en la diversidad alfa y su contribución en la inferencia funcional de la microbiota estudiada (Hughes et al., 2001). A pesar de la información detallada que se puede obtener mediante el procesamiento y predicción funcional realizada con las herramientas bioinformáticas, se recomienda ser prudente al emitir conclusiones sólidas dado que existen un gran número de unidades taxonómicas operativas (OTU) que no se pueden asignar a un género o familia especifica (Lee et al., 2019).
Finalmente es importante mencionar que las herramientas más utilizadas para el análisis de datos metagenómicos asociados al microbioma de aves de corral han sido MGRAST, QIIME y MOTHUR (tabla 3). Estas han permitido definir grupos funcionales y taxones bacterianos importantes en las diferentes investigaciones. En el estudio realizado por Danzeisen et al. (2011), se identificaron unidades taxonómicas operativas OTU exclusivas de los grupos de tratamiento con monensina, virginamicina y tilosina. Estas incluyen los géneros Anaerotruncus sp, Subdoligranulum sp y Sedimentibacter sp. De acuerdo a la literatura este tipo de microorganismos ha sido asociado con la promoción del crecimiento y la salud microbiana y podrían ser utilizados como marcadores moleculares de un estado intestinal saludable. De igual manera, en la investigación realizada por Ma et al. (2015), el ensamblaje de metagenomas combinado con el análisis de anotación taxonómica ejecutado en MEGAN y el análisis funcional utilizando KEEG; pudo evidenciar un nivel significativamente alto de genes de resistencia a la tetraciclina, eritromicina y aminoglucósidos (macA-macB y tetA-tetR), en las heces de aves adultas comparado con las muestras fecales de humanos y cerdos. Por otra parte se determinó la presencia de elementos móviles (aadA5, dfrA17) en heces fecales de pollos y en microorganismos patógenos humanos. Estos resultados indican la relación entre diferentes genes de resistencia antibióticos encontrados en una variedad de entornos y huéspedes.
Adicionalmente, Xiong et al. (2018), determinaron la abundancia de los genes de resistencia a antibióticos usando ARGs-OAP: herramienta en línea para la detección de genes de resistencia a antibióticos a partir de datos metagenómicos. El análisis bioinformático facilito la comprensión de cambios en la sucesión de la microbiota cecal de pollos y las variaciones en los genes de resistencia antibióticos cuando el huésped es tratado con niveles bajos y terapéuticos de clortetraciclina. Por ejemplo se encontró una disminución de genes a la tetraciclina (tetA y tetW) y la inhibición de genes resistentes a múltiples medicamentos (mdtA, mdtC, mdtK, ompR) con la dosis terapéutica de tetraciclina. Estos hallazgos son fundamentales en la optimización de los esquemas terapéuticos para microorganismos patógenos presentes en aves de corral y soporte para abordar estrategias terapéuticas alternativas a los mismos.
CONCLUSIONES Y PERSPECTIVAS FUTURAS
Fortalecer el conocimiento sobre el microbioma de aves de corral mediante estudios metagenómicos facilita comprender y obtener información detallada sobre la dinámica de las comunidades microbianas y rol de estas en el metabolismo y el estado de salud y bienestar de las aves.
Es importante mencionar que los estudios realizados se enfocan principalmente en identificar el perfil de las poblaciones bacterianas presentes, lo anterior puede estar influido por la enorme diversificación de cada sección del tracto gastrointestinal. Además de las variaciones y desviaciones con respecto a los métodos utilizados para la extracción de ADN, la selección de las regiones hipervariables del gen 16S y la caracterización en general dificultan la comparación de las investigaciones realizadas lo cual conduce a resultados no comparables. En consecuencia, los resultados obtenidos de la identificación de comunidades bacterianas asociadas a diferentes estrategias de alimentación y a la influencia de microorganismos patógenos no pueden ser en su totalidad concluyentes por las diferencias y desviaciones encontradas en los experimentos.
Por lo tanto, para incentivar las investigaciones de la microbiota del tracto gastrointestinal, se debe incluir un método estandarizado, con características similares a las establecidas en el protocolo de investigación del microbioma humano. De esta manera, el estudio de la microbiota del tracto gastrointestinal de aves de corral facilitaría la compresión de las funciones y del papel de los microrganismos en la mediación del crecimiento del huésped bajo diversas condiciones ambientales, como variación en los nutrientes, exposición a patógenos, estrés, entre otros. Con la información obtenida se puede implementar y optimizar la productividad y calidad del huésped teniendo presente que es una de las fuentes principales de proteína del ser humano.
Por otra parte, el uso de secuenciación de próxima generación en estudios metagenómicos en aves de corral proporciona un análisis a gran escala de la biodiversidad y abundancia de microorganismos. Facilitando un análisis completo y complejo de las comunidades microbianas es posible acceder a información en un único análisis sobre la presencia, cantidad, tipo, virulencia, predicción de roles nutricionales y resistencia a antibióticos de un determinado grupo de microorganismos. Por otra parte, este nuevo enfoque metagenómico ha permitido el desarrollo de análisis genómicos funcionales completos para delinear los requisitos genéticos requeridos para la colonización, infección y supervivencia de microorganismos patógenos en condiciones ambientales a las que este se encuentra expuesto. El desarrollo y la aplicación de tales herramientas novedosas de la metagenómica funcional pueden revelar vulnerabilidades desconocidas previamente, las cuales pueden explorarse para desarrollar nuevas intervenciones y romper la cadena de transmisión de microorganismos patógenos en la industria avícola.