INTRODUCCIÓN
Entre los hidrocarburos aromáticos, los hidrocarburos monocíclicos: benceno, tolueno, etilbenceno y xileno (comúnmente denominados BTEX) se han considerado como los principales contribuyentes en el deterioro del agua y el aire (El-Naas, Acio, & El Telib, 2014). En comparación con otros hidrocarburos, los BTEX tienen una solubilidad en agua considerablemente mayor (Faizal et al., 2005). La solubilidad en agua es un factor esencial que determina el destino y el transporte de estos compuestos en el subsuelo; en el caso de los BTEX, su solubilidad les permite viajar fácilmente desde derrames o filtración de tanques de almacenamiento a aguas subterráneas mediante procesos de advección y dispersión, y de ahí a fuentes de agua potable (Ahmed, Sik Ok, Jeon, & Ra, 2019; Hazrati et al., 2016). De acuerdo con la OMS, estos compuestos son considerados neurotóxicos y mutagénicos en la salud humana (Baghania et al., 2017); y la Agencia de Protección Ambiental de Estados Unidos (EPA, por sus siglas en inglés) los ha categorizado como uno de los contaminantes prioritarios para la salud humana y medio ambiente (Na, Moon, & Kim, 2005).
En condiciones favorables, algunos microorganismos presentes en los ecosistemas contaminados tienen la capacidad de utilizar ciertos compuestos orgánicos tóxicos, incluyendo los BTEX, como fuente de energía y de carbono, llegando a mineralizar estos contaminantes a compuestos orgánicos simples como dióxido de carbono y agua (Ugochukwu & Fialips, 2017; Kuhad & Gupta, 2009). Actualmente, se ha identificado gran variedad de bacterias que pueden degradar BTEX bajo condiciones aerobias, bacterias del género Pseudomonas, Acinetobacter, Auriginosas, Comamonas, Bacillus, Burkholderia, Terrimonas, Rodoccocus, Candida (Dellagnezze, Gomes, & Maia de Oliveira, 2018; Claro et al., 2018; Khodaei et al., 2017).
Para la degradación aerobia de BTEX, se usa como compuesto modelo al tolueno, ya que es el compuesto de más fácil transformación debido a la presencia de su grupo sustituyente en el anillo, lo cual ofrece una ruta alterna de ataque en la cadena lateral u oxidación del anillo aromático (El-Naas, Acio, & El Telib, 2014). Existen cinco rutas metabólicas aeróbicas encontradas en bacterias para la degradación de tolueno, estas rutan son TOD, TOL, TOM, TBU y TMO (Ramos et al., 2004). La mayoría de las vías enzimáticas tienen la capacidad de transformar no solo tolueno, sino entre tres a cinco compuestos hidrocarbonados diferentes (Nagarajan & Loh, 2015).
Con el propósito de obtener microorganismos con capacidad de degradar compuesto xenobióticos peligrosos como el tolueno, en este estudio, se aislaron y caracterizaron bacterias endémicas colombianas con capacidad de utilizar este compuesto como única fuente de carbono.
MATERIALES Y MÉTODOS
Reactivos químicos y medios de cultivo
La fuente de carbono principal de este estudio fue tolueno (Merk, USA). Los medios de cultivo utilizados fueron; Medio mineral Caldo Bushnell-Haas (BH) (Bushnell & Haas, 1941) compuesto por KH2PO4 1g/L, K2HPO4 1 g L-1, NH4NO3 1 g/L, MgSO4 0,2 g/L, FeCl3 0,05 g/L y CaCl2 0,02 g/L de la marca Himedia. Para la preparación de medio sólido para crecimiento de bacterias en cajas Petri se le añadió agar-bacto a una concentración de 15 g/L al medio BH; Medio Luria Bertani (LB) compuesto por triptona 10 g/L, extracto de levadura 5 g/L y cloruro de sodio 5 g/L de la marca Pronadisa. Adicionalmente, se usó medio LB agar, con la misma composición más 15 g/L de agar de la marca BF Biosciences.
Muestreo de suelos contaminados con hidrocarburos
Las muestras de suelo contaminado fueron obtenidas en tres lugares contaminados con hidrocarburos del departamento Valle del Cauca, Colombia, estos muestreos fueron realizados entre los meses noviembre 2018 a febrero 2019. Muestreo en el municipio de San Pedro (3°59’N, 76°13’W), realizado en las riberas de la quebrada Arrieta donde en el año 2012, se derramó más de 25.000 galones de gasolina a causa de un atentado terrorista a un poliducto de la empresa Ecopetrol; muestreo realizado en Palmira, Vía Candelaria (3° 30’N, 76° 18’W), en una estación de servicio con venta de gasolina, Diesel y ACPM; muestreo realizado en Cali (3° 28’N, 76° 30’W), en taller mecánico informal que repara los automóviles en el andén y jardín contiguo. En cada lugar de muestreo se tomaron 15 g de suelo superficial a 10 cm de profundidad. La colecta contó con el permiso Otrosí No.16 (17 de septiembre de 2018) al Marco del Contrato de Acceso a Recursos Genéticos y sus Productos Derivados No. 121 del 22 de enero de 2016.
Las muestras se almacenaron en bolsas plásticas estériles y fueron transportadas al laboratorio de Sanidad y Microbiología Agrícola de la Universidad Nacional de Colombia Sede Palmira, donde fueron tamizadas para retirar piedras, animales y material orgánico. Posteriormente, las muestras fueron correctamente rotuladas y almacenadas en tubos falcón estériles de 50 mL a 4°C, hasta su utilización.
Aislamiento de bacterias con capacidad de degradar tolueno
Las muestras de suelo fueron puestas a aclimatación disponiéndose en matraz de 250 mL, con tolueno fase vapor, se adicionó 1 mL del hidrocarburo en una burbuja de vidrio al interior del matraz, el tolueno se evapora y entra en contacto con la muestra (ver Figura 1). Las muestras, se llevaron a incubación con agitación orbital por 8 días, a una temperatura de 28 °C. Después de la aclimatación, se tomaron 4 g de cada muestra de suelo y se mezclaron homogéneamente con 8 mL de agua destilada estéril. A partir de esta solución se tomó 3mL de sobrenadante para una dilución del 10% en 27 mL del medio Bushnell-Hass (BH), en matraz de 250 mL con tolueno fase vapor y se llevó a incubación con agitación durante 5 días, a una temperatura promedio de 28 °C. Posteriormente, se realizó transferencia de 3 mL de cultivo a 27mL de medio fresco de BH con tolueno fase vapor, dispuesto en burbujas dentro del matraz, se llevó a incubar a temperatura de 28 °C, con agitación durante dos días. Pasadas las 48 horas, nuevamente 3 mL de cultivo se pasaron a 27mL de medio BH fresco con tolueno fase vapor, y se llevó a incubar a las mismas condiciones, otras 48 h (Figura 1). Finalmente, diluciones seriadas del segundo cultivo se sembraron en placas de agar BH y se incubaron en un desecador de tapa hermética con un vaso de precipitado que contenía tolueno líquido, a una temperatura de 28 °C durante 48 h. Las colonias seleccionadas fueron sembradas nuevamente en medio líquido para confirmar la utilización de tolueno. Las bacterias con capacidad de utilizar tolueno fueron almacenadas en glicerol al 30% a -20 °C. Se utilizó como control negativo, una inoculación en matraz que contenía medio fresco BH sin tolueno fase vapor.

Figura 1 Matraz Erlenmeyer con burbuja de vidrio incrustada en la tapa de plástico, utilizado en los experimentos de tolueno fase vapor como fuente de carbono. El tolueno se adiciona a la burbuja que porta un hueco por donde el tolueno se evapora y entra en contacto con el medio de cultivo /suelo, dependiendo del ensayo a realizar.
Ensayos de crecimiento de bacterias en tolueno directo
Las bacterias aisladas de cada lugar de muestreo: Palmira, San Pedro, Cali, se colocaron en matraces con caldo BH y tolueno fase vapor, durante 48 h, con agitación y a una temperatura de 28 ºC. Pasados las 48 horas, se tomaron de este cultivo 300 µL para cada réplica, y se realizó una dilución de 1/100 en 3 mL de medio BH con tolueno directo, a una concentración del 1%, en tubos de vidrio capacidad 15 mL. Los cultivos fueron incubados en un equipo con agitación orbital, a 28 ºC por 48 h. El crecimiento se analizó por la aparición de turbidez, medida a una densidad óptica OD a 600 nm empleando el equipo espectrofotómetro Spectroquant Pharo 300 de Merck. Los cultivos se realizaron en triplicado y con control negativo sin inóculo. El nivel alto de absorbancia fue el criterio de evaluación para el crecimiento de los microorganismos en el medio BH usando tolueno como única fuente de carbono.
Caracterización fenotípica
Se evaluaron las características morfológica y bioquímicas de las bacterias aisladas en medio LB después de 48 h de incubación a 28°C. Para la evaluación de las bacterias se tomó de base el Manual de Bergey’s® (Palleroni, 2015; Garrity, Bell, & Lilburn, 2005). En cuanto a morfología de colonias, se detalla forma, tamaño, color, consistencia. Se realizaron pruebas bioquímicas como: tinción de Gram, catalasa, motilidad, agar lisina-hierro (LIA), urea, gelatina, utilización de carbohidratos (glucosa, sacarosa, lactosa, manitol), crecimiento en medio salino (1%, 5%, 20%) y medio King B (KB).
Caracterización genotípica
Amplificación, secuenciación y análisis bioinformático del gen ribosomal 16S
Inicialmente se extrajo el DNA mediante una lisis rápida (Green & Sambrook, 2001). Para la caracterización molecular, se utilizó el análisis del gen ribosomal 16S, donde se amplificó por PCR un fragmento de 1.5 kb empleando los siguientes cebadores: 16S-1492R (5’-GGY-TAC-CTT-GTT-ACG-AC-TT-3’) y 16S-27F (5’-AGA-GTT-TGA-TCM-TGG-CTC-AG-3’). La mezcla de reacción del PCR se realizó en 25 μl, cada compuesto tuvo la siguiente concentración final: 1X de tampón de amplificación Taq, 5mM de mezcla de dNTP, 5 mM de cebador directo 16S-1492R, 5 mM de cebador inverso 16S-27F y 1 U de ADN polimerasa Taq. Las condiciones de PCR utilizadas fueron: 96°C, 3 min; y 25 ciclos de amplificación de desnaturalización a 96°C, 30 s; alineamiento 65°C, 1 min; extensión 72°C, 2 min; extensión final 72°C durante 10 min (Faizal et al., 2005). Los productos de PCR se visualizaron mediante electroforesis en gel de agarosa al 0,8% con buffer TAE 1X (Tris- Acetato-EDTA), y tinción de bromuro de etidio (10 ng/μL) y se purificaron con el Kit Promega Wizard® SV Gel and PCR Clean-Up System empleando el protocolo del fabricante. Posteriormente, se enviaron a secuenciar por el método de Sanger a la empresa Macrogen en Corea del Sur. Para el envío de las muestras se obtuvo el Permiso de Exportación de Especímenes de Diversidad Biológica No. 01649 de la Autoridad Nacional de Licencias Ambientales (ANLA).
Las lecturas de secuenciación del gen ribosomal 16S de cada bacteria fueron ensambladas en el programa CLC Main Workbenchv7.0 (Qiagen®). Las secuencias de nucleótidos obtenidas se analizaron contra las secuencias depositadas en la base de datos 16S ribosomal RNA (Bacteria and Archaea) de GenBank empleando el programa Nucleotide Blast (Blastn) disponible a través del Centro Nacional de Biotecnología (NCBI). Para encontrar las relaciones filogenéticas de las bacterias aisladas, se realizó un árbol filogenético utilizando el método del vecino más cercano (Neighbor Joining-NJ) mediante el software MEGA X (Kumar, Stecher, & Tamura, 2016). La validez de las relaciones filogenéticas representadas en los árboles obtenidos fue analizada estadísticamente utilizando el método del Bootstrap con 1000 repeticiones.
RESULTADOS Y DISCUSIÓN
Aislamiento de bacterias con capacidad de metabolizar tolueno
Las bacterias aisladas se cultivaron en medios con tolueno fase vapor cómo única fuente de carbono para su crecimiento, replicación y supervivencia. Se aislaron 35 cepas en cada lugar de muestreo; derrame por atentado-San Pedro, Gasolinería- Palmira, Taller mecánico-Cali. De las colonias crecidas en las placas suplementadas con tolueno, se eligieron 9 o 10 colonias individuales por cada lugar de muestreo, basados en su crecimiento en BH agar con tolueno vapor, se lograron aislar un total de 29 colonias. Las colonias fueron nombradas como: 3A16, 1B12, 3A8, 1A4, 1A17, 3B4, 3A21, 3B8, 2X16 y 1X3 para las aisladas del lugar de derrame; 1K6, 2K23, 1K20, 2L10, 2K12, 2K13, 1K18, 1K5, 1L24, 1L23 para las aisladas en la gasolinera y 2D13, 6V1R, 6V5R, 6V13, 6V14R, 2V25, 6V10, 6V25, 6V7 para las aisladas en el Taller mecánico.
Identificación morfológica y bioquímica de las bacterias aisladas
Las bacterias aisladas fueron agrupadas en G1, G2 y G3 de acuerdo con sus diferencias morfológicas y bioquímicas (Tabla 1).
Tabla 1 Caracterización morfológica y bioquímica de aislamientos bacterianos que utilizan tolueno como fuente de carbono.
+, crecimiento; -, no crecimiento: +p, crecimiento ligero
El grupo G3, donde se agruparon las diez aisladas de la gasolinería, las diez aisladas del lugar del atentado y la cepa 2D13 aislada del taller mecánico, se identificaron como bacilos Gram-negativos. Las colonias en la placa de agar LB fueron medias, circulares irregulares, planas, de bordes regulares, consistencia cremosa, color amarillo tenue. Las pruebas bioquímicas mostraron que son móviles, aerobias estrictas, con resultado positivo para catalasa, motilidad y descarboxilación de la lisina, crecimiento en medio salino (1% y 5% NaCl), utilización del carbohidrato glucosa, lactosa y sacarosa y capacidad de producir pigmentos en medio King B. Este medio es selectivo para Pseudomonas, pues esta especie tiene la capacidad de producir pigmentos como pioverdina (fluoresceína) y piocianina. La capacidad de hidrolizar gelatina, ureasa y la producción de sulfuro resultó negativa. El grupo G1, donde se dispuso a la bacteria 6V25 y 2V25 del muestreo del taller, fueron identificadas como bacilo Gram-negativos, colonias circulares irregulares, planas, circulares de bordes regulares, consistencia cremosa y color blanco. Las pruebas bioquímicas mostraron que a diferencia del grupo G3, las bacterias del G1 tuvieron un resultado positivo para producción de ureasa. Finalmente, el grupo G2 compuesto por las cepas 6V5R, 6V7, 6V1R y 6V1R de taller mecánico, presentaron diferencias bioquímicas al no utilizar el carbohidrato lactosa y sacarosa y no tener crecimiento en medio salino 5% NaCl.
Identificación molecular de bacterias aisladas
Para conocer la identidad de cada bacteria previamente aislada de suelos contaminados con hidrocarburos, se realizó la amplificación y secuenciación de un fragmento de 1,5 kb del gen ribosomal 16S (Figura 2).
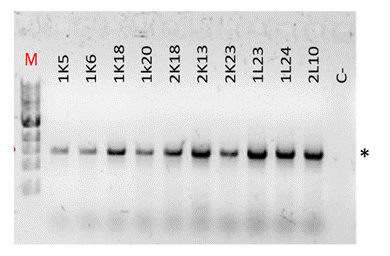
Figura 2 Amplificación del gen ribosomal 16S en 10 bacterias que metabolizan tolueno aisladas en Gasolinería. Se amplificó un fragmento de 1,5 kb empleando los cebadores universales 16S-1492R / 16S-27F y se visualizó en un gel de agarosa al 0,8%. M, marcador de peso molecular Ladder de Biolabs New England. C- representa el control negativo. El asterisco indica el tamaño de 1,5 kb.
Se obtuvieron secuencias de nucleótidos del gen ribosomal 16S entre 1337 - 1417 bp de longitud. El análisis de la secuencia genética con Blastn mostró que todas las bacterias aisladas de la gasolinería presentaron la mayor identidad (99,7%) con Pseudomonas taiwanensis DSM 21245 (NR_116172) (Wang et al., 2010). Del lugar de derrame se identificaron bacterias con porcentajes de identidad altos a tres especies diferentes de Pseudomonas: 3A16 y 3B8 con porcentajes de identidad superior a 99,9% a Pseudomonas plecoglossicida NBR 103162 (Nakagawa et al., 2013); las cepas 1A4, 1A17, 3A8, 3A21, 1B12, 1X3 y 2X16 con porcentajes de 99,4% con Pseudomonas taiwanensis DSM 21245, y finalmente, la cepa 3B4 tuvo una identidad de 98,99% a Pseudomonas resinovorans strain_ATCC_14235 (NR_112062) (Anzai et al., 2000). Para las bacterias aisladas del taller mecánico, se encontraron porcentajes altos de identidad, 99,35%, con Pseudomonas plecoglossicida NBR 103162, para las cepas 6V13, 6V5R, 6V7 y 6V1R; identidades de 99,2% con Pseudomonas taiwanensis DSM 21245 para las cepas 6V10, 6V14 y 2D13; y por último, identidades de 99,64% con Stenotrophomonas maltophilia strain Iam 12423 (Tanaka, Miyazaki, & Sugawara, 2011) para las cepas 6V25 y 2V25.
El análisis de la secuencia de nucleótidos del gen ribosomal 16S no permitió la identificación a nivel de especie de las bacterias aisladas en este trabajo, por lo que fueron denominadas Pseudomonas sp. y Stenotrophomonas sp. Las secuencias del gen ribosomal 16S obtenidas de cada bacteria fueron subidas a la base de datos GenBank con los siguientes números de accesión: Pseudomonas sp. 2K12 (GenBank OM757847), Pseudomonas sp. 2K18 (OM757848), Pseudomonas sp. 1K20 (OM757849), Pseudomonas sp. 1K5 (OM757850), Pseudomonas sp. 1K6 (OM757851), Pseudomonas sp. 1L23 (OM757852), Pseudomonas sp. 1L24 (OM757853), Pseudomonas sp. 2K13 (OM757854), Pseudomonas sp. 2K23 (OM757855), Pseudomonas sp. 2L10 (OM757856), Pseudomonas sp. 1A17 (OM757857), Pseudomonas sp. 1A4 (OM757858), Pseudomonas sp. 1B12 (OM757859), Pseudomonas sp. 1X3 (OM757860), Pseudomonas sp. 2X16 (OM757861), Pseudomonas sp. 3A16 (OM757862), Pseudomonas sp. 3A21 (OM757863), Pseudomonas sp. 3A8 (OM757864), Pseudomonas sp. 3B4 (OM757865), Pseudomonas sp 3B8 (OM757866), Pseudomonas sp. 2D13 (OM757867), Stenotrophomonas sp. 2V25 (OM757868), Pseudomonas sp. 6V10 (OM757869), Pseudomonas sp. 6V13 (OM757870), Pseudomonas sp. 6V14R (OM757871), Pseudomonas sp. 6V1R (OM757872), Stenotrophomonas sp. 6V25 (OM757873) Pseudomonas sp. 6V5R (OM757874) y Pseudomonas sp. 6V7 (OM757875).
El resultado arrojado por el análisis molecular del gen ribosomal 16S está relacionado con el resultado obtenido de las pruebas bioquímicas. Las bacterias aisladas que fueron agrupadas con el grupo G3 en la caracterización bioquímica, estuvieron relacionadas con P. taiwanensis en la identificación molecular, también la cepa 3B4 relacionada con P. resinovorans se agrupó en G3 de acuerdo con su respuesta a las pruebas bioquímicas. Las cepas 6V25 y 2V25, obtuvieron una identidad del 100% entre ellas y 99,636% de identidad con S. maltophilia, tuvieron diferencias bioquímicas y por eso se agruparon en G1 en su análisis. Todas las bacterias relacionadas con P. plecoglossicida del taller mecánico, se agruparon en el grupo G2, sin embargo, las cepas relacionadas con esta misma especie en el lugar de derrame no presentaron el mismo comportamiento bioquímico, se puede inferir entonces que a pesar de su alta identidad en el gen ribosomal 16S, los demás genes que codifican sus rutas metabólicas son diferentes. Esto indica que se requiere analizar a nivel molecular las rutas metabólicas de degradación de tolueno de cada bacteria, así como su capacidad de metabolizar otros compuestos xenobióticos (BTEX).
Se han reportado cepas de P. taiwanensis que contienen un gen que codifica para una proteína de la familia de proteínas de membrana de transporte de la membrana externa (OMPP1 / FadL / TodX), que participan en el catabolismo del tolueno y la degradación de hidrocarburos aromáticos (WP_013971392.1), esta secuencia genética se ha observado en varias cepas de Pseudomonas como P. monteilii, P. plecoglossicida, P. putida entre otras. Hasta la fecha, se conoce que P. taiwanensis, puede utilizar una amplia gama de fuentes de carbono, en los que se incluyen alcoholes de cadena corta y media y tolerancia a solventes como octanol, estireno y tolueno. La información genética disponible sugiere un espectro de tolerancia aún más amplio al poseer proteínas de membrana homólogas a dos proteínas bombas de eflujo de solvente orgánico bien caracterizadas, TtgABC y TtgGHI (Köhler et al., 2013), estas proteínas también han sido encontradas en el genoma de algunas cepas de P. plecoglossicida y P. resinovorans (QLB53749.1; AJG15514.1; AXM96879.1).
Para evaluar las relaciones filogenéticas de las bacterias aisladas, se construyó un árbol filogenético (Figura 3), mediante el método Neighbor-Joining, incluyéndose varias especies degradadoras de hidrocarburos, especialmente BTEX, reportadas en la literatura. Las bacterias cercanas a P. plecoglossicida y P. taiwanensis estuvieron estrechamente relacionadas a Pseudomonas sp. KA-08, esta bacteria que probó ser capaz de degradar benceno, tolueno y xileno, y tolerar dichos compuestos en altas concentraciones (Di Martino, López, & Raiger Iustman, 2012). También tuvieron relación con Pseudomonas sp. PS60, una bacteria aislada en Casanare- Colombia, con capacidad de degradar hidrocarburos en consorcios bacterianos (Lozano et al., 2008).
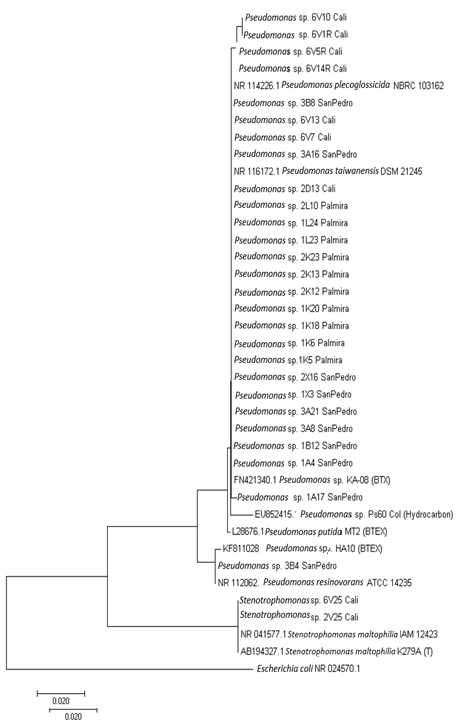
Figura 3 Árbol filogenético obtenido a partir de las secuencias del gen que codifica para el RNA ribosomal 16S, mediante el método Neighbor-Joining. La bacteria Escherichia coli (Número de accesión NR 024570) se usa como grupo externo.
En el árbol filogenético, la bacteria Pseudomonas sp. 3B4 cercana a P. resinovorans ATCC 14235 (identidad 99,9%), se ubicó en una rama diferente a las bacterias aisladas identificadas como cercanas a P. taiwanensis y P. plecoglossicida. Este resultado va de acuerdo con lo reportado en el manual de Bergey’s, ya que P. resinovorans pertenece al grupo de P. aeruginosa (Garrity, Bell, & Lilburn, 2005) diferente a las demás aisladas pertenecientes al grupo de P. putida. La bacteria Pseudomonas sp. 3B4 mostró cercanía con la cepa reportada en la literatura, Pseudomonas sp. HA10 con un porcentaje de identidad del 97%, esta cepa tiene la capacidad de degradar BTEX (Hassan & Aly, 2018). Adicionalmente se ha encontrado que P. resinovorans, tiene capacidad de degradar eugenol, un miembro de los compuestos de la clase alilbencenos, para producir vainilla y ácido vanílico (Ashengroph et al., 2011; Hong et al., 2017).
Stenotrophomonas maltophilia y las bacterias de este estudio cercanas a esta especie, Stenotrophomonas sp. 2V25 y Stenotrophomonas sp. 6V25, se encuentran en una rama distinta a la de las demás bacterias, identificadas como Pseudomonas. S. maltophilia es capaz de degradar compuestos aromáticos sulfónicos, herbicidas (ácido 3,6-dicloro-2-metoxibenzoico), compuestos BTEX y benzoatos (Palleroni, 2015). S. maltophilia K279A reportada en la literatura cercana a Stenotrophomonas sp. 2V25 y Stenotrophomonas sp. 6V25, con un porcentaje de identidad del 99,9% mostró capacidad para degradar tolueno, benceno y otros hidrocarburos (Mukherjee & Roy, 2013).
Crecimientos de bacterias en medio suplementado con tolueno directo 1%
Al analizar el comportamiento de las bacterias aisladas en tolueno directo como fuente de carbono, todas las bacterias aisladas en el taller mecánico obtuvieron valores superiores a las aisladas del lugar del derrame por atentado y la gasolinería. Se obtuvo un crecimiento mayor al 1,600 de absorbancia (densidad óptica 600 nm) para todas las aisladas del taller, siendo Pseudomonas sp. 2D13, Pseudomonas sp. 6V7 y Pseudomonas sp. 6V1R las bacterias que obtuvieron mayor crecimiento: 2,419, 2,418 y 2,227 respectivamente. Mientras que, para las aisladas en el lugar de atentado, se presentaron densidades ópticas entre 1,390 a 0,240, siendo el mayor crecimiento de Pseudomonas sp. 3A16, Pseudomonas sp. 1B12 y Pseudomonas sp. 3A8; Pseudomonas sp. 1,396, Pseudomonas sp. 1,359 y 1,303. Las aisladas de la gasolinería tuvieron los crecimientos más bajos, inferiores a 0,650, siendo Pseudomonas sp. 1K6 la que mostró mayor crecimiento (0,648) (Figura 4).
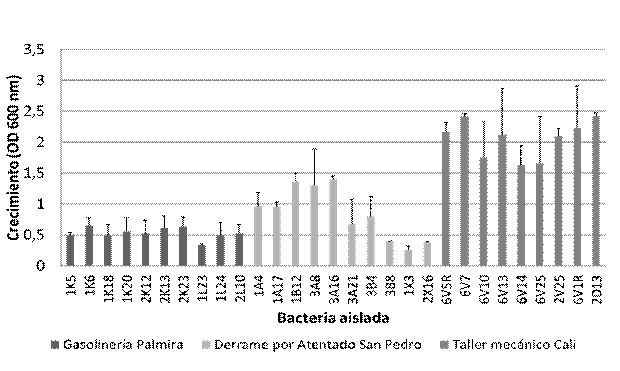
Figura 4 Crecimiento de bacterias aisladas con tolueno directo como fuente de carbono 1%. Las barras muestran el promedio obtenido para cada bacteria y la línea la desviación estándar.
El género Pseudomonas se ha reportado en la literatura como uno de los géneros con mayor adaptación genética para la metabolización de tolueno (Hendrickx et al.,2006; Tan, Tang, & Joannou, 1993). Bacterias del género Stenotrophomonas como las cepas Stenotrophomonas sp. 6V25 y Stenotrophomonas sp. 2V25, ya han sido reportadas como capaces de crecer en un medio de sal mineral que contenga tolueno y benceno como únicas fuentes de carbono (Mukherjee & Roy, 2013; Bashandy, Abd-Alla, & Dawood, 2020). También se ha reportado su capacidad para degradar fenol, cloroformo, tricloroetileno (TCE) (Patel & Patel, 2020).
Es probable que este crecimiento de las bacterias del taller mecánico se deba a que es el sitio de muestreo con mayor probabilidad de contaminación por hidrocarburos proveniente de gasolina, aceite de motor y lubricantes usados por el personal del lugar. El montallantas es informal, no cuenta con las regulaciones y normativas apropiadas para el uso y manejo de estos compuestos y sus desechos, como si lo tiene la gasolinería de la ciudad de Palmira. El suelo del muestreo ha estado expuesto a estos hidrocarburos en forma de vapor y líquidos alrededor de varios años, mismo tiempo que lleva en funcionamiento el taller mecánico, adicionalmente frente al taller mecánico, situada a pocos metros se encuentra una bomba de gasolina que lleva en funcionamiento más de 8 años (información suministrada por la comunidad del sector). Estos resultados confirman que la exposición repetida o constante a productos derivados del petróleo en un lugar, generalmente aumenta la capacidades y posibilidad adaptativa de los microorganismos y, a su vez aumenta la tasa de degradación al ser expuestos nuevamente un compuesto (Martinez et al., 2015). Los talleres automotriz y cambio de aceite, son ampliamente utilizados para el aislamiento de microorganismos con capacidades de degradar hidrocarburos (Delgadillo-Ordoñez et al., 2017).
Si bien las cepas aisladas del derrame en San Pedro, no obtuvieron resultados tan altos como los reportados en las aisladas del taller mecánico, se pudieron observar densidades ópticas superiores a 1,000 (OD 600 nm). Es importante resaltar que la emergencia por contaminación en este lugar fue un evento fortuito ocurrido en el año 2012, siete años antes de la fecha de muestreo, en fechas posteriores no se reportó en este municipio otro incidente de tal magnitud. Sin embargo, se encontraron en este ensayo, valores de crecimiento altos para algunas bacterias, se infiere que, a pesar del paso de los años, los microorganismos que lograron sobrevivir y adaptarse a la emergencia, replicaron en su descendencia la información genética requerida para la metabolización del tolueno, quedando así a disposición de las bacterias en el momento que las condiciones ambientales pudieran repetirse. Se conoce que la adición de hidrocarburos en un ecosistema da lugar a cambios en el tamaño y la composición de la comunidad microbiana. Además, en los lugares con derrames de hidrocarburos en dónde no se realiza extracción o mitigación física del hidrocarburo contaminante, el proceso de degradación microbiana es el mecanismo principal para la eliminación de hidrocarburos derramados (Benka-Coker & & Ekundayo, 1997). Chaerun et al., 2004, estudiaron el comportamiento de bacterias con capacidad de degradar hidrocarburos en lugares donde ha habido eventos accidentales de derrame de hidrocarburos, 5 años después del acontecimiento, encontrando que la morfología, la población de las bacterias y su capacidad para degradar los hidrocarburos no presentaba diferencias significativas al compararlos con los datos iniciales en el año del derrame.
CONCLUSIÓN
En este estudio, se han aislado y caracterizado bacterias de los géneros Pseudomonas y Stenotrophomonas con capacidad de crecer y metabolizar tolueno, compuesto xenobiótico modelo para la degradación de los BTEX. Presentándose los mayores crecimientos en las bacterias aisladas de un taller mecánico informal, lugar donde las aisladas están en constante exposición a la contaminación de hidrocarburos, dado por la manipulación humana de aceites, gasolina y lubricantes. Las bacterias encontradas en este estudio pueden ser utilizadas en un futuro en consorcios bacterianos para procesos de remediación en suelos y aguas contaminadas.















