INTRODUCCIÓN
El proceso de lodos activados es el tratamiento biológico más utilizado para la depuración de las aguas residuales urbanas en el mundo (Daigger, 2014; Xia et al., 2018). A pesar de que este proceso depende principalmente de la actividad metabólica de comunidades microbianas, aún es difícil incorporar el conocimiento de estas comunidades a la comprensión de este proceso biotecnológico. Lo anterior explica por qué este proceso se continúa operando como un modelo de caja negra (De los Reyes et al., 2015; Xia et al., 2018). Por lo tanto, se requieren más esfuerzos para comprender la dinámica de esta comunidad, pues sin duda es más compleja de lo que asumen los modelos actuales y, sus fluctuaciones pueden afectar el desempeño del sistema.
El progreso en las técnicas de secuenciación masiva ha permitido aproximarse a la composición taxonómica de la comunidad microbiana de los lodos activados y describir el potencial funcional algunas de estas comunidades (Dueholm, 2022; Guo et al., 2017; Tian & Wang, 2020; Xie et al., 2021). Además, se ha avanzado en el análisis de la relación entre la composición de la comunidad bacteriana y las variables ambientales, de operación y de funcionamiento entre diferentes plantas de tratamiento de aguas residuales (PTAR) (Chen et al., 2020; Wu et al., 2019). A pesar del carácter dinámico de los sistemas de lodos activados, se ha prestado menos atención a la variabilidad temporal de la estructura de la comunidad y su potencial funcional. Por lo tanto, conocer la dinámica temporal de las comunidades bacterianas y su impacto sobre el potencial funcional es un paso fundamental en la comprensión de la microbiología de lodos activados.
En general, las comunidades bacterianas de las PTAR pueden estar sometidas a fluctuaciones en la composición del afluente en función de la proporción de descargas domésticas, industriales y de escorrentía. Por lo tanto, es lógico pensar que la estructura de la comunidad bacteriana responda a esas fluctuaciones generando cambios en su composición taxonómica (Amanatidou et al., 2016; Sato et al., 2016; Tan et al., 2021). Dado que diferentes taxones son capaces de llevar a cabo una misma función (i.e. redundancia funcional) (Kuypers et al., 2018; Louca et al., 2018), se ha observado que la composición taxonómica de la comunidad bacteriana no necesariamente conduce a cambios en el funcionamiento del sistema de tratamiento (Chen et al., 2020; Fan et al., 2018; Fernandez-Gonzalez et al., 2016); contribuyendo de esta manera a la estabilidad funcional del sistema. Por lo tanto, para avanzar en la asociación de la dinámica de la comunidad bacteriana al proceso de tratamiento es preciso enfocarse en los cambios a nivel funcional más que en aquellos que ocurren a nivel taxonómico.
La metagenómica es una herramienta que se ha utilizado para obtener perfiles funcionales de comunidades microbianas; pero, esta aproximación resulta ser una alternativa muy costosa cuando se busca analizar múltiples muestras de comunidades complejas (Agrawal et al., 2019). En respuesta a lo anterior se han venido implementando herramientas como Tax4fun2 y PICRUSt; que permiten predecir el perfil funcional de la comunidad basándose en el gen marcador ARNr 16S. Aunque se han llevado a cabo varios estudios para caracterizar el potencial metabólico de comunidades bacterianas en las PTAR usando PICRUSt (Ahmed et al., 2017; Fan et al., 2018; Lin et al., 2019), pocos estudios han explotado el potencial de esta herramienta para el análisis temporal de los perfiles metabólicos de comunidades complejas como aquella que funciona en el proceso de lodos activados.
En este trabajo se implementó el uso de la herramienta PICRUSt2, para analizar la variación del potencial metabólico de la comunidad bacteriana del lodo activado a lo largo de un ciclo anual y relacionar dicha dinámica con algunas variables ambientales y operacionales de una PTAR que trata el agua proveniente de un alcantarillado que combina aguas residuales industriales, domésticas y de escorrentía.
MATERIALES Y MÉTODOS
Descripción de la planta y muestreo
La planta de tratamiento de aguas residuales elegida para este estudio trata un caudal promedio de 1,3 m3-s-1 proveniente de un sistema de alcantarillado que combina aguas residuales industriales, domésticas y pluviales. Para este trabajo, se recolectaron un total de 66 muestras de lodo activado en el tanque de aireación de la PTAR con una frecuencia de muestreo diaria, aproximadamente durante una semana en cada mes, entre enero y octubre de 2018 (exceptuando julio); rango temporal que cubre la variabilidad climática en la región. Por cada muestra se tomaron dos réplicas para la extracción de ADN. Para esto, se centrifugaron por duplicado 13 mL de muestra a 4.000 rpm por 10 minutos y se transfirieron 0,5 g del pellet a tubos de 5 mL del kit DNeasy® Power-Soil® (Qiagen). Estas submuestras fueron almacenadas a -20° C hasta la posterior extracción del ADN. La empresa a cargo de la operación de la planta suministró los datos correspondientes a edad de lodos, concentración de DBO5 del afluente y porcentaje de remoción de DBO5. Los datos de precipitación se tomaron del SIATA (Sistema de Alerta Temprana del valle de Aburrá) (SIATA, 2018).
Extracción de ADN, amplificación y secuenciación del gen ARNr 16S y procesamiento de las secuencias.
Se utilizó el kit comercial DNeasy® PowerSoil® (Qiagen) para extraer el ADN comunitario siguiendo las instrucciones del fabricante; se amplificó y secuenció en el Institute of Urban Environment (Chinese Academy of Sciences) la región V4 del gen ARNr 16S de la comunidad bacteriana presente en las muestras de lodo. Para ello, se utilizó el cebador forward 515F-Y (5'- GTGYCA-GCMGCCGCGGTAA), modificado del cebador forward 515F-C de Caporaso et al. (2012) y, el cebador reverse 926R (5'-CCGYCAATTYMTTTRAGTTT) propuesto por Quince et al. (2011). Los productos de amplificación fueron secuenciados usando Illumina MiSeq con protocolo paired-end. Posteriormente, se siguió la ruta de trabajo de DADA2 (Callahan et al., 2016) a través de la plataforma QIIME2 (Quantitative Insights Into Microbial Ecology) (Bolyen et al., 2019) para procesar los datos crudos y generar secuencias apareadas de alta calidad libres de quimeras, para cada muestra. De este procedimiento se obtuvo una tabla de ASVs (Amplicon Sequence Variants - 100% de identidad), a partir de la cual se generó una tabla de OTUs (Operational Taxonomic units) utilizando el método de agrupamiento vsearch de-novo con una identidad del 99% (Rognes et al., 2016).
Predicción de la composición funcional de la comunidad bacteriana del lodo activado
Se utilizó la herramienta bioinformática PICRUSt2 (Douglas et al., 2020) para predecir el perfil funcional de la comunidad con base en las ASV obtenidas. Para esto, se corrió la línea de procesamiento picrust2_pipeline.py, se obtuvieron los identificadores ortólogos funcionales (i.e. números KO) y se realizó la asignación funcional de cada uno utilizando la base de datos KEGG Pathway (Kyoto Encyclopedia of Genes and Genomes) (Kanehisa et al., 2019).
Análisis estadístico
De manera exploratoria, se utilizó el análisis de ordenación PCoA (análisis de coordenadas principales) para visualizar los patrones de distribución de los datos tanto de la composición taxonómica como de la composición funcional de la comunidad bacteriana del lodo activado. Para esta ordenación, realizada en RStudio, se utilizó el índice de Bray-Curtis, además de un análisis de similitud (ANOSIM) con el fin de evaluar la significancia.
El análisis temporal de los procesos de obtención de energía, asociados al metabolismo del nitrógeno y del azufre de la comunidad bacteriana, fue realizado utilizando la herramienta Statistical Analysis of Metagenomic (and other) Profiles (STAMP) (Parks et al., 2014). Por otra parte, se analizó la relación entre la variación en la abundancia de los números KO de algunas rutas metabólicas y el cambio en las variables ambientales y de operación de la PTAR (DBO5 a la entrada del tanque de aireación, precipitación acumulada y edad de lodo), usando el índice de correlación de Spearman en RStudio.
RESULTADOS
Comparación entre la composición taxonómica y la composición funcional de la comunidad bacteriana
A partir de la amplificación y secuenciación de la región hipervariable V4 del gen ARNr 16S, en las 66 muestras, se obtuvo un total de 16.674 ASVs y un total de 2.426 OTUs. PICRUSt2 permitió inferir el perfil funcional de la comunidad bacteriana presente en cada una de las muestras. Así, fue posible explorar simultáneamente los patrones de beta-diversidad taxonómica y de beta-diversidad funcional de la comunidad bacteriana estudiada.
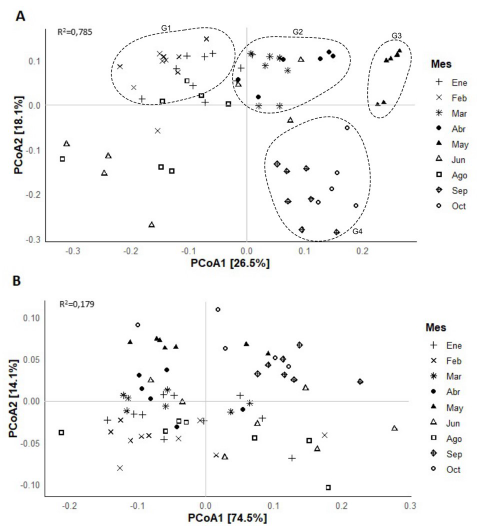
La distribución de la diversidad taxonómica mostró un patrón contrastante al de la diversidad funcional (figura 1). En el caso de la composición taxonómica (figura 1A), se observó la ordenación de las muestras en cuatro grupos: G1 (enero y febrero), G2 (marzo y abril), G3 (mayo) y G4 (septiembre y octubre). Por su parte, en la ordenación de la composición funcional (figura 1B), se observó una agrupación de las muestras solo en mayo, septiembre y octubre. Esto fue coherente con el resultado de la prueba ANOSIM, que evidenció una alta segregación entre los grupos para la composición taxonómica (ANOSIM - R2 =0,785); diferente a lo encontrado para la composición funcional, donde se observó una mayor dispersión entre las muestras de cada mes (ANOSIM - R2 = 0,179).
Análisis de la composición funcional de los procesos de obtención de energía
La predicción del potencial funcional arrojó un total de 6.123 números KO, con un rango de mediciones de NSTI (Nearest Sequenced Taxon Index) entre 0,04 y 0,15 con un promedio de 0,07±0,03, lo que corrobora la confiabilidad de los resultados de la predicción. Luego de realizar la asignación funcional de los números KO predichos se encontraron funciones pertenecientes a 256 rutas metabólicas.
En el conjunto del total de muestras, se encontró que la mayoría de los números KO (48%) pertenecen a la categoría "Metabolismo" del nivel 1 de KEGG, seguida de "No incluida en ruta o Brite" (26%), compuesta por todos los genes que no han sido asociados a una ruta específica de KEGG, lo que implica que se desconoce la función de gran cantidad de genes inferidos para la comunidad de lodo activado estudiada. Detallando un poco más, se encontró que las funciones más abundantes dentro de la categoría "Metabolismo" fueron aquellas relacionadas con procesos metabólicos centrales para las células (i.e. metabolismo de carbohidratos, metabolismo de aminoácidos, metabolismo de cofactores y vitaminas y metabolismo energético).
Algunos de los procesos de obtención de energía importantes para las comunidades bacterianas y de influencia en las plantas de tratamiento de agua residual se encuentran contenidos en las categorías "Metabolismo del nitrógeno" y "Metabolismo del azufre" del nivel 3 de KEGG. Para estas se encontraron 43 y 83 números KO, respectivamente. Con relación al metabolismo del nitrógeno, se hallaron genes para todos los procesos (nitrificación, desnitrificación, reducción desasimilatoria del nitrato, reducción asimilatoria del nitrato y fijación del nitrógeno) exceptuando anammox. En el caso del metabolismo del azufre se encontraron genes de reducción asimilatoria del sulfato, oxidación y reducción desasimilatoria del sulfato y sistema SOX.
Al explorar la dinámica temporal de la abundancia relativa de los números KO asociados con los procesos mencionados anteriormente, se observó que dicha abundancia fluctuó en el tiempo y que el patrón de variación fue diferente entre los procesos (figura 2). Para nitrificación, desnitrificación y reducción desasimilatoria de nitrato se encontró un descenso en la abundancia de genes para los meses de septiembre y octubre (figura 2A, 2B y 2C, respectivamente); contrario a lo visto para reducción asimilatoria de nitrato donde en estos mismos meses hubo un aumento en la abundancia (figura 2D). Por otro lado, la mayor proporción de los números KO, en los procesos relacionados con el azufre, se encontró en los meses de mayo y octubre (figura 2E y 2F).
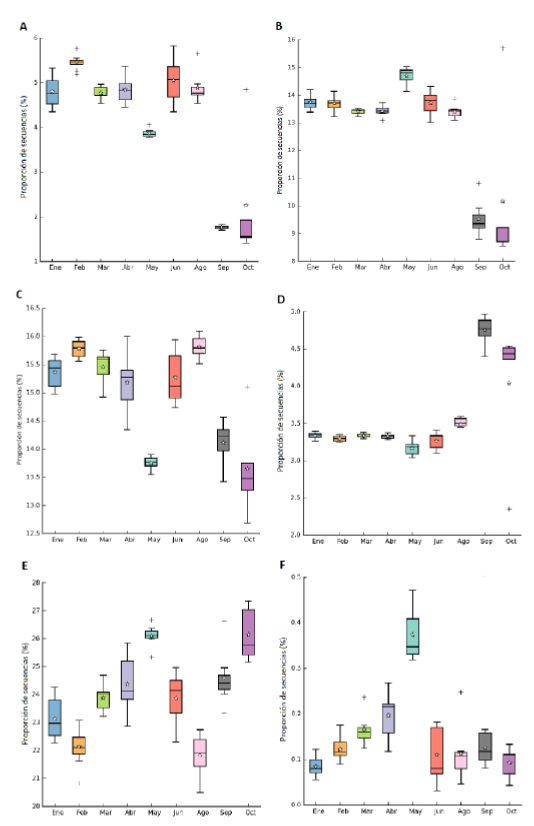
Figura 2 Patrones de variación mensual de la abundancia relativa de los números KO relacionados con las rutas de los ciclos del nitrógeno y azufre (proporción con respecto al total de números KO del metabolismo del nitrógeno y del azufre, respectivamente). A. Nitrificación. B. Desnitrificación. C. Reducción desasimilatoria del nitrato. D. Reducción asimilatoria del nitrato. E. Reducción asimilatoria del sulfato. F. Oxidación y reducción desasimilatoria del sulfato.
También se observó que funciones como nitrificación, reducción asimilatoria de nitrato y oxidación y reducción desasimilatoria de sulfato estuvieron poco representadas en las muestras y que, aunque sus abundancias fluctuaron en el tiempo, siempre se mantuvieron por debajo de 6%, 5% y 0,5%, respectivamente (figura 2A, 2D y 2F). Mientras que, desnitrificación, reducción desasimilatoria de nitrato y reducción asimilatoria de sulfato fueron los procesos más abundantes (figura 2B, 2C y 2E).
Relación entre los procesos de obtención de energía y las variables ambientales y de operación
En el comportamiento anual de la precipitación (figura 3B) se evidencia que los meses de abril, mayo, septiembre y octubre presentaron en 2018 los niveles más altos de precipitación. Esos meses corresponden al período del año en el que la concentración de DBO5 en el afluente de la PTAR presentó los niveles más bajos (figura 3A). Por otro lado, en los meses de agosto, septiembre y octubre, la PTAR trabajó a una mayor edad de lodos en comparación con los otros meses (figura 3C).
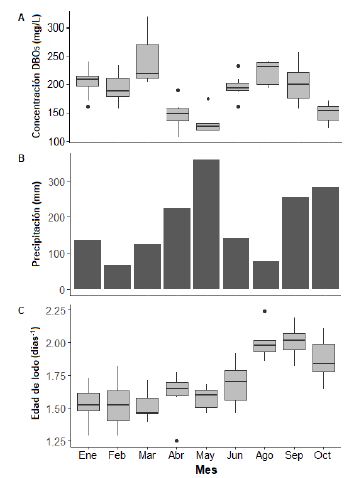
Figura 3 Comportamiento temporal de las variables ambientales y de operación de la planta estudiada A. Concentración de materia orgánica en el afluente (DBO5, mg-L-1) B, Precipitación acumulada (mm). C. Edad de lodo (días-1).
Se realizó un análisis de correlación con el fin de explorar los parámetros de la planta que podrían estar relacionados con la variación temporal en la abundancia relativa de los números KO descrita anteriormente. Se pudo observar que la precipitación acumulada y la edad de lodos se relacionaron significativamente con la mayoría de los procesos (figura 4). Se encontró una relación positiva entre la precipitación y la abundancia de números KO asociados con el metabolismo del azufre. En contraste, la precipitación se relacionó negativamente con la abundancia de KO correspondientes a la reducción desasimilatoria de nitrato y a la nitrificación. Adicionalmente, se evidenció que la concentración de materia orgánica (DBO5) del afluente se correlacionó positivamente con la abundancia de KO asociados a la reducción desasimilatoria y asimilatoria de nitrato y, negativamente con la abundancia de aquellos asociados con la reducción asimilatoria de sulfato. Por otra parte, se observó que la edad de lodos se relacionó negativamente con la abundancia de KO de las rutas de reducción desasimilatoria de nitrato, nitrificación y desnitrificación y solo positivamente con la abundancia de KO asociados con la reducción asimilatoria de sulfato.
DISCUSIÓN
La estabilidad funcional de sistemas biotecnológicos como las PTAR depende de la comunidad microbiana que lleva a cabo la remoción de materia orgánica del agua residual. En algunos estudios experimentales con biorreactores se ha encontrado un rendimiento estable del sistema a pesar de la variabilidad observada en la composición taxonómica de la comunidad bacteriana (Fernandez-Gonzalez et al., 2016; Wang et al., 2011). Este comportamiento podría explicarse por el fenómeno conocido como redundancia funcional, lo cual implica que diferentes especies pueden realizar una misma función (Kuypers et al., 2018; Louca et al., 2018). Al comparar la variación en la composición taxonómica y funcional de la comunidad bacteriana del lodo activado se encontró que la diversidad funcional fue más estable a lo largo del año, mientras que la composición taxonómica en enero-febrero fue diferente a la de marzo-abril y a la de mayo, septiembre y octubre (figura 1). Este comportamiento concuerda con los resultados obtenidos por Chen et al. (2020) y Fan et al. (2018) en otras PTAR, y apoya la idea de que los cambios a nivel taxonómico no siempre conducen a cambios a nivel funcional de la comunidad. Estos resultados sugieren que la composición funcional de la comunidad bacteriana del lodo activado podría ser un mejor predictor del funcionamiento del sistema de tratamiento. Por lo tanto, el esfuerzo por entender los factores ambientales y operacionales que pueden afectar el desempeño de los sistemas de tratamiento de aguas residuales debería enfocarse en explorar las relaciones entre dichos factores y aspectos funcionales de la comunidad bacteriana de los lodos activados.
Los compuestos del nitrógeno, en especial el amonio, son después de la materia orgánica carbonácea los componentes más abundantes del agua residual municipal. Curiosamente, aunque la PTAR estudiada no contaba en ese momento con la configuración necesaria para la remoción de nitrógeno, se encontró un mayor porcentaje de números KO asociados con las rutas anóxicas de reducción desasimilatoria de nitrato y desnitrificación, en comparación con el proceso aerobio de nitrificación. Este resultado podría explicarse por la amplia distribución que tienen los genes asociados a estas rutas anóxicas en diferentes grupos filogenéticos del dominio Bacteria (Kuypers et al., 2018). Adicionalmente, hay que tener en cuenta que este estudio describe solamente el potencial funcional porque se basa en una aproximación predictiva basada en los datos del gen ARNr 16S. Por lo tanto, se desconoce hasta qué punto los genes de una u otra ruta se están expresando bajo las condiciones de la PTAR. Sin embargo, se puede inferir que la comunidad bacteriana de esta PTAR tiene el potencial metabólico necesario para realizar el proceso de desnitrificación si se dieran condiciones anóxicas.
Por otra parte, la forma más oxidada del azufre, el ion sulfato, está comúnmente presente tanto en aguas residuales industriales como domésticas (Delgado Vela et al., 2018; Hao et al., 2014). Teniendo en cuenta la disponibilidad de sulfato en el afluente y la configuración aeróbica del sistema de lodos activados de la PTAR, resulta coherente el haber encontrado una mayor proporción de números KO asociados con la reducción asimilatoria del sulfato en comparación con aquellos relacionados con las reacciones desasimilatorias del mismo. Lo anterior sugiere que la comunidad bacteriana de los lodos activados de esta PTAR tiene un mayor potencial para incorporar el sulfato en biomasa, que para respirarlo anaeróbicamente y producir sulfuro de hidrógeno mediante la reducción desasimilatoria del sulfato.
El hecho de que el sistema de alcantarillado reciba aportes de agua lluvia, expone a la comunidad microbiana del lodo activado a cambios repentinos en el afluente, que pueden alterar el proceso de depuración del agua residual (Amanatidou et al., 2016; Sato et al., 2016). En este contexto, es interesante evidenciar cómo durante los meses más lluviosos (mayo, septiembre y octubre) disminuyó la abundancia de genes relacionados con las rutas de la reducción desasimilatoria de nitrato y de la nitrificación (figuras 2A y 2C), pero aumentó el número de genes asociados con el metabolismo del azufre (figuras 2E y 2F). Lo anterior, concuerda con los patrones opuestos que se observan en las correlaciones entre la precipitación y la abundancia de genes de ambos tipos de rutas (figura 4). Adicionalmente, cabe notar el comportamiento inverso que observamos en la relación entre la abundancia de genes asociados con la reducción desasimilatoria del nitrato y la concentración de materia orgánica (DBO5) en el afluente, en comparación con la correlación con la precipitación; en el caso de la DBO5 la relación es positiva, mientras que con la precipitación es negativa. Este mismo comportamiento inverso se observó en las correlaciones que se obtuvieron con la abundancia de genes asociados con la reducción asimilatoria de sulfato; en este caso la correlación de estos genes con la precipitación es positiva, pero negativa con la concentración de DBO5 del afluente (figura 4). Esa diferencia entre el efecto de la precipitación y el de la concentración de materia orgánica en el afluente sobre la abundancia de los genes podría explicarse por la asociación que hay entre estas dos variables; el agua lluvia al combinarse, en el alcantarillado, con el agua residual causa una dilución en la concentración de materia orgánica del afluente. En cualquier caso, los resultados sugieren que los niveles de precipitación, en el área de drenaje del sistema de alcantarillado de la PTAR, influencian de manera diferencial el potencial funcional de la comunidad bacteriana del lodo activado.
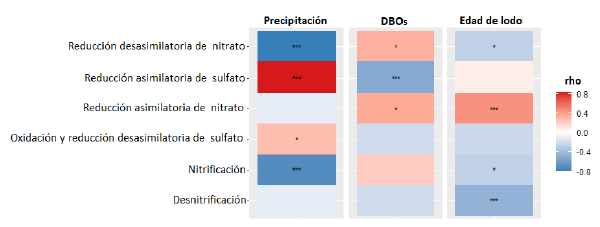
Figura 4 Correlaciones positivas (rojo) y negativas (azul) entre la abundancia de genes de los procesos metabólicos evaluados y las variables precipitación (mm), DBO5 (a la entrada del tanque de aireación -mg-L-1) y edad de lodo (días-1), usando el índice de correlación de Spearman. Los asteriscos corresponden al nivel de significancia: *, p<0,05; **, p<0,01; ***, p<0,001.
Teniendo en cuenta que la edad de lodos brinda una idea del tiempo de retención de los microorganismos en el sistema, se espera que cambios en este parámetro de operación en la PTAR tengan un efecto sobre la diversidad funcional de la comunidad bacteriana estudiada (Amanatidou et al., 2016; Pala-Ozkok et al., 2013). Por lo tanto, el análisis de correlación reveló que un incremento en la edad de lodos favorece la abundancia de los genes asociados a procesos como reducción asimilatoria de nitrato, a la vez que desfavorece la abundancia de otros genes como aquellos relacionados con reducción desasimilatoria de nitrato, desnitrificación y nitrificación (figura 4). Sin embargo, llama la atención el encontrar una relación negativa entre la edad de lodos y la abundancia de genes relacionados con la nitrificación, pues se esperaría que un mayor tiempo de retención favoreciera la proliferación de bacterias de crecimiento lento como las nitrificantes (Siripong & Rittmann, 2007). Por otra parte, se evidenció un efecto combinado entre la edad de lodos y la precipitación sobre la diversidad funcional de la comunidad bacteriana. Esta interacción permite explicar por qué la dinámica temporal de la abundancia de genes es diferente para los meses de lluvia que difieren en la edad de lodos (ver detalle en figura 2, septiembre-octubre vs mayo). Esto lleva a plantear la necesidad de realizar estudios posteriores, con datos que permitan generar modelos de regresión múltiple en donde se cuantifique el efecto de las interacciones de los diferentes parámetros sobre la diversidad funcional de la comunidad bacteriana de este tipo de sistemas.
CONCLUSIÓN
A partir de la predicción del metagenoma se logró describir la dinámica temporal de ciertas funciones de la comunidad bacteriana del lodo activado a escala real. Esta aproximación permitió comparar las composiciones taxonómica y funcional de la comunidad bacteriana; con lo cual se evidenció que cambios a nivel taxonómico no siempre conducen a cambios a nivel funcional. Además, con el análisis de la dinámica temporal de la abundancia de genes relacionados con el ciclo del nitrógeno y del azufre, se mostró que la concentración de materia orgánica en el afluente tiene efectos opuestos sobre estas funciones metabólicas de la comunidad. Por lo tanto, es importante profundizar en el análisis de los factores que influencian la dinámica de la composición funcional para entender mejor los mecanismos que favorecen el funcionamiento estable del sistema de lodos activados.