INTRODUCCIÓN
En la mayoría de las células eucariotas, la presencia de ARN de doble cadena (ARNdc) o ARN bicatenario, genera una respuesta de silenciamiento específico de secuencia conocida como ARN de interferencia - ARNi (RNA interference - RNAi), silenciamiento de genes pos-transcripcional o knockdown génico, el cual se ha convertido en una de las mejores herramientas biotecnológicas para analizar la funcionalidad de los genes (Senthil-Kumar y Mysore, 2010).
La fuente de ARNdc que dispara el mecanismo de silenciamiento puede ser exógena (generalmente virus) o endógena (a partir de genes nucleares), lo cual produce la hidrólisis del ARNdc en pequeños fragmentos que activan ribonucleasas, que posteriormente atacarán moléculas de ARN de cadena sencilla (ARNcs) que contienen secuencias complementarias. Este mecanismo es altamente conservado, ya que regula la expresión de genes que codifican proteínas e interviene en la resistencia a ácidos nucleicos patógenos (Hannon, 2002).
Los primeros indicios de este mecanismo, aunque sin ser reconocido como tal, fueron reportados por Napoli et al. (1990), quienes introdujeron copias del gen que codifica para la chalcona sintasa en petunias (Petunia spp.), una enzima responsable de la coloración púrpura de sus flores. El resultado esperado eran plantas púrpuras; sin embargo, encontraron flores blancas con niveles de la enzima 50 veces menores a los de las plantas originales, indicando una cosupresión del gen endógeno de la enzima. En 1992, Romano y Macino en un trabajo con el hongo Neurospora crassa, observaron que la sobreexpresión del gen relacionado con la producción de caroteno produjo un fenotipo albino. Este fenómeno ocurría a nivel post-transcripcional, por lo que fue denominado "silenciamiento de genes postranscripcional" o PTGS. Posteriormente, trabajos en Caenorhabditis elegans demostraron que el PTGS era debido a un proceso de ARN de interferencia-ARNi (Fire et al., 1998).
Más tarde, Whangbo y Hunter (2008), señalaron que en eucariotas existen dos mecanismos mediados por ARNi. El primero, ARNi de autonomía celular, en el que el silenciamiento está limitado a las células que generan el ARNdc o que están directamente expuestas a un ARNdc exógeno. El segundo, ARNi de autonomía no celular, que se presenta cuando el efecto se propaga a lo largo de los límites celulares y puede actuar en células o tejidos distantes al lugar donde se inició.
El ARNi de autonomía no celular, se divide a su vez en ARNi ambiental, que es observado en respuesta a moléculas de ARN externas presentes en el medio ambiente (Wang et al., 2017) y se ha descrito en gusanos planos, garrapatas, abejas y algunos nemátodos, pero no en vertebrados (Huvenne y Smagghe, 2010), y en ARNi sistémico, que comienza localmente mediante un complejo sistema de señalización y que se expande de célula a célula en forma controlada, siendo exclusivo de organismos multicelulares (Fire et al., 1998) y es importante para restringir las infecciones virales en plantas (Taliansky et al., 2021).
Dada la gran importancia del uso de la tecnología basada en ARNi en diversos campos, con esta revisión se hace una recopilación de información actualizada en cuanto a su aplicación para el control biológico de plagas y fitopatógenos, con el fin de demostrar su gran potencial para la producción de bioplaguicidas efectivos y ambientalmente seguros.
Silenciamiento génico mediante ARNi
El silenciamiento por ARN está dado principalmente por la acción de varias macromoléculas: 1. una molécula desencadenante de ARNdc que activa el mecanismo; 2. la proteína de la familia de las ARNasa III denominada Dicer en animales, o Dicer-like1 (DCL) en plantas (Kurihara y Watanabe, 2004) que cataliza el primer paso del mecanismo de ARNi; 3. un complejo RISC (RNA-induced silencing complex) compuesto por ribonucleo-proteínas al que se ensamblan los fragmentos generados por Dicer (Hammond et al., 2001) y cuyo componente activo son las enzimas Argonautas (AGO), que son proteínas de unión a ARN pequeños no codificantes (Hutvagner y Simard, 2008), y 4. una ARN polimerasa dependiente de ARN (RdRp) (RNA-dependent RNA polymerase) que contribuye a amplificar la señal de inducción del mecanismo mediante la generación de ARN secundarios (Singh et al., 2016).
De manera general, el mecanismo de ARNi comienza cuando la enzima Dicer reconoce y corta el ARNdc en fragmentos de doble cadena de 20-25 nucleótidos (ARNip y miARN), con grupos fosfato en el extremo 5' y dos nucleótidos libres en el extremo 3'. El ARNip dúplex se une a la proteína AGO, y una vez ensamblado en el complejo RISC, es desenrollado y sólo una de las dos cadenas, la cadena guía, dirige el proceso de degradación del ARNm complementario, mientras que la otra cadena, la cadena pasajera, es clivada, liberada y rápidamente degradada durante el proceso de activación de RISC (Gregory et al., 2005), dándose el silenciamiento en la expresión de genes (Dominska y Dykxhoorn 2010; Singh et al., 2016.
Tipos de ARN interferente
Los ARN endógenos pequeños derivan del procesamiento de sus precursores y se agrupan de acuerdo con diferencias en biogénesis y función. Existen ARN pequeños derivados de sus precursores de ARN de cadena sencilla, unos con estructura en horquilla ARNhp (hairpin RNA), que en plantas pueden ser divididos en los micro-ARN y los no microARN; y los ARN derivados de precursores de ARNdc, que corresponden a ARNip (small interference RNA - siRNA) (Axtell, 2013, Tabla 1).
Tabla 1 Clasificación de ARN endógenos pequenos en plantas.
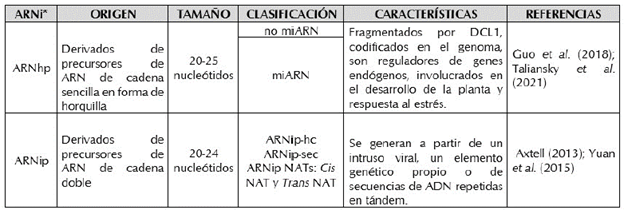
*ARNhp: ARN en forma de horquilla; ARNip: ARN interferente pequeño; miARN: micro ARN; ARNip-hc: ARNip heterocromático; ARNip-sec: ARNip secundario; ARNip NATs: ARNip de transcritos naturales antisentido; DCL1: Dicer tipo 1.
Adicionalmente, se reconocen tres categorías principales de ARN pequeños de acuerdo con su origen, estructura, proteínas efectoras asociadas y funciones biológicas: ARN interferentes pequeños (ARNip); microARN (miARN) y ARN asociados a PIWI - piARN. Estos últimos han sido descritos solamente en células animales y se asocian específicamente con proteínas PIWI (P-element induced wimpy testis), las cuales son miembros de la familia de las proteínas AGO (Axtell, 2013; Ozata et al. 2019). Entre estos, los ARNip y miARN son los más ampliamente distribuidos, ya que actúan en células somáticas y líneas germinales en un amplio rango de especies eucariota.
Protección frente a fitopatógenos
El ARNi le confiere protección a la planta hospedadora frente al desarrollo de infecciones (Koch y Kogel, 2014) y desempeña un papel importante en el control de la expresión de genes frente a respuestas inmunes como mecanismo de resistencia, por ejemplo, contra la replicación viral (Li y Wang, 2019; Jin et al., 2021). Asimismo, protege el genoma de la planta frente a elementos genéticos móviles como los transposones de tipo endógeno (Matzke et al., 2004; Ramakrishnan et al., 2022).
La aplicación de este proceso natural define una tecnologia llamada "Silenciamiento de genes inducido al hospedero" (Host Induced Gene Silencing - HIGS), la cual es una herramienta biotecnológica para la protección vegetal, que combina una alta selectividad para el organismo blanco con mínimos efectos secundarios en comparación con el uso de tratamientos químicos (Wang et al., 2017).
Por ejemplo, se ha demostrado que Botrytis cinerea, es capaz de absorber ARNdc y ARNi ambiental (Wang et al., 2017), por lo que la aplicación por aspersión de moléculas de ARNi dirigidas contra este patógeno en la superficie de verduras, frutas y flores inhibe la enfermedad causada por el hongo. Esta estrategia se denomina "Silenciamiento génico inducido por aspersión" (Spray-Induced Gene Silencing - SIGS) la cual permite el uso de moléculas exógenas de ARNdc y ARNip que son sintetizadas in vivo o in vitro en sistemas alternativos para su posterior administración tópica en los cultivos (Wang et al., 2017).
Otro caso eficaz, se ha reportado en el control de enfermedades en monocotiledóneas. La aspersión con ARNdc dirigido al gen CYP51 de Fusarium graminearum redujo eficazmente los síntomas de la enfermedad en hojas de Hordeum vulgare (Koch et al., 2016). Un ejemplo similar, realizado con F. asiaticum, en plantas de trigo (Triticum aestivum) mostró que la aspersión de ARNdc dirigido al gen miosina 5 (Myo 5) del patógeno, redujo la virulencia e interrumpió el ciclo de vida del hongo (Song et al., 2018). Otros estudios también mostraron que en plantas de Brassica napus y Arabidopsis thaliana afectadas por el patógeno Sclerotinia sclerotium, el tratamiento permitió disminuir el tamaño de las lesiones provocadas por el hongo (McLoughlin et al., 2018).
Durante una infección viral en plantas, el ARNdc viral funciona como inductor del silenciamiento génico. Este, puede provenir de la replicación viral (en virus ARN), transcripción bidireccional del genoma viral (en virus ADN), emparejamiento intramolecular del ARN viral, o de la biosíntesis de ARNdc por acción de ARN polimerasa dependiente de ARN (RdRp) (Boualem et al., 2016; Taliansky et al., 2021). En este caso, los productos virales generados por el mecanismo de silenciamiento se esparcirán sistémicamente por el floema y los plasmodesmos de la planta desde el sitio de inicio de la infección hacia tejidos distantes, lo que desencadena un silenciamiento de autonomía no celular (Boualem et al, 2016).
Se han descrito mecanismos de protección contra infecciones virales a partir de ARNdc producido de manera in vivo e in vitro. Por ejemplo, la aplicación de ARNdc producido in vitro y dirigido hacia la proteína de cápside (coaf protein - Cp) del virus del mosaico del tomate (Tomato Mosaic Virus) confirió protección a las plantas de tomate (Solanum lycopersicum) (Rego-Machado et al., 2020). Adicionalmente, ARNdc derivados de TMV (Tobbaco Mosaic Virus) producidos in vivo en Pseudomonas syringae dieron protección a plantas de tabaco (Nicotiana beníhamiana) contra la infección (Niehl et al., 2018).
Por otra parte, el uso del "Silenciamiento de Genes Inducido por Virus" (Virus Induced Gene Silencing - VIGS) ha permitido la identificación de las funciones de un gran número de genes relacionados con el crecimiento y desarrollo de las plantas, respuestas de defensa y tolerancia a condiciones ambientales, interacciones planta-microorganismo y estrés abiótico (Gupta et al., 2013; Dommes et al., 2018).
Protección frente a insectos plaga
La investigación en ARNi en el campo de la entomología, ha aportado en el estudio de diferentes funciones, regulación y expresión de genes mediante el empleo de diferentes modelos de insectos. El enfoque hacia el control de plagas ha sido dirigido al bloqueo de genes específicos involucrados en el desarrollo, la reproducción u otros, vitales en la supervivencia del insecto (de Andrade y Hunter, 2016). Por lo anterior, es necesario conocer el ciclo de vida y la fisiología del insecto blanco y también algunas variables como el método de administración y dosis, propagación sistémica del ARNi, tamaño de las moléculas de ARNdc, duración del efecto silenciador, sistemas de producción masiva y formulaciones, entre otras (Jain et al., 2020; Borah y Konakalla, 2021).
Producción y aplicación
La aplicación de la tecnología ARNi depende de la calidad, estabilidad y eficiencia de la producción a gran escala del ARNdc. En este sentido, existen diferentes métodos para su obtención: la síntesis enzimática, que involucra una transcripción in viíro mediada por la polimerasa del bacteriófago T7 y la síntesis en sistemas in vivo con células de E. coli HT115 (DE3) deficientes en ARNasa III, transformadas con el plásmido que contiene el inserto del gen de interés usualmente flanqueado por promotores que permiten la transcripción de cada una de las cadenas del ADN (Tenllado et al., 2003). El uso de bacterias recombinantes proporciona ventajas debido a su fácil manipulación, rápido crecimiento y capacidad de contener plásmidos en su interior (Papić et al., 2015). Otros sistemas eficientes para la obtención de ARNdc y ARNhp se han desarrollado en P. syringae (Niehl et al., 2018) y Saccharomyces cerevisiae (Zhong et al., 2019). Existen diversas estrategias para el control de fitopatógenos e insectos plagas, las cuales incluyen enfoques transgénicos (HIGS) y no transgénicos (SIGS) (Figura 1).
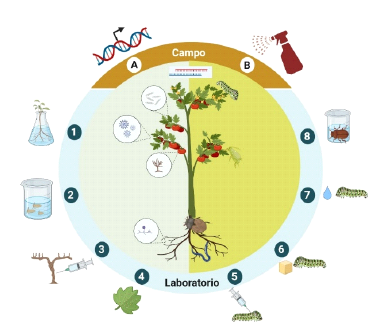
Figura 1 Diferentes estrategias para el control de fitopatógenos e insectos plaga a partir del uso de ARNi en campo y en ensayos de laboratorio. (A) HIGS. (B) SIGS. El lado izquierdo representa los fitopatógenos más comunes (bacterias, virus y hongos), mientras que el derecho muestra invertebrados plaga. Los números indican las principales estrategias de aplicación de ARNi en laboratorio. (1) Inmersión de raíces. (2) Inmersión de semillas. (3) Inyección de ARNi en tallo. (4) Inoculación mecánica de ARNi en hojas con el uso de abrasivos. (5) Inyección de ARNi en invertebrados. (6) Administración en dieta artificial. (7) Administración por gota. (8) Inmersión de insectos. Imagen creada en BioRender.com
El uso de HIGS permite la obtención de cultivos transgénicos que desarrollarán inmunidad contra el patógeno, mediante la expresión de ARNdc endógenos (Wang et al., 2017). Sin embargo, debido a las limitantes que existen para el desarrollo de plantas transgénicas (carencia de protocolos de transformación estandarizados, dificultad en la transformación genética de algunas especies de plantas, altos costos, preocupación sobre el uso de organismos genéticamente modificados (OGM)), se desarrolló una metodología basada en SIGS, la cual es más específica y respetuosa con el medio ambiente y menos perjudicial para la salud del consumidor (Wang y Jin, 2017; Wang et al., 2017).
Tabla 2 Ensayos experimentales basados en ARNi dirigidos a patógenos de plantas.
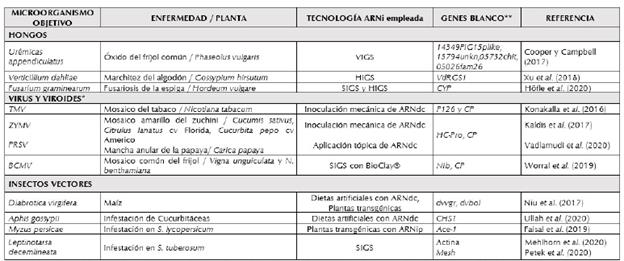
*TMV: Virus del mosaico del tabaco; ZYMV: virus del mosaico amarillo del zuchini; BCMV: virus de la necrosis del mosaico común del frijol, PRSV: virus de la mancha anular de la papaya; ** 14349FICISplike: proteína tipo fosfatasa; 13794unkn: gen de función desconocida; 057J2ch/t: quitinasa; 05026íam26: hidrolasa; VdR.CS): regulador de señalización de proteína C; CYP: citocromo P450 lanosterol C-14α-desmetilasa; CP: proteína de cápside; Pf26: proteína supresora de silenciamiento; HC-Pro: proteína del componente auxiliar Nlb: inclusión nuclear b; dvvgr y dvbol: genes involucrados en reproducción; CHI : quitina sintasa 1; Ace-I: Acetilcolinesterasa 1
La aplicación externa de ARNdc desnudos, es susceptible a la degradación por el efecto de las enzimas en el intestino de los insectos y factores ambientales (Taning et al., 2019). Por esto, el desarrollo de moléculas de ARNdc protegidas por liposomas, polímeros o nanopartículas, garantiza su integridad al ser consumidas por el insecto (Zhang et al., 201; Mitter et al., 2017). Otro tipo de aplicaciones exógenas utilizadas en laboratorio o en experimentos de pequeña escala, incluyen remojo de raíces o semillas, inyección de tronco, absorción de pecíolo y cebos insecticidas (Dalakouras et al., 2019, Liu et al., 2020).
Algunos de los numerosos ensayos experimentales realizados para entender la genómica funcional de las plantas se presentan en la Tabla 2, cuyos resultados promisorios podrían ser la base para futuros bioplaguicidas disponibles comercialmente en el mercado.
Bioseguridad y consideraciones regulatorias de los bio-plaguicidas basados en ARNi
Antes de su comercialización, los bioplaguicidas basados en ARNi deben ser aprobados por parte de las entidades regulatorias de cada país, por lo que se deben desarrollar estudios de bioseguridad, impacto medio ambiental de las moléculas y evaluación de efectos secundarios en microorganismos no objetivo (Taning et al., 2019).
La Autoridad Europea para la Seguridad Alimentaria (EFSA, European Food Safeíy Auíhoriíy) tiene directrices más exigentes, a partir del enfoque "basado en el proceso" en el que el uso de cualquier técnica de ingeniería genética presenta inconvenientes de seguridad y necesita supervisión regulatoria. Ninguna tecnología con ARNdc para el cultivo de plantas ha sido presentada a las autoridades europeas hasta ahora (Jalaluddin et al., 2018). De esta forma, la EFSA ha publicado varias revisiones sistemáticas para respaldar la evaluación de riesgos al usar ARNi en plantas (Christiaens et al., 2018; Dávalos et al., 2019), las cuales proporcionan una descripción general del ARNi y serán útiles para el desarrollo de lineamientos regulatorios para bioproductos basados en SIGS (Taning et al., 2019).
Tabla 3 Productos basados en ARNi liberados al mercado o en espera de aprobación por entidades regulatorias.
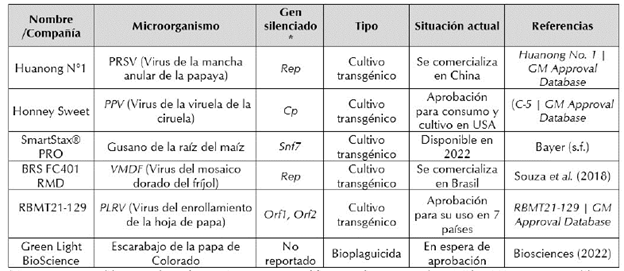
*Rep: gen que codifica para la replicasa; Cp: gen que codifica para la proteína de cápside; Snf7: gen que codifica para la sacarosa no fermentativa 7, Rep: gen que codifica para la proteína de replicación viral; Orfl: Replicasa; Orfl: helicasa.
En Colombia, los alimentos destinados al consumo humano a partir de OGM, solo pueden ser comercializados hasta que cuenten con la autorización por parte del Instituto Nacional de Vigilancia de Medicamentos y Alimentos (INVIMA). El proceso debe cumplir con los requerimientos de la Resolución 4525 de 2005, que evalúa la información técnico-científica relacionada a un evento asociado a OGM. Asimismo, la evaluación de riesgos para la toma de decisiones sobre la autorización o no de un alimento derivado de una planta genéticamente modificada para consumo humano en Colombia, también se regula por convenios internacionales, como el Protocolo de Cartagena sobre Seguridad de la Biotecnología (Ley 740 de 2002) y el Codex Alimentarius (Decreto 977 de 1998, Alimentos transgénicos, s. f.).
Casos exitosos de uso de ARNi en agricultura
En los últimos años, las solicitudes de aprobación de patentes relacionadas con el uso de la tecnología ARNi en plantas, se ha incrementado casi 100 veces (Jalaluddin et al., 2018). Estas patentes se distribuyen entre la industria, la academia y agencias de gobierno, e incluyen métodos para la producción de ARNi, aplicaciones, formulaciones y cultivos transgénicos. En la Tabla 3, se reportan algunos de los productos disponibles en el mercado o que están próximos a su comercialización.
CONCLUSIONES Y PERSPECTIVAS
Los bioplaguicidas constituyen un mercado pequeño (aproximadamente US $3000-4000 millones) en comparación con el mercado de pesticidas a nivel global (US $61000 millones). A pesar de esto, se percibe un rápido crecimiento y se prevé que tengan un aumento anual entre el 10 y 20% (Marrone, 2019).
Los plaguicidas basados en ARNi, son más ecológicos y específicos, lo que evita el daño a otras especies benéficas (de Schutter et al., 2022). Su uso y comercialización constituye una poderosa herramienta para sustituir o disminuir el uso de plaguicidas químicos, cuyos efectos tóxicos para el ecosistema y el ser humano, ha llevado a la resistencia de diversas plagas a estos productos y a una gran contaminación ambiental.
De acuerdo con lo anterior, los trabajos de investigación deben centrarse en:
Diseñar moléculas de ARNdc más estables y desarrollar estrategias eficientes para su liberación en las células blanco, ya que las ARNasas endógenas del intestino de los insectos degradan el ARNdc (Kourti et al., 2017).
Entender los mecanismos de resistencia de los insectos a bioplaguicidas con ARNi, para el diseño de tecnologías efectivas (Khajuria et al., 2018).
Indagar cómo se filtran y degradan las moléculas de ARNdc en el medio ambiente, y si representan un riesgo para microorganismos no objetivo.
Generar acuerdos y alianzas estratégicas entre la academia, el sector privado y el gobierno, con el fin de comercializar bioproductos basados en ARNi y establecer directrices regulatorias claras que permitan el uso de esta tecnología (Taning et al., 2019).















