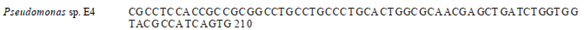INTRODUCCIÓN
El polietilenglicol 400 (PEG) es un fluido no volátil sintético empleado como sustancia química (Fusako, 2005), a temperatura de 17-22°C es líquido, mientras que a temperaturas más bajas puede ser semisólido. Su solubilidad en el agua es su característica más significativa, lo que lo hace ideal para usarse en múltiples aplicaciones (Suhaimi et al., 2021), en Colombia es generalmente utilizado en productos farmacéuticos y como agente osmótico para reducir el estrés hídrico en los floricultivos.
Se ha atribuido al PEG efectos ambientales negativos como la toxicidad en plantas tanto por los iones metálicos como el aluminio, como por la retardada transferencia de oxígeno a la raíz (Suhaimi et al., 2021). En animales, los compuestos muestran evidencia de genotoxicidad (Biondi et al., 2002) y se ha probado que el PEG puede causar hipersensibilidad en personas si estas tienden a tener reacciones alérgicas a medicamentos (Wylon et al., 2016). Pero el mayor problema con los polímeros sintéticos es la persistencia prolongada en el medio ambiente debido a sus estructuras químicas específicas, propiedades fisicoquímicas y una larga vida incluso en ambientes hostiles (Fusako, 2010).
Los macroorganismos aplicados en mecanismos de tratamiento biotecnológico, como lo es la biodegradabilidad aerobia, ofrecen una solución eficaz para reducir los impactos ambientales generados por esta sustancia (Garzón et al., 2017). Tanto la degradación aeróbica como la anaeróbica muestran efectividad que varía según la simbiosis de microorganismos utilizados y el peso molecular del PEG (Huang et al., 2003). Investigaciones de microorganismos en condiciones aeróbicas y anaeróbicas arrojan el efecto del peso molecular - Mw en la eficiencia de los procesos de biodegradación de polietilenglicol donde se encontró que la degradación aeróbica es más eficiente que la anaeróbica por medio de las capacidades de degradación de PEG (Huang et al., 2005).
La biodegradación de PEG por microorganismos ha sido sugerida como el método futuro para remediar vertimientos contaminados con este polímero (Suhaimi et al., 2021). Investigaciones con agua residual contaminada con concentraciones elevadas de PEG 400, presentan una tasa de reducción de hasta un 97% al adicionar biomasa (Chen & Yangdong, 2015; Huang et al., 2003).
Las Pseudomonas se encuentran entre los degradadores más citados para una amplia gama de polímeros plásticos (Wilkes & Aristilde, 2017), por su parte Rhizobium sp. es un género bacteriano caracterizado por su habilidad para producir nódulos en las raíces de las leguminosas, y vivir en asociación simbiótica (Contreras, Iriarte & Muñoz, 2007), es utilizado frecuentemente en asociación junto con otras bacterias de los géneros Flavobacterium y Pseudomonas para transformar PEG (Takeuchi et al., 1992).
Esta investigación buscó encontrar un método eficiente para la eliminación de PEG 400 utilizando la simbiosis entre Pseudomonas sp. y R. trifolii en reactor a escala de laboratorio.
Ruta metabólica del PEG
La biodegradación de un polímero depende de sus propiedades físicas y químicas como el peso molecular y la cristalinidad (Pathak, 2017). El primer criterio para la biodegradación es el procesamiento enzimático (Fusako, 2010). Las enzimas responsables de la degradación de polímeros se clasifican en dos grupos: despolimerasa extracelular y despolimerasa intracelular. Dado que el PEG no es un compuesto natural sino un poliéter sintético, los genes responsables de su metabolismo son un mosaico de genes evolucionados a partir de genes con otras funciones metabólicas celulares (Tani et al., 2007)
Investigaciones de la descomposición aeróbica de PEG han demostrado que la metabolización se lleva a cabo a través de la oxidación por medio de enzimas: una deshidrogenasa de alcohol (aldehído deshidrogenasa) y una enzima bacteriana atacan primero un grupo de alcohol terminal y luego dividen un enlace de éter terminal que rompe el éter produciendo glioxilato - GOA como se observa en la Figura 1 (Suhaimi et al., 2021 - Kawai, 2010).
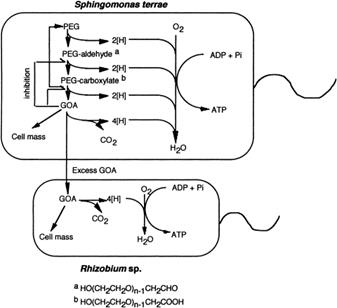
Nota. Se debe a la eliminación de un metabolito tóxico, el ácido glioxílico - GOA que se forma durante el proceso de biodegradación. Tomado de Biodegradations of Polyethers (Polyethilene Glycol, Polypropylene Glycol, Polytetramethylene Glycol, and Others) por Fusako, 2005, Miscellaneous Biopolymers and Biodegradation of Polymers.
Figura 1 Mecanismo de degradación de PEG por cultivo mixto simbiótico Gram Negativo S. terrae y Rhizobium sp.
Thélu et al., en 1980 informan que los extractos libres de células de Pseudomonas sp. crecen en PEG 400 ácido 2-etoxiacético deshidrogenado o derivados de dimetiletilo de diEG y tetraEG, donde la formación transitoria de un doble enlace en una unidad terminal de glicol, seguido de hidratación se representa así: R-O-CH2CH2OH → [R-O- CH=CHOH]→R-O-CHOHCH2OH.
Por otra parte, las alcano hidroxilasas - alkB son las enzimas bacterianas clave en la degradación aeróbica de los alcanos. Estas enzimas hidroxilan los alcanos a alcoholes, que luego se oxidan a ácidos grasos y se catabolizan a través de la vía de oxidación bacteriana (Nie et al, 2014). De Pseudomonas sp. se puede obtener la secuencia de nucleótidos de la alkB (Yoon, Jeon & Nam, 2012):
La actividad deshidrogenasa del ácido diglicólico - DGA presente en Rhizobium, es la responsable de dividir el enlace éter liberando el GOA de todos los sustratos (Enokibara & Kawai, 1997).
MATERIALES Y MÉTODOS
Cultivo bacteriano
A partir de una muestra de borra de petróleo se aisló y mantuvo la cepa de Pseudomonas sp. en Agar Pseudomonas. Desde la raíz de Trifolium repens, se aisló la cepa de Rhizobium trifolii y se mantuvo en Agar Yeast Manitol - YMA, ambas donadas por el laboratorio de Biotecnología Ambiental de la Universidad Distrital Francisco José de Caldas.
Condiciones ambientales
Para precisar las condiciones óptimas de crecimiento de los microorganismos se aplicó una matriz de trabajo 3x33 con condiciones variables de temperatura a 37, 15 y 25°C, concentración de PEG 400 al .1, 1 y 2%, pH de 5.00, 7.00, y 9.00. Para tres réplicas de cada variable, se usaron tubos de ensayo con 10ml de CSMEP y 108 cel/ml/tubo de cada uno de los microorganismos. Los tubos fueron incubados durante siete días. Al finalizar el periodo de incubación se realizó análisis de turbidez y conteo de viabilidad por recuento en placa estándar.
Los parámetros de operación de los reactores fueron agitación constante a 300 rpm, temperatura de 25ºC, tiempo de operación 20 días y pH 7.00 constante.
Ensamblaje de reactores
Se prepararon tres reactores tipo Batch: BK1, BK2 y BK3, con agitación magnética en Biobase MST-H550-Pro®, recipientes herméticos con volumen de trabajo de 2l, tapas con rejilla de HDPE y papel filtro Whatman® con 185mm de diámetro y 8μm de retención de partículas.
Los reactores se cargaron con medio de cultivo así: BK1 se inoculó con una concentración de 8x108 células/ml de Pseudomonas sp., 4x108 células/ml de R. trifolii, sales mínimas a partir de soluciones stock 10X: macroelementos 2ml/100ml y microelementos 1ml/100ml, se adicionó peptona, PEG 400 al .1% y se aforó con agua destilada. Al reactor BK2 se le adicionó la misma cantidad de células de las dos bacterias que a BK1, PEG 400 al .1%, sales mínimas y agua destilada. El reactor BK3 contiene los mismos componentes que BK1, excepto el inóculo bacteriano, de manera que actuara como testigo comparativo del comportamiento del PEG 400. En la tabla 1 se aprecia el contenido de los componentes de cada reactor.
Tabla 1 Componentes inoculados en los reactores.
| Componentes | BK1 | BK2 | BK3 |
|---|---|---|---|
| Bacteria Pseudomonas | + | + | - |
| Bacteria Rhizobium | + | + | - |
| PEG .1% | + | + | + |
| Peptona .1% | + | - | + |
| Sales mínimas | + | + | + |
Nota. Se representa (+) como presencia y (-) como ausencia de componentes.
De cada reactor se tomaron muestras de 10ml, se procedió a realizar conteos directos en cámara de Neubauer donde se identificó el número de generaciones, tiempo de duplicación y la velocidad específica de crecimiento, luego se centrifugaron en HERMLE-Z 446® a 4000rpm por 10min y, por último, se realizó el análisis de la DQO.
Medición de PEG
La DQO es la cantidad de oxidante específico que reacciona con la muestra bajo condiciones controladas, se empleó por considerarse efectiva para medir la variación de PEG 400 en una solución (Sánchez, 2020).
Con una concentración de PEG 400 de .1% y factor de dilución 1/25, se utilizó el método de digestión en sistema cerrado, este es válido para valores de DQO entre 40-400mg/L (APHA et al, 2017). Para mayores valores, se requieren diluciones, como es el caso del presente estudio.
Se emplearon tubos de reacción de 16 x 100mm, un termoreactor VELP Scientifica® y tres réplicas de cada muestra, tomando una muestra por reactor para cada medición. Los volúmenes por tubo de digestión fueron 2.5ml de muestra, 1.5ml de solución digestora y 3.5ml de reactivo de ácido sulfúrico. Para la preparación de soluciones en cantidades y volúmenes idóneos, se siguió la metodología expuesta por Cárdenas, 2015.
RESULTADOS
Biocinética
Una vez realizada la incubación en BK1, BK2 y BK3, bajo las condiciones establecidas en la matriz, se inició la toma de 14 muestras para conteo bacteriano y nueve mediciones para DQO en un lapso de 20 días. Las lecturas se muestran en la tabla 2.
Tabla 2 Crecimiento microbiano en BK1 y BK2.
| Día | Conteo células promedio | Sustrato (ml/L) | Velocidad específica crecimiento (μ) | |||
|---|---|---|---|---|---|---|
| BK1 | BK2 | BK1 | BK2 | BK1 | BK2 | |
| 1* | 8.0E+06 | 8.0E+06 | 2 | 2 | - | - |
| 8.0E+06 | 8.0E+06 | 2 | 2 | 1.4 | 1.3 | |
| 4 | 3.2E+07 | 2.8E+07 | 2 | 1.85 | 3.2E-01 | 3.3E-02 |
| 5 | 4.4E+07 | 3.2E+07 | - | - | 2.0E-01 | 3.4E-02 |
| 6 | 9.6E+07 | 3.8E+07 | 2 | 1.7 | 8.2E-03 | - |
| 8 | 1.0E+08 | 3.8E+07 | 1.85 | 1.5 | - | 6.4E-03 |
| 11 | 1.0E+08 | 4.0E+07 | 1.7 | 1.4 | 2.3E-02 | - |
| 12 | 1.2E+08 | 4.0E+07 | - | - | 4.6E-02 | - |
| 13 | 2.0E+08 | 4.0E+07 | 1.07 | 1.4 | - | - |
| 14 | 2.0E+08 | 4.0E+07 | - | - | -8.1E-03 | -7.5E-03 |
| 15 | 1.8E+08 | 3.6E+07 | .8 | 1.2 | 7.5E-03 | 7.0E-03 |
| 18 | 2.0E+08 | 4.0 E+07 | .5 | 1.07 | - | - |
| 19 | 2.0E+08 | 4.0 E+07 | - | - | -1.2E-02 | -1.2E-02 |
| 20 | 1.6E+08 | 3.2 E+07 | .15 | 1.07 | - | - |
*La medición del día 1 se refiere al inició la fermentación, ocho horas después se realizó una siguiente medición.
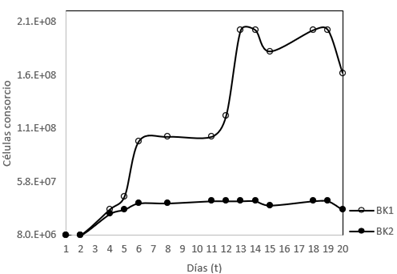
Se alcanzaron 4.64 generaciones bacterianas en BK1 y 2.32 en BK2 donde el tiempo de duplicación de cada generación fue de 2.37 y 4.74 respectivamente. En cuanto a la velocidad específica de crecimiento bacteriano es inconstante, puede deberse a que las bacterias incorporan la mayoría de sus nutrientes por mecanismos de transporte activo, que implica una concentración citoplasmática máxima (Parés & Juárez, 2020), así los cambios en las concentraciones de sustrato alteran la velocidad de crecimiento.
DQO
A partir del consumo del titulante sulfato ferroso amoniacal - FAS en cada muestra, se realizó el cálculo de la DQO. El consumo promedio de FAS por el blanco de referencia fue 2ml, obteniéndose los resultados de la tabla 3.
Prueba de hipótesis
Los valores a evaluar corresponden al cambio de las medias de la DQO en BK1 y BK2. Con mediciones en nueve días, se aplicó la prueba t-student con nivel de confianza del 95% para una hipótesis nula con medias iguales de DQO y medias diferentes como hipótesis alterna.
Al realizar la prueba de hipótesis, no se encontraron diferencias significativas entre las medias de BK1 y BK2, pero sí de sus varianzas. Por ende, para comprobar los cambios en las mediciones de DQO se tomaron los datos desde la quinta medición, cuando se elimina por completo la peptona en BK1 y por ende cambia la media.
Se calcularon las nuevas estadísticas cuyos resultados indican que el estadístico de prueba se encuentra entre la zona de rechazo de la hipótesis nula, por consiguiente, se rechaza Ho puesto que la media de DQO1 no es igual a la media de DQO2.
DISCUSIÓN DE RESULTADOS
Cinética de Crecimiento en los reactores
Al comenzar la fase exponencial en BK1, cerca al séptimo día y antes de entrar a una fase estacionaria el consorcio disminuye su crecimiento, debido a la adaptación del medio al consumo exclusivo de PEG 400 como fuente de carbono, después del quinto día, en BK1, la peptona se ha consumido completamente. En el día 11 se eleva su crecimiento hasta llegar al número máximo de células en el medio y en el día 19 se presenta decrecimiento hasta llegar a la muerte de la población bacteriana. Asimismo, hasta el sexto día BK2 tiene un crecimiento bacteriano permanente hasta el día seis, luego, hasta el día 14, pasa a un crecimiento constante y del día 15 al 19 un incremento celular hasta el descenso que manifiesta el comienzo de la fase de muerte celular, este proceso se advierte en la Figura 2.
El número de generaciones y tiempo de duplicación durante la fase lag en los reactores, es de aproximadamente el doble para ambos casos en BK1, debido a que contiene peptona, que actúa como impulsadora de crecimiento además de ser óptima fuente de nitrógeno.
En los reactores tipo Batch ocurre que el crecimiento bacteriano es inverso al sustrato, en el estadio en el cual se reproducen mayoritariamente las bacterias es en la fase lag, representada desde la segunda medición hasta la doceava como se muestra en los resultados de la tabla 2 y figura 2. A medida que aumenta el número de células por agotamiento del sustrato y por acumulación de productos tóxicos en la bioxidación, la tasa especifica de crecimiento disminuye y el crecimiento declina (Romero, 2008).
Se advierte que los microorganismos asimilan a tasas de consumo variadas las diferentes fracciones de PEG 400 como respuesta a las diferentes afinidades que presentan las bacterias hacia los compuestos disponibles, la cual depende de la estructura y el peso molecular (García & Aguirre, 2014). Esto explica el prolongado tiempo que le toma al consorcio consumir el sustrato.
También, se evidencia que un cultivo mixto de microorganismos trabajando en consorcio, es más eficiente en la degradación de PEG 400 que si se realizan cultivos individuales. En la biodegradación de poliéter realizada mediante consorcios bacterianos cada microorganismo desempeña uno de los papeles necesarios para la degradación final.
Medición de la Degradación de PEG 400
Una razón de seleccionar el método de reflujo cerrado titrimétrico es porque se requieren menos reactivos y se genera menos desechos peligrosos (APHA et al, 2017). El método utilizado prueba la disminución del poliéter, cuando se compara su concentración inicial con las siguientes concentraciones a medida que transcurre el tiempo en los fermentadores.
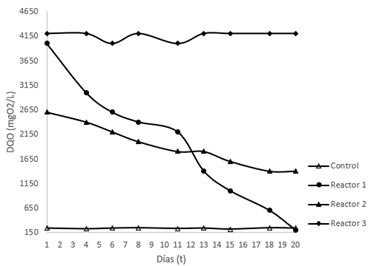
Nota. Se aprecia la decadencia de la DQO en los reactores BK1 y BK2, el control es biftalato de potasio - BP y el blanco BK3.
Figura 3 Medida de DQO en los reactores BK1, BK2 y BK3.
BK1 desciende aceleradamente la concentración del poliéter a diferencia de BK2 que decrece de manera más lenta, lo que muestra que la disminución de PEG es inversamente proporcional al crecimiento bacteriano. La eficiencia de BK1 es de 98.5% y de 88.5% la de BK2 en los 20 días, valores que pueden considerarse como óptimos de remoción del sustrato. El sustrato es consumido lentamente, este método percibe cambios en concentraciones reducidas de diversas sustancias por lo que es adecuado para medir los cambios de una sustancia en un sistema.
La prueba fundamental en la precisión del método se evidencia en la estabilidad del blanco BK3 debido a la ausencia de microorganismos. El resultado analítico del control se encuentra dentro del rango aceptable ± 25% para 200 mgO2/L (IDEAM, 2007).
El método de digestión en sistema cerrado utilizado, en comparación con los demás métodos de DQO presenta mejores condiciones analíticas respecto a su precisión, exactitud, recuperación e incertidumbre (Blanco, 2012).
Confrontación metodológica
De manera análoga, una investigación realizó la medición de la degradación de polietileno por medio de la diferencia de peso de la muestra que estuvo en contacto con un consorcio microbiano que incluye Rhizobium (Figura 4). En este método conocido como “Soil Burial Test”, la disminución en el porcentaje de pérdida de peso se debe a la invasión de microorganismos en el sustrato y la absorción de la humedad por parte de las muestras (Gautam & Kaur, 2013).
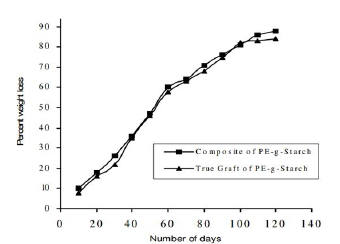
Nota. Tomado de Soil burial biodegradation studies of starch grafted polyethylene and identification of Rhizobium meliloti therefrom. Por Gautam & Kaur, (2013). Journal of Environmental Chemistry and Ecotoxicology.
Figura 4 Porcentaje de pérdida de peso en función del número de días en contacto con Rhizobium meliloti.
El porcentaje de remoción de DQO que se representa en la figura 5, obedece a los resultados del presente estudio. El comportamiento de la DQO explica la biodegradación por acción de la simbiosis bacteriana.
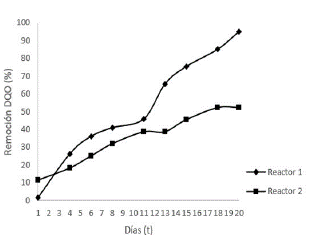
Nota. Porcentaje de remoción en BK1 con peptona como impulsador de crecimiento y en BK2 menor remoción sin peptona.
Figura 5 Porcentaje de disminución de DQO en incubación con Pseudomonas sp. y Rhizobium trifolii.
La figura 6 muestra que el modelo de aguas residuales con PEG 400 puede ser biodegradable con oxidación anaeróbica al hacer uso de lodo digerido, producto de una planta de tratamiento de aguas residuales que contiene una gran cantidad de biomasa, la cual utiliza el PEG como única fuente de carbono y energía. La degradación del poliéter se determinó por el análisis de la DQO y el carbono orgánico total - TOC (Chen & Yangdong, 2015).
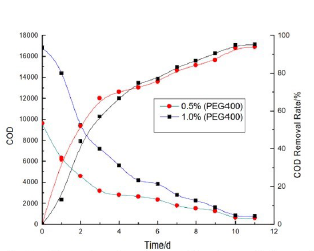
Nota. Tomado de Anaerobic Degradation of Polyethylene Glycol 400. Por Chen & Yangdong, 2015. International Conference on Manufacturing Science and Engineering.
Figura 6 Porcentaje de remoción de DQO durante oxidación con lodo anaeróbico.
En la figura 7, el polietilenglicol de bajo peso molecular - LMWPE se biodegrada al inocularse al medio bacterias mesófilas del género Pseudomonas, donde se demuestra que la biodegradación está definida por la diferencia entre la cantidad de CO2 acumulado con presencia y ausencia de LMWPE sobre la cantidad de CO2 acumulado cuando todos los carbonos se han mineralizado (Yoon, Jeon & Nam, 2012).
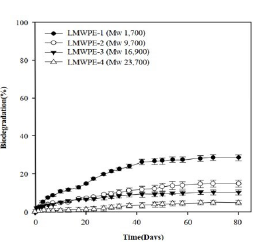
Nota. Tomado de Biodegradation of Polyethylene by a Soil Bacterium and AlkB Cloned Recombinant Cell. Por Yoon, Jeon, Nam, 2012. Journal of Bioremediation & Biodegradation.
Figura 7 Porcentaje de degradación de polietileno con compost inoculado con Pseudomonas sp.
En general, las figuras 4, 5, 6 y 7 presentan un patrón sigmoide donde la transición se produce con base en el metabolismo de los microorganismos presentes en el ambiente de reacción. La degradación de los poliéteres es lenta y está condicionada principalmente por su peso molecular, tiempo de retención hidráulico y una simbiosis acertada.
La eficiencia de remoción de los polímeros varía del 30-98.5% aproximadamente, en la figura 7, sucede una menor eficiencia de remoción (30%) puesto que la actividad de biodegradación disminuye con el aumento del peso molecular que fluctúa entre 1,700-23,700Mw (Yoon, Jeon & Nam, 2012). En la figura 5, la eficiencia del PEG con 400Mw llega al 98.5%.
Los resultados de estas investigaciones también indican la variación de los mecanismos de reacción de una simbiosis a otra que, aunque degradan continuamente y tienen tendencia similar, su comportamiento difiere a medida que avanza el tiempo.
Prueba de hipótesis de comparación de medias
Con una confiabilidad del 95% existen evidencias para aceptar la prueba de hipótesis nula que afirma que no hay diferencia significativa entre los reactores BK1 y BK2 a partir del quinto día.
El valor crítico, es el punto que divide la región de aceptación y la región de rechazo de la hipótesis nula. 1 - α, que se encuentra en la región entre -2.447 y 2.447, límites establecidos por la tabla de la distribución t-student.
CONCLUSIONES
El PEG 400 fue biodegradado en más del 95% por el consorcio de Pseudomonas sp. y R. trifolii en 20 días, de manera que esta simbiosis actúa con eficacia en la disminución del poliéter. Su progresivo crecimiento celular indica que las bacterias tomaron el PEG 400 como única fuente de carbono, es decir, como alimento para reproducirse, aunque la peptona es necesaria para acelerar el crecimiento de los microorganismos y así, reducir su tiempo de duplicación celular.
En una de las investigaciones referenciadas, la eficiencia de remoción de los poliéteres fue cercana al 30% puesto que la biodegradación disminuye con el aumento del peso molecular y una eficiencia máxima del 98.5% obedece a unas condiciones de reacción idóneas con una conveniente simbiosis de microorganismos.
El método de evaluación de la disminución de PEG 400, DQO por reflujo cerrado permite observar los cambios en la concentración del poliéter dentro de los reactores y así refleja un consumo del contaminante evaluado.