Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista U.D.C.A Actualidad & Divulgación Científica
Print version ISSN 0123-4226
rev.udcaactual.divulg.cient. vol.16 no.1 Bogotá Jan./June 2013
CIENCIAS DE LA SALUD-Artículo de Reflexión
TÉCNICA NO CONVENCIONAL DE EXTRACCIÓN DE ADN A PARTIR DE TEJIDO EMBEBIDO EN PARAFINA PARA USO EN LA REACCIÓN EN CADENA DE LA POLIMERASA
UNCONVENTIONAL TECHNIQUE OF DNA EXTRACTION FROM PARAFFIN EMBEDDED TISSUE FOR USE IN THE POLYMERASE CHAIN REACTION
Giovanna Meza1, Juan Carlos Ulloa2, Ana María Uribe3 María Fernanda Gutiérrez4
1Bact. M.Sc. Profesor asistente, Facultad de Ciencia y Tecnología. Universidad de Ciencias Aplicadas y Ambientales U.D.C.A. Calle 222 No.55-37, Bogotá, Colombia. Correo electrónico: gmeza@udca.edu.co
2Microbiol. Ph.D. Profesor Departamento de Microbiología, Facultad de Ciencias Básicas. Pontificia Universidad Javeriana. Carrera 7 # 40 - 62, Bogotá, Colombia. Correo electrónico: julloa@javeriana.edu.co
3Médica, Patóloga, Hospital San Ignacio. Profesor Asistente. Universidad Javeriana. Cra 7 # 40-62 Edificio de Patología.
4Bact. Ph.D. Profesor titular Departamento de Microbiología. Facultad de Ciencias Básicas. Pontificia Universidad Javeriana. Carrera 7 # 40 - 62, Bogotá, Colombia. Correo electrónico: mfgutier@javeriana.edu.co
Rev. U.D.C.A Act. & Div. Cient. 16(1): 35 - 41, 2013
RESUMEN
Los tejidos embebidos en parafina son muestras que reposan en los bancos de patología y almacenan importante información genética, útil para estudios retrospectivos de epidemiología y de patología molecular y para el diagnóstico de enfermedades en humanos y en animales. Dentro de las metodologías descritas para extraer ADN de tejidos en parafina, pocas resultan exitosas a la hora de obtener un material genético, con la cual se pueda amplificar fragmentos superiores a 200pb, por la técnica de PCR. Este trabajo expone la experiencia del laboratorio de biología molecular de la U.D.C.A, relacionada con la aplicación de una metodología poco utilizada para extraer ADN de tejido embebido en parafina y óptimo para amplificar por PCR un segmento de 600pb. Fueron utilizadas dos técnicas ampliamente referenciadas para este tipo de muestras, como son el protocolo de extracción fenólica y el del estuche comercial de QIAGEN® y una tercera metodología comúnmente empleada para extraer ADN de muestras de sangre, pero no para para tejidos en parafina, como lo es la técnica de extracción por sales o salting out. La capacidad amplificable del ADN extraído, se evidenció al obtener un fragmento de 600pb de un gen constitutivo humano. Los resultados mostraron que con la extracción fenólica y el estuche comercial de QIAGEN®, se consiguió el 21% de amplificaciones y con el método de extracción por sales, el 91%. Este trabajo muestra que una técnica no convencional de extracción de ADN a partir de tejido embebido en parafina puede ofrecer mejores resultados que metodologías tradicionales.
Palabras clave: Extracción, ADN, protocolos, Reacción en cadena de la Polimerasa, patología molecular.
SUMMARY
The paraffin-embedded tissues are samples resting in the pathology banks, storing important genetic information, useful for retrospective studies of epidemiology and molecular pathology and diagnosis of diseases in humans and animals. Among the methods described for extracting DNA from tissues in paraffin, few are successful in obtaining the genetic material which can be amplified above 200bp fragments by the PCR technique. This paper describes the experience of U.D.C.A's molecular biology laboratory, related to the application of a methodology recently used to extract DNA from paraffin-embedded tissue and which permits to be optimal amplified by PCR of a fragment to 600pb. Two techniques were used, extensively referenced to these samples, such as the phenol extraction protocol and the commercial kit from QIAGEN®, and also a third commonly used method for extracting DNA from blood samples, but not for tissue in paraffin, the technique of extraction or salting out salts. The extracted DNA amplifiable ability was evidenced to obtain a fragment of 600pb establishing a human gene. The results showed that extraction with phenol and commercial kit from QIAGEN ® achieved amplifications of a 21%, while the salt extraction method a 91%. This work reveals that an unconventional technique of DNA extraction from paraffin-embedded tissue can offer better performance than traditional methodologies.
Key words: Extraction, DNA, PCR, protocols, molecular pathology.
INTRODUCCIÓN
Los tejidos embebidos en parafina (TEP), se almacenan en los bancos de histopatología después de su estudio macro y microscópico. El análisis de los ácidos nucleicos, contenido en dichas muestras, se ha convertido en una herramienta importante para el diagnóstico e investigación en patología, en epidemiología y en endocrinología molecular (Armas et al. 2006; Thyagarajan et al. 2005; Kizys et al. 2012), en genética molecular post mortem, en detección de enfermedades infecciosas (Morales et al. 2005), en estudios de malformaciones congénitas en fetos humanos (Santos et al. 2008) y para todo tipo de estudios retrospectivos (Huijsmans et al. 2010), razón por la cual, la utilización de una técnica de desparafinación y de extracción de ADN, a partir de TEP, se hace cada vez más necesario.
Los proceso de extracción de ADN a partir de TEP incluye tres pasos fundamentales: el primero, la desparafinación, donde se utilizan sustancias químicas, como el xileno y el etanol o procesos físicos, como el calor (Zafra et al. 2004; Cannavo et al. 2012), que buscan eliminar la parafina del tejido en estudio; el segundo paso es la digestión del tejido desparafinado, mediante el uso de enzimas, como la proteinasa K o de soluciones alcalinas a altas temperaturas (Tufan et al. 2004), que desnaturalizan las proteínas del tejido y degradan los fosfolípidos celulares, liberando el ADN (Gilbert et al. 2007). El tercer paso busca la purificación del material genético extraído mediante soluciones, como el fenol- clo- rorformo- isoamilalcohol (Morales et al. 2005; Coura et al. 2005), etanol y resinas quelantes, como Chelex- 100 (Armas et al. 2006; 2011) o las de la casa comercial de QIAGEN®. Todas ellas ofrecen extraer un ADN óptimo para estudios moleculares; sin embargo, a la hora de utilizar dicho material en la técnica de PCR, los resultados pueden no ser favorables.
Debido a la naturaleza del TEP, muchas son las variables que repercuten en el éxito de las técnicas de extracción de ADN, desde el momento de la preservación del tejido para los estudios de patología (Armas et al. 2006), hasta la utilización del ADN, en la técnica de PCR. La literatura reporta que sustancias fijadoras del tejido, como el formol, los alcoholes o los aldehídos ocasionan un entrecruzamiento de las hebras de ADN, modifican las bases y degradan parcialmente la molécula (Tufan et al. 2004; Jiménez et al. 2007). Otras variables, como el tiempo de fijación, el tamaño del tejido fijado, la edad del bloque, la temperatura, el pH de los reactivos, los solventes orgánicos usados en la desparafinación, entre otros, también pueden afectar la calidad de los ácidos nucleicos extraídos y, por lo tanto, la eficacia en la técnica de Reacción en Cadena de la Polimerasa (PCR), en contraste, con el ADN de alta calidad obtenido a partir de tejidos frescos o de muestras de sangre (Gilbert et al. 2007). Por lo anterior, uno de los principales retos en el laboratorio es la utilización de una metodología de extracción de ADN a partir de TEP, que permita obtener una molécula óptima para su utilización en la técnica de PCR.
Con este trabajo, se pretende dar a conocer la experiencia del laboratorio de biología molecular de la Universidad de Ciencias Aplicadas y Ambientales U.D.C.A, en la búsqueda de una metodología para extraer el ADN, a partir de tejido mamario fijado en formalina y embebido en parafina, que cuente con los parámetros de calidad establecidos para pruebas moleculares y que, adicionalmente, permitiera amplificar por PCR un fragmento de 600pb.
Según la literatura, fueron seleccionadas tres metodologías de extracción de ADN a partir de TEP, dos de ellas consideradas como las técnicas más frecuentemente reportadas, cada una con principio químico diferente. La primera, se basa en la extracción y en la purificación del ADN con solventes orgánicos, como el fenolcloroformo y temperaturas de reacción entre 90 y 100°C (Shi et al. 2002; Coura et al. 2005) y, la otra, en la extracción mediante unión selectiva del ADN a columnas quelantes, del estuche comercial de QIAGEN® (Thyagarajan et al. 2005; Gilbert et al. 2007); sin embargo, también se ensayó una tercera metodología ampliamente utilizado para extraer ADN a partir de muestras de sangre, pero no para tejidos embebidos en parafina, como es la metodología de extracción por salado o "Salting out", seleccionada por su disonibilidad en los laboratorios de biologia molcular, facilidad procedimental y bajo costo.
MATERIALES Y MÉTODOS
En el presente estudio, se realizaron 136 ensayos de extracción de ADN a partir de bloques de tejidos mamarios embebidos en parafina, almacenados desde el 2007 hasta el 2009, en el laboratorio de patología, del Hospital San Ignacio de Bogotá, Colombia. Los bloques de parafina fueron sometidos a tres técnicas de extracción de ADN: la técnica de extracción fenólica (fenol- cloroformo), la de extracción por columnas quelantes del estuche comercial de QIAGEN® y la técnica de extracción por sales o Salting out.
Método de extracción fenólica (fenol- cloroformo), descrito por Shi et al. (2002): Se pesaron entre cinco a diez miligramos de TEP y se adicionó un mililitro de xyleno, se incubó a 98°C durante 15 minutos, se centrifugó y se descartó el sobrenadante; al sedimento, se le adicionó un mililitro de etanol absoluto, se agitó manualmente y se dejó en reposo por 30 minutos; posteriormente, se centrifugó a 10.000 rpm durante 11 minutos, se descartó el sobrenadante y se le adicionó 1mL de etanol al 70% y 1mL de buffer fosfato salino, nuevamente se centrifugó a 12.000 rpm durante diez minutos; se descartó nuevamente el sobrenadante y al sedimento se le agregaron 500μL de tampón de lísis que contenía: proteinasa K (20 mg/mL), tris-HCL (1M), 2μL de EDTA (0,5M) y 100μL de SDS (10%); la mezcla se incubó durante toda la noche a 52°C en agitación continua, posteriormente, se le agregaron 500μL de solución de fenol- cloroformo - isoa- milalcohol (25:24:1) y se centrifugó a 12.000 rpm por cinco minutos. El sobrenadante fue removido cuidadosamente a otro tubo y se agregó 0,1 volúmen de acetato de sodio (3M), se homogeneizó manualmente, se adicionó un volumen de isopropanol y se dejó en almacenamiento a -20°C durante toda la noche; luego, se centrifugó y el sedimento que contenía el ADN extraído fue lavado con etanol (70%); luego de secar el ADN, se le adicionó 30μL de buffer TE1X.
Método de extracción por columnas quelantes del estuche comercial de QIAGEN® (Spin- Column Protocol): Se cortaron 25g de TEP y se depositaron en tubos de 2mL con 1200μL de xyleno, se agitó en vortex y se centrifugó a 10.000 rpm durante cinco minutos, a temperatura ambiente. El sobrenadante se pasó a otro tubo y se adicionó 1200μL de etanol absoluto hasta eliminar el xyleno y el etanol. El tejido desparafinado, se resuspendió en 180μL de buffer ATL (no se conoce su composición) y se agregó 20μL de proteinasa K, se mezcló nuevamente y se incubó a 56°C durante toda la noche; luego, se adicionó 200μL de buffer AL (no se conoce su composición), se mezcló y se agregó 200μL de etanol absoluto; esta mezcla se depositó en la columna DNeasy Mini Spin y se centrifugó a 8000 rpm durante un minuto. La columna se depositó en un nuevo tubo de recolección y se adicionó 500μL de buffer AW1; posterior a la centrifugación, la columna fue transferida a otro tubo de recolección y se agregaron 500μL de buffer AW2, se centrifugó a 14.000rpm durante tres minutos. La columna con el material genético extraído, se depositó en otro tubo de recolección y se agregó directamente sobre la membrana 200μL de buffer AE, se incubó a temperatura ambiente durante un minuto y se centrifugó a 8000 rpm por un minuto más, para eludir el ADN.
Técnica de Extracción por sales o Salting out, según Santos et al. (2008): Mediante micro disección manual con una hoja de bisturí estéril fueron recolectados, en microtubos de 1,5mL, entre 15 a 24mg de tejido mamario embebido en parafina (TEP), de las áreas identificadas microscópicamente, con mayor celularidad. Estos cortes, se sometieron al proceso de desparafinación, agregando 500mL de xyleno (CN.158693 de la casa comercial. MPB), se incubó durante 10 minutos en baño serológico (Haake FSL) a 65°C y se centrifugó a 10.000 revoluciones por minuto (rpm) (Biofga BDC), durante siete minutos; posteriormente, con una pipeta pasteur de vidrio estéril, se extrajo cuidadosamente el sobrenadante; este paso se repitió cinco veces. Al tejido desparafinado, se le agregó 1000mL de etanol absoluto y se llevó a incubación en bloque de calor a 37°C durante diez minutos; posteriormente, se centrifugó a 10.000 rpm durante cinco minutos y con la pipeta pasteur de vidrio, se eliminó el sobrenadante y se agregó 1000mL de etanol al 70%, se incubó en bloque de calor (Canlab- BAXTER) a 37°C por diez minutos y nuevamente se centrifugó a 10000 rpm, durante cinco minutos. Finalizado el proceso de desparafinación, se dejó secar el tejido por unos minutos en bloque de calor a 37°C y se agregó 50mL de agua HPLC y 1000mL de solución amortiguadora o tampón TNE (17mM Tris HCl pH: 8,0; 50mM NaCl y 7mM EDTA), se homogeneizó y se centrifugó por diez minutos a 10.000 rpm. Posteriormente, se añadió 1000mL de buffer de lísis (10mM de Tris HCl pH: 8,0; 0,5% de SDS; 5mM de EDTA) y se adicionó 10mL de proteinasa K (N.C. 95H014715 C.C. SIGMA) (20mg/mL), se mezcló con vórtex (Thermolyne BDC) durante cinco segundos y se sometió a incubación en baño serológico con agitación continua, a 55°C durante toda la noche. 500mL de acetato de amonio 8M y EDTA 1mM fueron agregados, se agitó fuertemente durante 15 segundos y un segundo más en vórtex, se centrifugó a 10000rpm, durante 15 minutos; 900mL del sobrenadante fueron transferidos a un tubo nuevo y se adicionaron 600mL de isopropanol frío, se mezcló 20 veces por inversión suave y se centrifugó a 10000rpm, durante 15 minutos. El sobrenadante fue eliminado y al sedimento se le agregaron 300mL de etanol frío al 70%, nuevamente se centrifugó a 10000rpm por ocho minutos y, cuidadosamente, se eliminó el sobrenadante; el procedimiento se repitió una vez más. El pellet obtenido, se dejó secar a 50°C por 15 minutos y se reconstituyó en 30mL de TE 1X.
Las tres metodologías fueron ejecutadas bajo las mismas condiciones de laboratorio y con los mismos equipos previamente referenciados.
Cantidad y calidad del ADN: Todas las muestras de ADN fueron analizadas mediante un equipo nanodrop 1000, que permitió calcular la concentración de ADN obtenido y la relación A260/A280 y A260/A230, que informa el nivel de la pureza de la molécula. El tamaño del ADN fue analizado por electroforesis en geles de agarosa al 1% teñidos con bromuro de etidio, utilizando una cámara de electroforesis marca Tyler (Canadá) y una fuente de poder Power Pac Basic (BioRad), visualizado en un transiluminador (2UV, Higt Performance).
Capacidad amplificable del ADN: La capacidad amplificable del ADN extraído fue evaluada mediante la detección del segmento de un gen constitutivo, conocido en la literatura como housekeeping que, en este caso, fue el gen que codifica para la enzima Gliceraldehído 3-Fosfato Deshidrogenasa (GAPDH). Para su amplificación, se utilizó un estuche de PCR de la casa comercial de Invitrogen® (C.N. 10966-020) y un juego de cebadores (PF): 5' TTGCAACTGTTTTAGGA- CTTT 3': y (PR): 5'AGCATTGGGAAATGTTCAAGG 3', que flanquea una región con una talla molecular de 600pb. Para la reacción de PCR, se utilizaron 2,5mL de buffer PCR (1X); 1,0mL de MgCl2 (2mM); 1,0mL de dntps (0,8mM); 1,0mL de PF (0,4mM); 1,0mL de PR (0,4mM); 0,3mL de Taq Platinum® (Invitrogen, Brasil) y 3mL de ADN molde y se completó con agua HPLC, hasta alcanzar un volumen final de 25mL por reacción. Como control positivo de la prueba, se utilizó un ADN humano, donado por el grupo de investigaciones biomédicas y de genética de la U.D.C.A, quienes previamente habían amplificado el fragmento génico en mención y, finalmente, se utilizó un blanco de agua (grado HPLC) en lugar del ADN molde, como control negativo del ensayo. La amplificación, se llevó a cabo en un termociclador PTC-100 (MJ RESEARCH, Canada), con un el siguiente programa: 94°C durante 30 segundos; 52°C durante 30 segundos y 72°C por 40 segundos, en 40 ciclos. El producto amplificado fue analizado en un gel de agarosa al 2%, utilizando una cámara de electroforesis marca Tyler (Canadá) y una fuente de poder Power Pac Basic (BioRad), visualizado en un transiluminador (2UV, Higt Performance).
RESULTADOS Y DISCUSIÓN
Concentración y pureza del ADN: Las características ideales del ADN que se va a emplear para estudios moleculares es que posea una buena concentración, una alta pureza y el menor grado de degradación (Armas et al. 2011); sin embargo, estos tejidos antes de ser embebidos en parafina son fijados en formalina, sustancia que preserva el tejido, pero que degrada los ácidos nucleicos (Cannavo et al. 2012) y genera un entrecruzamiento con las proteínas del tejido, por lo cual, hace aún mas complicada la eliminación de proteínas, tipo histonas de los ácidos nucleicos (Kizys et al. 2012). Adicionalmente, el ADN se debe encontrar libre de otras sustancias, como lípidos y carbohidratos, propios del tejido y de residuos provenientes de las soluciones utilizadas en el proceso extracción y desparafinación.
Mediante los protocolos convencionales de fenol-cloroformo y del estuche comercial de QIAGEN®, se alcanzaron concentraciones de ADN, que oscilaron entre 21 y 3057 ng/mL y con el protocolo de extracción por salado, las concentraciones fueron de 5,0 a 4500 ng/mL. Thyagarajan et al. (2005) sugieren tener en cuenta la cantidad de células que contenga el tejido recolectado en los microtubos de reacción, para extraer una adecuada cantidad de ADN. En este caso, las muestras utilizadas fueron de tejidos de glándula mamaria en bloques de parafina, debido a que algunos de los proyectos de investigación que se desarrollan en el laboratorio de biología molecular de la U.D.C.A, están relacionados con el estudio de cáncer de seno. Y conociendo que la glándula mamaria contiene mucho tejido graso fue necesario realizar el análisis microscópico del tejido, mediante las láminas de histopatología, para establecer la porción del TEP con la mayor celularidad posible. Las muestras de TEP utilizadas en este estudio correspondían a diferentes patologías de la glándula mamaria, por cuanto el contenido celular de cada bloque fue diferente, explicando esto, el amplio rango en la concentración de ADN reportada, a pesar de haber utilizado la misma cantidad de tejido en los tres protocolos. Un aspecto metodológico relacionado con la concentración del ADN es la utilización de la proteinasa K en el paso de la lisis celular, momento, en el cual, los ácidos nucleicos se liberan de las células, quedando vulnerables al efecto de los solventes orgánicos. Estudios realizados por Huijsmans et al. (2010) refieren que la proteinasa K es indispensable para la óptima digestión y extracción de ADN; en este trabajo se utilizó la proteinasa K en los tres protocolos, pero en concentraciones diferentes, es así como en las técnicas de extracción fenólica y QIAGEN se utilizaron 50μL/mL y 100uL/mL de proteinasa K, respectivamente, mientras que en la técnica de extracción por sales fue de 2μL/mL, observando que en los protocolos tradicionales se requiere mayor concentración de proteinasa K, que en la técnica de extracción por sales, lo que supone una mayor digestión del tejido, mayor concentración de ADN, pero mayor grado de fragmentación de la molécula.
En cuanto a los índices de pureza, la relación A260/A280 que da cuenta de la contaminación con proteínas fue similar en las tres metodologías, 1,8 en los protocolos convencionales y 1,9 en la técnica de extracción por sales, siendo ideales valores entre 1,8 y 2,2 (Santos et al. 2008), indicando que los procesos de precipitación y de separación de proteínas fueron óptimos con los tres protocolos y superaron los valores reportados por otros autores (Armas et al. 2011). La relación A260/A230 mostró cifras de 1,1, para los protocolos de extracción fenólica y QIAGEN y de 1,9, para la técnica de extracción por sales, con valores de referencia alrededor de 2,0. Este índice muestra contaminación con sales caotrópicas (Santos et al. 2008), carbohidratos, fenoles o compuestos aromáticos. El xyleno y el fenol- cloroformo fueron sustancias utilizadas en los procesos de desparafinación y extracción de ADN, por la metodología de extracción fenólica, por lo que se cree pudieron afectar negativamente los resultados del índice 260/230 (no se concoce la naturaleza de los buffer ATL, AW1, AW2, del estuche comercial de QIA- GEN), en contraste con el protocolo de extracción por sales, donde se utilizó menores cantidades de xyleno y no requiere de fenol - cloroformo, por lo tanto, el valor del índice fue cercano al valor ideal.
Capacidad amplificable del ADN: Se diseñó housekeeping para controlar internamente la optimización de la molécula extraída para su uso en la técnica de PCR, se seleccionó el gen constitutivo humano que codifica para la enzima Gliceraldehído 3- fosfodeshidorgenasa (GADPH), procedimiento previamente estandarizado en el laboratorio de biología molecular. El ADN fue considerado óptimo para su uso en la técnica de PCR, al amplificar un único fragmento de 600pb del gen GAPDH. Los ensayos de PCR-GAPDH realizados con el ADN extraído a partir de los protocolos convencionales (extracción fenólica y QIAGEN) fueron positivos en el 21% de los ensayos, mientras que con el ADN extraído por la técnica de extracción por salado, alcanzaron el 91%. Indicando que la técnica de extracción por salado fue la metodología más eficiente a la hora de conseguir un material genético óptimo para su uso en la técnica de PCR. Estos resultados muestran que la eficacia del ADN extraído de TEP para la PCR, puede estar más relacionada con la calidad del ADN que con la cantidad obtenida.
El porcentaje de amplificaciones conseguidas en este estudio y el tamaño diana amplificado supera los resultado de trabajos anteriores, siendo uno de los más exitosos el de Cannavo et al. (2012), quienes reportan 100% de amplificaciones, con un fragmento de 650pb del gen de la actina, en 46 ensayos realizados mediante la técnica de extracción de sílica magnética (NucliSENS easy MAG); Tabanifar et al. (2008) muestran el 82% de resultados positivos al amplificar un fragmento de 200pb del gen constitutivo de la β- globina con ADN extraído con el protocolo Chelex- 20; Armas et al. (2011), el 100% de amplificaciones del mismo gen, pero en sólo en 10 muestras trabajadas con el método de chelex -100 y Santos et al. (2008), logra amplificar un fragmento de 500pb, utilizando la extracción por sales. Lo anterior es de relevancia, pues la literatura menciona que la mayoría de los fragmentos que se pueden amplificar con ADN proveniente de TEP, oscilan entre 50 a 300pb (Zafra et al. 2004; Huij- smans et al. 2010) y pocas veces se logra con fragmentos superiores a éstos (Armas et al. 2006; Jiménez et al. 2007); no obstante, con el protocolo de extracción por sales, se consiguió amplificar el fragmento de 600pb del gen GAPDH (Figura 1).
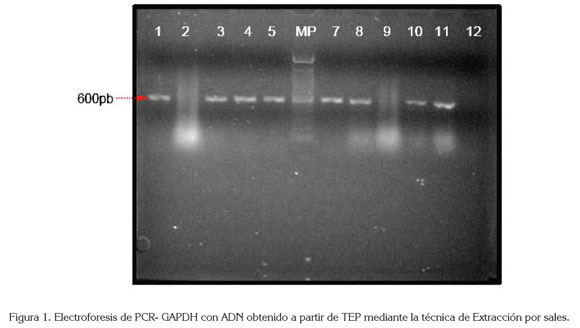
Un elemento importante al considerar la eficiencia del ADN extraído para el PCR es la integridad del material genético obtenido que puede interferir con la hibridación de los oligonucleótidos a la cadena molde (Huijsmans et al. 2010), dando resultados negativos (Figura 1, carriles 2 y 9) que, en este trabajo, fueron el 79% de los ensayos de PCR- GAPDH, con el protocolo de extracción fenólica y QIAGEN, mientras el 9%, con la técnica de extracción por sales. Es posible que estas diferencias obedezcan a factores, como la utilización de xyleno para desparafinar, la temperatura de reacción, las concentraciones de proteinasa K y los remanentes de solventes orgánicos que inhiben la PCR. Cannavo et al. (2012) mencionan que el 100% de amplificaciones del PCR fue posible solamente en aquellos TEP que se desparafinaron sin agregar el xyleno, en este caso, en los tres protocolos, se utilizó el xyleno, aún cuando en menor proporción en la técnica de extracción por sales. Las temperaturas oscilaron entre 100 y 120°C en los protocolos tradicionales, pero en la técnica de extracción por salado las temperaturas fueron entre 37 y 65°C; adicionalmente, en este protocolo se utilizaron alcoholes en concentraciones descendientes y procedimientos paulatinos de rehidratación del tejido desparafinado, lo cual, disminuyó el daño causado al ADN extraído (Coura et al. 2005). La proteinasa K en altas concentraciones y los remanentes de solventes orgánicos, como fenoles y alcoholes que pueden contaminar el ADN, no solamente pueden afectar la estructura del ADN, sino que son potenciales inhibidores de la reacción de PCR (Jiménez et al. 2007) incrementando, de este modo, el porcentaje de resultados falsamente negativos.
Con base en los resultados obtenidos en este estudio, se recomienda que a la hora de evaluar una metodología eficiente de extracción de ADN a partir de TEP, para su uso en técnicas moleculares, es necesario diseñar un control interno del PCR, que permita amplificar un fragmento de un gen constitutivo del genoma extraído y cuya longitud sea superior al fragmento génico que se desea estudiar. En este trabajo, se seleccionó un fragmento de 600pb del gen constitutivo humano de la enzima GAPDH por tratarse de un ADN humano. Las muestras positivas a este PCR fueron consideradas aptas para otros ensayos de PCR de interés en otras investigaciones con tejido mamario, pues indican que el ADN obtenido carece de inhibidores de la reacción y que es posible amplificar con él otros fragmentos de menor longitud.
Hoy por hoy, los investigadores están optando por la utilización de estuches comerciales que ofrecen facilidad en los procedimientos de laboratorio, pese a que muchos de ellos no permiten obtener una adecuada cantidad de ADN y, menos aún, si se trata de muestras almacenadas por más de tres meses (Kizys et al. 2012) y, en cambio, sí generan altos costos monetarios para las investigaciones y alto impacto ambiental; sin embargo, los resultados obtenidos en este trabajo nos permite sugerir a la comunidad científica revisar cuidadosamente el abanico de posibilidades en técnicas de laboratorio, según sus intereses, pues no siempre lo más fácil puede ser lo mejor para las investigaciones y presupuestos. Y dado el avance en la biología molecular y el recurso genético que poseen las muestras embebidas en parafina es necesario estudiar el principio químico de cada metodología, pues aun cuando no sea la más convencional, puede ser la más conveniente para el propósito de la investigación. En este caso, la técnica de extracción por sales es un procedimiento que se encuentra disponible en los laboratorios de biología molecular, requiere de reactivos de bajo grado de toxicidad para la salud humana y para el medio ambiente, los reactivos son de bajo costo, con respecto a otros y su fácil ejecución, son el valor agregado a la hora de evaluar el éxito de esta técnica sobre otras, ampliamente utilizadas para extraer ADN de TEP.
De acuerdo a los resultados obtenidos, se puede concluir que la técnica más eficiente para obtener ADN, a partir de tejido mamario embebido en parafina y amplificar un fragmento de 600pb por PCR, fue la técnica de extracción por salado, a pesar de ser una metodología poco convencional para extracción de ADN a partir de TEP.
Agradecimientos. Los autores expresan sus agradecimientos a la microbióloga Prunella Fernández, de la Universidad Javeriana, por el apoyo prestado en el manejo de equipos y lectura de resultados. Conflicto de intereses: Los autores consideran que no existe ningún conflicto de intereses que pueda afectar los resultados del trabajo. Financiación: Este proyecto fue financiado por la Universidad de Ciencias Aplicadas y Ambientales U.D.C.A, Bogotá- Colombia.
BIBLIOGRAFÍA
1. ARMAS, Y.; CAPÓ, V.; GONZÁLEZ, E.; MEDEROS, L.; DÍAZ, R. 2006. Extracción de ADN de tejidos embebidos en parafina por Chelex- 100. ADN de tejidos en parafina por resina quelante. Rev. Esp. Patología. 39(3):171-174. [ Links ]
2. ARMAS, Y.; CAPO, V.; LÓPEZ, L.; MEDEROS, L.; DÍAZ, R. 2011. Comparación de tres métodos de extracción de tejidos embebidos en parafina. Biotecn. Apl. 28:40-43. [ Links ]
3. CANNAVO, I.; LOUBATIER, C.; CHEVALLIER, A.; GIORDANEGRO, V. 2012. Improvement of DNA extraction for human papillomavirus genotyping from formalin- fixed paraffin-embedded tissues. BioRes. 1(6):333-337. [ Links ]
4. COURA, R.; PROLLA, C.; MEURER, L.; ASHTON, P. 2005. An alternative protocol for DNA extraction from formalin fixed and paraffin wax embedded tissue. J. Clin. Path. 58:894-895. [ Links ]
5. GILBERT, M.; HASELKORN, T.; BUNCE, M.; SÁNCHEZ, J.; LUCAS, S.; JEWELL, L. 2007. The isolation of nucleic acids from fixed, paraffin- embedded tissues- which methods are useful when? PLos One. 2(6):1-112. [ Links ]
6. HUIJSMANS, C.; DAMEN, J.; VAN DER LINDA, J.; SAVEL KOUL, P.; HERMANS, M. 2010. Comparative analysis of four methods to extract DNA from paraffin- embedded tissues: Efect on downstream molecular applications. BioMed Central. 3:239-248. [ Links ]
7. JIMÉNEZ, G.; VILLALOBOS, M.; JIMÉNEZ, E.; PALMA, W. 2007. Determinación de la efectividad de cinco protocolos de extracción de ADN a partir de material parafinado para estudios moleculares. Rev. Méd. U. Costa Rica. 1(1):10-19. [ Links ]
8. KIZYS, M.M.L; CARDOSO, M.G.; LINDSEY, S.C; HARADA, M.Y; SORAES, F.A.; MELO, M.C.C.; MONTOYA, M.Z.; KASAMATSU, T.S.; KUNII, I.S.; GIANNOCCO, G.; MARTINS, J.R.M.; CERUTTI, J.M.; MACIEL, R.M.B.; DIAS-da-SILVA, M.R. 2012. Optimizing nucleic acid extraction from thyroid fine-needle aspiration cells in stained slides, formalin-fixed/ paraffin-embedded tissues, and long-term stored blood samples. Arq. Bras. Endocrino.l Metab. 56(9):618- 626. [ Links ]
9. MORALES, A.; MARTÍNEZ, I.; CARLOS, A.; ÁLVAREZ, G.; ÁLVAREZ, M.; MALDONADO, J. 2005. Comparación de histopatología, cultivo y PCR en el diagnóstico de tuberculosis bovina. Rev. Cient. FCV-LUZ. 15(2):103-108. [ Links ]
10. SANTOS, M.; SAITO, C.; LINE, S. 2008. Extraction of genomic DNA from paraffin-embedded tissue sections of human fetuses fixed and stored in formalin for long periods. Path. Res. Practice. 204(9):633- 636. [ Links ]
11. SANTOS, S.; SÁ, D.; BASTOS, E.; GUEDES, H.; GUT, I.; GÃRTNER, F. 2008. An efficient protocol for genomic DNA extraction from formalinfixed paraffin- embedded tissues. Res. Vet. Sci. 86(3):1-6. [ Links ]
12. SHI, S.; COTE, R.; WU, L.; LIU, C.; DATAR, R. 2002. DNA Extraction from archival formalinfixed, paraffin- embedded tissue sections base don antigen retrieval principle: heating under the influence of pH. J. Histochem. Cytochem. 50(8):1005-1011. [ Links ]
13. TABANIFAR, B.; SALCHI, R.; ASGARANI, E.; FAGHIHI, M.; HCIDARPUR, M. 2008. An efficient method for DNA extraction from paraffin wax embedded tissues for PCR amplification of humman and viral DNA. Irani J. Path. 3(4):173-178. [ Links ]
14. THYAGARAJAN, B.; ANDERSON, E.; KONG, F.; SELK, F.; LYNCH, C.; GROSS, M. 2005. New approaches for genotyping paraffin wax embedded breast tissue from patients with cáncer: the Lowa women's health study. J.Clin. Pathol. 58: 955-961. [ Links ]
15. TUFAN, L.; BLI, F.; DUZCAN, E. 2004. Rapid and effective DNA amplification by polimerase chain reaction directliy from paraffin-embedded tissue. Aegean Path. 1:33-38. [ Links ]
16. ZAFRA, G.; FLÓREZ, O.; GONZÁLEZ, C. 2004. Influencia del método de desparafinación y el tiempo de almacenamiento en la extracción de DNA a partir de tejido de archivo. Salud UIS. 36:73-79. [ Links ]
Recibido: Enero 30 de 2013 Aceptado: Abril 22 de 2013













