INTRODUCCIÓN
La producción de cebolla junca o de cebolla de rama Allium fistulosum en Colombia, ocupa una de las áreas de mayor siembra dentro de las hortalizas. En el 2017, se estimó en 6.385ha, con una producción de 231.386t/ha. En el ámbito nacional, Nariño ocupa el cuarto lugar en producción de esta hortaliza, representada con 466ha y 2.005 unidades de producción, situándose por encima de otras especies importantes en la región, como tomate de mesa, lechuga, zanahoria, repollo, coliflor y brócoli (Gobernación de Nariño y Corporación Colombia Internacional, 2015).
La pudrición basal de la cebolla de rama ocasionada por Fusarium spp. es considerada la enfermedad más limitante en el cultivo. Los síntomas iniciales de plantas afectadas muestran en las hojas coloraciones amarillas, que se van tornando necróticas, especialmente en la punta, hasta alcanzar toda la hoja para, por último, marchitarse. En las raíces, se presentan coloraciones café oscuras, transparentes, adquiriendo una forma plana y hueca. Al realizar un corte longitudinal en los tallos, se observa una coloración que va de rojiza a café en la capa externa, la cual, se extiende hasta las hojas internas. En el tallo, se produce un micelio blanco que lo cubre por completo; con el tiempo puede llegar a invadir las raíces. En estados avanzados de la enfermedad, las plantas infectadas se arrancan fácilmente, observándose raíces atrofiadas y descompuestas (Kiehr & Delhey, 2005; Sañudo et al. 2003).
Las especies de este género afectan una amplia variedad de hospedantes; en plantas de la familia Alliaceae, se han registrado daños en los semilleros, como el mal del talluelo, la pudrición basal del bulbo de la cebolla (Fusarium oxysporum f. ssp. cepae) y la pudrición basal del ajo (Fusarium culmorum). Asimismo, los daños causados por estos hongos en plantas de cebolla pueden alcanzar el 40% de reducción de rendimientos (Schwartz & Mohan, 1995). Stankovic et al. (2007), encontraron a Fusarium proliferatum causando daños en cebolla y ajo; por su parte. Dugan et al. (2003) asocian a F. oxysporum, como el principal causante de la pudrición basal en ajo.
F. oxysporum cuenta con el mayor número de hospedantes y genera daños económicos en cultivos; sus síntomas son amarillamientos, marchitamiento vascular, que es seguido por la muerte de la planta. Este patógeno, se caracteriza por producir micelio de colores púrpura a blancos, de crecimiento rápido en medio de PDA o agar clavel, con tres tipos de esporas: microconidios, macroconidios y clamidosporas, encontrándose en condiciones diversas, desde suelos de tipo ártico como desérticos (Nelson et al. 1983; Arbeláez, 2000; Leslie & Summerell, 2006).
El reconocimiento de géneros y de especies puede ser muy complejo. Es así, como la identificación basada en la morfología presenta un importante grado de dificultad, sobre todo, para distinguir especies cercanas, con características fenotípicas muy similares. Los métodos moleculares se han convertido en una herramienta eficiente para confirmar la identidad de especies dentro del género, basada, especialmente, en marcadores específicos, que amplifican una región de su genoma (Unda et al. 2011). Por lo anterior, el objetivo del estudio fue aislar e identificar molecularmente las especies de Fusarium, encontradas en cebolla de rama, afectadas por pudrición basal, en los municipios productores de Nariño, como son Pasto, Potosí y Buesaco.
MATERIALES Y MÉTODOS
Localización. El presente trabajo, se realizó en las instalaciones de La Universidad de Nariño (Pasto-Nariño), en los laboratorios de Sanidad vegetal, microbiología y biología celular.
Material vegetal y Suelo. La colección de muestras de cebolla de rama con síntomas, como marchitamiento y pudrición basal, se efectuó en los municipios de Pasto (Buesaquillo, Cujacal y Santa Lucia); Potosi, (Mueses-Florida, Mueses, Lourdes, Guaracal) y Buesaco (Rosal del monte), referenciando geográficamente cada punto de muestreo. Se escogieron tejidos de plantas sintomáticas y asintomáticas, incluyendo raíces, tallos y hojas. Las muestras se etiquetaron, se envolvieron en papel toalla y aluminio y se depositaron en bolsas plásticas, para su transporte hasta los laboratorios. El material vegetal, se conservó en nevera, a 4°C, hasta su procesamiento. De igual manera, se seleccionaron muestras de suelo de cada sitio, tomando, aproximadamente, 100g de la zona cercana a la raíz.
Aislamiento de hongos
Aislamiento a partir de suelo. Se pesaron 10g de suelo por muestra y se agregaron 90mL de agua destilada estéril, para obtener una solución de 10-1 y, a partir de ésta, se obtuvieron soluciones hasta 10-4. De cada solución, se sembró un mililitro en cajas Petri esterilizadas, que contenían medio de cultivo PDA acidificado (Sañudo et al. 2003; Llanos & Sánchez, 1982).
Aislamiento a partir de tejido. Fragmentos de tejido de 5mm, previamente desinfestados, se colocaron en el medio de cultivo, para su posterior purificación, siguiendo la metodología citada por Castella et al. (1997) y Agrios (2005).
Purificación. En el mismo medio de cultivo, se purificaron colonias de los hongos, procedentes de los diferentes aislamientos; se incubaron a 28°C, de 7 a 10 días, hasta que el crecimiento de cada aislamiento completó la dimensión de la caja Petri. Para la obtención de cultivos monospóricos, se aplicó la metodología descrita por Nelson et al. (1983). En los aislamientos que no se observaron estructuras, se sembraron en medio Agar Clavel, de acuerdo con la metodología usada por Nelson et al. (1983).
Identificación. Los aislamientos, se identificaron mediante un microscopio Nikon de luz, por características morfológicas y criterios taxonómicos, propios del género Fusarium (Barnett, 1960; Nelson et al. 1983; Alexoupolus et al. 1996; Samson et al. 2004; Leslie & Summerell, 2006).
Caracterización molecular
Extracción de ADN. A partir de cultivos monospóricos, se recogió micelio con una espátula metálica estéril, depositándolo en un tubo Eppendorf de 2mL, punzando hasta deshacerlo. Luego, se añadieron 750μL de solución tampón (Tris-HCl 100mM pH 7.2; EDTA 100mM y dodecil sulfato sódico (SDS) al 10%), se agitó y se mantuvo en frío. En seguida, se agregaron 3μL de una solución de proteinasa K (20mg/mL), se agitó e incubó a 37°C, durante una hora. Posteriormente, se adicionaron 100μL de NaCl 5M. Luego, se agregaron 80μL de solución CTAB/NaCl (CTAB al 10% en NaCl 0,7M), se agitó en homogenizador vortex y se incubó a 65°C, durante 10 min.
Para la purificación del ADN, se adicionaron 700μL de cloroformo/alcohol isoamílico (24:1), se agitó y se centrifugó a 12000rpm, durante 10min. El sobrenadante, se transfirió a un nuevo tubo, al que se incorporó un volumen equivalente (entre 600-700μL) de fenol/cloroformo/alcohol isoamílico (25:24:1), el cual, se centrifugó, a 12000rpm, durante 5 min.
El sobrenadante, se transfirió a otro tubo, al que se le añadió entre 500-600μL de isopropanol, para precipitar el ADN; se agitó suavemente y se centrifugó a 12000rpm, durante 10 min. Se eliminó el isopropanol y se añadieron 500μL de etanol frío al 70%; se agitó y se centrifugó nuevamente durante 5 min. Finalmente, se eliminó el etanol y se secó el precipitado al vacío. El ADN, se suspendió en 50μL de tampón TE (Tris 10mM, EDTA 1mM, pH 8) y se conservó a -20°C.
PCR para amplificar la región ITS1 - ITS4. La región ITS del ADNr fue amplificada con los cebadores universales ITS1(5'- TCCGTAGGTGAACCTGCGG-'3) e ITS4 (5'TCCTCCGCTTATTGATATGC-3'), descritos por White et al. (1990); la mezcla para la amplificación, se realizó en un volumen de 25μL, con 5μL de 10X PCR tampón (100mM KCl, 100mM Tris HCl (pH 8.8); 500mM KCl; 3μL de 25mM MgCl2; 2mM de dNTP; 1μL de ADN, a una concentración de 100ng/μL en total 2,5 unidades de Taq ADN polimerasa (Biolabs inc®) y 1μL de 10μM de cada cebador ITS1-ITS4. La amplificación, se realizó en un termociclador programable Labnet®, siguiendo los ciclos de temperatura, así: desnaturalización inicial, a 95°C, por 1 min, seguido de 35 ciclos; desnaturalización, a 95°C, por 30 seg; alineación, a 55°C, por 1min; extensión, de 72°C, por 45 segundos y una elongación final, a 72°C, por 5 min. El producto, se visualizó por electroforesis en gel de agarosa a 1,2%, para obtener fragmentos de 490 pares de bases (pb).
PCR para amplificar la región TEF 1 alfa. Se utilizó el protocolo estándar propuesto por O'Donnell et al. (1998), para amplificación del gen Ef1 5´-ATGGGTAAGGA (A/G) GACAAGAC-3) y 5´-GA(G/A)GTACCAGT(G/C) ATCATGTT-3´.
La reacción de PCR, se realizó en un volumen final de 25μL, que contenían de 100ng de ADN; la mezcla de los productos de PCR, se hizo en un tubo Eppendorf, al cual, se agregó 12,5uL de Green Master Mix, 0.5uL de cada primer, 1uL de ADN y se completó con agua miliq. El programa de PCR comprendió una desnaturalización inicial a 95°C, por 2 min, seguido de 35 ciclos; desnaturalización, a 95°C, por 30 seg; alineación, a 56°C, por 30 segundos; extensión, de 72°C, por 1 min y una elongación final, de 72°C, por 10 min.
La electroforesis, se realizó con un gel de agarosa al 1,2% con Hydra green ® y TBE al 05X, con un marcador de peso molecular de 100pb (Thermo scientific gene ruler 100pb®); para su visualización, se usó un fotodocumentador (Enduro GDS Labnet, USA), que permitió observar la presencia de fragmentos, entre 600 y 700pb.
Secuenciación. Algunos productos de PCR fueron enviados a secuenciar al instituto de biotecnología de Corea, MACROGEN, para determinar su identidad, a nivel de especie, mediante el programa BLAST (Basic Local Alignment Search Tool), del NCBI (http://blast.ncbi.nlm.nih.gov/Blast.cgi).
Análisis filogenético. Para el caso del gen TEF, las secuencias fueron alineadas con el programa ClustaW. Los análisis filogenéticos, se realizaron con el coeficiente de similitud de Neighbor joining (NJ) y Máxima parsimonia (MP), con un bootstrap de 1.000 réplicas, usando el programa MEGA 6 (Tamura et al. 2013).
RESULTADOS Y DISCUSIÓN
Caracterización morfológica
Síntomas asociados a pudrición basal causada por Fusarium: Los síntomas observados en campo fueron: plantas de crecimiento bajo; hojas amarillentas y marchitas, dobladas hacia abajo; tejido de hojas basales con coloración rojiza, que se extiende hacia la parte interna del pseudotallo y raíces necrosadas de color café (Figura 1). Esta sintomatología de la encebolla larga fueron descritas también por otros autores (Conn et al. 2012; Ávila et al. 1996).

Figura 1 Síntomas de pudrición basal asociados a Fusarium en cebolla de rama (Allium fistolosum). a. Hojas externas con amarillamiento y dobladas; b. Estado de las plantas con pudrición avanzada; c. y d. Coloración rojiza en tejido basal y raíces con necrosamiento.
Dentro de las características macroscópicas, se observaron colonias con crecimiento rápido y de apariencia algodonosa, con coloraciones blanquecinas y rosadas en los primeros estados de desarrollo; en algunos aislamientos, con el paso de los días, se percibieron colores lilas, violeta oscuro, rosadas, rosado y blancas; en el reverso de la caja de Petri, se advirtieron coloraciones lila y curuba, con crecimiento uniforme en el centro de la caja y un tono más claro hacia el borde.
Fusarium oxysporum . Los aislamientos PO (001,002, 003, 004, 005, 007, 008, 009, 010, 011), PA (012,016, 017, 018, 019) y BU (006, 020), presentan características propias de la especie, como son macroconidias con tres septos, de forma alargada y ligeramente curvadas, el pie basal termina en un punto, las microconidias sin septos y con forma ovalada, partiendo, en algunos casos, de monofiálides cortas y se observó alta presencia de clamidosporas (Figura 2). Presentan un rápido crecimiento del micelio aéreo, con coloraciones púrpuras y blancas, con presencia de esporodoquios, forman micelio de color naranja, los colores varían entre claros y oscuros; en algunos aislamientos, se presentaron dos tipos de pigmentación.
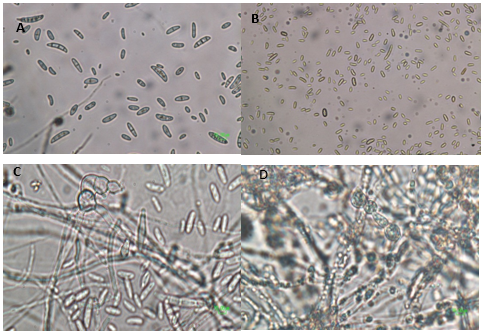
Figura 2 Características microscópicas de los aislamientos de F. oxysporum. a. Macroconidias; b. microconidias; c. Fiálides cortas; d. Clamidosporas.
Fusarium solani . Los aislamientos PA (013, 014, 015) presentan características, como macroconidias abundantes y redondas, similares a las de F. oxysporum, con 3-7 septos, de forma ancha y redonda; la célula apical es redonda y sin definición en el pie basal; las microconidias son abundantes, pueden presentar o no divisiones. Los conidióforos muestran monofiálides ramificados o no (Figura 3). La morfología de la colonia en PDA presentó un crecimiento rápido con abundante micelio aéreo; la superficie es cubierta por un micelio denso (pionnotes), que forma coloraciones crema. Algunas cepas pueden mostrar coloración púrpura oscuro.
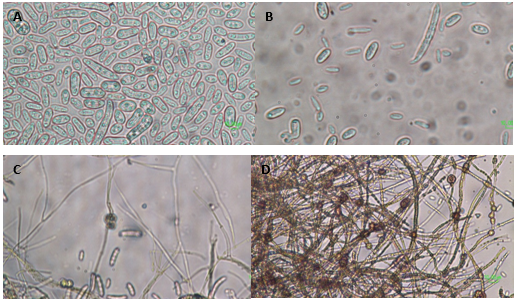
Figura 3 Características microscópicas de los aislamientos de F. solani. A. Macroconidias; b. microconidias; c. Fialides cortas; d. Clamidosporas.
En los 19 aislamientos de Fusarium spp., se observaron colonias con apariencia algodonosa y de color blanquecino y rosado, en los primeros días de crecimiento. Posteriormente, algunos, se tornaron de un color levemente púrpura, en el centro del mismo. Por el reverso de la caja de Petri, se percibió un color morado oscuro hacia el centro y de un tono más claro hacia el borde; colonias de color amarillo y blanco, también fueron observadas. Asimismo, la presencia de estructuras de reproducción y sobrevivencia, propias del género, como: macroconidias, microconidias y clamidosporas, fueron frecuentes en los diferentes aislamientos.
Tal como lo mencionan Leslie & Summerell (2006), se requiere de los criterios biológicos, morfológicos y moleculares, para asignar taxonómicamente la posición de las especies de Fusarium, lo que permite tener una caracterización más aproximada de las especies, que pueden estar involucradas en las enfermedades de las plantas.
Caracterización molecular
Análisis filogenético
Análisis de la Región ITS: Los cebadores ITS1-ITS4 amplificaron a 560pb. El uso de los cebadores ITS1 -ITS4 determina que los aislamientos PO001, PO002, PO010 y BU020, corresponden a F. oxysporum f. sp. lycopersici; los aislamientos PO003, PO004, PO005, PO007, PO008, PO011, PA012, PA017, PA018, PA019, a F. verticillioides (16); F. solani (013, 014, 015) y F. oxysporum f. sp. cepae (9).
La amplificación de las secuencias de Fusarium con los marcadores ITS y TEF1α, confirmaron la presencia del hongo en los aislamientos evaluados, asociados a la pudrición basal de la cebolla de rama.
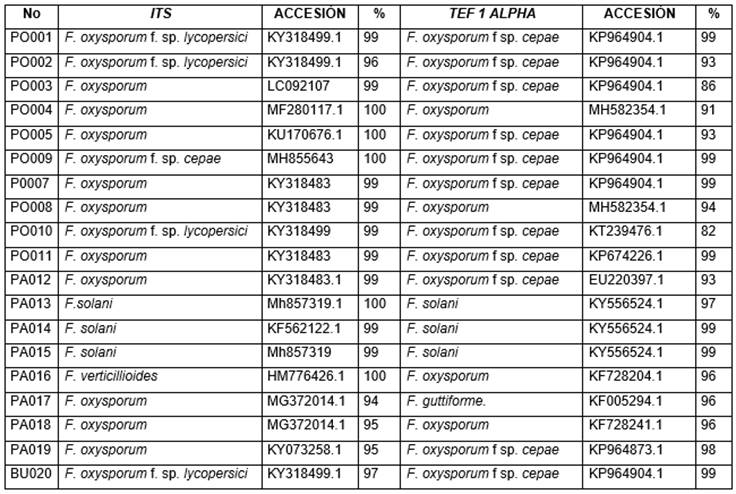
Las secuencias de los aislamientos con los cebadores ITS, evaluados de las tres zonas productoras de cebolla de rama, correspondieron al género Fusarium; se obtuvo un porcentaje de similitud de 94 a 100%. Cabe destacar que, de los 19 aislamientos evaluados, 14 correspondieron a F. oxysporum (PO001, PO002, PO003, PO004, PO005, PO007, PO008, PO010, PO011, PA012, PA017, PA018, PA019, BU020), datos que fueron previamente confirmados con la caracterización biológica y las secuencias de los aislamientos; PO001, PO002, PO010, BU020, se ubican dentro de F. oxysporum f. sp, lycopersici; los aislamientos PA013, PA014 y PA015, correspondieron a F. solani. El aislamiento PA016, a la especie F. verticillioides y el aislamiento PO009, a F. oxysporum f. sp. cepae, con un porcentaje de similitud del 100% (Tabla 1).
Tabla 1 Número de Accesión según Genbank y porcentaje de identidad de los aislamientos de Fusarium, usando los marcadores ITS y TEF 1α.
Análisis de la Región de TEF 1α: Con el cebador TEF 1α, se observó un fragmento esperado de 700pb. Las secuencias de los aislamientos PO001, PO002, PO005, PO007, PO009, PO010, PO011, PA012, PA019, BU020, se ubican en la especie F. oxysporum f. sp. cepae; los aislamientos PA013, PA014, PA015, como F. solani; F. oxysporum, con PA004, PA008, PA018 y el aislamiento 017, como F. guttiforme.
Los análisis de secuenciación para TEF1α, al realizar la alineación de las secuencias, se encontró que, 15 de los aislamientos evaluados, se clasifican como F. oxysporum (PO001, PO002, PO003, PO004, PO005, PO007, PO008, PO010, PO011, PA012, PA016, PA018, PA019, BU020); tres, como F. solani (PA013, PA014 y PA015) y uno, como F. guttiforme, demostrando, para estos marcadores, la alta variabilidad intraespecífica dentro de la población de Fusarium, causante de la pudrición basal de la cebolla.
En aliáceas, específicamente en ajo y cebolla, se han registrado, a nivel mundial, varias especies de Fusarium, como F. oxysporum, F. oxysporum f sp. cepae, F. solani, F. proliferatum, F. verticillioides, F. acuminatum, F. culmorum (Bayraktar et al. 2010; Cramer, 2000; Dissanayake et al. 2009; Dugan et al. 2003; Galván et al. 2008; Montes et al. 2003; Ochoa Fuentes et al. 2012; Stankovic et al. 2007).
Se demuestra una consistencia en el concepto filogenético, que se debe a que los aislamientos evaluados presentaron secuencias muy similares a las registradas en el GenBank, como F. oxysporum y F. oxysporum f. sp. cepae. Es así, que la base de la variabilidad para las especies de Fusarium, en las que no se identifica su teleomorfo o estado sexual, se podría asociar con mutaciones, es decir, que, cuando se presentan estos fenómenos, facilitan la presencia de genes saltarines o elementos transposones, como también la transferencia genética, por vía de anastomosis, lo cual, es un indicio de que en la reproducción asexual de Fusarium se establezcan líneas clonales dentro de su población y, por consiguiente, la variabilidad de la misma (Jacobson & Gordon, 1991; Kistler, 1997; Salazar-González et al. 2016).
Para determinar el porcentaje de identidad (superiores al 93%, para ITS y 82%, para TEF1α), las secuencias fueron analizadas, utilizando el coeficiente del vecino más cercano, con un bootstrap de 1000, en el cual, se obtuvo agrupamientos muy similares, tanto para el factor de elongación de la traducción (TEF1α) como para el espacio interno transcrito (ITS).
El análisis filogenético, usando cebadores de ITS, agrupó a F. oxysporum y F. oxysporum f. sp. Cepae, en un solo brazo (Figura 4), observándose una distancia mínima entre las especies. De igual manera, los aislamientos correspondientes a F. solani, se agruparon en otro brazo, mostrando al aislamiento PA013, con una distancia mayor a la presentada por PA014 y PA015, demostrado, para estos aislamientos, que se pueden agrupar en dos complejos muy específicos, como son el CSFO y CSFS (Aoki et al. 2014).
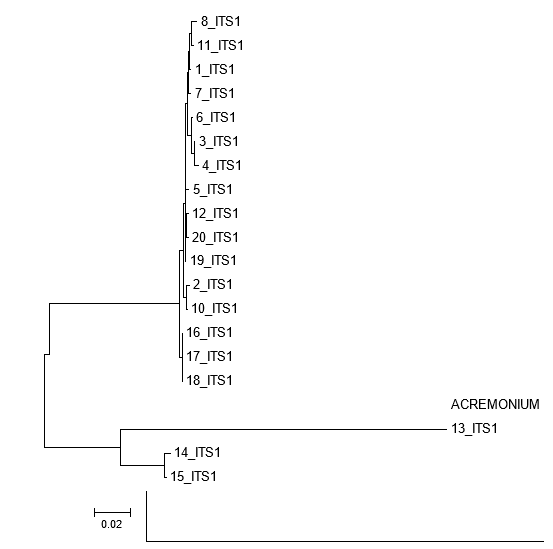
Figura 4 Árbol filogenético de los complejos de especies F. oxysporum y F. solani con el primer ITS, usando el coeficiente de similitud de Máxima parsimonia. El árbol se elaboró con un bootstrap de 1000 réplicas. Universidad de Nariño, Pasto, noviembre de 2018.
El uso del cebador TEF1α, permitió determinar que las secuencias correspondientes a F. oxysporum y F. oxysporum f. sp. cepae fueron homólogas, con alto grado de similitud y que la distancia encontrada agrupó a las secuencias en una sola especie (Figura 5). Autores, como Leslie & Summerell (2006), O’Donnell et al. (2008), Balajee et al. (2009), señalan que dentro del mismo complejo de especies es común encontrar diferencias genéticas, adquiridas de un origen filogenético diferente. La utilización del cebador TEF1α es amplia, ya que codifica una parte esencial de las proteínas que hacen parte de la maquinaria de traducción; a su vez, muestra una afinidad en las secuencias polimórficas evaluadas, siendo altamente informativo, a nivel de especie (O’Donnell et al. 1998; Geiser et al. 2004). En este estudio, se determinó que varios de los aislamientos del CSFO provienen de diferentes linajes.

Figura 5 Árbol filogenético de los complejos de especies F. oxysporum, F. solani, F. fujikoroi con el primer TEF 1α, usando el coeficiente de similitud de Máxima parsimonia. El árbol se elaboró con un bootstrap de 1000 réplicas. Universidad de Nariño, Pasto, noviembre de 2018.
Por otro lado, diez aislamientos de F. oxysporum f. sp. cepae son ubicadas en el CSFO, pero también sus secuencias son homólogas a las obtenidas a partir de aliáceas; esto determina, que existen secuencias muy cercanas en F. oxysporum, las cuales, no se agruparon en otros grupos, como se observó con el cebador TEF1α. La alta diversidad del genoma en las especies de F. oxysporum, se puede inferir, que se debe a los elementos transposones que ejercen un papel definitivo en la generación de diversidad, ya que su presencia y la continua actividad puede producir mutaciones espontáneas y, por consiguiente, variabilidad en el genoma (Daboussi et al. 1992; McDonald, 1993). Edel et al. (2001) consideran que la presencia del elemento transposon Fot1 es el causante de dicha diversidad. Además de la presencia de aislamientos pertenecientes al CSFO, también el estudio demuestra que tres de sus aislamientos pertenecen al CSFS, siendo uno de ellos con una distancia genética diferente a sus homólogos. Es posible que el aislamiento PA013 provenga de un linaje diferente.
En este estudio, la variación encontrada con el aislamiento PA013 con el cebador ITS, se explica por la complejidad que se genera, no solo en la morfología, sino que el complejo CSFS agrupa sus especies, teniendo en cuenta el origen geográfico y el hospedante afectado. Se clasificó, inicialmente, dentro de la sección Martiella, que produce enfermedades, como la pudrición de raíz y tallo de arveja, el síndrome de muerte súbita, en soya; pudrición de pie en fríjol y pudrición húmeda, en papa. En esta morfoespecie, se estima que existen alrededor de 60 especies filogenéticamente distintas (O’Donnell et al. 2008). En un estudio filogenético, morfológico y fenotípico en muerte súbita de la soya, se habían clasificado a las especies, inicialmente, dentro de formas especiales de F. solani, es decir, F. solani f. sp. glycines; se demostró, que estas no correspondían a las formas especiales, sino a siete nuevas especies filogenéticamente diferentes.
Esto agrupó a las especies de F. solani en clados, geográficamente delimitados, generando un sistema alfanumérico de nomenclatura (Nalim et al. 2011; O’Donnell et al. 2000; O’Donnell et al. 2008). Los estados teleomórficos, por ejemplo, F. striatum Sherb. [syn. = Haematonectria ipomoeae (Halst.) Samuels & Nirenberg] y F. neocosmosporiellum (syn. = Neocosmospora vasinfecta), se ubican en un cuarto clado y Nectria haematococca fue transferido a un nuevo género, Haematonectria (Rossman et al. 1999). Posteriormente, H. haematococca (Berk. & Br.) Samuels & Rossman fue recombinada como Neocosmospora haematococca (Berk. & Br.) Samuels, Nalim & Geiser y, finalmente, su anamorfo fue descrito como F. haematococcum Nalim, Samuels & Geiser, con diferencias filogenéticas distintas a F. solani (Nalim et al. 2011) Esta serie de confusiones y desacuerdos en cuanto a la clasificación de especies del complejo de F. solani han provocado propuestas para formar un nuevo género (Geiser et al. 2013).
Con el cebador TEF1α, se encontró el aislamiento PA017, correspondiente a F. guttiforme, que se ubica dentro del complejo de especies de F. fujikoroi, CSFF; esta especie está registrada como causante de la fusariosis de la piña en Sur y Centroamérica y aparece en complejos de enfermedades donde intervienen especies, como F. oxysporum, F. proliferatum, F. solani, F. graminearum, entre otras (Salazar-González et al. 2016). También es causante de enfermedades en piel y ojos en humanos (Migheli et al. 2010).
Los resultados obtenidos en este estudio proveen una detección confiable de diagnóstico de Fusarium, usando dos tipos de marcadores moleculares, que permiten establecer las diferencias intra e interespecíficas de las especies de este género. Asimismo, genera una herramienta valiosa que conduce a determinar, a futuro, cuál de las especies encontradas están relacionadas directamente con la patogenicidad en la planta.














