INTRODUCCIÓN
Los alimentos funcionales (AF) son fisiológicamente útiles para el hombre y los animales, dado que aportan nutrientes esenciales que ofrecen beneficios para la salud y reducen el riesgo de enfermedad (FAO, 2002; Aranceta & Gil, 2010). Los AF incluyen los probióticos, prebióticos y simbióticos, siendo los primeros, los más relevantes, por su incidencia, uso y beneficios en las actividades humanas y en los que recae la más sólida evidencia científica (Rodríguez et al. 2004; Arvanitoyannis & Van Houwelingen-Koukaliaroglou, 2005; Santillán-Urquiza et al. 2014).
La Organización de Alimentos y Agricultura (FAO) y la Organización Mundial de la Salud (OMS) definen a los probióticos como microrganismos vivos que, administrados en cantidades suficientes, proveen efectos benéficos en la salud del huésped (FAO, 2002; Mariño García et al. 2016). El Instituto Colombiano Agropecuario, ICA, mediante Resolución No. 00375 del 27 de febrero del 2004, exigió que los bioproductos, a partir de microrganismos que entran al mercado, deben estar caracterizados molecularmente, demandando su identificación, a nivel de género y especie (ICA, 2004), actividad que busca contrarrestar el creciente fenómeno de biopiratería, que experimenta la región (Acosta & Martínez, 2015; Silvestri, 2015).
Siguiendo tal lineamiento, en este trabajo, se establece la huella genómica basada en las secuencias del ADNr 16S, marcador más común, para realizar estudios taxonómicos en bacterias (Valenzuela-González et al. 2015) y del gen rpoB, cuyas secuencias resultantes suelen ser de mayor calidad que las del gen del ADNr 16s (Bou et al. 2011), a través del encriptamiento en un código de barras, para la protección intelectual y el control de calidad del probiótico Rumitec® (Agrosavia, 2018), para terneros neonatos.
MATERIALES Y MÉTODOS
Crecimiento de las cepas. Las tres cepas componentes del probiótico C2, B9 y G8, crecieron en medio caldo anaerobio glucosa-celobiosa al 0,1% p/v, para bacterias anaerobias ruminales, bajo la técnica de Roll Tube, descrita por Hungate (1969). Se realizaron 3 réplicas por cepa al 10%, a partir de inoculo proveniente de criopreservados del Banco de Germoplasma de Nutrición Animal de la Corporación Colombiana de Investigación Agropecuaria, Agrosavia. Estas fueron cultivadas a 39ºC, durante 18 horas.
Extracción de ADN de las cepas del probiótico. Se ensayaron dos metodologías de extracción de ADN: 1) Extracción basada en el protocolo de la Agencia Internacional para la Energía Atómica (Rodríguez et al. 2004) y 2) Extracción basada en el protocolo comercial Ultra Clean Kit Mo Bio® 2012. Se realizó cuantificación con espectrofotómetro NanoDrog® ND-1000 Spectrofotometer v. 3.3.0, usando 2uL de muestra, medidos a una absorbancia de 260 nanómetros.
Amplificación por Reacción en Cadena de la Polimerasa del 16s y el rpoB. Se usaron los oligonucleótidos 27F: 5'- AGA GTT TGA TC (A:C) TGG CTC AG -3'; 1492R: 3'- TAC GGY TAC CTT GTT ACG ACT T -5', para el ADNr 16s, con un tamaño de amplicón de 1490pb (James, 2010) y rpoB Long F: 5'- GCG AAC ATG CAA CGT CAG GC -3'; rpoB Long R: 3'- AGT GCC CAT ACT TCC AT -5', con un tamaño de amplicón de 600pb (Perumbakkan & Craig, 2011). Tanto la amplificación del 16s como del gen rpoB, se realizó sobre un volumen final de reacción de 25uL, (Rodríguez et al. 2004). Los programas de temperaturas fueron: desnaturalización inicial a 95°C x 2 y 9min; desnaturalización a 95°C x 30 y 20s; anillamiento a 57°C x 30s y 53°C x 40s; extensión a 72°C x 1 y 1,5min y extensión final a 72°C x 7 y 10min, respectivamente.
Los productos de PCR fueron visualizados en geles de agarosa al 1% p/v, teñidos con Sybr Safe Invitrogen® 0,5uL en 40mL de gel. Se sembraron 5uL de ADN con 2uL de Orange G. El tiempo de electroforesis fue de 30min, a 80 voltios, corrido en TAE 1x. El control negativo en todos los casos consistió en agua grado molecular. El marcador de peso usado fue 1Kb ladder, de Invitrogen®; de 12 a 0,25kb, 2uL por pozo. Todas las imágenes fueron registradas con el documentador Molecular imager® Gel DocTMSystem con imagen LabTM software y QuantityOne 4.6.5 software.
Análisis de restricción. Se usaron las endonucleasas de restricción de corte frecuente AluI, HaeIII, RsaI y MseI. Para el gen rpoB, se realizó una reacción de 10uL, 1uL de Buffer 10x, 0,1uL de BSA al 100%, 0,4uL de cada enzima, a 5U/uL, 5,0uL, de producto de PCR a ~400ng/uL y 3,5uL, de agua grado molecular. La reacción, se prolongó por 3 horas y media, a 37°C. La digestión, se visualizó en un gel de agarosa al 2,5% p/v, corrido a 100 voltios, durante 1 hora y media. Se sembró todo el producto de la digestión, con 3uL de buffer de carga. Con los productos de PCR del 16s, se realizó una reacción a 20uL, 2uL de Buffer 10x, 0,2uL de BSA al 100%, 0,5uL de cada enzima a 5U/uL, 10uL de producto de PCR a ~400ng/uL y 7,3uL, de agua grado molecular. La reacción, se prolongó por 3 horas y media, a 37°C. La digestión, se visualizó en un gel de agarosa al 2,5% p/v, corrido a 60 voltios, durante 2 horas. Todo se documentó con los programas Molecular imager® Gel DocTMSystem, imagen LabTM software y QuantityOne 4.6.5 software.
Secuenciación. Las reacciones de secuenciación, se realizaron en las direcciones 5' a 3' y 3' a 5', según lo estipulado por la empresa privada Corpogen® (Rodríguez et al. 2004; Vargas, 2020). El análisis de las secuencias, se realizó mediante BLASTN de la base de datos GeneBank, del Centro Nacional para Información en Biotecnología (NCBI) y del Ribosomal Data Project (RDP). Se tuvieron en cuenta los índices Bit scores y E-value, para los análisis (Ude et al. 2020; Altschul et al. 1997).
Encriptamiento en código de barras. Se codificaron las secuencias de los genes ADNr 16s y rpoB de cada cepa, con el programa BarCodeMagic® (Zhou & Piramuthu, 2017), usando la simbología tipo código 128, para la generación de códigos bidimensionales y QR estáticos.
RESULTADOS Y DISCUSIÓN
Crecimiento de las cepas y Extracción de ADN de los componentes del probiótico. Se alcanzó el crecimiento de colonias viables, a partir de las 6 horas posteriores a la inoculación; no obstante, la cepa B9 tuvo crecimiento negativo y fue reactivada en medio glucosa al 1%, previa a su inoculación en caldo anaerobio glucosa-celobiosa. Las otras cepas componentes del probiótico, se caracterizaron por manifestar un amplio rango de actividades metabólicas, desde celulolítica y hemicelulolítica hasta amilolítica y proteolítica (Aldana et al. 2009). Los crecimientos en caldo anaerobio glucosa-celobiosa al 0,1% p/v fueron exitosos para las cepas C2 y G8, componentes del probiótico. La metodología de la IAEA mostró ser eficiente en la cantidad de ADN recuperado; sin embargo, el tiempo total invertido en la extracción y la purificación es un factor en contra, más aún, cuando la metodología por Kit ofrece resultados de pureza de ADN superiores; por tal, se escogió la metodología del Kit Mo Bio, con base en la facilidad, la practicidad y la velocidad de la extracción, sumado a la pureza del ADN obtenido.
Amplificación por Reacción en Cadena de la Polimerasa del 16s y el rpoB. Se obtuvieron los amplicones en las bandas esperadas. No se observaron problemas de resolución de bandas ni artefactos en el gel; por tanto, los productos obtenidos sirvieron de insumo para las secuenciaciones. Debido al poco crecimiento de la cepa B9, se obtuvo poco ADN, lo que impidió sembrar pozos replicados durante las corridas en geles (Figura 1). Los programas de temperaturas del laboratorio de Microbiología Molecular de AGROSAVIA, para el ADNr 16S y de Perumbakkan & Craig (2011), para un fragmento del rpoB, son adecuados para la obtención de copias de las regiones de interés.
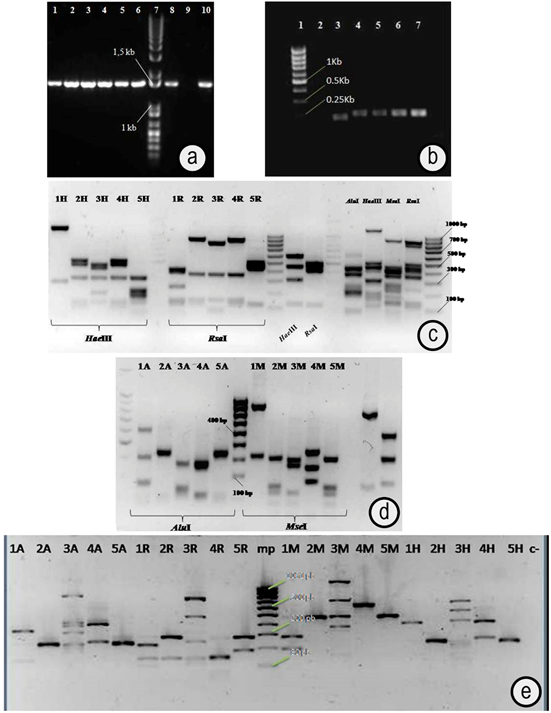
Figura 1 a. Amplificación de la región 16s del ADNr. 1: B9; 2 y 3: C2; 4 y 5: G8; 6: Bacillus sp.; 7: Marcador de peso 1Kb ladder Invitrogen®; 8: Paenobacillus sp.; 9: Control negativo; 10: Paenobacillus sp.; b. Amplificación de parte del gen rpoB (primers rpoB Long F y R, Perumbakkan & Craig (2011). 1: Marcador de peso 1Kb ladder Invitrogen®; 2: Control negativo; 3: B9; 4 y 5: C2; 6 y 7: G8; c. Perfil de restricción del ADNr 16s con las enzimas HaeIII (H), RSA I (R). 1: B9; 2: C2; 3: Bacillus sp.; 4: Paenobacillus sp.; 5: G8; d. Perfil de restricción del ADNr 16s con las enzimas AluI y MseI I. 1: B9; 2: C2; 3: Bacillus sp.; 4: Paenobacillus sp.; 5: G8; e. Perfil de restricción del gen rpoB. A: AluI, R: RsaI, M: MseI, H: HaeIII; 1: B9; 2: C2; 3: Bacillus sp.; 4: Paenobacillus sp.; 5: G8.
Análisis de restricción y secuenciación. En el perfil de restricción del ADNr 16S, la enzima HaeIII generó 19 bandas, con un polimorfismo del 15,8%; RsaI, 17 bandas, con un polimorfismo de 29,4%; AluI, 10 bandas, con un polimorfismo del 50% y, finalmente, MseI generó 15 bandas, con un polimorfismo del 20%. Por su parte, en el perfil de restricción del rpoB, la enzima HaeIII generó 9 bandas, con un polimorfismo del 33,3%; RsaI, 11 bandas, con un polimorfismo de 36,3%; AluI, 11 bandas, con un polimorfismo del 36,3% y MseI generó 9 bandas, con un polimorfismo del 55,5% (Figura 1).
Las enzimas escogidas para la PCR-RFLP han mostrado ser suficientemente discriminativas en varios trabajos (Mata et al. 2020; Fallahizadeh et al. 2019; Jiménez et al. 2011), generándose perfiles de restricción ricos en polimorfismos únicos, suficientemente diferenciables, como para establecer una huella genómica reproducible; no obstante, los amplicones son más informativos que las restricciones, razón, por la cual, se usaron para generar los códigos. La estrategia permitió codificar una información mucho más exacta y fiable, que ofrece reproducibilidad y moderada variación, en términos de identidad. Una vez se estableció la similitud de las secuencias editadas con el programa CLC-Workbench® contra las anotaciones de las bases de datos (Tabla 1), se corroboró la identidad de las cepas del probiótico. Dicha composición, ya se encuentra patentada y es pública (AGROSAVIA, 2018).
Tabla 1 Resultados de similitud y códigos de barras de las secuencias de los genes 16s y rpoB.
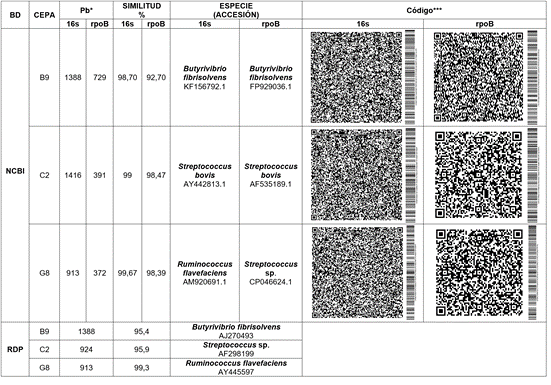
*: Pares de bases. **: cepa contaminada con enterobacterias, descartada. ***: para acceder a la secuencia del amplicón basta con escanear el código.
Código de barras. Ambos códigos contienen toda la secuencia de cada cepa para cada gen y, en el caso del código QR, está vinculada la simbología con los caracteres de origen (Tabla 1). La inclusión del código QR responde a las tendencias en las cadenas logísticas de mercadeo que, actualmente, se dan en el mundo (Gonzalez-Argote & Garcia-Rivero, 2016), donde la protección de los bioinsumos ha superado el nivel de especies-razas a la de individuo-secuencia. Con la conformación de estos códigos, se asegura un insumo importante, para contribuir a la estrategia de propiedad y protección intelectual de la mezcla probiótica, a la par, que se brinda una herramienta fiable de monitoreo de su pureza.















