INTRODUCCIÓN
Teniendo en cuenta que en el último siglo se viene presentando un incremento en el detrimento de la diversidad genética en los cultivos agrícolas y de la biodiversidad de los ecosistemas naturales (Angarita et al. 2017), se hace necesario concretar esfuerzos para la preservación de los recursos fitogenéticos de este importante recurso, que ocupa en Colombia, el tercer lugar con mayor demanda interna y el séptimo, de mayor producción (Asohofrucol, 2019).
Buena parte de los protocolos existentes, para aislar y purificar DNA de buena calidad, se basan en la técnica del bromuro de cetiltrimetilamonio (CTAB), por ser económico (Sahu et al. 2012). También existen en el mercado kits comerciales para la extracción de ADN, convenientes para obtener ADN de plantas, pero con mayores costos económicos (Whitehouse & Hottel, 2007), que se diferencian en rendimiento de ADN, tipo y cantidad de muestra, tecnología de aislamiento y volumen de elución; deben ser adecuados para la extracción de múltiples muestras y producir exiguos residuos perjudiciales (Pipan et al. 2018).
Las hojas de guayaba contienen compuestos polifenólicos, flavonoides (Camarena-Tello et al. 2018), ácidos fenólicos y ácidos hidroxicinámicos (Narváez-Cuenca et al. 2020). Los fenólicos y los polisacáridos, se unen o coprecipitan con los ácidos nucleicos (Azmat et al. 2012), lo cual, puede acarrear al deterioro, a la contaminación y al bajo rendimiento del ADN (Sika et al. 2015). Nerdo et al. (2004) reportan éxito parcial en dos de los ocho métodos que utilizaron para extraer el ADN de las hojas jóvenes de guayaba, al igual que Kumari et al. (2018) y Kumar et al. (2020).
Cuando se trabaja en lugares apartados y no se dispone de lo necesario para congelar las muestras, éstas deben ser deshidratadas, utilizando gel de sílice. La obtención de ácidos nucleicos de tejidos vegetales secos, se suele dificultar, ya que la mayoría de los protocolos están estandarizados para trabajar con muestras frescas (Kumari et al. 2018 y Kumar et al. 2020). Ante esto, el presente estudio tuvo como objetivo la comparación de dos métodos de extracción de ADN, a partir de hojas secas de Psidium guajava L.
MATERIALES Y MÉTODOS
Obtención del material vegetal. Se colectaron hojas en fase madura de 10 árboles de guayaba, ubicados en el campus central de la Universidad de Córdoba (8°47'31.0" N y 75°51'45.0" W), las cuales, se colocaron en una bolsa pequeña resellable, que tenía, previamente, sílica gel y, después, almacenadas a temperatura ambiente.
Obtención del ADN. Se evaluaron dos metodologías de extracción: el comercial Kit de extracción Wizard Genomic de Promega® (USA, Promega A1120) (Kit Promega) y el Mini-prep de Doyle & Doyle (1987) modificado (MMpD&D). En el primero, se siguieron las recomendaciones del fabricante y para el segundo método, se realizaron las siguientes modificaciones: se reemplazó el octanol por alcohol isoamílico, se redujo la centrifugación a 13.000 los rpm y se realizó la precipitación del ADN, con una solución de etanol absoluto + acetato de sodio en reemplazo del isopropanol.
Visualización del ADN. Se utilizó una electroforesis en gel de agarosa al 1% (p/v), utilizando TBE 1X teñido con Gel-Red 30X, que se visualizaron en un transiluminador DyNA Light™ UV (Labnet International Inc.) y documentados, mediante el fotodocumentador ENDUROTM GDS (Labnet International Inc.).
Cuantificación del ADN. El ADN obtenido fue cuantificado en un espectrofotómetro Colibri Microvolume Spectrometer (Titertek-Berthold, Berthold Detection Systems GmbH, Bleichstrasse, Pforzheim, Germany), se estimó la pureza del ADN, por medio de la relación de absorbancia (A260/A280nm).
Evaluación de la calidad del ADN. Se realizó mediante los marcadores mPgCIR03, mPgCIR16, mPgCIR09 y mPgCIR19 (Risterucci et al. 2005). La reacción en cadena de la polimerasa (PCR), se desarrolló en un volumen final de 25μL, que contenían 1U de de Taq polimerasa (Thermo Scientific, Uklam-USA), 10pM de cada primer (forward y reverse), 0,2mM de dNTPs, buffer de PCR 1X (GIBCO-BRL, Groningen, Netherlands), 2,5mM de MgCl2, 20ng/µL de ADN y agua estéril, hasta alcanzar el volumen final. El programa empleado es: 95°C, 3min; 95°C, 30s; 45°C, 30s; 72°C, 2min, con repetición de 40 ciclos del paso 2 al 4 y un paso final de extensión de 72°C, 10min. La PCR, se realizó en un termociclador Bioard T100 #1861096 (Los Ángeles, USA).
Amplificación del ADN. Para la verificación de cada producto PCR obtenido, se prepararon geles de agarosa al 1% (p/v), teñido con el reactivo GelRedTM, las cuales, se corrieron en una cámara de electroforesis horizontal Mini-Sub® Cell GT Bio-Rad SF1704467 (Los Ángeles, USA), a 100 V y 50 mA por 40 minutos. Los geles, se visualizaron en un Transiluminador DyNA Light UV (Labnet International Inc., Cary, North Carolina, USA); los registros, se tomaron en un Fotodocumentador ENDURO™ GDS (Labnet International Inc., Cary, North Carolina, USA).
Análisis estadístico. Se utilizó el software SPSS para Windows, versión 17.0 (SPSS Inc., 2008). Para cada uno de los métodos de extracción, se calcularon las medias y las desviaciones estándar (s). Para identificar el método que presenta diferencias significativas en la concentración y calidad de ADN, se utilizaron las pruebas de Tukey y Dunnet (Miller, 1996). Para el análisis de las amplificaciones de los marcadores, se comprobó la proporción de éxito, mediante la prueba de Chi-cuadrado (Halos et al. 2004).
RESULTADOS Y DISCUSIÓN
Los resultados del presente estudio mostraron la eficacia del MMpD&D al compararlo con el Kit Promega, en cuanto a la extracción, pureza y rendimiento del ADN obtenido y mejora lo reportado por otros autores para la misma especie, quienes extrajeron ADN de hojas frescas de guayaba. Los resultados del análisis de absorbancia mostraron una concentración promedio de ADN en las muestras tratadas con el MMpD&D de 9,76ng/µL (s 2,028); asimismo, para la pureza del ADN, el valor obtenido en la relación A260/A280 fue 1,8 (s 0,128), mientras la concentración promedio de ADN obtenido con el Kit Promega fue 155,9ng/µL (s 91,8), mientras para la pureza del ADN el valor obtenido en la relación A260/A280 fue 1,12 (s 0,04) (Tabla 1).
Tabla 1 Concentración y pureza del ADN de hojas secas de Psidium guajava L. obtenido con los protocolos utilizados.

Los ADN obtenidos mediante el kit Promega, se mostraron pegajosos, viscosos, oscuros, lo cual, indicaría la presencia de polifenoles de elevada densidad, que se co-purifican con el ADN, afectando la calidad del ácido nucleico obtenido (Deshmukh et al. 2007); además, Rojas et al. (2011) afirman que el color oscuro del pellet final evidencia la contaminación. Mediante el MMpD&D, se obtuvo un ADN incoloro, mostrando que se logró separar en el ADN componentes, como proteínas, polisacáridos y polifenoles. De igual forma, para la extracción de ADN, se puede adicionar el acetato de sodio por su utilidad para separar polisacáridos (Michiels et al. 2003).
Si bien las cantidades de ADN obtenidos en los dos métodos utilizados reveló una menor concentración de ADN al utilizar el MMpD&D respecto al Kit Promega (Tabla 1), estos valores sugieren que durante la extracción hay pérdida de ADN por factores, como la maceración con nitrógeno líquido, ya que al romperse las membranas los componentes celulares son liberados y la acción de polifenoles hace que gran parte del ADN se degrade o precipite con ellos (Cota et al. 2006). Los resultados revelaron que el MMpD&D presentó un mayor nivel de pureza de 1,8 en comparación con el Kit Promega, que mostró valores de 1,12 (Tabla 1). Según Gerstein (2001), un ADN puro y de calidad debe estar en un rango de 1,7 a 2,0, para la relación de absorbancias 260/280; valores mayores a 2,0 indican la presencia de ARN y valores menores a 1,7, contaminación con proteínas.
Al aplicar la prueba de Tukey, los resultados obtenidos en la concentración de ADN en ambos métodos mostraron diferencias significativas, lo cual, indica que, en ambos casos, no fue posible obtener buena cantidad de ADN. Al comparar los resultados mediante la prueba de Dunett, se encontraron diferencias significativas y mayores concentraciones de ADN para el método Kit de Promega (Tabla 1); se encontraron diferencias en cuanto a la relación A260/A280. Es importante resaltar que la relación A260/A280 fue de 1,12 (Tabla 1) para el método comercial, sugiriendo la probable presencia de contaminantes, tales como proteínas (Manning, 1991), mientras dicha relación fue de 1,8 para el MMpD&D, obteniéndose así un ADN puro y de calidad.
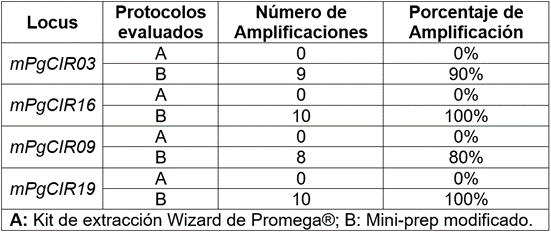
Como se observa en la tabla 2, el mayor porcentaje de éxito en la amplificación, se obtuvo con el MMpD&D para los marcadores mPgCIR16 y mPgCIR19 (100%), seguido por el marcador mPgCIR03 (90%) y el marcador mPgCIR09 (80%). Los resultados obtenidos con el Kit de Promega, no mostraron amplificaciones, produjo ADN de mala calidad para obtener productos de PCR, lo cual, se pudo deber a que el ADN pudiera presentar contaminantes o bien se ha degradado; resultado similar a lo expresado por Ferreira & Grattapaglia (1998), quienes expresaron que la presencia de metabolitos secundarios en el ADN, inhiben la acción de la Taq polimerasa.
Las modificaciones realizadas al MMpD&D para la extracción de DNA de P. guajava permitieron optimizar un protocolo para obtener DNA de buena calidad y cantidad, a partir de hojas secas; obtener mayor calidad en la pureza y la integridad del DNA y la total amplificación de los marcadores moleculares utilizados.