INTRODUCCIÓN
El tomate de árbol Solanum betaceum Cav. es una especie originaria de Sur América, donde se encuentran sus principales centros de distribución, como la zona Andina de Perú, Bolivia, norte de Argentina y sureste de Brasil (Díaz Granada et al. 2017). Es una alternativa de producción, que se ha generalizado, gradualmente, entre productores de clima frío y frío-moderado, en la región natural Andina de Colombia, siendo una especie, cuyo fruto posee características organolépticas aceptadas entre los consumidores de los mercados, tanto nacional como internacionales (Acosta-Quezada et al. 2016). Entre 2007 y 2018, en el país, se presentó un incremento del 26,5 % en el área cultivada de tomate de árbol, con una superficie cosechada en el 2018, de 10.161 ha, una producción de 174.230 t y rendimientos entre 15,5 y 20,2 t ha-1 (Agronet, 2021).
Existe una escasa oferta de genotipos mejorados para la siembra en frutales de Colombia, como el tomate de árbol; es por eso, que los agricultores obtienen semilla tomando frutos de las plantas y los cultivos resultantes son fenotípicamente heterogéneos, además, genera en la especie pérdida de variabilidad genética, ya que, para procesos de selección, se debe partir de una base genética amplia (Lobo Arias, 2006; Lagos-Santander et al. 2013).
Los factores más importantes en la producción de variabilidad en plantas son: la hibridación, la recombinación y la mutación (espontanea o inducida); sin embargo, la ocurrencia de las mutaciones de forma natural es demasiado baja para fines prácticos, por lo tanto, mutágenos físicos y químicos han probado ser útiles para incrementar las mutaciones y la variabilidad (Donini & Sonnino, 1998). Las mutaciones causan un cambio repentino en un carácter, el cual, es heredable y, por lo tanto, transmisible a la progenie, además de tener un gran potencial y servir como una aproximación complementaria en el mejoramiento de cultivos (Gandhi et al. 2014).
El dietil sulfato (DES) es ampliamente reportado como un agente químico capaz de causar alteraciones genéticas en distintas especies. Bhagwat & Duncan (1997) utilizaron diferentes dosis de ácido de sodio, dietil sulfato y etilmetano sulfonato en ápices de banano Musa spp., cultivados in vitro, con el objetivo de evaluar su efectividad induciendo mutaciones; además de producir variantes tolerantes a Fusarium oxysporum, encontrando que los individuos expuestos a los diferentes tratamientos mostraron menos de 10 % de infección de F. oxysporum.
Para identificar la variabilidad generada, a través de la mutagénesis inducida en especies cultivadas, se conocen diferentes metodologías para el análisis de la composición genética, las cuales, permiten tomar decisiones en el manejo de los recursos genéticos de las plantas e identificar, de forma precisa, los individuos, aún en fases tempranas de desarrollo (Estrada Salazar et al. 2010). Dentro de las metodologías están los marcadores moleculares, que se basan en la reacción en cadena de la polimerasa (PCR) y, dentro de ellos, se pueden distinguir varios métodos, que difieren entre sí por su poder de resolución, es decir, el nivel de polimorfismo que puedan detectar (Muñoz Flórez et al. 2008).
Uno de los marcadores convencionales y de fácil aplicación son los microsatélites amplificados al azar-RAMs (por sus siglas en inglés), propuestos inicialmente por Zietkiewicz et al. (1994), para el estudio de la diversidad en plantas y en animales y combinan propiedades, tanto de los marcadores de amplificación aleatoria de ADN polimórfico- RAPDs (por sus siglas en inglés), como de secuencias simples repetidas o microsatélites- SSRs (por sus siglas en ingles). Los estudios de diversidad dirigidos con marcadores moleculares RAMs han mostrado ser viables y sus resultados confiables, en muchas especies, entre las cuales, se pueden mencionar a Physalis peruviana (Morillo Paz et al. 2011), Guadua angustifolia (Rugeles-Silva et al. 2012), Musa spp (Muñoz F. et al. 2012), Prunus persica (Morillo Coronado et al. 2014), Prunus sp (Ben Tamarzizt et al. 2015), Physalis ixocarpa (Delgado-Alvarado et al. 2018), entre otros.
El objetivo del presente trabajo fue evaluar, a través de marcadores RAMs, las variaciones moleculares presentes en plántulas de S. betaceum provenientes de semillas, previamente sometidas a diferentes concentraciones del agente mutante dietil sulfato (DES).
MATERIALES Y MÉTODOS
Material vegetal. Las semillas de tomate proceden de una planta cultivar Manzano, con 14 meses de establecida en campo, una variedad seleccionada por el agricultor; su fruto es de tamaño grande 120 a 200 g, color de pulpa y arilo de semilla naranja; contenido de solidos solubles de 9,35 a 10,32 °Brix, contenido de jugo por fruto entre 26 a 31 mL y un rendimiento de 5,21 a 9,32 t ha-1 (Lagos-Santander et al. 2013). Las semillas, se trataron con diferentes concentraciones y tiempos de incubación del agente mutagénico dietil sulfato (DES). Se utilizaron 25 semillas por repetición, con cuatro repeticiones, para un total de 100 semillas por tratamiento.
Inducción de mutaciones. En los laboratorios de Biología Molecular y de Cultivo de Tejidos Vegetales del Grupo de Investigación en Producción de Frutales Andinos, GPFA, de la Universidad de Nariño, se desarrolló la fase de inducción de mutaciones. Las semillas de tomate de árbol, previamente desinfectadas y sumergidas en ácido giberélico, se secaron a temperatura ambiente y luego se sometieron a los tratamientos, que incluyeron 0,5, 1,0, 1,5 y 2 % de concentración de DES y se incubó durante 8 y 16 horas, a temperatura ambiente, con agitación suave. En total, se obtienen 10 tratamientos que corresponden a T1: 0 %DES-8h, T2: 0,5 %DES-8h, T3: 1 %DES-8h, T4: 1,5 %DES-8h, T5: 2 %DES-8h, T6: 0 %DES-16h, T7: 0,5 %DES-16h, T8: 1,0 %DES-16h, T9: 1,5 %DES-16h y T10: 2 %DES-16h.
Para determinar el efecto de los tratamientos sobre la variación genética, se sembraron los tratamientos en medio de cultivo in vitro (Murashige & Skoog, 1962).
Extracción de ADN. Tomando el ADN total de 50 plántulas de tomate por tratamiento a los 35 días después de la germinación (tratadas con DES y plantas no tratadas), se aisló con el kit extracción de ADN DNeasy Plant Mini Kit (QIAGEN). La cuantificación y la pureza de los ácidos nucleicos, se determinó con el espectrofotómetro NanoDrop One de Thermo Scientific.
Amplificación de marcadores RAMs. Para evaluar la variabilidad genética producida por los tratamientos mutantes, se probaron seis cebadores RAMs, los cuales, corresponden a: AG (5´HBH(AG)7A 3´), CA (5´DBDA(CA)7 3´), TG (5´HVH(TG)7T 3´), ACA (5´BDB(ACA)5 3´), CCA (5´DDB(CCA)5 3´) y CGA (5´DHB(CGA)5 3´), donde: H = A, T o C; B = G, T o C; V = G, A o C y D = G, A o T.
Los perfiles de amplificación de la PCR fueron los siguientes: desnaturalización inicial de 95 ºC, durante 5 minutos; 37 ciclos de desnaturalización, a 95 ºC, por 30 segundos; hibridación: 50 ºC (Primer AG), 41 ºC (Primer CA), 55 ºC (Primer TG), durante 45 segundos; 58 ºC (Primer ACA), 61 ºC (Primer CGA) y 55 ºC (Primer CCA), durante 50 segundos. La extensión, se hizo a 72 ºC, por 2 minutos y la extensión final, a 72 ºC, durante 7 minutos. Para la amplificación, se utilizó un termociclador MultigeneTM OptiMax (Labnet, USA). Los productos amplificados fueron separados por electroforesis vertical, en geles de poliacrilamida, al 7,5 % y se construyó una matriz binaria de presencia y ausencia, donde 1 es presencia y 0 ausencia de bandas.
Se determinó el número de alelos polimórficos, la heterocigosidad observada, la heterocigosidad esperada y el número de alelos por cebador. Para el análisis, se utilizó el complemento de Excel, GenAlex versión 6 (Peakall & Smouse, 2006). Dado que el cebador CCA fue 100 % monomórfico en las plantas evaluadas, no se incluyó en el análisis estadístico. Las estimaciones de similitud, se obtuvieron mediante el índice de Dice (1945) y la construcción de grupos y la generación del dendrograma, se realizó por el método UPGMA (Unweighted Pair Group Method with Arithmetic Mean), que permite buscar la distancia más pequeña en la matriz de distancias genéticas y agrupar las unidades que lo conforman, como una sola unidad taxonómica independiente (Hart, 1983), para los cuales, se utilizó el programa Numerical Taxonomy System (NTSYSpc) version 2.02g (Rohlf, 1988).
RESULTADOS Y DISCUSIÓN
Los cinco cebadores RAMs analizados generaron 37 loci, de los cuales, siete se obtuvieron en CA y TG; cinco en AG; seis en ACA y 12 en CGA. Igualmente, se lograron 1.258 registros con 594 unos y 664 ceros; estos valores son inferiores a los encontrados en estudios que determinaron genotipos con mayor diversidad genética, como en mora (Rubus glaucus) (Morillo C. et al. 2005) y en cacao (Theobroma cacao L) (Morillo C. et al. 2014); sin embargo, los niveles de diversidad identificados se consideran aceptables, teniendo en cuenta que se utilizó la progenie de una sola planta. El número de alelos (Na) varió entre 1,86 y 2,0, con un promedio de 1,97 ± 0,97 y el número de alelos efectivos (Ne), entre 1,0 y 1,60, con una media de 1,40 ± 0,05, mientras que considerando todos los loci, el Ne osciló entre 1,0 (CA5) y 1,99 (ACA1) (Tablas 1 y 2). Según lo reportado por Magallán Hernández et al. (2009), se presentan alelos raros dentro de la población evaluada, a causa de que el Na es mayor al Ne; el número de alelos por locus polimórfico es la medida más directa de la diversidad y se refiere al número de estados variantes de un locus dentro del genoma, ya sean las formas alternativas de una proteína, el número de sitios dentro del genoma que pueden ser cortados con enzimas de restricción, o a la variación en el número de secuencias repetitivas en el ADN, entre otras.
Tabla 1 Parámetros de diversidad genética de los loci obtenidos con cinco marcadores RAMs en plántulas de Solanum betaceum, provenientes de semillas tratadas con dietil sulfato-DES.
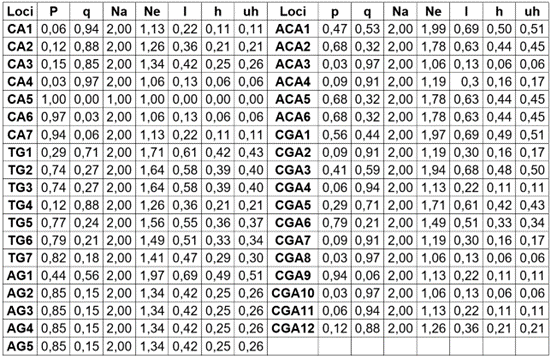
Na = número de alelos diferentes; Ne = número de alelos efectivos = 1/(p2+q2); I = Índice de información de Shannon = -1*(p*Ln(p)+q*Ln(q)); h = heterocigosidad = 1- (p2 + q2); uh = heterocigosidad insesgada = (N / (N-1)) * h; %P = porcentaje de loci polimórficos (%P = 1-[(1/m)∑p2]); p = frecuencia de banda presente y q = 1 - p. Fst = índice de diferenciación genética.
Tabla 2 Parámetros de diversidad genética obtenidos con cinco marcadores RAMs en plántulas de Solanum betaceum, provenientes de semillas tratadas con dietil sulfato-DES.
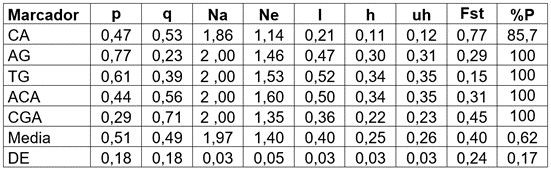
Na = número de alelos diferentes; Ne = número de alelos efectivos = 1/(p2+q2); I = Índice de información de Shannon = -1*(p*Ln(p)+q*Ln(q)); h = heterocigosidad = 1- (p2 + q2); uh = heterocigosidad insesgada = (N / (N-1)) * h; %P = porcentaje de loci polimórficos (%P = 1-[(1/m)∑p2]); p = frecuencia de banda presente y q = 1 - p. Fst = índice de diferenciación genética.
A excepción de CCA, los loci identificados mostraron una naturaleza polimórfica. Solo CA presentó el 87,5 % de loci polimórficos; los demás obtuvieron el 100 %. El porcentaje de loci polimórficos en la población evaluada fue del 97,30. Los loci más polimórficos correspondieron a TG1, TG2, TG3, TG5, TG6, AG1, ACA1, AC2, ACA5, ACA6, CGA1, CGA3, CGA5 y CGA6, que mostraron una diversidad génica o una heterosis media insesgada (uh), entre 0,34 y 0,51, con una media de 0,44 (Tabla 2), que permite establecer que estos marcadores son útiles para obtener una mayor discriminación entre los tratamientos mutantes en Solanum betaceum. En un estudio relacionado con marcadores codominantes, como los microsatélites (SSRs), en esta especie, los niveles del Índice de Contenido Polimórfico (PIC), para un mismo microsatélite, estuvieron en un rango de 0,08 y 0,51. Los resultados de este estudio revelan una reducida diversidad genética en las plantaciones de S. betaceum, en el Ecuador y constituyen un primer paso para determinar futuras acciones en el mejoramiento de este cultivo (Peñafiel L. et al. 2009), como puede ser la inducción de mutaciones en cultivares existentes.
En la tabla 2, se puede observar una síntesis de los parámetros de diversidad genética obtenidos con cinco marcadores RAMs, a través de los tratamientos mutantes en S. betaceum. Los valores de heterosis insesgada promedia (uh) y los índices de diferenciación genética (Fst) indican altos niveles de variación en los tratamientos. Llama la atención el marcador CA, que presenta una moderada uh, pero un alto Fst, lo cual, se debe a que de los 238 registros de presencias/ausencias que se presentó en la población muestreada, puede diferenciar entre aquellos que contienen unos loci de aquellos que no lo manifiestan. Así, en un grupo uno, están C1, C2, C3 y C4, que solo presentan bandas en un 0,09 % de 136 registros y, en el otro, C5, C6 y C7, que exhibieron bandas en un 97 % de 102 registros, a través de los 34 individuos evaluados.
Los demás marcadores muestran altos valores de variación genética, reflejados en altos valores de uh y Fst. Si se tiene en cuenta el criterio de Wright (1978), que indica que valores de Fst entre 0,05 y 0,15 muestran una diferenciación moderada, entre 0,15 y 0,25 alta y mayor a 0,25 muy alta, demuestra que existieron cambios entre los grupos de plantas tratadas y no tratadas, tal como se verá más adelante. En la tabla 3, se muestran los valores de similitud (diagonal y encima de la diagonal) y disimilitud (debajo de la diagonal), de Dice (1945), obtenidos por los diferentes tratamientos de DES. Las observaciones 1, 2 y 3 fueron blancos (0 %DES-8 h), al igual que 18, 19, 20 y 21 (0 %DES-16 h). Las observaciones 4 y 5 correspondieron a T2 (0,5 %DES-8 h), de 6 a 9 a T3 (1 %DES-8 h), 10, 11 y 12 a T4 (1,5 %DES-8 h), 13 a 17 a T5 (2 %DES-8 h), 22 y 24 a T7 (0,5 %DES-16 h), 25 a 29 a T8 (1,0 %DES-16 h), 31 y 32 a T9 (1,5 %DES-16 h), 33 y 34 a T10 (2 %DES-16 h).
Tabla 3 Valores de similitud (diagonal y encima de la diagonal) y disimilitud (debajo de la diagonal) de Dice (1945), para las plantas de Solanum betaceum, obtenidas de semillas tratadas con dietil sulfato-DES y evaluadas con cinco marcadores del tipo RAMs.

T= tratamiento; T1(0-8h) = 1, 2, 3; T2(0,5-8h) = 4, 5; T3(1-8h) = 6, .., 9; T4(1,5-8h) = 10, 11, 12; T5(2-8h) = 13, 14, .., 17; T6(0-16h)= 18, 19, 20, 21; T7(0,5-16h)= 22, 23, 24; T8(1,0-16h)= 25, .., 29; T9(1,5-16h)= 31, 32, 32; T10(2,0-16h)= 33, 34.
Los valores de similitud que están por encima de la diagonal (Tabla 3) tienen un total de 34t (34-1)/2 = 561 registros, sobre este conjunto de datos, tal como lo muestran los parámetros de diversidad genética (Tablas 1 y 2). Como se dijo anteriormente, las plantas evaluadas surgen de semillas tomadas de una sola planta al azar de un cultivo de tomate de árbol var. Manzano, sometidas a diferentes concentraciones de DES; de ahí, que los valores de similitud permiten establecer los cambios y las diferencias entre plantas tratadas y no tratadas.
Los valores de distancia (Tabla 3) y la proporción de la presencia de bandas con los cinco cebadores RAMs en cada uno de los tratamientos DES en Solaum betaceum (Tabla 4), reflejan los cambios en las frecuencias de los loci identificados entre plantas tratadas y no tratadas. En las observaciones no tratadas (1, 2, 3, 18, 19, 20 y 21), los marcadores se manifestaron con las mismas proporciones, a excepción del marcador CGA, que cambia sus proporciones en las observaciones 1, 2 y 18. Estas variaciones, se deben al sistema de reproducción mixta que tiene la especie (Ramírez & Kallarackal, 2019), que permite manifestar algunos niveles de variación en progenies de una sola planta.
Tabla 4 Proporción de la presencia de bandas con los cinco cebadores RAMs en cada uno de los tratamientos de dietil sulfato-DES, en Solaum betaceum.
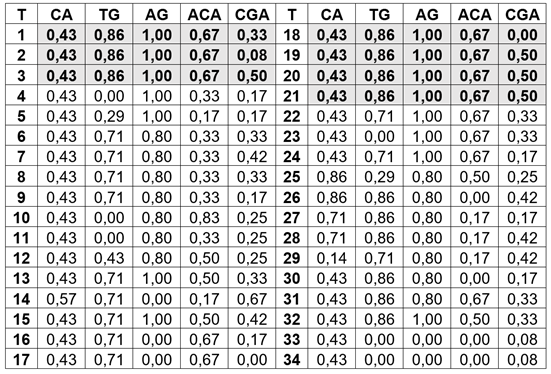
Es claro que muchos de los tratamientos con DES son semejantes a los tratamientos no tratados, pero con variaciones en los niveles de similitud; por ejemplo, las distancias genéticas o disimilitudes (debajo de la diagonal) (Tabla 3) son más grandes en concentraciones de 2 % de DES, por 8 y 16 horas, aunque las distancias son de diferente magnitud, que oscilan entre 0,4 y 0,9, confirma que existieron cambios de diferente magnitud entre plantas tratadas y no tratadas e incremento de la variabilidad genética, aunque con pequeños cambios, que dieron como resultado una mayor frecuencia de distancias entre 0,1 a 0,4. Cambios similares observaron Mudibu et al. (2011), en plantas de soya, tratadas con rayos Gamma.
En el dendrograma de la figura 1, se muestran las relaciones genéticas entre los tratamientos DES de Solanum betaceum, construido con base en el coeficiente de similitud de Dice (1945) y el método de agrupamiento UPGMA, obtenido de cinco cebadores RAMs. A una similitud de 0,70, se identificaron tres grupos: en el grupo 1 (G1), se ubicaron el 70,6 % de la población analizada; en el grupo 2 (G2), el 20,6 % y en el grupo 3 (G3), el 5,9 %. La observación 14, que corresponde a T5 (2 %DES, 8 h), no se ubicó en ningún grupo. Esta planta, se caracterizó por no producir bandas en el marcador AG y reducir la proporción de presencia de bandas en TG y CGA. Algo similar ocurrió en el G3, con las observaciones 33 y 34 del T5 (2 %DES, 16 h), pero con la diferencia que no manifestaron bandas en los tres marcadores antes mencionados (Tablas 4 y 5). La similitud de estas tres observaciones respecto a las no tratadas oscila entre 0,3 y 0,6 (Tabla 3), lo cual, confirma el efecto del DES en el cambio de las frecuencias de los marcadores utilizados en esta investigación. En términos generales, la variación entre las 34 observaciones evaluadas es alta, pero las distancias genéticas moderadamente pequeñas, tal como se encontraron en soya tratada con rayos Gamma (Mudibu et al. 2011). De hecho, la heterosis total es relativamente alta (50 %), para la progenie de una planta, cuyas muestras tratadas presentan similitudes altas, que van entre 0,90 y 1,0.
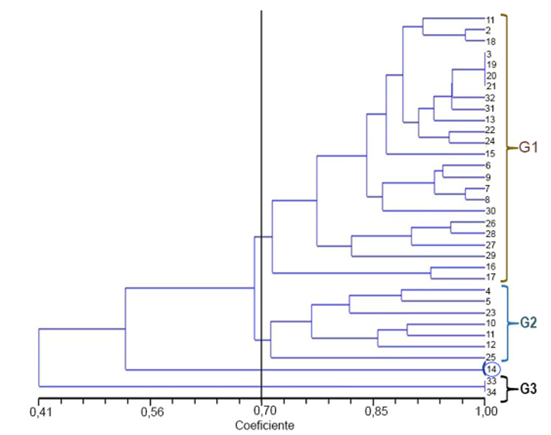
Figura 1 Dendrograma de las relaciones genéticas entre los tratamientos de dietil sulfato-DES de Solanum betaceum, construido con base en el coeficiente de similitud de Dice (1945) y el método de agrupamiento UPGMA, obtenido de cinco cebadores RAMs.
Tabla 5 Parámetros de diversidad genética obtenidos con cinco marcadores RAMs en plántulas de Solanum betaceum, provenientes de semillas tratadas con dietil sulfato-DES.
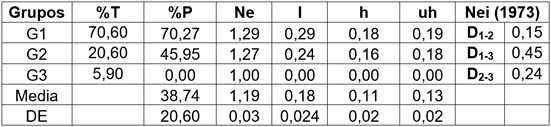
%T = tamaño del grupo respecto a la población en porcentaje; %P = porcentaje de loci polimórficos (%P = 1-[(1/m)∑p2]); Ne = número de alelos efectivos = 1/(p2+q2); I = Índice de información de Shannon = -1*(p*Ln(p)+q*Ln(q)); h = heterocigosidad = 1- (p2 + q2); uh = heterocigosidad insesgada = (N / (N-1)); D.= distancias genéticas insesgadas.
El análisis genético de los tres grupos formados (Figura 1) indica que el porcentaje de loci polimórficos en el G1 fue de 70,27 %; en el G2 de 45,95 % y de cero, en el G3. El índice de heterosis insesgada (uh) fue alta para los grupos dos y tres, con valores de 0,19 y 0,18, en su orden. El grupo 3 presentó una alta homogeneidad, con un uh de cero; es de aclarar que en el G1 se encuentran todos los controles (0 %DES-8 h y 0 %DES-1 h). Este grupo presenta una distancia baja con G1, moderada alta con G2 y moderada con G3. El número de alelos efectivos (Ne) en los tres grupos es similar y oscila entre uno y 1, 3 (Tabla 5). Se comprueba que los cambios producidos por el agente DES fueron de baja magnitud; sin embargo, produjeron cambios en los niveles de variación genética, tal como lo indican los altos niveles de los parámetros de diversidad genética y el porcentaje de loci polimórficos. Es necesario, comprobar, en la siguiente generación de esta población mutante, si existen diferencias a nivel fenotípico. Al respecto, Prina et al. (2010) indican que en la generación mutante (M2), se deben identificar los cambios causados por los agentes mutantes y es necesario iniciar, en esta fase, el proceso de selección.
Basado en los resultados obtenidos, se puede concluir que los marcadores RAMs fueron útiles para detectar cambios entre plantas provenientes de semillas tratadas con el agente mutagénico dietil sulfato (DES). Los niveles de variación genética entre los tratamientos con DES fueron más altos que los tratamientos sin DES. Las similitudes de Dice fueron bajas entre plantas tratadas y no tratadas y fueron altas entre plantas no tratadas. Los cambios producidos por el agente mutante DES fueron de baja magnitud; sin embargo, produjeron cambios en los niveles de variación genética en la población estudiada.















