INTRODUCCIÓN
Los dípteros, en especial, las moscas calipteradas de las familias Calliphoridae, Muscidae, Fanniidae y Sarcophagidae, son relevantes en la sucesión entomológica asociada a procesos de descomposición en cadáveres (Byrd & Tomberlin, 2020). La identificación taxonómica, a nivel de especie, es fundamental para el estudio de su biología, bionomía e historia evolutiva (De Queiroz, 2007) y, en el campo forense, para estimar el intervalo post-mortem, posibles traslados del cadáver, entre otras aplicaciones (Byrd & Tomberlin, 2020). Tradicionalmente, se han utilizado caracteres morfológicos para la identificación de los especímenes; sin embargo, la alta diversidad de especies (Stork, 2018), su similitud morfológica, la escasez de especialistas y de claves morfológicas, dificultan esta tarea (Packer et al. 2009). El estudio del ADN y su aplicación en la identificación y confirmación molecular de especies permite superar estas dificultades y avanzar en el conocimiento de su biología, diversidad genética, entre otros aspectos X (Packer et al. 2009).
La obtención del ADN en una concentración y pureza apropiada es fundamental en el flujo de trabajo de un laboratorio de biología molecular para garantizar resultados óptimos, de acuerdo con la aplicación requerida (Kuhn et al. 2017; Psifidi et al. 2015). Para lograrlo, la técnica de extracción ideal debería ser económica, simple, proveer suficiente cantidad y calidad, así como no utilizar agentes peligrosos para el ambiente. La concentración de ADN final no solo depende del tipo de tejido utilizado y su tamaño, sino también de variables propias del método de extracción, que determinan, a su vez, la pureza del material genético (Ausubel et al. 2003). Existen métodos de extracción no comerciales y comerciales. Aunque los métodos comerciales logran recuperar ADN de alta calidad son costosos para el trabajo rutinario (Niu et al. 2008), en especial, en países en desarrollo. En este contexto, se han propuesto algunos métodos no comerciales que logran obtener ADN con resultados adecuados, a un bajo costo por muestra (Niu et al. 2008; Saavedra-Matiz et al. 2013; Xin & Chen, 2012).
Uno de los métodos no comerciales que se utiliza, como base para extraer ADN en moscas, es el de precipitación salina, propuesto por Collins et al. (1987), originalmente, aplicado en mosquitos del género Anopheles y modificado para el estudio de moscas (Cadavid, 2018). Este método, se caracteriza por ser más amigable con el medio ambiente que aquellos que utilizan fenol-cloroformo u otros reactivos peligrosos (Panigrahy et al. 2022).
Con relación al tipo de tejido, el par de patas medias es el tejido seleccionado, debido a que, usualmente, no son utilizadas para la identificación morfológica en estas moscas, lo que permite conservar los caracteres utilizados en taxonomía tradicional (Nakano & Honda, 2015; Aristizábal-Botero et al. 2016); sin embargo, algunos autores prefieren utilizar una parte de mayor tamaño, como el abdomen o el tórax, para lograr obtener mayor concentración de ADN (Guo et al. 2012). El tamaño del ejemplar, a nivel de especie, a su vez, se relaciona con el dimorfismo sexual, donde, en general, las hembras tienden a mayor tamaño que los machos, factor adicional a considerar durante la selección de especímenes (Benítez, 2013; Nuñez Rodríguez & Liria, 2017; Cortés-Suarez et al. 2021).
El primer paso del protocolo de Cadavid (2018) requiere una maceración del tejido, la cual, se puede lograr manualmente, con un macerador plástico o de manera semiautomática, consistente en una previa cristalización del tejido con nitrógeno líquido y posterior agitación mecánica del tejido con perlas hasta su pulverización en un agitador. Un reporte previo sugiere que el método semiautomático mejora significativamente la concentración de ADN obtenida, con relación a los métodos manuales, a partir de fitoplancton (Yuan et al. 2015); sin embargo, no se conocen datos en moscas. El impacto de otras variables del proceso de extracción de ADN relacionadas con el tamaño de los especímenes, los cuales, pueden diferir en promedio entre las especies o la cantidad de etanol utilizada para la precipitación de ADN, no han sido probadas en este modelo.
El objetivo de este estudio fue comparar la concentración y la pureza del ADN obtenido de moscas de tres especies, pertenecientes a tres familias de importancia forense, cuando se utiliza el protocolo de Cadavid (2018), con modificaciones en el tiempo de incubación inicial en la fase de lisis, la maceración: manual versus agitación con perlas de vidrio o el volumen de alcohol utilizado durante la fase de precipitación de ADN. Adicionalmente, se evaluaron las diferencias potenciales en los resultados entre especímenes machos y hembras y entre el uso de las patas medias versus el abdomen, como tejido de partida para la extracción.
MATERIALES Y MÉTODOS
A partir de la Colección Entomológica Tecnológico de Antioquia, CETdeA, se seleccionaron especímenes de Peckia collusor (Curran & Walley, 1934) (Sarcophagidae), Biopyrellia bipuncta (Wiedemann, 1830) (Muscidae) y Fannia pusio (Wiedemann, 1830) (Fanniidae), los cuales, estuvieron conservados en etanol al 80 %, entre 3 y 8 años. Los especímenes, se identificaron morfológicamente con claves taxonómicas especializadas (Carvalho & Mello-Patiu, 2008; Buenaventura & Pape, 2013; Durango & Ramírez-Mora, 2019; Grisales & de Carvalho, 2019). Se procesó un total de 180 especímenes, divididos en cinco grupos experimentales (Tabla 1). El ADN, se obtuvo mediante el protocolo descrito por Cadavid (2018), que consiste en cuatro fases lisis: i) maceración del tejido, mediante la cristalización del tejido con nitrógeno líquido y maceración de las muestras, en un homogenizador de tejidos (Tissuelyser ll, QIAGEN), durante cinco minutos, a 300 frecuencias por segundo, exceptuando el grupo experimental, donde se comparó la maceración con perlas vs manual; ii) ruptura celular, mediante el buffer de lisis (NaCl 0,1 mol/l; Sucrosa 0,1 mol/l; Tris 0,1 mol/l; EDTA 0,05 mol/l y SDS 0.5 %); iii) incubación de las muestras con el buffer de lisis en baño seco, a 56 oC, durante 2 horas o toda una noche, según el grupo experimental y iv) precipitación del ADN con etanol en diferentes volúmenes (1:1, 1:3, 1:5 y 1:8).
Tabla 1 Grupos experimentales y muestras utilizadas para la extracción de ADN de moscas.
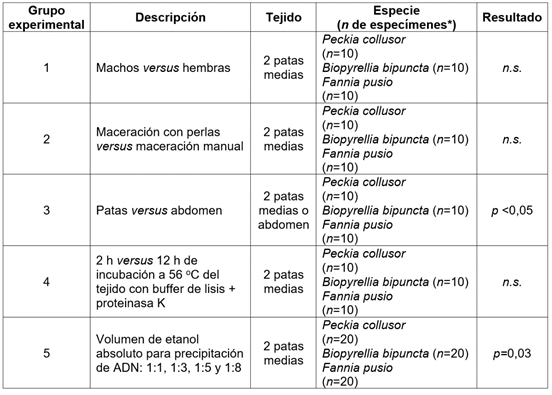
* El número de especímenes en cada una de las especies fue equitativamente dividido, de acuerdo con la descripción de cada grupo experimental.
n.s.: no se encontraron diferencias estadísticamente significativas.
Se reporta el p valor de las comparaciones, en donde se encontraron diferencias estadísticamente significativas.
La medición de la concentración de ADN (ng/µl) y su pureza (A260/280), se realizó para cada muestra por triplicado, en un espectrofotómetro NanoDrop®, con un volumen de 2 μl por cada muestra ND-1000 (Thermo Scientific, USA), según las instrucciones del fabricante y el dato analizado corresponde al promedio de las mediciones por muestra. Para los primeros cuatro grupos experimentales, los datos se compararon mediante la prueba de Mann-Whitney-Wilcoxon, mientras que las diferencias potenciales en los datos del grupo cinco, se evaluaron con la prueba de Kruskal-Wallis. Adicionalmente, se evaluaron las posibles diferencias en cantidad de ADN obtenido entre las especies mediante ANOVAs de dos factores. Se aplicó la prueba de comparaciones múltiples de Tukey en los casos de existencia de diferencias significativas (p <0,05), para discriminar las diferencias entre las medias. Para el análisis estadístico, se utilizaron los programas GraphPad Prism 5 (GraphPad Software Inc., San Diego, CA, EUA) y PAST v. 3.25 (Hammer et al. 2001).
RESULTADOS Y DISCUSIÓN
Para los grupos experimentales uno y dos (sexo y tipo de maceración, respectivamente), no se encontraron diferencias estadísticamente significativas en la concentración de ADN, ni en su pureza (p >0,05). El grupo experimental tres (patas versus abdomen) presentó diferencias en la cantidad (p <0,05), pero no en la pureza de ADN (p =0,78); la extracción de ADN a partir de abdomen rindió casi cinco veces más ADN (Me= 508,3 ng/µl), en comparación con la de las patas (Me= 108,9 ng/µl). Para el grupo experimental cuatro, el tiempo de incubación de la muestra en baño seco, a 56 ºC, tampoco generó diferencias significativas en la cantidad y la pureza del ADN recuperado.
Finalmente, el grupo experimental cinco, donde se comparó el efecto de diferentes volúmenes de etanol absoluto para la precipitación de ADN, rindió diferencias significativas, tanto en la cantidad (p =0,03) como en la pureza de ADN (p <0,0001); la dilución 1:8 mejoró sustancialmente el ADN obtenido en las muestras analizadas. El análisis de varianza de dos factores mostró diferencias estadísticamente significativas en la concentración de ADN recuperada en todos los grupos experimentales, de acuerdo con la especie (p <0,0001). A partir del par de patas medias, se recuperó, en promedio, 22,6 ng/µl de ADN por espécimen de Fannia pusio, mientras que para Biopyrellia bipuncta y Peckia collusor, tres (67,7 ng/µl) y diez veces más (227,7 ng/µl), respectivamente.
Este trabajo evidenció el impacto de variable extrínsecas, tales como la especie, relacionada con su tamaño corporal, así como el tipo de tejido utilizado para la extracción de ADN, en la concentración de ADN obtenido. Además, el impacto de variables intrínsecas del método de extracción, como el volumen de etanol absoluto utilizado durante la fase de precipitación de ADN, en la eficiencia de la extracción de ADN en moscas de importancia médico-legal, utilizando el protocolo descrito por Cadavid (2018). Por otra parte, se evidenció que otros factores, como el sexo, el tipo de maceración y el tiempo de incubación en la fase de lisis, no resultaron en diferencias significativas en la concentración y la pureza del ADN obtenido.
Las diferencias en el tamaño corporal de las tres especies evaluadas explican los resultados observados en la concentración de ADN obtenida; por ejemplo, Fannia pusio es una mosca pequeña, con una longitud entre 2,5 a 3,5 mm, mientras que las especies de la familia Sarcophagidae, a la que pertenece Peckia collusor, pueden medir entre 2 y 25 mm (Carvalho & Mello-Patiu, 2008; Paseto et al. 2019). Adicionalmente, en correspondencia con el tamaño del tejido, en todas las especies analizadas, se obtuvo mayor concentración de ADN cuando se usó el abdomen, que a partir del par de patas medias. Aun así, la cantidad de ADN obtenida a partir del par de patas medias (108,9 ng/µl), se considera suficiente para el éxito de la técnica PCR, con la ventaja de preservar el espécimen y conservar información de estructuras, que son de gran relevancia taxonómica, aspecto óptimo en el marco de estrategias de identificación molecular (Prendini et al. 2002; Ratnasingham & Hebert, 2007); no obstante, se requiere la evaluación de otras variables que pueden afectar la PCR, como la presencia de inhibidores en el extracto de ADN obtenido. Los valores A260/280, una medida de la pureza del ADN extraído, en todos los grupos experimentales, fue inferior a 1,8, dato que sugiere contaminación potencial por proteínas o sales (Boesenberg-Smith et al. 2012). Aunque la pureza del ADN con este método de precipitación salina no es alta, ha sido de utilidad para la amplificación exitosa de diversos fragmentos de ADN nucleares y mitocondriales, tanto en moscas como en otros dípteros (Gómez et al. 2015), lo que indica que no representa una limitante para implementar este protocolo de forma rutinaria.
Los resultados de este estudio sugieren que el incremento de la relación entre el volumen de etanol y el extracto de ADN en la fase de precipitación del ADN mejora, sustancialmente, la concentración final de este ácido nucleico en las muestras. La molécula de ADN está cargada negativamente, debido a los residuos de fosfato y se encuentra altamente hidratado en una fase acuosa. El etanol altera las capas de hidratación del ADN y permite que los residuos de fosfato formen enlaces iónicos con cationes en el solvente y cuando la concentración de etanol se aproxima al 70 % en presencia de una alta concentración de sales, las fuerzas repulsivas entre las cadenas de polinucleótidos, se reducen hasta precipitar el ADN (Green & Sambrook, 2016), resultando en un incremento en la eficiencia del método utilizado en este estudio.
Es de resaltar que el tiempo de incubación del tejido a 56 °C con buffer de lisis y proteinasa K, no afectó la cantidad de ADN recuperado. Este resultado permite disminuir el tiempo de incubación en el baño seco de las muestras, con la consecuente ventaja de reducir el gasto de energía del equipo y el tiempo empleado durante la extracción de ADN. Similarmente, los hallazgos no evidenciaron que el método de maceración semiautomático mejorara el rendimiento en la concentración de ADN obtenido; este resultado es contrastante con un estudio previo en artrópodos, donde este tipo de maceración mejora el rendimiento (Ammazzalorso et al. 2015). Aun así, para la extracción de ADN de moscas con el protocolo de Cadavid (2018), se recomienda utilizar el método semiautomático, debido a que se reduce el tiempo del protocolo en este paso, el riesgo de contaminación de las muestras y se eliminan factores de riesgo relacionados con el desarrollo de síndrome del túnel carpiano (El-Helaly et al. 2017) y otras tendinopatías, potencialmente, asociados a actividades, como la maceración manual de las muestras durante este proceso.
La extracción de ADN a partir de un tejido de mayor tamaño, así como la reducción del tiempo de incubación durante la fase de lisis celular y el incremento en el volumen de etanol absoluto en la fase final de precipitación de ADN, mejoran significativamente la concentración de ADN obtenido, al utilizar este protocolo no comercial en moscas de interés médico-legal. Este método modificado es más amigable con el medio ambiente en comparación con otros métodos no comerciales, basados en fenol-cloroformo y otros reactivos más tóxicos y, además, permitió la recuperación de ADN a partir de especímenes almacenados en etanol al 80 %, con una antigüedad de hasta ocho años, insumo clave para el desarrollo de estudios de identificación y sistemática molecular.















