1. INTRODUCCIÓN
1.1 Consumo de leche
La leche desempeña un papel fundamental que inicia desde el periodo de lactancia en las distintas especies de mamíferos. La producción preservada de lactasa intestinal presenta, desde una perspectiva evolutiva, un rasgo que se asocia a la duración del proceso de amamantamiento y a la dependencia de la leche como un nutriente en el desarrollo de los mamíferos [1]. La sustitución de la leche en la dieta se ha visto limitada a causa del desafío que representa hallar una bebida que provea la amplia gama de nutrientes que esta ofrece[2].
La leche bovina, obtenida del rumiante Bos taurus, presenta el mayor porcentaje de consumo a nivel mundial con un 85 %; no obstante, la ingesta de leche derivada de otras especies rumiantes como Bubalus bubalis (búfalo), Capra hircus (cabra) y Ovis aries (oveja) corresponde a un 11 %, 2.3 % y 1.4 %, respectivamente. [3]. Estos porcentajes resultan ser bastante fluctuantes, pues, en la actualidad, factores como la preferencia de un tipo de leche frente a otra, la elección con base en el valor nutricional, o inclusive el aumento en la frecuencia de alergias a la leche bovina, inclinan la balanza hacia el consumo de leche proveniente de otros organismos rumiantes.
1.2 Fracción sólida de la leche
El consumo de leche de distintas especies representa una fuente de nutrientes variable, pues su contenido y valor nutricional no son del todo similar. La variedad en la composición química de la leche depende de numerosos factores: especie del mamífero, rasgos genéticos, variantes fisiológicas, entre otras [4]. En general, la leche es una suspensión de diferentes nutrientes entre los cuales se encuentran proteínas, grasas, azúcares, vitaminas y minerales. La fracción sólida de la leche (nutrientes grasos y no grasos) corresponde a un valor alrededor de 15 %, dentro del cual las proteínas representan un valor mayor al 3 %, en cuanto a sólidos no grasos [5].
Teniendo en cuenta que los sustratos de las enzimas proteolíticas corresponden a la fracción sólida no grasa, esta investigación se centra en la descripción de las diferentes concentraciones de proteínas en los distintos tipos de leche. Los valores de concentración se presentan en la Tabla 1.
Tabla 1 Concentración de proteínas (g/L) en la leche de bovino, oveja, cabra y búfalo
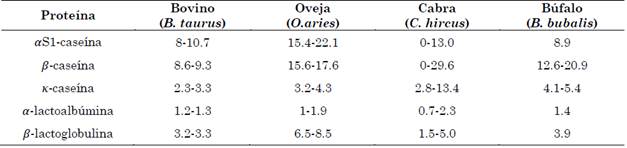
Fuente: Tabla obtenida y modificada de [6].
Las proteínas de la leche se agrupan en dos grandes categorías: caseínas y seroproteínas, siendo las primeras las de mayor abundancia. Las caseínas se caracterizan por la formación de micelas como consecuencia de la agrupación de las mismas [5]. Dentro de las caseínas se encuentran la αS1-caseína, β-caseína y la κ-caseína. Entre las seroproteínas, o proteínas del suero, se encuentran la α-lactoalbúmina y la β-lactoglobulina. Estas proteínas son, en general, menos solubles que las caseínas y cumplen diversas funciones biológicas como constituyentes del complejo enzimático productor de lactosa. Es indispensable resaltar que la síntesis endógena de estas proteínas, como la de cualquier otro polipéptido, depende en gran parte del gen de la especie que codifique para este. Siendo así, se establece que las proteínas derivadas de los rumiantes anteriormente mencionados presentan cierto grado de similitud, más no son idénticas en su totalidad. Para las cuatro especies, existe una isoforma de cada caseína y cada seroproteína.
1.3 Péptidos Bioactivos (PBA)
Existe un abundante número de alimentos con propiedades nutracéuticas que se atribuyen a los productos de su digestión proteolítica; estos fragmentos aminoacídicos poseen un alto potencial de acción biológica y se denominan péptidos bioactivos (PBA) [7]. La leche se caracteriza por ser un alimento precursor de una gran variedad de PBA. Entre los productos de hidrólisis de las proteínas de la leche se reportan péptidos con acción inhibitoria de ACE, antioxidantes, inmunomoduladores, opioides, antimicrobianos, antihipertensivos y antidiabéticos [8], [9]. Ahora bien, la acción biológica del péptido depende de la secuencia de aminoácidos constituyentes, y esta a su vez, está determinada por los puntos de clivaje en el sustrato precursor del péptido por parte del catalizador proteolítico. La hidrólisis por proteasas ajenas al sistema digestivo humano produce PBA con acción biológica distinta a los que producirían las proteasas digestivas.
Las caseínas y seroproteínas anteriormente mencionadas, constituyen la fracción proteica de la leche, y, por tanto, son sustratos para las proteasas del sistema digestivo. Dado el grado de homología variable entre las isoformas de las proteínas presentes en la leche de bovino, oveja, cabra y búfalo, es válido afirmar que los puntos de clivaje, y consecuentemente los PBA liberados, variarán de una especie a otra.
1.4 Estudios e investigaciones in silico
La experimentación in vitro e in vivo son herramientas indispensables en las disciplinas científicas; no obstante, los recientes avances de la bioinformática han permitido desarrollar sistemas que complementan estas investigaciones mediante simulaciones computacionales de fenómenos químicos y biológicos. El estudio de PBA requiere montajes experimentales extensos y costos que limitan las posibilidades de investigación [10] mientras que las investigaciones in silico, o de simulación computacional, facilitan la identificación de fragmentos aminoacídicos a partir de métodos que se fundamentan en: análisis de la relación estructura-función de una secuencia polipeptídica, simulaciones de docking molecular y procesos de optimización estadística [11], [12].
La experimentación in silico no es independiente de la experimentación in vitro e in vivo. El estudio de los PBA se basa en el conjunto de estas tres metodologías a partir de procesos que comparen la producción, identificación y validación de péptidos mediante hidrólisis in vitro, cribado de bioactividad in silico y validación in vivo [13]. El desarrollo de estudios in silico es esencial para la identificación de fenómenos moleculares que posteriormente puedan ser comprobados y validados en organismos vivos. Siendo así, la presente investigación se enfoca en la evaluación de los PBA obtenidos a partir de procesos de digestión in silico catalizados por pepsina, tripsina y quimotripsina, empleando como sustratos a las isoformas de las caseínas y seroproteínas presentes en la leche de bovino (B. taurus), oveja (O. aries), cabra (C. hircus) y búfalo (B. bubalis
2. METODOLOGÍA
2.1 Obtención de la secuencia de aminoácidos de las proteínas
A partir de la base UniProt, se obtuvo para cada isoforma un formato de tipo FASTA con la secuencia de aminoácidos correspondientes. UniProt agrupa una serie de bancos de datos bioinformáticos que ofrecen una correlación entre la secuencia y la función de distintas proteínas registradas [14]. El primer análisis se basó en la evaluación de la homología y la comparación de las propiedades fisicoquímicas de las proteínas presentes en la leche de las cuatro especies. El estudio de homología se realizó a partir de la base de datos Clustal Omega del EMBL-European Bioinformatics Institute (EBI). Los valores de los parámetros fisicoquímicos fueron obtenidos a partir de la herramienta ProtParam en la base de datos ExPASy del Swiss Institute of Bioinformatics (SIB). Tanto Clustal Omega como ProtParam permiten extraer datos calculados a partir de la secuencia aminoácidos de un determinado polipéptido [15].
2.2 Digestión in silico y evaluación de los PBA producto de la proteólisis
La evaluación de las proteínas como precursoras de PBA se realizó mediante la base BIOPEP-UWM de la Universidad de Mazury en Olsztyn, Polonia. Las proteínas fueron sometidas a digestión in silico bajo la acción de enzimas presentes en un proceso de digestión in vivo: Pepsina (EC 3.4.23.1) a pH 1.3, Tripsina (EC 3.4.21.4) y Quimotripsina (EC 3.4.21.1).
Los PBA resultantes de la digestión fueron registrados en las Tablas 3 a 7. Para cada proteína se calcularon tres parámetros planteados por [16], a partir de los cuales se realizó un análisis comparativo. Estos parámetros fueron calculados mediante (1), (2) y (3). La frecuencia de liberación de fragmentos peptídicos bioactivos (A E ) se halló a partir de (1):
Donde d corresponde al número de péptidos bioactivos con actividad específica liberados por acción de determinadas enzimas y N corresponde al número de aminoácidos presentes en una secuencia. La frecuencia relativa porcentual de liberación de fragmentos bioactivos (W) se obtuvo mediante (2):
Donde A E corresponde a los valores de frecuencia obtenidos a partir de (1), a corresponde al número total de biopéptidos liberados y N corresponde al número de aminoácidos presentes en el sustrato. Los resultados de (2) fueron registrados en las Figuras 1 a 5.
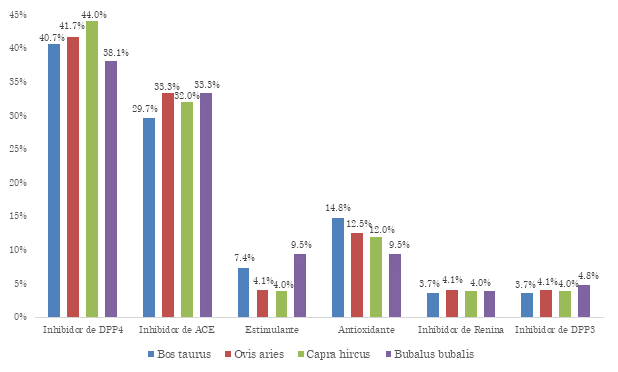
Fuente: elaboración propia.
Figura 1 Frecuencia relativa porcentual de liberación de PBA con αS1-caseína como sustrato. La evaluación estadística muestra p>0.05 en los análisis de varianza y de medias
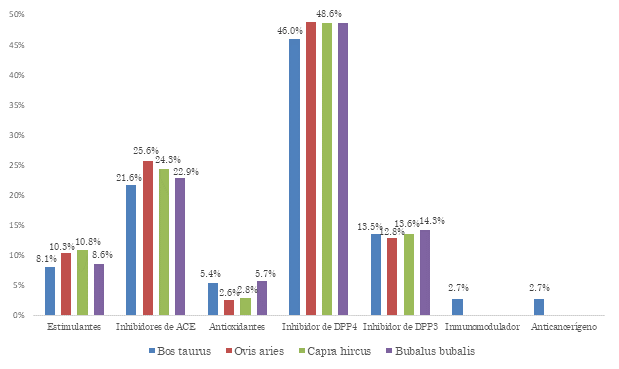
Fuente: elaboración propia.
Figura 2 Frecuencia relativa porcentual de liberación de PBA con β-caseína como sustrato. La evaluación estadística muestra p>0.05 en los análisis de varianza y de medias
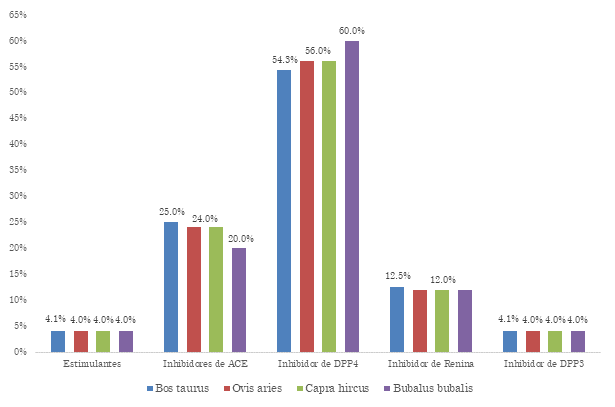
Fuente: elaboración propia.
Figura 3 Frecuencia relativa porcentual de liberación de PBA con κ-caseína como sustrato. La evaluación estadística muestra p>0.05 en los análisis de varianza y de medias
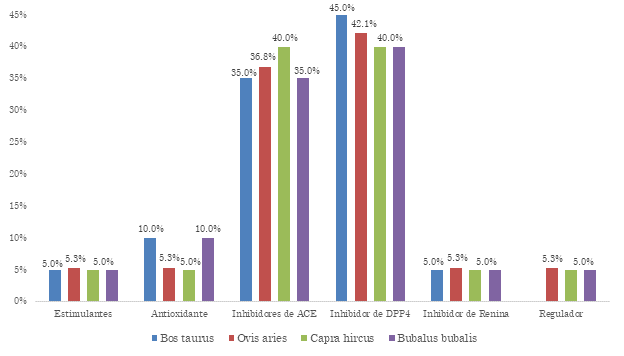
Fuente: elaboración propia.
Figura 4 Frecuencia relativa porcentual de liberación de PBA con α-lactoalbúmina como sustrato. La evaluación estadística muestra p>0.05 en los análisis de varianza y de medias
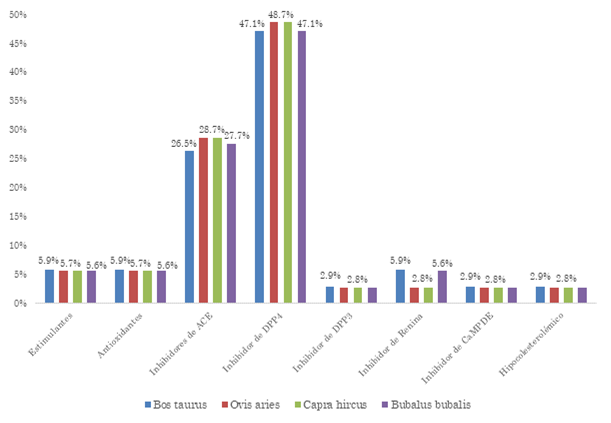
Fuente: elaboración propia.
Figura 5 Frecuencia relativa porcentual de liberación de PBA con β-lactoglobulina como sustrato. La evaluación estadística muestra p>0.05 en los análisis de varianza y de medias
Posteriormente, se realizó una sumatoria de los péptidos liberados del conjunto de proteínas pertenecientes a una única especie para determinar un perfil total de PBA obtenidos de la leche de cada organismo. De este modo, fue posible calcular la frecuencia media mediante (3):
Donde a t corresponde al número de péptidos bioactivos liberados de un conjunto de proteínas de una misma especie por acción de enzimas específicas y N t corresponde al número total de aminoácidos presentes en las proteínas de la leche de cada especie. Finalmente, los datos obtenidos de (3) fueron sometidos a un análisis de varianza de un solo factor (ANOVA) y a un análisis de medias realizado mediante la prueba de Tukey.
3. RESULTADOS Y DISCUSIÓN
3.1 Evaluación de las propiedades fisicoquímicas de las isoformas
Como se observa en la Tabla 2, el registro de variación en el número de aminoácidos para una misma proteína entre las diferentes especies es mínimo. La mayor disimilitud se presenta en la αS1-caseína de B. bubalis respecto a las isoformas de las otras especies; una diferencia de ocho residuos en la αS1-caseína de B. bubalis puede tener una repercusión considerable al comparar el número de PBA liberados contra los PBA de la misma proteína obtenidos de las otras especies. Las demás isoformas varían entre especie por un numero de residuos no superior a dos.
Tabla 2 Número de residuos y propiedades fisicoquímicas de las proteínas: masa molecular, punto isoeléctrico e hidropaticidad
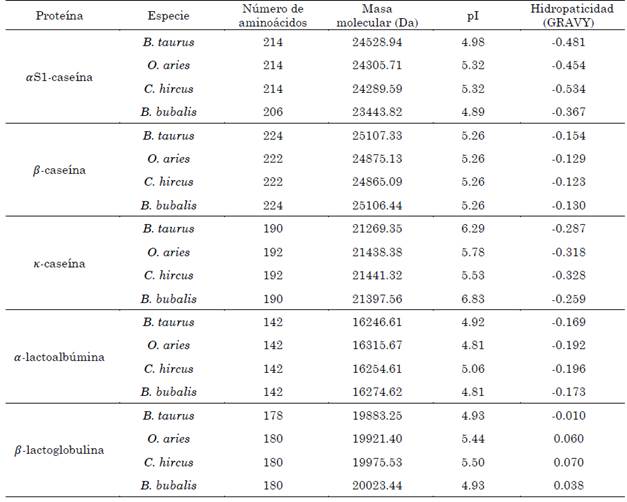
Fuente: datos obtenidos a partir de [17].
La diferencia de masa molecular alude a la variación de residuos entre las secuencias polipeptídicas. Las enzimas proteasas seleccionadas presentan alta esteroespecificidad frente a los aminoácidos que componen el enlace peptídico que se busca hidrolizar; la pepsina, en condiciones de pH<2, muestra afinidad por el clivaje de enlaces constituidos por Phe, Tyr, Trp y Leu; por otro lado, la tripsina presenta alta afinidad por residuos de Lys y Arg; y, finalmente, la quimotripsina es afín a los enlaces péptidos compuestos por Trp, Tyr y Phe [18]. Siendo así, se establece que los residuos variables en las isoformas suponen que cada proteína presentará diferencias en los puntos de clivaje proteolítico y en la secuencia en los péptidos producto de la digestión.
Entre las propiedades fisicoquímicas reportadas, punto isoeléctrico (pI) e hidropaticidad, se evidencia un alto grado de similitud en los valores obtenidos. La escala de hidropaticidad empleada refleja la solubilidad de una proteína en una solución acuosa, clasificando como solubles a los polipéptidos con valores inferiores a cero. En los resultados se observa que, a excepción de las isoformas de β-lactoglobulina de O. aries, C. hircus y B. bubalis, todas las proteínas presentan un grado considerable de hidrofilicidad. Dadas las condiciones acuosas en las que se lleva a cabo los procesos digestivos, se destaca la solubilidad del sustrato como una propiedad que facilita los procesos proteolíticos de las enzimas digestivas.
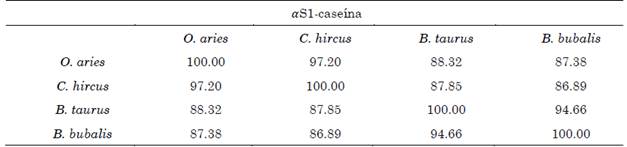
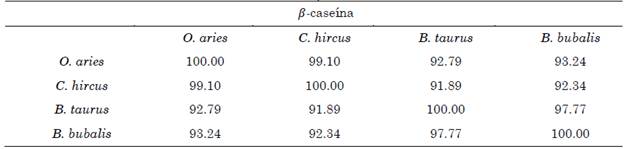
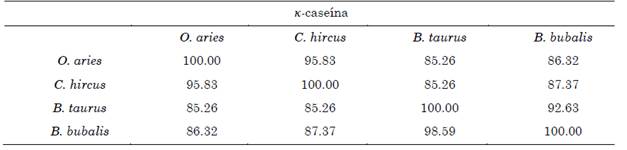
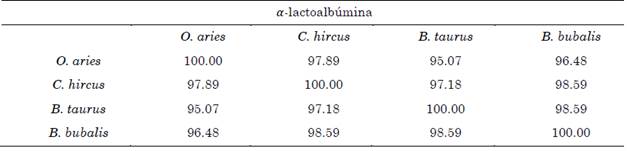
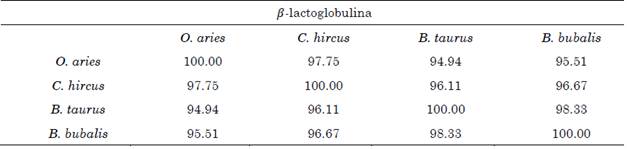
3.2 Evaluación de la homología entre las isoformas
Las Tablas 3 a 7 presentan el grado de homología entre las isoformas correspondientes a cada organismo. La representación en forma de matriz permite obtener la relación entre las cuatro especies de forma simultánea. Para las cinco proteínas estudiadas se cumple que: el bovino (B. taurus) y el búfalo (B. bubalis) presentan isoformas con un grado de homología mayor a 90 %, y la cabra (C. hircus) y la oveja (O. aries) presentan isoformas con un grado de homología mayor a 95 %. Pese a que las cuatro especies de rumiantes pertenecen a la familia Bovinidae, B. taurus y B. bubalis son especies pertenecientes a la subfamilia Bovinae mientras que C. hircus y O. aries pertenecen a la subfamilia Antilopinae [20]. Diferentes estudios se han centrado en dilucidar la relación filogenética entre las especies estudiadas por medio de análisis moleculares. Como concluyen [21], a partir de la evaluación de la homología de la secuencia de nucleótidos que codifican para la κ-caseína se determinó: un 92.7 % de homología entre O. aries y B. taurus y un 96.7 % de similitud entre B. bubalis y B. taurus. Adicionalmente, se encontró, con excepción de leves polimorfismos, una secuencia idéntica entre O. aries y C. hircus.
3.3 PBA obtenidos de la digestión in silico mediante BIOPEP UWM
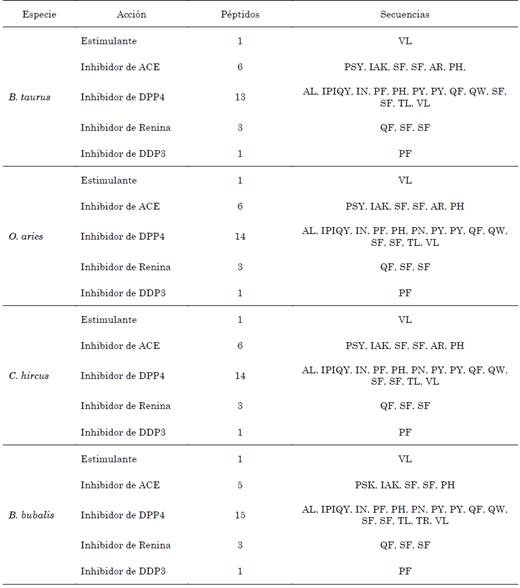
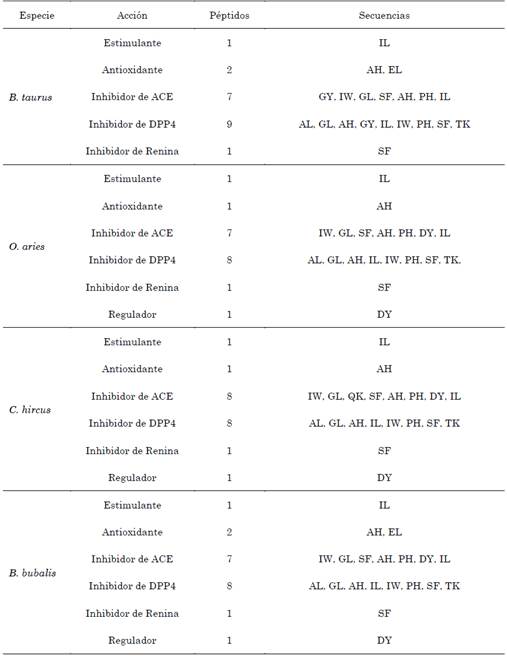
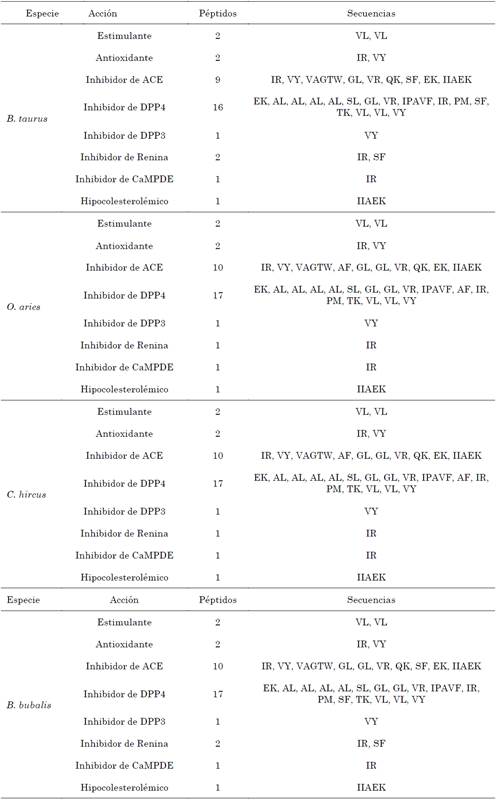
El proceso de digestión catalizado por las enzimas expuestas anteriormente se realizó mediante la base de datos BIOPEP-UWM donde, a partir de la secuencia de las proteínas presentadas en formato tipo FASTA, se obtiene el número de PBA, su acción biológica y su respectiva secuencia. Los aminoácidos se presentan en nomenclatura de una letra. Los PBA resultantes se muestran en las Tablas 8 a 12.
3.4 Frecuencia relativa porcentual de liberación de PBA bajo proteólisis in silico catalizada por Tripsina, Quimotripsina y Pepsina (pH 1.3)
A partir de los datos presentados en las Tablas 8 a 12, se procedió a calcular los valores de frecuencia de liberación de PBA mediante (1). Con base en estos resultados fue posible hallar la frecuencia relativa porcentual de obtención de PBA calculada mediante (2). Los valores de frecuencia relativa porcentual permiten realizar una comparación a nivel de porcentaje entre la frecuencia de liberación de PBA a partir de cada isoforma. Los resultados obtenidos se presentan en las Figuras 1 a 5.
3.5. Análisis de frecuencias relativas porcentuales de liberación de PBA
Los valores de la frecuencia relativa porcentual permiten evidenciar la variación en la producción de PBA entre las cuatro especies. A partir del análisis de varianza, se obtuvo un valor de p>0.05. Asimismo, al realizar la prueba de Tukey se determinaron valores <HSD, por ende, se concluye que no existe una diferencia estadísticamente significativa (p>0.05) en los valores de frecuencia relativa porcentual entre las cuatro especies.
Los resultados presentados en las Figuras 1 a 5 se analizan a continuación de forma específica para cada sustrato; es decir, de forma particular para cada isoforma del bovino (B. taurus), oveja (O. aries), cabra (C. hircus) y búfalo (B. bubalis). No obstante, se destaca que, para las cinco proteínas, la clase de PBA con mayor frecuencia relativa porcentual de liberación en las cuatro isoformas corresponde a los inhibidores de DPP4.
Para la αS1-caseína, la isoforma de la cabra presenta la mayor frecuencia relativa porcentual de liberación de inhibidores de DPP4 con un valor de 44.00 %. Las isoformas de la oveja y el búfalo presentan la mayor frecuencia relativa porcentual, 33.30 %, de liberación de PBA con acción inhibitoria de ACE. La isoforma del bovino, con un valor de 14.80 %, presenta la mayor frecuencia relativa porcentual de liberación de PBA con acción antioxidante. La isoforma del búfalo presenta la mayor frecuencia relativa porcentual de liberación de PBA con acción estimulante, 9.50 %. Los PBA con acción inhibitoria de Renina y DPP3 presentan una mayor frecuencia relativa porcentual de liberación a partir de la isoforma del búfalo, 4.80 %.
En cuanto a los PBA obtenidos de la β-caseína, la isoforma de la oveja presenta la mayor frecuencia relativa porcentual de liberación de inhibidores de DPP4 con un valor de 48.70 %.
La isoforma de la oveja también presenta la mayor frecuencia relativa porcentual de liberación de inhibidores de ACE con un valor de 25.60 %. Por otro lado, la mayor frecuencia relativa porcentual para PBA inhibidores de DPP3 y PBA estimulantes corresponde a: el búfalo con un 14.26 %, y la cabra con un 10.80 %, respectivamente. La mayor frecuencia relativa porcentual para PBA antioxidantes corresponde a la isoforma del búfalo con un valor de 5.70 %.
Para la κ-caseína, la isoforma del búfalo presenta la mayor frecuencia relativa porcentual de liberación de inhibidores de DPP4 con un valor de 60.00 %. La isoforma del bovino presenta la mayor frecuencia relativa porcentual para cuatro clases de PBA: inhibidores de ACE, con un 25.00 %; inhibidores de Renina, con un 12.46 %; e inhibidores de DPP3 y estimulantes, ambos con un 4.13 %.
Respecto a los PBA producto de la hidrólisis de la α-lactoalbúmina, la isoforma del bovino, con un valor de 45.00 %, presenta la mayor frecuencia relativa porcentual de liberación de inhibidores de DPP4. La isoforma de la cabra también presenta la mayor frecuencia relativa porcentual de liberación de inhibidores de ACE con un valor de 40.00 %. Los PBA con acción antioxidante presentan la mayor frecuencia relativa porcentual en las isoformas del búfalo y del bovino, con un valor de 10.00 % para ambos. La isoforma de la oveja presenta la mayor frecuencia relativa porcentual de liberación de PBA reguladores, inhibidores de renina y estimulantes, todos con un valor de 5.26 %.
Por último, para la β-lactoglobulina, en cuanto a los PBA de tipo inhibidores de DPP4 y ACE, las isoformas de la oveja y la cabra presentan la mayor frecuencia relativa porcentual con un valor de 48.70 % y 28.70 %, respectivamente. La isoforma del bovino presenta la mayor frecuencia relativa porcentual para PBA antioxidantes, estimulantes e inhibidores de Renina, todos con un valor de 5.90 %. Así mismo, los PBA hipocolesterolémicos, inhibidores de CaMPDE e inhibidores de DPP3 presentan la mayor frecuencia relativa porcentual en la isoforma del bovino con valores de 2.90 %, 2.90 % y 2.93 %, respectivamente.
La caracterización e identificación in silico de productos peptídicos con acción biológica es actualmente un área de investigación fluctuante debido a que la clasificación de un péptido como PBA requiere que su bioactividad sea validada in vitro. Por esta razón, a medida que se identifican nuevos agentes biológicos derivados de las proteínas de la leche, las bases como BIOPEP-UMW deben actualizarse y añadir los nuevos PBA.
Hacia el año 2009, [22] realizaron un estudio in silico de las proteínas presentes en la leche bovina. En esta investigación se evidenció una frecuencia relativa porcentual del 2.40 % en cuanto a la liberación de PBA inmunomoduladores derivados de la β-caseína de B. taurus. La simulación proteolítica se llevó a cabo empleando diferentes enzimas, algunas ajenas al sistema digestivo humano; por ende, algunos de los valores de frecuencia relativa porcentual de liberación de PBA difieren frente a los resultados obtenidos en el presente estudio. No obstante, en concordancia con esta investigación, se concluye que la mayor parte de los productos peptídicos con acción biológica son dipéptidos o tripéptidos y se enfatiza en la β-caseína, κ-caseína y α-lactoalbúmina de B. taurus como potenciales fuente de PBA al ser sometidas a proteólisis catalizada por tripsina. Adicionalmente, concluyen que las enzimas que producen la mayor cantidad de péptidos con acción biológica corresponden a la tripsina y la quimotripsina.
Asimismo, la investigación realizada por [23] se centró en la digestión in silico de la αS1-caseína y la β-caseína de C. hircus. Los datos obtenidos muestran frecuencias relativas porcentuales de 44.86 % y 46.85 % referente a la liberación de inhibidores de ACE, 61.21 % y 81.53 % frente a la liberación de inhibidores de DPP4, y 11.6 % y 6.73 % referente a péptidos antioxidantes. Pese a que los resultados difieren levemente de los valores de frecuencias relativas porcentuales determinadas en este estudio, se destacan, al igual que en la presente investigación, a la β-caseína y la αS1-caseína de C. hircus como una potencial fuente de péptidos inhibidores de DPP4 con una mayor producción frente a los inhibidores de ACE.
En general, el estudio de los derivados bioactivos de la proteólisis de la leche se lleva a cabo mediante un amplio rango de proteasas que, en su mayoría, no están presentes en el sistema digestivo humano; por este motivo, los estudios in silico que emplean enzimas proteolíticas humanas son escasos. Complementario a la investigación por simulación computacional, [24] identificó, mediante experimentación in vitro con enzimas digestivas, una elevada presencia de péptidos antioxidantes e inhibidores de ACE. Por otra parte, afirma que los polimorfismos en los sustratos afectan los PBA derivados de la digestión. De forma similar [25] compararon los productos bioactivos de los derivados lácteos de bovino, oveja y cabra mediante experimentación in vitro empleando enzimas digestivas. Al igual que en la presente investigación, los resultados mostraron que, a pesar de las variaciones en los PBA y el distinto grado de homología entre las isoformas de las especies, no hay una diferencia estadísticamente significativa en la concentración de PBA liberados, además, se identificó que independiente al origen de la proteína, todas presentaron una alta concentración en solución de PBA inhibidores de ACE.
3.6 Análisis de medias y análisis de varianza para los valores de frecuencia media
La evaluación de frecuencia media permite realizar una comparación cuantitativa entre los cuatro tipos de leche. De forma semejante a lo que se evidenció en la evaluación de la frecuencia relativa porcentual, el análisis de varianza de un solo factor (p>0.05) y el análisis de medias (<HSD) permitieron concluir que no existe una diferencia significativa entre las frecuencias medias de liberación de PBA durante la digestión del conjunto total de proteínas presentes en la leche de bovino (Bos taurus), oveja (Ovis aries), cabra (Capra hircus) y búfalo (Bubalus bubalis).
3.7 Evaluación de los valores de frecuencia media de liberación de PBA
La Figura 6 presenta las once clases de PBA obtenidos y su respectiva frecuencia media calculada a partir de (3). Los PBA estimulantes actúan a través de una serie de mecanismos dependientes de la secuencia de los mismos, entre ellos se ubican el aumento de la absorción de glucosa en tejidos musculares y el aumento de liberación de sustancias vasoactivas [26].
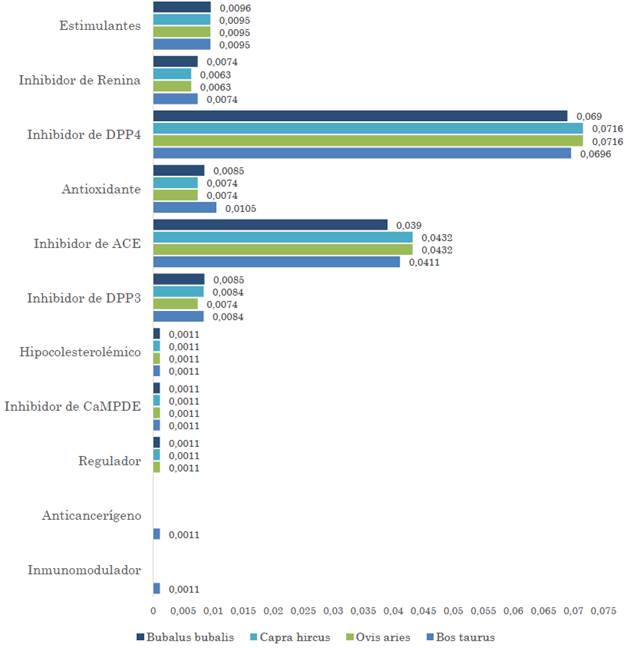
Fuente: elaboración propia.
Figura 6 Frecuencia media de PBA obtenidos de la digestión de las proteínas presentes en la leche de cada especie. La evaluación estadística presenta un valor de p>0.05 en los análisis de varianza y de medias
Los PBA inhibidores de Renina actúan mediante la alteración del eje hormonal Renina-Angiotensina-Aldosterona; siendo así, la inhibición de la Renina suprime la acción de la Angiotensina-II reduciendo sus efectos en el aumento de la presión sistémica [27].
Los PBA inhibidores de DPP4, como su nombre lo indica, inhiben la acción catalítica de la enzima Dipeptidil Peptidasa-4 [28]. Los PBA antioxidantes se asocian a diferentes mecanismos mediante los cuales ejercen su función protectora, entre ellas se encuentran: la inhibición de la peroxidación lipídica, activación de la vía Nrf2 y la inducción de enzimas antioxidantes [29]. Los PBA inhibidores de ACE reducen la actividad catalítica de la Enzima Convertidora de Angiotensina (ACE), por esta razón, ejercen un efecto regulador en la presión sanguínea [30]. Los PBA inhibidores de DPP3, de forma similar a los inhibidores de DPP4, suprimen la acción de la Dipeptidil Peptidasa-3 y, consecuentemente, inducen respuestas favorables en términos de hemodinámica [31]. Los PBA hipocolesterolémicos tienen efectos reductores sobre los niveles de colesterol sanguíneo [32]. Los PBA inhibidores de CaMPDE (Fosfodiesterasa dependiente de Calmodulina) inhiben la respectiva enzima en vías de señalización intracelular [33]. Los PBA reguladores, particularmente el dipéptido con secuencia DY (ver Tabla 5), tienen una función reguladora de flujo iónico a través de sistemas de membranas [34]. En las clasificaciones de PBA con carácter anticancerígeno e inmunomodulador se encuentra únicamente un péptido, PGPIPN (ver Tabla 3), el cual ha sido aislado in vitro y se ha determinado su acción antitumoral e inmunomoduladora [35].
Adicionalmente, se destaca que la mayor parte de los PBA son dipéptidos; siendo así, estos son fácilmente absorbidos por transportadores de tipo SLC15A1 sin ser previamente hidrolizados por acción de endopeptidasas o aminopeptidasas presentes en las microvellosidades de los enterocitos [36].
Los resultados muestran, para las proteínas de la leche de las cuatro especies, una mayor frecuencia media de liberación de PBA con carácter inhibidor de DPP4. Seguido a esto, la producción de PBA con acción inhibitoria de ACE presenta la segunda frecuencia media más elevada para las cuatro especies. Los PBA estimulantes, inhibidores de Renina, inhibidores de DPP3 y antioxidantes presentan una frecuencia media considerable cuyo mayor valor, entre las cuatro especies, corresponde a: búfalo, bovino y búfalo por igual, bovino, y búfalo, respectivamente. Los PBA con acción hipocolesterolémica, inhibitoria de CaMPDE y reguladora tienen una frecuencia media relativamente baja en comparación con los demás PBA. Las proteínas de la leche de bovino no muestran producción de péptidos reguladores al ser sometidas a digestión. Los PBA con acción anticancerígena e inmunomoduladora se obtienen únicamente de la digestión de proteínas de la leche de bovino, sin embargo, presentan una frecuencia considerablemente baja.
En concordancia con la presente investigación, diversos estudios afirman que las proteínas presentes en la leche son una potencial fuente de dipéptidos y tripéptidos con acción biológica; la α-lactoalbúmina, κ-caseína y la β-caseína de la leche de bovino fueron reportadas en estudios in silico e in vitro como proteínas precursoras de un gran número de derivados peptídicos bioactivos [37], [38]. Estas proteínas, junto a la αS1-caseína y a la β-lactoglobulina del bovino, fueron blanco de estudios donde se determinó, al igual que en la presente investigación, una producción elevada de PBA con potencial inhibitorio de DPP4 [39]-[41].
3.8 Comparación de PBA derivados de las isoformas presentes en la leche de bovino, oveja, cabra y búfalo
Los resultados de la investigación muestran un perfil de PBA para cada tipo de leche, donde los valores máximos de cantidad y frecuencia por cada clase de péptido se encuentran distribuidos de la siguiente forma: 9 péptidos estimulantes para las cuatro especies y una frecuencia media levemente mayor para el búfalo con un valor 0.096, 7 péptidos inhibidores de Renina con una frecuencia media de 0.0074 para el bovino y el búfalo, 68 péptidos inhibidores de DPP4 con una frecuencia media de 0.0716 para la cabra y la oveja, 10 péptidos antioxidantes con una frecuencia media de 0.0105 para el bovino, 41 péptidos inhibidores de ACE con una frecuencia media de 0.0432 para la cabra y la oveja, 8 péptidos inhibidores de DPP3 para el bovino, la cabra y el búfalo, este último con una frecuencia media levemente mayor con un valor de 0.0085. Se obtuvo 1 péptido con acción hipocolesterolémica y 1 péptido inhibidor de CaMPDE a partir de la β-lactoglobulina de las cuatro especies. Se evidencia la producción de 1 péptido regulador obtenido de la leche de búfalo, cabra y oveja únicamente. Finalmente, se obtuvo 1 péptido con acción inmunomoduladora y anticancerígena, este, como se mencionó anteriormente, fue producto de la digestión de la β-caseína del bovino.
Los estudios referentes a PBA producidos mediante hidrólisis catalizada por enzimas presentes en organismo humano se han centrado en la leche de bovino debido a su elevado porcentaje de consumo a nivel mundial. Se han reportado investigaciones donde se evalúa el potencial productor de PBA de las proteínas de la leche de búfalo, camello, cabra, oveja, yegua y yak [42] donde se obtuvo péptidos opioides, inhibidores de ACE, antitrombóticos, citotóxicos, entre otros. Sin embargo, el proceso de proteólisis fue llevado a cabo por enzimas ajenas al sistema digestivo humano.
Recientemente se ha destacado la leche de especies como la oveja, la cabra y el búfalo como un nutriente viable que sustituye la leche bovina [43]-[45]. Por este motivo, resulta pertinente una evaluación comparativa del perfil de PBA obtenidos de las proteínas de leche de bovino, oveja, cabra y búfalo.
Diferentes investigaciones han evaluado los PBA obtenidos de las isoformas de cada especie de forma particular. En [46] concluyen que los péptidos derivados de la β-caseína y las α-caseínas de la leche de cabra inducen una mejoría en la resistencia a la insulina. Por su parte, en [47] demostraron que los polimorfismos en la αS1-caseína del búfalo afectaban el proceso de digestión, y consecuentemente los PBA liberados. De forma similar a los resultados de esta investigación, se halló un porcentaje considerable de PBA con acción inhibitoria de ACE. [48] determinaron, a partir de experimentación in vitro, un elevado número de PBA con acción inhibidora de ACE producto de la hidrolisis de proteínas de la leche de oveja.
3.9 Aplicación de proteínas de la leche como potencial fuente de administración de moléculas con efecto terapéutico
Entre los diversos ámbitos de relevancia de los PBA, se destaca su interacción con la DPP4. La Dipeptidil Peptidasa-4 (DPP4) es una enzima de tipo serin-proteasa que puede hallarse en forma soluble en el plasma sanguíneo conservando su acción catalítica de proteólisis [49]. Esta enzima hidroliza oligopéptidos presentes en la sangre y líquidos intersticiales; por tal motivo, esta proteína ha sido blanco de investigación en el diseño de moléculas inhibitorias que contrarresten su acción de degradación [50]. Los fármacos inhibidores de DPP4 son una opción terapéutica en pacientes con Diabetes Mellitus Tipo 2, esto debido a su acción supresora en el proceso de degradación de péptidos hipoglucemiantes como GLP-1 y GIP, asociados al efecto incretina [51]. El uso de inhibidores de DPP4 en el manejo de pacientes diabéticos permite evadir los riesgos asociados a otras formas de regulación glicémica [52].
Adicional al manejo homeostático de la glucosa, se ha destacado un efecto protector sobre el sistema cardiovascular y renal por parte de estas moléculas [53], principalmente mediante propiedades antihipertensivas y antiinflamatorias [54], [55], [56] plantean que el consumo de alimentos con proteínas precursoras de inhibidores de DPP4, como las proteínas en la carne de cerdo, pueden ser un complemento dietario en paciente con alteraciones en la regulación de la glicemia. De forma similar, la ingestión de extractos proteicos de la leche sugiere un mecanismo de administración enteral de inhibidores de DPP4 [57], no obstante, se requiere una evaluación detallada de la efectividad y los mecanismos de acción in vivo que quedan sujetos a futuros estudios e investigaciones.
4. CONCLUSIONES
Las caseínas y seroproteínas presentes en la leche de bovino, oveja, cabra y búfalo muestran grados de homología distintos. Consecuentemente, al ser hidrolizadas por acción simultanea de la pepsina, tripsina y quimotripsina se observan diferentes puntos de clivaje y productos de digestión.
A partir de simulación proteolítica en BIOPEP-UWM es posible evidenciar once clases de PBA: estimulantes, inhibidores de Renina, inhibidores de DPP4, antioxidantes, inhibidores de ACE, inhibidores de DPP3, hipocolesterolémicos, inhibidores de CaMPDE, reguladores, anticancerígenos e inmunomoduladores. Cada clase de PBA se produce en frecuencias variables dependientes de la isoforma. Los PBA inhibidores de DPP4 presentan la mayor frecuencia de liberación en las cuatro especies.
Pese a la diferencia en los valores de frecuencia, los cuatro perfiles totales de PBA correspondientes a la leche de cada especie no muestran una diferencia significativa (p>0.05). El grado de inhibición o estimulación in vivo, así como el efecto terapéutico de cada PBA producto de la digestión de las proteínas de la leche, está sujeto a futuros estudios e investigaciones.