1. INTRODUCCIÓN
El cáncer de próstata es el cuarto más frecuente en el mundo, con más de un millón doscientos mil nuevos casos y más de trescientas mil muertes cada año [1]. Existen diversas herramientas diagnósticas como la resonancia magnética, ecografía transrectal, pero con reportadas limitaciones de especificidad en la tarea de detección, caracterización y prognosis de la enfermedad [2]. Es por ello por lo que hoy en día las biopsias constituyen el principal método para cuantificar la agresividad de la enfermedad, mediante un análisis histológico de imágenes microscópicas. Estas imágenes son obtenidas mediante la tinción de muestras con hematoxilina y eosina (H y E), que permiten destacar y caracterizar la distribución arquitectural de las células y la geometría atípica de estructuras glandulares [3].
El sistema de puntuación de Gleason es el principal método de apoyo para los patólogos en cuanto a la cuantificación, la estandarización del diagnóstico y la descripción de patrones característicos relacionados con la evolución de la enfermedad. Para ello, este sistema se basa principalmente en el análisis de estructuras glandulares, definiendo su estándar en dos escalas de puntuación. La primera escala está dedicada a caracterizar variaciones atípicas en patrones visuales con puntuaciones entre uno y cinco. En una segunda escala de Gleason se determina el grado de afectación y progresión de la enfermedad, según la suma de los valores predominantes de la primera escala. Esta segunda escala de diagnóstico está acotada entre valores de seis y diez. Específicamente, en esta segunda escala se obtiene una valoración de la muestra mediante la suma entre los dos grados más comunes en la muestra. Sin embargo, el procedimiento de este sistema es tedioso, estimándose que para un experto puede tomar hasta tres días en el etiquetado de seis a quince muestras de una sola biopsia [4]. Además, en la rutina clínica este procedimiento es totalmente dependiente de la interpretación visual del experto, lo que introduce una gran variabilidad en el diagnóstico. Diversos estudios han reportado valores bajos y moderados de concordancia, que van desde puntajes promedios de 0.55 (sobre 30 muestras y tres patólogos) hasta valores bajos de 0.33 (sobre 20 muestras y 24 patólogos) [5]-[7].
En la literatura han emergido diferentes propuestas computacionales y sistemas de apoyo que buscan mitigar esta variabilidad y subjetividad en el diagnóstico. Por ejemplo, en [8] se utilizaron características texturales y morfológicas para clasificar epitelio benigno, estroma benigno, grado 3 y grado 4 de Gleason. [9] proponen la clasificación de tejido benigno y maligno utilizando una descomposición piramidal de las histologías. En este trabajo, se extraen características de textura en cada descomposición para su posterior clasificación, utilizando una clasificación en cascada. En [10] se propone un esquema de segmentación de glándulas aplicando un filtro de varianza que calcula características asociadas al tamaño y la forma de las glándulas para generar un índice, proporcional a la malignidad del cáncer. Estos enfoques, sin embargo, no suelen generalizar completamente la enfermedad al limitarse a un cierto número de características predefinidas, y dependen de las normalizaciones de color, el tamaño de las muestras y el aumento, entre otras dependencias.
En años recientes, los enfoques basados en aprendizaje profundo han mostrado grandes resultados en diferentes campos de aplicación, incluyendo la histología. Por ejemplo, en [11] se entrenó una red neuronal convolucional (CNN) en un conjunto de más de 800 imágenes para la clasificación de parches benignos, Gleason 3, 4 y 5. En [12] se implementó una red InceptionV3, que hace uso de la factorización de kernel y las conexiones residuales para profundizar sin sufrir desvanecimiento de gradiente. Esta red se utilizó para clasificar los grados de Gleason 3 y 4 en parches histológicos.
Otros enfoques en redes neuronales incluyen técnicas de detección de regiones de interés [13] y segmentación de glándulas [14]. Estas estrategias se centran en una caracterización local de la enfermedad, dividiendo las imágenes histológicas en parches, lo cual va en contravía o resulta insuficiente, según lo definido en la escala de Gleason. También se han realizado esfuerzos orientados a la segmentación de glándulas cancerígenas en imágenes histológicas. Por ejemplo, [15] proponen un método de segmentación utilizando tres redes convolucionales para la detección de tumor, detección de tejido no epitelial y, finalmente, para la segmentación de las glándulas prostáticas. Por otra parte, en el trabajo de [16] se implementaron cuatro arquitecturas de redes neuronales convolucionales: FCN-8s, dos variantes de SegNet y U-Net multiescala, para la segmentación semántica de tumores de alto y bajo grado en glándulas, según la escala de Gleason.
Este trabajo introduce una estrategia para segmentar y estratificar patrones en imágenes histológicas según la escala de Gleason. En este sentido, los puntajes de Gleason son asociados a patrones espaciales particulares, que pueden ser segmentados como regiones coherentes en una imagen histológica. Se implementó una estrategia multinivel utilizando representaciones con el método Mask R-CNN, un esquema de segmentación supervisado basado en CNN. En un primer nivel de procesamiento, se toman todos los segmentos delineados por patólogos, asociados a diferentes grados de Gleason, y se ajusta una representación profunda de tipo Mask R-CNN. Esta arquitectura entrenada genera delineaciones regionales, siguiendo un grado de Gleason y con una probabilidad asociada de predicción. Sin embargo, para ciertas regiones se pueden obtener múltiples segmentaciones para una misma región debido a la alta variabilidad del problema. Entonces, se definió una segunda representación de tipo Mask R-CNN dedicada únicamente a definir segmentaciones en regiones desafiantes, con múltiples candidatos de grado para una misma localización. Finalmente, en un tercer nivel de representación, se entrenó una representación con un enfoque regional y granular, tomando como regiones de entrada segmentos con mayor atención, en las capas de atención, de la representación del primer nivel. La superposición de los tres niveles de representación conlleva a la segmentación final. Esta estrategia puede soportar los diagnósticos en la escala de Gleason, así como también permite proponer marcaciones iniciales a los patólogos para agilizar su tarea de análisis.
2. METODOLOGÍA
Los patrones espaciales que demarcan un grado específico de Gleason agrupan estructuras histológicas que pueden ser regiones características de un estadio particular de la enfermedad. Bajo esta hipótesis, en este trabajo se propuso un método de segmentación para modelar las anotaciones de grados de Gleason, pretendiendo ser una herramienta de soporte para la rutina de análisis de los patólogos. El esquema de segmentación propuesto utiliza como módulo de modelamiento una representación profunda de tipo Mask R-CNN, que ha demostrado robustez para obtener segmentaciones semánticas en diferentes escenarios.
Teniendo en cuenta la complejidad del problema de caracterización de grados de severidad de cáncer, aquí se implementó una estrategia progresiva que entrena diferentes versiones de Mask R-CNN para obtener una segmentación final. En el primer nivel de representación, se entrenó una red regional utilizando anotaciones delineadas por un experto patólogo en imágenes histológicas. En el segundo nivel de representación, fue entrenada una red con anotaciones que reportan una probabilidad alta, dada por el primer nivel de representación, para diversos grados de Gleason. Una última estimación de los grados de Gleason fue recuperada desde las activaciones convolucionales de una representación profunda, que indican patrones de atención dados por la Mask R-CNN para la localización de regiones de interés en placas histológicas, extrayendo nuevos indicadores y definiendo nuevas regiones de forma local. El consenso de las tres segmentaciones da origen a la segmentación propuesta para soportar la tarea de análisis de placas histológicas. La metodología propuesta se detalla en la Figura 1.
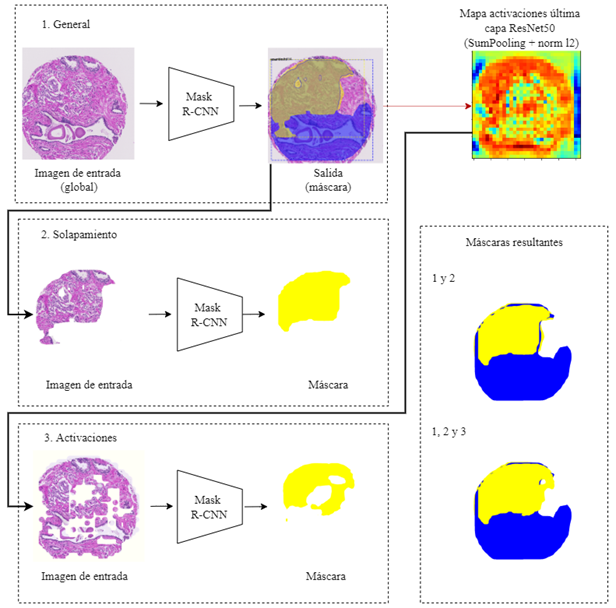
Fuente: elaboración propia.
Figura 1 Estrategia propuesta utilizando un enfoque multinivel de tres representaciones Mask R-CNN
2.1. Unidad regional de representación: Mask R-CNN
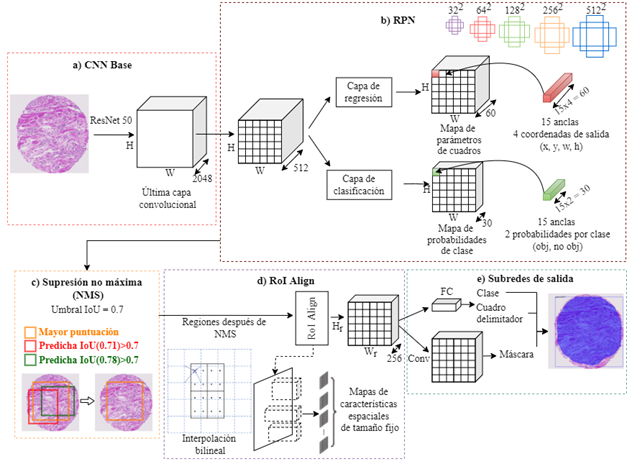
Con el fin de obtener una segmentación regional, en este trabajo se utilizó como unidad de representación la arquitectura Mask R-CNN, entrenada con diferentes conjuntos de datos y utilizando activaciones intermedias como pseudo-segmentaciones que guían la delineación final propuesta en la metodología [17]. En la Figura 2 se ilustra el esquema del funcionamiento de esta arquitectura. A continuación, se describen las etapas de la Mask R-CNN.
CNN base. En la primera fase, las imágenes de entrada se mapean a una representación profunda. Sin embargo, una principal limitación para entrenar redes profundas en el escenario histológico es el limitado número de muestras para lidiar con la amplia variabilidad y las relaciones visuales complejas que se representan. Es por ello por lo que, en este trabajo, como base de representación visual convolucional, se decidió utilizar una arquitectura residual ResNet-50. Esta red logra aprender jerárquicamente patrones visuales de los estadios de Gleason y resaltar las principales características que pueden estar correlacionadas con la severidad de la enfermedad. Esta fase es ilustrada en la Figura 2-a.
RPN (region proposal network). Es una representación de alto nivel, dada en la capa cercana a la predicción, utilizada para realizar la cuantificación de regiones de interés (RoIs), y su posterior segmentación. Específicamente, se extraen las activaciones de la última capa. Posteriormente, estas activaciones son nuevamente convolucionadas en el marco Mask R-CNN por una red donde se extraen patrones visuales. La red entrenada sobre esta capa tiene como único objetivo generar posibles regiones que contengan un objeto, llamadas regiones propuestas. Para cada posición de la ventana deslizante en las dos últimas capas convolucionales, la RPN puede predecir múltiples propuestas de región delimitadas por rectángulos de anclaje. Estos rectángulos de anclaje están centrados en cada posición de la ventana deslizante de las capas anteriormente mencionadas, y consisten en diversas escalas que indican tamaños de la región, y diferentes relaciones de aspecto que indican proporciones entre la anchura y la altura de una región. Una ilustración de los rectángulos de anclaje está disponible en la Figura 2-b. Cada región propuesta por RPN devuelve una puntuación relativa a la presencia del objeto en esa región concreta y las coordenadas que representan la región propuesta.
Supresión no máxima (NMS). Para todas las regiones predichas por RPN, se realiza un cálculo para seleccionar solamente un conjunto de regiones significativas. Este cálculo es basado en la intersección sobre la unión (IoU) entre la región predicha con mayor puntuación y las demás regiones predichas, con un umbral experimental establecido de 0.7. Aquellas regiones que superan ese umbral son descartadas, eliminando así las detecciones repetidas para una misma instancia. Una ilustración de esta fase está disponible en la Figura 2-c.
Alineamiento de las regiones propuestas (RoI Align). Cada coordenada de región es normalizada para representarla en función del mapa de características. Luego, cada región de interés (RoI) con respecto al mapa de características es alineada, usando interpolación bilineal. Una ilustración de esta fase es ilustrada en la Figura 2-d.
Subredes de salida. Finalmente, para cada RoI alineada, se resuelve un problema de clasificación (etiqueta de la región), un problema de regresión de caja delimitadora (índices espaciales de la región) y una rama para la predicción de máscara de segmentación.
2.2. Primer nivel: representaciones primarias basadas en anotaciones
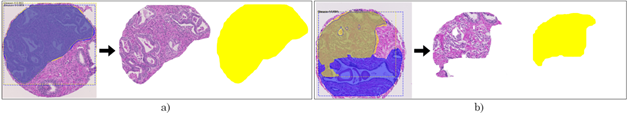
En el primer nivel de representación, para todo el conjunto de datos de entrenamiento, cada imagen de entrada o imagen histológica completa se introduce en un primer modelo Mask R-CNN que se denominó Mask R-CNN General. Esta red se entrenó utilizando las anotaciones completas realizadas por un único experto patólogo, capturando de esta manera patrones globales de la muestra histológica, por lo que su resultado fueron máscaras globales con anotaciones de áreas asociadas a un puntaje de Gleason. Esta unidad Mask R-CNN se entrena en un esquema multiclase (4 clases), lo que permite generar segmentaciones según los grados de Gleason más probables dados los patrones visuales y las segmentaciones dadas en el entrenamiento. En términos generales, este primer nivel de segmentación logra obtener segmentaciones globales, con una buena asociación, cuando solo existe un grado de Gleason en toda la placa histológica (la Figura 3 muestra un ejemplo de predicción de máscara). Por lo anterior, se puede inferir que esta representación no es suficiente para diferenciar regiones pequeñas y para detectar patrones locales en imágenes histológicas que puedan contener distintos grados de Gleason, razón por la cual es necesario definir estrategias complementarias que permitan redefinir regiones de máscaras globales, o que puedan ubicar otras asociaciones de grados diferentes en segmentos más pequeños de la imagen. Por ejemplo, una de las características de este nivel es la generación de múltiples máscaras sobrelapadas y con un alto nivel de probabilidad. Estas asociaciones sobrelapadas fueron determinadas como las más desafiantes, por lo cual se propuso un nuevo nivel de segmentación que involucrara únicamente estas regiones para dar mayor complejidad al conjunto de entrenamiento y forzar a la representación a separar estas segmentaciones. En la siguiente subsección se define la solución propuesta para estos casos de solapamiento de etiquetas.
Fuente: elaboración propia.
Figura 3 Ejemplos para el primer nivel de representación. a) Ejemplo de dato de entrenamiento: imagen histológica completa con su respectiva máscara de anotación (azul - Gleason 3) realizada por un experto patólogo. b) Ejemplo de dato de prueba: imagen histológica completa y su máscara predicha (amarillo - Gleason 4)
2.3. Segundo nivel: representaciones específicas en regiones de frontera de Gleason
En algunos casos, la salida generada por el primer nivel de representación son dos máscaras superpuestas, es decir, dos máscaras que etiquetan una región específica con dos grados diferentes de Gleason. Por lo tanto, se implementó un segundo modelo para dar solución a estos casos de solapamiento y para decidir la etiqueta correspondiente a esas regiones (ver Figura 4). Para el entrenamiento de esta segunda red, se realizó una extracción de los fragmentos de la imagen histológica y su máscara, relativos a la región de solapamiento entre las dos etiquetas predichas por la primera red. Para la prueba solo se tomaron las áreas relativas al solapamiento de las imágenes histológicas para predecirlas. Así, este segundo modelo predice y decide la etiqueta correspondiente en aquellas regiones que reportan una alta probabilidad para diferentes estadios de Gleason.
2.4. Tercer nivel: redefinición de fronteras según activaciones convolucionales
Uno de los desafíos predominantes en la segmentación de grados de Gleason tiene que ver con la definición local de las fronteras de la región. A pesar de los dos niveles de segmentación definidos previamente, se pudo observar carencia local en la definición de algunas regiones, esto debido al carácter global de la Mask R-CNN. Por lo tanto, como un tercer nivel de procesamiento, se exploraron activaciones y representaciones intermedias de la representación profunda para extraer nuevos indicadores locales para modificar las segmentaciones de un grado de Gleason específico.
Las redes neuronales convolucionales detectan y extraen, en cada capa, características específicas de los objetos presentes en las imágenes, eliminando así características más complejas en capas más profundas. Ciertas características que se resaltan en cada capa convolucional corresponden al impacto que tienen en los patrones visuales de una imagen, que logran que las neuronas se activen con mayor magnitud. Así, las activaciones son asociadas a las regiones en donde, para cierta capa convolucional, existe mayor significancia en las imágenes.
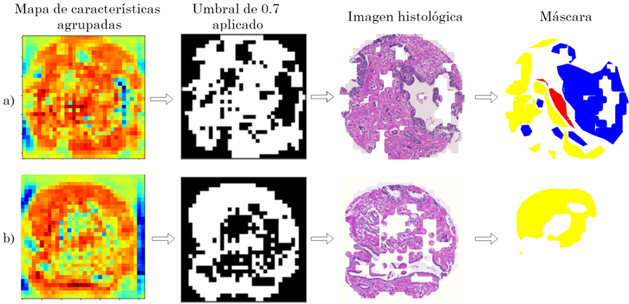
Específicamente en este trabajo, se llevó a cabo un tercer nivel de representación Mask R-CNN, en donde se tuvieron en cuenta las activaciones de la última capa de la red convolucional base (mostrada en la Figura 2-a). Esta última capa proporciona información sobre los patrones en los que se enfoca el método Mask R-CNN para localizar las regiones y predecirlas, y donde se centra la RPN para proponer regiones de interés. Esta capa tiene un total de 2048 activaciones de tamaño 32 x 32. Las activaciones de la última capa de la ResNet-50 del primer modelo se agruparon mediante SumPooling, y las características sumadas se normalizaron en l2 [18]-[19]. De este modo, se obtuvo el mapa de características agrupadas (MCA). A continuación, en el MCA, se tomaron los píxeles superiores a un umbral de 0.7, y se tomaron las regiones proporcionales a ese umbral para las imágenes histológicas y sus máscaras para el entrenamiento (ver Figura 5). Con estas regiones se entrena un nuevo nivel Mask R-CNN permitiendo una descripción más granular de las regiones. Para la prueba, sólo se tomaron las regiones proporcionales al umbral de activaciones en las placas histológicas, prediciendo así una nueva máscara, determinada a partir de las regiones de las activaciones convolucionales de la primera red.
2.5. Fusión de representaciones regionales
El presente trabajo se limitó a realizar una fusión de los resultados de las máscaras de anotación, obtenidas en los diferentes niveles, a una proporción lineal. Es decir, se implementó una fusión tardía de las máscaras obtenidas en cada nivel de representación. Sin embargo, se pueden pensar múltiples maneras de explorar representaciones multinivel y fusión de máscaras predichas. La fusión de máscaras generadas en cada nivel de representación se realizó de la siguiente manera: la máscara generada por el segundo nivel de representación se superpone en la salida anterior, es decir, la máscara generada por el primer nivel, generando una segunda máscara global a partir de la fusión de los resultados de las dos primeras redes. La máscara de salida predicha por el tercer y último nivel de representación, igualmente se superpone en el resultado anterior, es decir, la máscara acoplada por la predicción de las dos primeras redes, creando una máscara global final, generada a partir de nuevas anotaciones (ver Figura 1).
2.6. Conjunto de datos
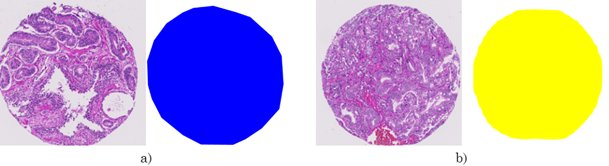
La estrategia de segmentación propuesta se entrenó y validó en un conjunto de datos publicados en el repositorio de Harvard Dataverse [20]. Este conjunto de datos contiene un total de 886 imágenes de muestras de tejido prostático, teñidas con H y E a una resolución de 40x, con un tamaño de 3100 x 3100 píxeles. Cada una de las imágenes fue digitalizada con un escáner de portaobjetos digital Hamamatsu C9600 NanoZoomer 2.0-HT [21]. Cada una de las imágenes tiene su respectiva máscara de anotación según la escala de Gleason, y están agrupadas en cinco microarreglos de tejidos (TMAs, por sus siglas en inglés). La asignación de las etiquetas de las regiones fue realizada por un uropatólogo (en este trabajo se denominará patólogo 1) en todo el conjunto de datos y por un uropatólogo adicional (en este trabajo se denominará patólogo 2) únicamente en el subconjunto de prueba. Esta disposición de las anotaciones viene dispuesta previamente en el conjunto de datos públicos. De hecho, se consideran los dos patólogos con un mismo nivel de formación. Cada etiqueta en el conjunto de datos tiene un color distintivo para identificar cada clase en la escala de Gleason de la siguiente manera: verde para benigno, azul para Gleason 3, amarillo para Gleason 4 y rojo para regiones con Gleason 5 (ver Figura 6). En cuanto al diseño experimental, los autores del repositorio proponen una partición de los datos, la cual es seguida fielmente en este trabajo.

Fuente: elaboración propia.
Figura 6 Ejemplos del conjunto de datos del repositorio Harvard Dataverse. Muestras histológicas y encima sus máscaras de anotación etiquetadas por un experto patólogo en la escala de Gleason
En este caso se utilizaron cuatro TMAs para el entrenamiento con un total de 641 imágenes histológicas y un TMA para la prueba, que corresponde a 245 imágenes histológicas.
2.7. Configuración de la estrategia
Para evaluar el esquema de segmentación propuesto se utilizó la ResNet-50 con el gradiente descendente estocástico (SGD), con una tasa de aprendizaje de 0.001. La RPN se utilizó para proponer RoIs en cinco tamaños (32 x 32, 64 x 64, 128 x 128, 256 x 256 y 512 x 512) y en tres relaciones de aspecto de anclas (1:2, 1:1 y 2:1). Se utilizó el umbral de NMS determinado por defecto en el marco Mask R-CNN, 0.7, debido a que es un valor que permite evitar repetidas detecciones de una misma instancia, disminuyendo el costo computacional al descartar regiones para etapas posteriores. Este valor discriminativo ha sido ampliamente validado sobre conjuntos de datos en imágenes naturales y en este trabajo se decidió adoptar.
En cuanto a las subredes de salida, el regresor de cuadro delimitador y el clasificador de objeto estaban compuestos por capas totalmente conectadas (FC) y el predictor de máscara era una red totalmente convolucional, compuesta por cuatro capas de convolución consecutivas de 3 x 3, una capa de deconvolución de 2 x 2 y una última capa de convolución de 1 x 1 en secuencia. En el proceso de entrenamiento se inició la representación en el dominio de imágenes naturales (pesos del conjunto de datos COCO [22]), y se entrenó con 100 épocas de 1000 iteraciones cada una. Para entrenar el enfoque propuesto, se realizó un aumento de datos para enriquecer el conjunto de entrenamiento. Este aumento de datos consistió en una rotación de 90º de todas las imágenes histológicas con su respectiva transformación a las máscaras de entrenamiento, teniendo un total de 1282 imágenes para este conjunto. En cuanto al enfoque, para el tercer y último nivel de representación, en el mapa de características de la última capa de la CNN Base (ResNet-50) se aplicó la estrategia SumPooling, y una posterior normalización en l2 para agrupar y fusionar las activaciones, generar nuevas regiones a partir de estas activaciones y entrenar este último modelo. El repositorio del enfoque propuesto está disponible en: https://gitlab.com/bivl2ab/research/2021-andres-gleason.
2.8. Validación estadística
La validación del esquema propuesto siguió el esquema de entrenamiento-evaluación (train-test), de acuerdo con las particiones sugeridas por los autores del conjunto de datos evaluado. En este trabajo se asume que las regiones generadas por el modelo se corresponden con anotaciones de grados histológicos, determinada por expertos patólogos, por lo tanto, la validación fue enfocada principalmente en la evaluación de las segmentaciones generadas con respecto a las anotaciones de referencia realizadas por dos expertos patólogos. Las métricas de evaluación regional de la segmentación se describen a continuación:
Intersección sobre Unión (IoU). La intersección sobre unión (IoU) es una métrica que permite validar el nivel de sobrelapamiento entre la delineación realizada por un patólogo (S) y la segmentación propuesta (Ŝ) [23]. En este caso, el grado de superposición entre las máscaras es definido, en términos de relación de conjuntos, como se muestra en (1).
Donde el numerador define el área de la intersección, y el denominador es el área de la unión entre las dos máscaras de segmentación S y Ŝ. En el caso de la segmentación binaria o multiclase, una imagen puede contener diferentes máscaras de segmentación para cada clase, por lo tanto, se calcula la media de IoU (mIoU) de una imagen tomando la IoU de cada máscara de clase segmentada, presente en la imagen original, y realizando un promedio de todas las IoU calculadas.
Coeficiente de Dice. El coeficiente Dice también mide la similitud de dos muestras mediante la superposición [24]. Esta métrica brinda información sobre el nivel de sobrelapamiento con mayor énfasis en los verdaderos positivos estimados en la segmentación.
Esta relación entre las máscaras está definida como se muestra en (2).
El puntaje Dice, por lo general, tiende a ser mayor que el valor de IoU, debido a que es dos veces el área de superposición sobre el número total de píxeles en ambas segmentaciones.
Media de la precisión promedio (mAP). En esta métrica primero se calcula la precisión P (número de píxeles correctamente segmentados) para cada una de las máscaras de referencia (ground-truth) [25]. En este caso se utiliza un valor de IoU, acotado por un umbral α. En este sentido, si IoU > α, el valor de la precisión será 1, de lo contrario, será 0.
Luego de obtener la precisión para cada máscara, se calcula la precisión promedio (AP, por sus siglas en inglés) entre dos imágenes con sus máscaras de segmentación, como se describe en (3).
En donde P es la precisión entre la máscara delineada por un patólogo S y la máscara predicha (segmentación propuesta) Ŝ, y N mask es el número total de máscaras de ground-truth presentes en una imagen. Por último, mAP es dado como la media de varias AP, es decir, la media de todas las precisiones promedio del conjunto de imágenes, y es representada como se muestra en (4).
Donde AP es la precisión promedio y N img es el número total de imágenes.
Área bajo la curva de la precisión y la sensibilidad (AUPRC). Esta métrica evalúa la similitud entre máscaras de segmentación teniendo en cuenta los valores de precisión y sensibilidad (recall) [26]. En este caso, la precisión toma en cuenta la proporción de píxeles correctamente etiquetados con respecto a los píxeles que fueron erróneamente anotados como una segmentación. Por otra parte, la sensibilidad toma en cuenta la proporción de píxeles correctamente anotados como segmentación con respecto a los píxeles que dejaron de ser anotados como segmentación. Los valores que determinan una segmentación positiva o negativa se pueden variar con respecto al umbral de IoU y por lo tanto obtener un espectro de valores de precisión-sensibilidad. Gráficamente se genera una curva, sensibilidad frente a precisión, representada por cada uno de estos valores. El área bajo esta curva define el valor de la métrica AUPRC, midiendo así el balance de la precisión y la sensibilidad.
3. EVALUACIÓN Y RESULTADOS
3.1. Evaluación cualitativa y observacional
La evaluación de la estrategia aquí desarrollada se llevó a cabo para el conjunto de máscaras predichas y acopladas por los tres niveles de representación, comparado con el conjunto de anotaciones de los patólogos 1 y 2. El entrenamiento de cada nivel de representación tomó aproximadamente un tiempo total de 22 horas, en un equipo de cómputo de alto rendimiento que contaba con una tarjeta gráfica NVIDIA TITAN RTX 24GB. Una vez entrenada la representación, la inferencia en cada muestra tomó aproximadamente 2 segundos. En la Figura 7 y en la Figura 8 se presentan algunos ejemplos de resultados visuales en máscaras multiclase (que poseen dos o más anotaciones de grados de Gleason) y en máscaras con una única anotación. Cada ejemplo muestra las máscaras de segmentación predichas, generadas y acopladas por los tres niveles de representación en secuencia, y las máscaras de anotación de los patólogos 1 y 2.
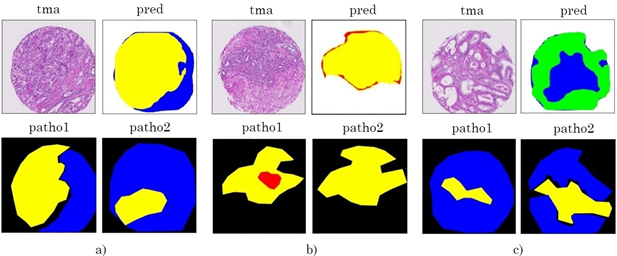
Fuente: elaboración propia.
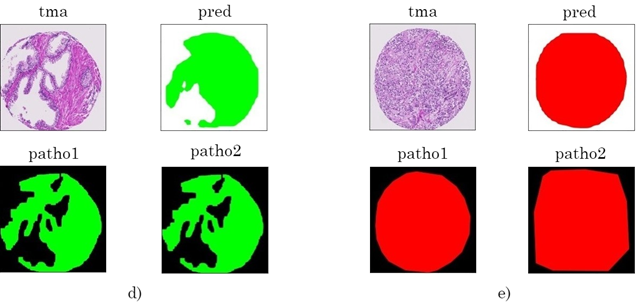
Figura 7 Resultados de máscaras multiclase. Para cada ejemplo se muestra la imagen histológica, la máscara predicha por los tres niveles de representación y la delineación realizada por ambos patólogos. En cada subconjunto tma representa la imagen original, pred la imagen predicha por el método propuesto y patho1, patho2 las anotaciones realizadas por el patólogo 1 y el patólogo 2, respectivamente
La alta variabilidad descrita en los protocolos de anotación se puede ilustrar en la Figura 7-a. Las máscaras de los patólogos 1 y 2 presentan diferencias regionales en su anotación, debido a la persistente subjetividad. Sin embargo, la máscara final predicha presenta una recuperación en las anotaciones de puntajes de Gleason 3 y 4 y una óptima segmentación comparada con la máscara del patólogo 1. En distintos casos se presentan mejoras en la segmentación debido al tercer nivel de representación, como por ejemplo la región en amarillo (Gleason 4). Del mismo modo, en la Figura 7-b, la máscara predicha presenta una recuperación en la anotación de Gleason 4 (amarillo), ya que, como se puede observar, el primer nivel de representación predijo toda la muestra como Gleason 5 (color rojo). Este ejemplo permite evidenciar la complejidad de la tarea de anotación, pero también la utilidad de comprender diferentes niveles de representación. En la figura 7-c se reportan algunas limitaciones del método propuesto debido a la similitud estructural que existe entre grados contiguos (por ejemplo, Benigno y Gleason 3), y a la complejidad de estratificar las imágenes histológicas y diferenciar sus patrones.
Cabe resaltar que en varias muestras la segmentación se realizó de manera precisa en contraste con las máscaras de referencia (ground-truth) que contienen una única anotación de grado de Gleason, principalmente en muestras que presentan anotaciones de grados bajos (benigno y Gleason 3), como se puede observar en la Figura 8. Esto puede deberse a que el modelo logra una buena diferenciación en los patrones glandulares para estos estadios a causa del tamaño y la separación de las glándulas. En los casos d) y e), la segmentación se realizó con una gran aproximación en contraste con las anotaciones de ambos patólogos, y la estratificación fue realizada de forma con éxito para los grados Benigno y Gleason 5, respectivamente.
3.2. Evaluación cuantitativa
Esta evaluación se realizó para el conjunto de máscaras predichas por los tres niveles de representación. Cada conjunto fue evaluado en comparación con el subconjunto de prueba anotado por los patólogos 1 y 2. Igualmente, se calcularon las métricas entre las máscaras anotadas por los dos expertos patólogos, con el fin de obtener medidas de referencia al momento de realizar la evaluación del enfoque propuesto en este trabajo. Las métricas se calcularon para cada clase de cada máscara de referencia (ground-truth). Para cada dato de prueba, el valor de una métrica estuvo dado por el promedio de los resultados por clase.
En la Tabla 1 se resumen los resultados obtenidos para el conjunto total de evaluación, utilizando el enfoque propuesto con diferentes niveles de representación y comparándose con respecto a cada uno de los patólogos de forma independiente. Para la validación se tuvo en cuenta las métricas de AUPRC, mIoU y Dice, que reflejan la capacidad del método propuesto en tarea regional de segmentación, con respecto a la referencia de los patólogos expertos.
Como línea base para determinar el alcance del trabajo, inicialmente se decidió hacer una comparación entre las marcaciones de ambos patólogos. Como se esperaba, las marcaciones tienen una moderada coincidencia regional con niveles limitados de sobrelapamiento entre las marcaciones. Cabe destacar que el método con un mejor puntaje de AUPRC es el método propuesto Mask 2L, que integra dos redes Mask R-CNN, pero que no incluye la validación con las activaciones de la representación profunda, logrando así un 0.8 ± 0.18 con respecto a un primer patólogo y 0.76 ± 0.15 con respecto a un segundo patólogo.
Tabla 1 Resultados de máscaras globales generadas a partir de los tres niveles de representación. Los resultados obtenidos en cada nivel de representación se compararon con las máscaras del subconjunto de evaluación, para los patólogos 1 y 2. Para cada métrica se reportan los valores promedio y desviación estándar
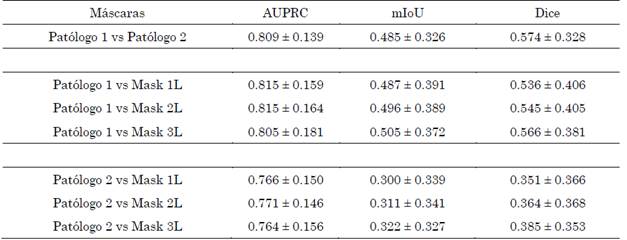
Fuente: elaboración propia.
A pesar de los desafíos inherentes para obtener una máscara ideal que determine el área de un estadio particular de Gleason, resulta relevante en este trabajo determinar la eficacia en localizar apropiadamente estos estadios. Es por ello que en una segunda evaluación se procedió a validar la propuesta con la métrica mAP, pero sometida a diferentes umbrales de la intersección de las regiones. En este caso, la métrica valida si por lo menos existe una intersección del 10 % (mAP@0.1) entre la predicción y la referencia. De este modo, puede estimar y ponderar mejor la localización que la determinación de áreas del grado de Gleason. La Tabla 2 presenta los resultados obtenidos con diferentes umbrales en la métrica mAP, siendo destacable el resultado del enfoque propuesto, utilizando la configuración completa (Mask 3L) en mAP@0.1 y mAP@0.2, respectivamente.
Tabla 2 Resultados de mAP para cada conjunto de máscaras resultantes de los tres diferentes niveles de representación. Cada uno se comparó con las máscaras del subconjunto de prueba, para los dos diferentes patólogos
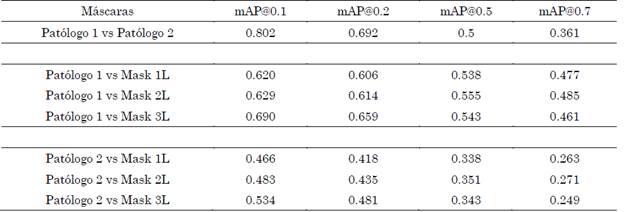
*mAP@0.1: mAP con valor para el umbral de IoU de 0.1, mAP@0.2: mAP con valor para el umbral de IoU de 0.2, mAP@0.5: mAP con umbral de IoU de 0.5, mAP@0.7: mAP con umbral IoU de 0.7.
Fuente: elaboración propia.
Asimismo, se hizo un análisis por grado de forma independiente, calculando las métricas para cada grado de Gleason. En este caso, para cada estadio se tomó su respectiva anotación presente en las máscaras de referencia y se comparó con respecto a la anotación de las máscaras finales de la metodología propuesta Mask 3L (ver Tabla 3). Con esta comparación se busca indagar sobre las capacidades particulares del enfoque propuesto, con respecto a los patrones visuales que agrupan cada grado de Gleason. Se observó que el patrón aprendido de mayor asociación es el grado benigno, que resulta el más frecuente y de patrones texturales relativamente constantes en todo el conjunto de datos. De hecho, para este grado existe un puntaje significativo tanto en AUPRC como en métricas relacionadas con la segmentación regional, alcanzado puntajes de Dice superiores al 80 %.
Tabla 3 Resultados entre el conjunto máscaras de los patólogos 1 y 2 por grado de Gleason, de forma independiente, y el conjunto de máscaras finales generadas por las tres redes implementadas
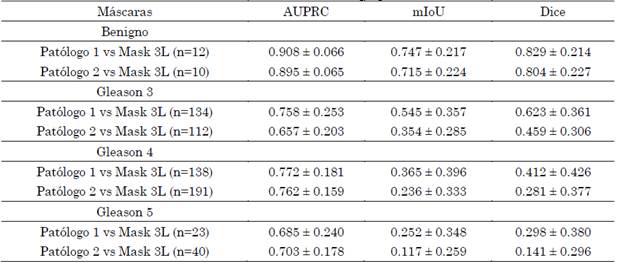
* El número de máscaras utilizado para cada gradode Gleason anotado por cada patólogo está representadopor n
Fuente: elaboración propia.
Se realizó una evaluación discriminada por grados de Gleason y teniendo en cuenta diferentes niveles de umbral de intersección de regiones, en la cuantificación del mAP. En la Tabla 4 se reportan los resultados para los diferentes niveles de Gleason. Como es de esperarse, el nivel benigno, con patrones planos y poca variabilidad tiene una alta correspondencia tanto en la localización (mAP@0.1), como en puntajes asociados con la segmentación (mAP@0.7).
Tabla 4 Resultados de mAP obtenidos para máscaras de patólogos 1 y 2 por grado de Gleason, de forma independiente, y el conjunto de máscaras finales generadas por las tres redes implementadas
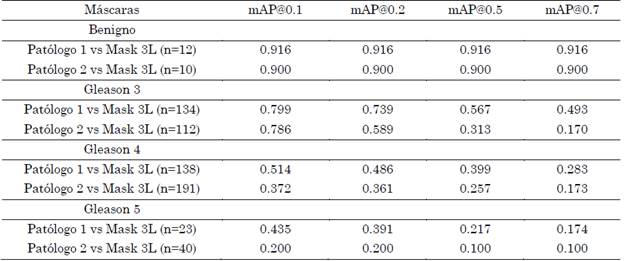
*El número de máscaras utilizado para cada grado de Gleason anotado por cada patólogo está representadopor n.
Fuente: elaboración propia.
También es notable el desempeño logrado en Gleason 3, sobre todo en términos de localización del estadio. Los demás grados presentan notables limitaciones y su caracterización puede requerir muchas más anotaciones, así como también la delineación por parte de múltiples expertos.
4. DISCUSIÓN
En el presente trabajo se introdujo una representación profunda dedicada a la segmentación de patrones visuales con correspondencia a los estadios de Gleason. La representación propuesta se fundamenta en la arquitectura Mask R-CNN que ha demostrado notables resultados en la segmentación semántica de instancias naturales. Sin embargo, debido a la complejidad de la tarea histopatológica, esta representación resulta insuficiente utilizando una única etapa de aprendizaje. Es por ello por lo que en este trabajo se introduce un esquema con diferentes niveles de representación, basado en la totalidad de datos de entrenamiento (primer nivel), utilizando las máscaras más desafiantes para el modelo (segundo nivel), y definiendo un esquema basado en las regiones de mayor atención visual (tercer nivel). Esta estrategia de refinamiento conduce a una mejora en el desempeño, en lo que se refiere al sobrelapamiento entre máscaras de referencia y predichas, evidenciada en los resultados de las métricas mIoU y Dice. Este último nivel, además, pretende reducir un poco el sesgo marcado por las anotaciones del patólogo. De hecho, el método propuesto alcanzó en su configuración Mask 2L un AUPRC del 0.8 ± 0.18. También, en general, se obtiene una asociación más cercana de las máscaras predichas con respecto al patólogo 1, esto debido a que este experto fue el mismo que hizo las anotaciones en el conjunto de datos de entrenamiento. Sin embargo, las métricas de sobrelapamiento logran un mejor desempeño utilizando tres niveles de representación (Mask 3L), lo cual puede tener ventajas en cuanto a la generalización de la propuesta.
En lo referente al análisis y estratificación de los estadios del cáncer, una de las limitaciones inherentes es la moderada concordancia entre expertos patólogos para definir las fronteras de afectación y los estadios asociados en cada nivel. Este hecho ha sido ampliamente fundamentado en los diversos estudios de concordancia y variabilidad en la delimitación de regiones y diagnóstico de placas histopatológicas [5]-[7]. Para soportar esta tarea, previamente se han propuesto diversos enfoques basados en la extracción de características en imágenes histológicas para su posterior clasificación [8]-[10]. Sin embargo, estos enfoques se limitan a un número de características predefinidas y, por consiguiente, no generalizan completamente la enfermedad. Otros enfoques se centran en la extracción de parches histológicos para su posterior clasificación [11]-[12]. No obstante, estos enfoques son limitados a una caracterización local de regiones sin tener muchas veces en cuenta las estructuras celulares que se correlacionan con la enfermedad. En contraste, el enfoque propuesto utiliza regiones completas, con sentido histopatológico que abarca diferentes estructuras celulares para realizar la aproximación en la anotación automática. En este sentido, la herramienta propuesta puede ser importante para proponer regiones con un estadio de Gleason particular, el cual puede luego ser modificado más fácilmente por un experto. De acuerdo con los resultados reportados, este hecho es notable para hacer marcaciones rápidas de tejido benigno, durante la exploración histopatológica, para que luego el experto defina regiones más específicas de otros grados afectados. En cuanto a los estadios de Gleason 3, 4 y 5, se observa un apropiado comportamiento en términos de AUPRC, pero con notables limitaciones para el grado cinco, hecho asociado al número de muestras disponibles para el entrenamiento y la variabilidad en este nivel de la enfermedad.
También, en la literatura se han propuesto algunas aproximaciones que buscan segmentar glándulas y estructuras celulares específicas [15]-[16]. Este trabajo resulta interesante para apoyar la tarea diagnóstica, pero los datos específicos de entrenamiento hacen tediosa la labor, con conjuntos de datos limitados, siendo una actividad compleja el incremento dinámico del conjunto de datos. Estas herramientas también, al limitarse en las estructuras celulares conocidas, impiden explorar nuevas relaciones o patrones arquitecturales de la célula que puedan ser autoaprendidos por los algoritmos, además de definir un estadio de Gleason particular. El trabajo propuesto podría complementar esta herramienta al brindar información con respecto a la localización específica de estadios de Gleason (ver Tabla 3).
Como se esperaba, en la validación de la localización con respecto al patólogo 2, se resalta un notable puntaje en mAP@0.1 y mAP@0.2, pero con una notable pérdida cuando se consideran intersecciones superiores.
El presente trabajo tiene un amplio potencial para ser implementado como herramienta de soporte en etapas preliminares para proponer regiones y grados de Gleason que posteriormente sean ajustados y validados por patólogos. También tiene notables ventajas como herramienta para el soporte clínico, pero acotado a una intervención posterior por parte del patólogo para tomar decisiones en las delineaciones finales de los grados de Gleason. Por ejemplo, en estadios tempranos y para tejido benigno, el enfoque propuesto puede mostrar considerables ventajas para hacer la tarea automática, agilizando el proceso de análisis y permitiendo a los expertos enfocarse en regiones de las placas más críticas. Por otra parte, la visualización de características durante la delineación, en el tercer nivel de la representación propuesta, puede apoyar la tarea del patólogo y reducir el sesgo en las anotaciones. Sin embargo, el presente enfoque tiene una amplia dependencia de las anotaciones aprendidas por el patólogo 1, lo cual lo hace susceptible a delimitaciones fijas sobre patrones comunes marcados por el experto, los cuales pueden distar ampliamente del patólogo 2, como se reportaron en la sección de resultados.
5. CONCLUSIONES
Este trabajo presentó una estrategia de segmentación semántica de los estadios de Gleason, usando representaciones de aprendizaje profundo, organizados en un esquema de múltiples niveles de representación. Como base de representación se implementó la arquitectura Mask R-CNN utilizando tres niveles de aprendizaje que logran capturar los principales patrones visuales que se asocian a los estadios, en imágenes histopatológicas. Los resultados obtenidos muestran comportamientos favorables, con niveles de intersección regional aproximados a los patólogos de referencia y constituyéndose en una herramienta de potencial implementación para el soporte a la delineación y análisis clínico. De hecho, se destacan las segmentaciones de tejido benigno y de grado 3, los cuales pueden ser depurados por la herramienta para un análisis más eficiente y dedicándose en niveles más críticos por parte de los patólogos. Trabajos futuros incluyen la validación con el mismo conjunto de datos, pero anotado por un mayor número de patólogos, permitiendo introducir una mayor flexibilidad en la representación aprendida. A partir de ello, se espera formular estrategias que ajusten las diferentes anotaciones con respecto a la experticia de cada patólogo involucrado en el estudio. También se planea explorar nuevas arquitecturas convolucionales dedicadas a la segmentación semántica que incluyan solapamientos y activaciones que puedan aportar a las representaciones visuales con correspondencia a los grados de Gleason.