Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Infectio
Print version ISSN 0123-9392
Infect. vol.10 no.3 Bogotá July/Sept. 2006
ARTÍCULO ORIGINAL
Caracterización molecular de cepas toxigénicas de Staphylococci aisladas de operarios de plantas de alimentos
Molecular characterization of toxigenic Staphylococci strains isolated from food handlers
MARÍA CONSUELO VANEGAS,
AIDA JULIANA MARTÍNEZ,
MAYRA VIVIANA MEDRANO
Laboratorio de Ecología Microbiana y de Alimentos, Universidad de Los Andes, Bogotá, D. C., Colombia.
Fecha de recepción: 19/05/2006; fecha de aceptación; 10/07/2006
Resumen
Objetivo. Utilizar la técnica de amplificación aleatoria de ADN polimórfico ( random amplification of polymorphic DNA, RAPD para caracterizar molecularmente cepas de Staphylococci productoras de toxinas, aisladas de operarios de plantas de producción de alimentos. Materiales y métodos. Se utilizaron 31 aislamientos enterotoxigénicos de Staphylococci para la extracción y cuantificación de ADN. Posteriormente, se realizó un ciclo general de amplificación utilizando los oligonucleótidos HLWL-74 y arbitrario, seguido por la visualización de los productos de RAPD por electroforesis en gel de agarosa. Resultados. Para los dos oligonucleótidos utilizados, se observaron de 1 a 15 bandas, dos linajes, divididos en tres conglomerados (A, B y C). El oligonucleótido arbitrario generó 10 bandas polimórficas (66,66%) y el oligonucleótido HLWL-74 arrojó 13 bandas altamente polimórficas (86,66%). Cada oligonucleótido mostró un agrupamiento diferente de cada una de las cepas, lo cual muestra una alta diversidad de aislamientos de Staphylococci presentes en humanos. Conclusiones. Se presentó una alta diversidad molecular en cuanto a aislamientos de garganta, nariz y manos de un mismo individuo, así como en todos los aislamientos analizados, lo cual demuestra que las cepas enterotoxigénicas de Staphylococci encontradas en los operarios analizados tienen una alta diversidad molecular.
Palabras clave: Staphylococci, RAPD, operarios, polimorfismos, toxinas.
Abstract
Objective. To use Randomly Amplified Polymorphic DNA (RAPD) to compare molecularly enterotoxigenic Staphylococci strains isolated from people working in food processing plants. Materials and methods. 31 Staphylococci enterotoxigenic isolates were used for extraction and quantification of DNA, followed by a general amplification cycle with the HLWL-74 and arbitrary oligonucleotides with visualization of the RAPD products by agarose gel electrophoresis. Results. The two oligonucleotides used generated 1 to 15 bands, two mayor lineages divided in three clusters (A, B, and C). The arbitrary oligonucleotide generated 10 polymorphic bands (66.66%), the HLWL-74 oligonucleotide generated 13 polymorphic bands (86.66%). Each oligonucleotide generated a different type of grouping with respect to each of the strains analyzed. This shows a high diversity between the human isolates of Staphylococci. Conclusion. A high molecular diversity was present amongst throat, nose and hands isolates from the same person, and among the analyzed isolates; this demonstrates a high molecular diversity in Staphylococci enterotoxigenic isolates. Key words: Staphylococci, RAPD, food handlers, polymorphisms, toxins.
Fecha de recepción: 19/05/2006; fecha de aceptación; 10/07/2006
INTRODUCCIÓN
Diferentes especies de Staphylococci, tales como S. aureus, S. intermedius, S. epidermidis, S. hyicus y S. warneri, pueden producir enterotoxinas (1). La intoxicación por S. aureus se ha considerado, a nivel mundial y en Colombia, como la segunda causa de enfermedades transmitidas por alimentos (ETA), después de Campylobacter o Salmonella (2-5). Esta intoxicación alimentaria se desarrolla pocas horas después del consumo de alimentos o bebidas contaminadas con una o más de las toxinas de Staphylococci preformadas. La intoxicación causa gastroenteritis con síntomas como náuseas, vómito, dolor abdominal, con diarrea o sin ella, de recuperación relativamente rápida que varía entre 1 a 3 días, y de baja mortalidad (6).
Por lo anterior, en la mayoría de los países las personas no reportan la intoxicación, lo cual dificulta la obtención de datos epidemiológicos confiables de la incidencia de la misma (7). Además, en países como Estados Unidos se ha estimado una incidencia anual de 185.000 casos con 1.750 hospitalizaciones (8). En el sector económico y en el industrial es una de las más importantes a nivel mundial, con costos de $1,5 billones (2, 6, 8). Las toxinas preformadas de Staphylococci son proteínas sencillas, codificadas en islas de patogenicidad, las cuales han sido designadas, de acuerdo con su antigenicidad, desde toxinas de Staphylococci preformadas tipo A a O. La mayoría se han considerado como superantígenos y pueden ser producidas por S. aureus o por otras especies en alimentos ricos en proteínas y grasas como jamones, quesos, sándwiches, etc. (6, 8).
Los alimentos pueden contaminarse por manejo inadecuado o malas condiciones de producción y almacenamiento, o ambas. La causa más frecuente de contaminación es por los operarios que preparan y sirven los alimentos (manipuladores de alimentos) (3), debido a que S. aureus forma parte de la flora usual de la nasofaringe y piel sin causar necesariamente enfermedad en humanos (9).
Diferentes estudios epidemiológicos de S. aureus aislado de alimentos, humanos y de animales se han basado en técnicas fenotípicas como biotipificación, tipificación por bacteriófagos, pruebas de sensibilidad o resistencia a antibióticos. Igualmente, otros autores han realizado estudios moleculares de diferentes cepas de Staphylococci utilizando técnicas como electroforesis en campo pulsado ( pulsed field gel electrophoresis, PFGE), relacionando los aislamientos ya sea de humanos o animales con el origen del brote y estableciendo agrupaciones genotípicas, con el fin de caracterizar las bacterias. Los RAPD han sido utilizado extensamente para diferenciar aislamientos de Staphylococci (10) a nivel mundial.
Sin embargo, en Colombia no existen datos sobre la variabilidad genotípica de las cepas enterotoxigénicas de Staphylococci (11), y no hay información de este método en nuestro medio con respecto a los aislamientos de Staphylococci provenientes de humanos.
El objetivo del presente trabajo es la utilización de dos oligonucleótidos (HLWL-74 y arbitrario) mediante la amplificación aleatoria de AND polimórfico (RAPD) para determinar diferencias genotípicas entre las cepas de Staphylococci productoras de toxinas, aisladas de personas que trabajan en manipulación de alimentos, lo anterior con el fin de tener un tamizaje inicial en el conocimiento molecular de las cepas que están circulando en nuestro medio para que, en un futuro, sean caracterizadas molecularmente con técnicas más precisas. Los resultados de este trabajo son de gran importancia porque es el primer estudio en nuestro medio en el que se caracterizan molecularmente cepas toxigénicas de Staphylococci, lo cual permite determinar la diversidad de las mismas y, a su vez, los resultados pueden ser utilizados para relacionar cepas causantes de brotes de intoxicación alimentaria y cepas de humanos.
MATERIALES Y MÉTODOS
Aislamientos bacterianos
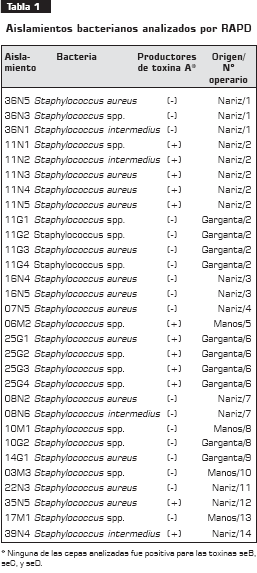
Se estudiaron 31 aislamientos de Staphylococci, de operarios de alimentos de diferentes plantas de producción de alimentos; 16 aisladas de nariz, 4 de manos y 11 de garganta. Los 31 aislamientos se caracterizaron toxigénicamente en un estudio anterior para la producción de toxinas seA, seB, seC y seD (tabla 1). Las bacterias se conservaron a -70°C hasta su análisis.
Extracción, cuantificación y preservación de ADN
Se realizaron cultivos en BHI (Oxoid) a 37°C por 18 horas y la extracción de ADN bacteriano se realizó a partir de 1 ml del cultivo. Se utilizó el estuche para aislamiento de ADN procariótico ( ProDNA 2003, Corporación Corpogen) y se siguieron las instrucciones del fabricante. El ADN extraído se reconstituyó en 50 µl de solución de resuspensión del estuche. Posteriormente, se cuantificó el ADN en el espectro-fotómetro ( Biomate 3, Thermospectronic, Rochester NY, USA) y se tomaron lecturas a 260, 280 y 320 nm para estimar la pureza y cantidad del ADN. Una vez establecida la cantidad de ADN y ajustado a concentración de 20 ng/ul, se conservó a -20°C hasta su análisis.
Condiciones de RAPD
La técnica fue aplicada para los 31 aislamientos. Se utilizaron dos oligonucleótidos sintetizados por Promega Corporation de 10 pares de bases HLWL-74 (5'-ACG TAT CTG C-3') (12, 13, 14) y arbitrario de 10 pb (5'-GAG CTC GTG T-3') reportado por la Unidad de Servicio de Ácidos Nucleicos y Proteínas de la University of British Columbia como estándar para reacciones de RAPD (3). El ciclo general con el cual se trabajó fue estandarizado en el laboratorio, con base en protocolos previamente reportados en la literatura (15-18). Se llevó a cabo una denaturación inicial, 94°C: 5 minutos, seguida por 30 ciclos de 94°C por 30 segundos, 37°C por 45 segundos, 72°C por 1 minuto y un ciclo final de 72°C por 5 minutos. Los ciclos de amplificación se llevaron a cabo en un termociclador Gene CyclerTM (Bio-Rad Laboratories, Inc., United States of America).
Se trabajó un volumen final de 25 µl que contenía 0,1 U/µl Taq polimerasa ( TucanTaq; Corporación Corpogen); 3 mM MgCl2, 0,2 mM de cada dNTP ( Promega Corporation), 1,5 µM de iniciador y 105 ng de ADN. Cada reacción se realizó dos veces para cada aislamiento y cada oligonucleótido.
La visualización de los productos de amplificación se hizo en geles de agarosa ( Bio-Rad Labora-tories, Inc., United States of America) al 0,7% preparados en tampón TBE 1X, se corrieron en cámaras Wide Mini y Wide Sub Cell GT ( Bio-Rad Laboratories, Inc. United States of America). Los geles se tiñeron con bromuro de etidio, 10 mg/ml por 45 minutos. Todas las reacciones se prepararon con un control negativo (agua libre de nucleasa) y con marcador de peso molecular de ADN de fago digerido con EcoRI+ HindIII ( Promega Corporation). Los productos se visualizaron en Chemi-Doc XRS system ( Bio-Rad Laboratories).
Los datos se analizaron con el programa Quantity One Chemi-Doc XRS system ( Bio-Rad Laboratories).
Para cada oligonucleótido se construyó una matriz binaria (1 = banda presente, 0 = banda ausente) teniendo en cuenta solamente las bandas más fuertes y de mayor resolución. Finalmente, se construyó un dendrograma empleando el programa SYN-TAX 2000, con la herramienta de agrupación jerárquica algoritmo UPGMA y el coeficiente de correlación de Jaccard para cada oligonucleótido.
RESULTADOS
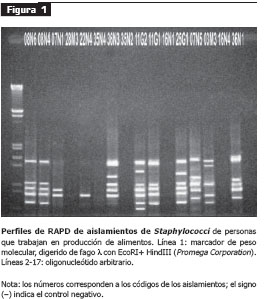
Los dos oligonucleótidos generaron un número de bandas considerables. Para la determinación de la variabilidad genética sólo se tuvieron en cuenta aquellas bandas que tenían mayor intensidad. El perfil de bandeo mostró un rango de 1 a 15 bandas (figura 1 y figura 2). El oligonucleótido arbitrario generó 10 bandas polimórficas (66,66%) y el oligonucleótido HLWL-74 generó 13 bandas, lo cual determinó mayor polimorfismo (86,66%) entre las cepas. No se detectaron productos de amplificación en los controles negativos.
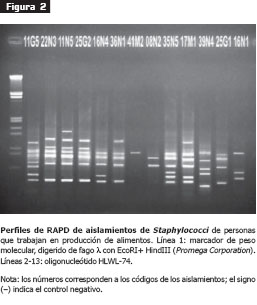
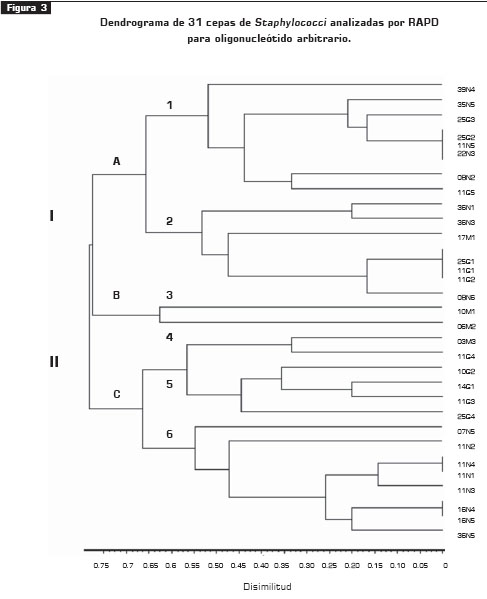
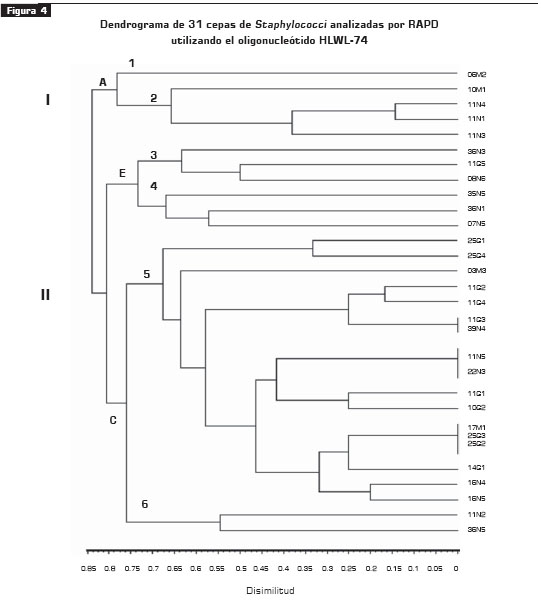
Los dendrogramas, construidos para cada oligonucleótido por medio del programa SYN-TAX, mostraron dos linajes (I, II), agrupados en tres grupos mayores (A-C) o seis subtipos (tipo1 a tipo 6) (figuras 3 y figura 4).
Los operarios portadores de cepas toxigénicas seA se agruparon en los dos linajes para los dos oligonucleótidos (HLWL-74 y arbitrario) (figuras 3 y figura 4). Con el oligonucleótido HLWL-74, tres de cinco aislamientos (11N4, 11N1, 11N3) seA del operario 2 (tabla 2) se agruparon en el linaje I, grupo A y en el linaje II conglomerado C para el oligonucleótido arbitrario, al comparar los aislamientos de garganta del mismo operario 2 (11G1, 11G2, 11G3, 11G4 y 11G5) (tabla 1) , se observó que se agruparon en los linaje I y II grupo C para los oligonucleótidos arbitrario y HLWL-74. Con respecto a los demás aislamientos de nariz, se observó que, al utilizar los dos oligonucleótidos, estas cepas estuvieron agrupadas en los dos linajes.
Se observó que solamente los aislamientos del operario 3 (16N4, 16N5) (tabla 1), el cual no era portador de cepas toxigénicas, se agruparon en el conglomerado C al utilizar los dos oligonucleótidos, hecho que no se observó con los otros aislamientos.
En cuanto a los aislamientos de garganta del operario 6 (25G1, 25G2, 25G3 y 25G4) (tabla 1), portador de enterotoxina seA, se observó que estuvieron agrupados bajo el linaje II, conglomerado C, al utilizar el oligonucleótido HLWL-74, y bajo el linaje I conglomerado A, con el oligonucleótido arbitrario. Los aislamientos de garganta (10G2 y 14G1) correspondientes a los individuos 8 y 9 (tabla 1) se agruparon en el linaje II, conglomerado C al utilizar los dos oligonucleótidos; los aislamientos restantes se agruparon en los dos linajes. Los aislamientos de manos (10M1 y 06M2) (tabla 1) estuvieron agrupados bajo el linaje I, conglomerado B, con un porcentaje de disimilitud de 63% con respecto a los demás aislamientos con el oligonucleótido arbitrario y, en el linaje I, conglomerado A, con un porcentaje de disimilitud de 77% con el oligonucleótido HLWL-74.
Los principales reservorios de Staphylococi son los canales de la nasofaringe y la piel. Constantemente, las empresas procesadoras de alimentos experimentan problemas asociados con el elevado número de contaminación a causa de este microorganismo, el cual puede generar gastroenteritis en los consumidores de estos alimentos originados por la producción de enterotoxinas termoestables (6, 19). Para fines de control específico de este microorganismo, es importante el conocimiento del origen de la contaminación. Las técnicas moleculares como RAPD se han utilizado en otros estudios con el fin de demostrar la fuente de contaminación y las rutas por las cuales el microorganismo es transmitido al alimento (20). De igual forma, la genotipificación de Staphylococci se ha convertido, según otros autores, en parte de sistemas de inspección, para el estudio de brotes y la relación entre cepas, ya que al igual que en Colombia la intoxicación por este patógeno no se reporta y no se conoce la relación que existe entre los aislamientos de humanos, principales reservorios de Staphylococci (19, 20). Por esta razón, estudiamos 31 aislamientos de Staphylococci obtenidos de operarios manipuladores de alimentos con el fin de determinar las relaciones entre estos mediante la técnica de RAPD. Esta técnica, junto con la electroforesis en campo pulsado (PFGE), y el polimorfismo de longitud de fragmentos amplificados ( amplified fragment length polymorphism AFLP), entre otras, se ha utilizado en otros estudios de genotipificación de Staphylococci en los cuales se ha reportado que esta técnica, aunque con menor poder discriminatorio que la técnica de PFGE, ha mostrado una buena sensibilidad y reproducibilidad, además de ser menos costosa (6, 10).
Los perfiles moleculares obtenidos utilizando los dos oligonucleótidos generaron bandas altamente reproducibles. Cada oligonucleótido mostró un agrupamiento para cada una de las cepas, lo cual demuestra una alta diversidad genética entre los aislamientos de Staphylococci que forman parte de la flora usual en humanos. Los resultados indican que hubo un mayor porcentaje de polimorfismos (86,66%) con el oligonucleótido HLWL-74 que con el oligonucleótido arbitrario (66,66%). Por lo tanto, se sugiere para futuros estudios con RAPD utilizar el oligonucleótido HLWL-74. Los resultados de la caracterización molecular de los aislamientos indican que un individuo puede ser portador de varios tipos de aislamientos diferentes; por ejemplo, las cepas de nariz de un mismo individuo productoras de toxina estuvieron agrupadas bajo el mismo conglomerado y presentaron patrones particulares de bandeo, al igual que los aislamientos de garganta no productores de toxina de este mismo individuo fueron agrupadas en un solo conglomerado. Así mismo, los aislamientos de garganta productores de toxina seA provenientes del individuo VI se agruparon bajo un mismo conglomerado, mientras que los demás aislamientos no se agruparon en un conglomerado exclusivo. Las cepas de manos se agruparon bajo el linaje I para ambos oligonucleótidos con un porcentaje mayor a 60% de disimilitud.
Estos resultados sugieren que puede haber flujo genético entre cepas de un mismo individuo, lo cual es muy importante, ya que estas cepas pueden generar diferentes tipos de brotes los cuales no están reportados epidemiológicamente en nuestro país.
En conclusión, este estudio es un primer acercamiento a la tipificación molecular de cepas de Staphylococci en humanos. La presencia de cepas aisladas de garganta, nariz y manos con diferentes tipos moleculares en un mismo individuo revela diversa información genética que, eventualmente, puede dar origen a nuevas variantes de estas cepas debido al flujo genético. Estas diferencias moleculares pueden reflejar características fisiológicas y ecológicas que pueden conferir ventajas adaptativas, las cuales finalmente podrían traducirse en cepas productoras de enterotoxinas más resistentes a las condiciones ambientales.
La presencia de enterotoxina seA en algunos aislamientos revela que las personas saludables son un reservorio de cepas enterotoxigénicas, lo cual puede ser un riesgo de salud pública, ya que estas cepas pueden pasar a los alimentos contaminándolos y generando brotes que, en la actualidad, son desconocidos por la ausencia de reportes epidemiológicos.
Para futuros estudios, se recomienda determinar la relación entre cepas provenientes de humanos y de alimentos implicados en brotes con el fin de establecer relaciones genéticas más específicas, lo cual será muy útil para estudiar la epidemiología de intoxicaciones estafilocócicas en Colombia. Además, se recomienda continuar con la implementación de programas de salud ocupacional en las industrias con el propósito de tener especial interés en las personas que manipulan alimentos y, de esta forma, asegurar la inocuidad de los alimentos.
Conflicto de intereses
Declaramos que la investigación a partir de la cual se originó este artículo no tiene ningún tipo de conflicto de interés.
Correspondencia: María Consuelo Vanegas, Laboratorio de Ecología Microbiana y de Alimentos, LEMA, Carrera 1 Nº 18A-10, J209, Bogotá, D. C. Colombia. Teléfono: 339-4949, extensiones 2784/2792.
REFERENCIAS
1. Escartin FE. Microbiología e inocuidad de los alimentos. Universidad Autónoma de Querétaro; 2000. p. 322. [ Links ]
2. Fueyo JM, Martín MC, González-Hevia MA, Mendoza MC. Enterotoxin production and DNA fingerprinting in Staphylococcus aureus isolated from human and food samples. Relations between genetic types and enterotoxins. Int J Food Microbiol 2001;67:139-45. [ Links ]
3. Berrada H, Soriano JM, Mañes J, Pico Y. Real-time quantitative PCR of Staphylococcus aureus and application in restaurant meals. J Food Prot 2006;69:106-11. [ Links ]
4. Inghan SC, Engel RA, Fanslau MA, Shoeller EL, Searls G, Buege DR, Zhu J. Fate of Staphylococcus aureus on vacuum-packaged ready-toeat meat products stored at 21°. J Food Prot 2005;68:1911-5. [ Links ]
5. Bennett RW. Staphylococcal enterotoxin and its rapid identification in foods by enzyme-linked immunosorbent assay-based methodology. J Food Prot. 2005:1264-70. [ Links ]
6. Martín MC, Fueyo JM, González-Hevia MA, Mendoza MC. Genetic procedures for identification of entertotoxigenic strains of Staphylococcus aureus from three poisoning outbreaks. Int J Food Microbiol 2004;94:279-86. [ Links ]
7. Smyth DS, Keneddy J, Twohig J, Miajlovic H, Bolton D, Smyth CJ. Staphylococcus aureus isolates from Irish domestic refrigerators possess novel enterotoxin and enterotoxin-like genes and are clonal in nature. J Food Prot 2006;3:508-15. [ Links ]
8. Pinto B, Chenoll E, Aznar R. Identification and typing of food-borne Staphylococcus aureus by PCR–based tecniques. Syst Appl Microbiol 2005;28:340-52. [ Links ]
9. Fueyo J, Fueyo M, Mendoza C, Álvarez MA, Martín MC. Relationships between toxin gene content and genetic background in nasal carried isolates of Staphylococcus aureus from Asturias, Spain. FEMS Microbiology Letters 2005;243:447-54. [ Links ]
10. Reinoso E, Bettera S, Frigerio C, DiRenzo M, Calzolari A, Bogni C. RAPD-PCR analysis of Staphylococcus aureus strains isolated from bovine and human hosts. Microbiolo Res 2004;159:245-55. [ Links ]
11. Perdomo AM. Estimación de la incidencia de las enfermedades transmitidas por alimentos (ETA) en el periodo 1992-2003 (trabajo de grado). Bogotá: Pontificia Universidad Javeriana; 2004. [ Links ]
12. Lawrence L, Gilmour A. Characterization of Listeria monocytogenes isolated from poultry products and from the poultry-processing environment by random amplification of polymorphic ADN and multilocus enzyme electrophoresis. Int J Food Microbiol 1991;12: 14-70. [ Links ]
13. Inoue S, Katagiri K, Terae M, Maruyama T. RAPD and actA genertyping of Listeria monocytogenes isolates from human listeriosis, the intestinal content of cows and beef. Microbiol Immunol 2001;45:127-33. [ Links ]
14. Nucleic Acid Protein Service Unit, University of British Columbia (Canada). Consultado en abril de 2006, http://www.michaelsmith.ubc.ca/services/NAPS/Primer_Sets. [ Links ]
15. Inoue S, Katagiri K, Terao M, Maruyama T. RAPD and actA genetyping of Listeria monocytogenes isolates from human listeriosis, the intestinal content of cows and beef. Microbiol Immunol 2001; 45: 127-33. [ Links ]
16. Yoshida T, Takeuchi M, Sato M, Hirai K. Typing Listeria monocytogenes by Random Amplified Polymorphic DNA (RAPD) fingerprinting. J Vet Med Sci 1999;61:857-60. [ Links ]
17. McPherson MJ, Moller SG. PCR: The basics. United Kingdom: BIOS Scientific Publishers Limited; 2000. p. 257-528. [ Links ]
18. Lawrence LM, Harvey J, Gilmour A. Development of a random polymorphic ADN typing method for Listeria monocytogenes. Appl Environ Microbiol 1993;59:3117-9. [ Links ]
19. Boerma JA, Clemens R, Brightwell G. Evaluation of molecular methods to determine enterotoxigenic status and molecular genotype of bovine, ovine, human and food isolates of Staphylococcus aureus. Int J Food Microbiol 2006;107:192-201. [ Links ]
20. El-Huneidi W, Bdour S, Mahasneh A. Detection of enterotoxin genes seg, seh, sei, and sej and of a novel aroA genotype in Jordanian clinical isolates of Staphylococcus aureus. Diag Microbiol Infect Dis 2006. [ Links ]