Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Infectio
Print version ISSN 0123-9392
Infect. vol.14 no.1 Bogotá Jan./Mar. 2010
1 Laboratorio de Biología Molecular y Patogénesis, Departamento de Ciencias Fisiológicas, Escuela de Ciencias Básicas, Facultad de Salud, Universidad del Valle, Cali, Colombia
Recibido: 05/02/2009; Aceptado: 25/01/2010
Resumen
Introducción: La mayor parte del genoma celular es accesible a la integración retroviral; sin embargo, se propone que este proceso no es aleatorio y es dependiente de cada retrovirus.
Objetivos: Identificar y caracterizar las regiones del genoma humano en donde ocurre la integración del virus de la inmunodeficiencia humana de tipo 1 (VIH-1) en células mononucleares de sangre periférica, macrófagos y células T de Jurkat infectadas.
Materiales y métodos: Se seleccionaron 300 secuencias de ADN humano obtenidas por el método de ligación mediada por PCR, previamente depositadas en el GenBank. Utilizando el programa BLAST, sólo 264 de ellas se incluyeron en el estudio, pues se pudo obtener información sobre localización cromosómica, genes anotados, secuencias repetidas, número de islas CpG y tiempo medio de replicación, entre otras variables genómicas. Estas secuencias se exportaron a otras bases de datos.
Resultados: El 53% (140/264) de las integraciones se registraron en bandas G. El 70,45% de los provirus se localizaron en los genes humanos anotados, mientras que el restante lo hizo en elementos repetidos. En general, la selección del sitio de integración se relacionó con las características locales genómicas y estructurales de la cromatina, entre las que se incluyen secuencias Alu-Sx y L1, densidad génica y de islas CpG, remodelación de la cromatina y tiempo de replicación. Éstas influenciarían la interacción eficiente del complejo de preintegración con los genomas celulares.
Conclusión: Se determinó que la integración del VIH-1 en los genomas celulares estudiados estaría condicionada por características diferenciales de la cromatina y por procesos epigenéticos que influirían la selección del sitio blanco de integración.
Palabras clave: virus de la inmunodeficiencia humana 1, integración retroviral, bioinformática, genoma humano.
Abstract
Introduction: Most of the infected host cell genome is available for retroviral integration; however, it has been proposed that this process does not occur at random and depends upon each type of retrovirus.
Objective: The objective is to identify and characterize differences in human genome regions of peripheral blood mononuclear cells, macrophages and Jurkat T cells in which integration of HIV-1 occurs.
Material and Methods: Three hundred human DNA genome sequences, previously deposited in the GenBank, were selected at random. Using program BLAST, only 264 of them were included in the study because relevant information about chromosomal position, associated genes, repetitive sequences, number of CpG islands and average replication time was available; these sequences were exported to other data bases for analysis.
Results: 53% (140/264) of integrations were located on G bands. 70.45% of provirus was located in human genes and the rest was located in repetitive elements. In general the integration site selection was correlated with genomics and structural characteristics of cell chromatin including Alu-Sx and L1 sequences, gene and CpG island densities, remodeling of chromatin, and replication time. All of them would influence the efficient interaction between the pre-integration complex and target cell genomes.
Conclusion: It was determined that HIV-1 integration in target cellular genomes would be conditioned by differential characteristics of associated chromatin and by epigenetic processes that would influence the selection of integration sites.
Key words: Human Immunodeficiency Virus 1, retroviral integration, bioinformatics, human genome
Introducción
El virus de la inmunodeficiencia humana de tipo 1 (VIH-1), identificado inicialmente por Montagnier y Barre-Sinoussi en 1983 (1), es un patógeno perteneciente a la familia Retroviridae, subfamilia Ortoretrovirinae, del género Lentivirus, que incluye también al VIH-2, el virus de la inmunodeficiencia de simios (VIS), el de la inmunodeficiencia bovina (VIB) y el de la inmunodeficiencia felina (VIF) (2). Se ha reportado un amplio rango de células que pueden infectarse in vitro, entre las cuales se incluyen las células CD4+ y CD8+, los linfocitos B, los megacariocitos, los astrocitos, los oligodendrocitos y las células dendríticas, entre otras (2,3).
La integración del ADN retroviral en el genoma del huésped es un paso esencial para su replicación. El ARN viral, antes de la integración, se asocia con la transcriptasa inversa, la integrasa y la proteína de la matriz (4-6), y recluta, en el citoplasma, una serie de proteínas celulares (7,8) para formar el complejo de preintegración. En el transporte de este complejo del citoplasma al núcleo, la proteína viral Vif (viral infectivity factor) (factor de contagio del virión) parece actuar como puente de unión entre el complejo de preintegración y los microtúbulos, promoviendo su transporte activo hacia el núcleo (9).
A diferencia de los oncorretrovirus, el VIH-1 puede infectar células quiescentes pues, mediante un proceso de translocación, el complejo de preintegración atraviesa la envoltura nuclear. En este paso intervienen tanto la Vpr (viral protein r) (10), la proteína de la matriz y la integrasa (11-13), como un complejo proteico heterodimérico compuesto por importina-a (Imp-a) e importina-b (Imp-b). Mientras que la Imp-a se une a secuencias específicas NLS (nuclear localization signals,) (señales de localización nuclear), la Imp-b participa en la translocación a través del complejo del poro nuclear (14). Tanto la proteína de la matriz como la integrasa contienen secuencias NLS e interaccionan con miembros de las importinas (15).
Se ha propuesto que la integración retroviral no es al azar y que, por el contrario, existen regiones del genoma en donde habría una de alta probabilidad de integración del ADN viral (16-19). Sin embargo, a pesar del conocimiento actual, el mecanismo por el cual se seleccionan estas regiones de mayor probabilidad todavía no se conoce completamente. Esto es porque la integración es un proceso multifactorial, el cual varía entre las especies de retrovirus y está influenciado principalmente por los procesos epigenéticos intrínsecos de la cromatina del huésped, y por las proteínas celulares y virales que se unen al sitio blanco (20-23).
Puesto que la integración retroviral depende del estado de conformación de la cromatina, un factor condicionante de la integración es la remodelación de la arquitectura de la cromatina interfásica. En las últimas dos décadas, se han obtenido pruebas sobre la territorialidad de la organización de la cromatina en el núcleo celular interfásico; existen porciones de cromatina que, en la interfase, conforman unas estructuras definidas denominadas territorios cromosómicos (24). Los resultados más recientes parecen confirmar la existencia de una organización no aleatoria de estos territorios cromosómicos en la interfase. Esta organización se correlaciona con el tamaño cromosómico y la densidad de los genes que contiene. Así, pues, hay una localización preferencial de los cromosomas pequeños hacia el interior del núcleo, mientras que los cromosomas grandes se observan más frecuentemente hacia la periferia (25).
Con el fin de determinar las características de la integración del VIH-1 y su dinámica en tres tipos de células humanas susceptibles de infección se analizó una serie de variables genómicas que definen el ambiente genómico de los sitios de integración del provirus VIH-1 en el genoma de estas células. El análisis partió de secuencias del genoma humano adyacentes a sitios de integración retroviral consignadas en bases de datos públicas, utilizando para ello herramientas bioinformáticas. Mediante este enfoque, se encontró que el VIH-1 tiene un patrón amplio de distribución en el genoma humano, el cual está condicionado por el tipo de célula. Sin embargo, se observó que la selección del sitio de integración está relacionada con las características genómicas y estructurales locales de la cromatina, específicas para cada tipo de célula. Estas características incluyen: secuencias Alu, densidad génica, densidad de islas CpG, estado topológico de la cromatina y tiempo de replicación, las cuales determinan que el complejo de preintegración pueda interaccionar eficientemente con el genoma celular.
Materiales y métodos
Selección de las secuencias del genoma humano
En este estudio de tipo descriptivo, se escogieron aleatoriamente, utilizando el método del testigo oculto, un grupo de 300 secuencias del genoma humano, en una extensión de 100 a 200 pb, que contenían extremos LTR (long terminal repeat) de los flancos de provirus VIH-1, provenientes de macrófagos, células mononucleares de sangre periférica y células T de Jurkatt infectadas. Estas secuencias habían sido previamente depositadas en el GenBank(http://www.ncbi.nlm.nih.gov) (26-28).
Alineamiento y análisis de las secuencias del genoma humano
Las secuencias adyacentes a los sitios de integración, se alinearon con aquéllas del genoma humano, utilizando el programa BLAST (http://www.ncbi.nlm.nih.gov/BLAST). Las que tenían grados de homologías mayores o iguales a 95%, fueron seleccionadas para este trabajo. La correspondiente información de las diferentes variables genómicas, se consignó en una base de datos en Excel. A partir de ésta, se efectuaron comparaciones de diferentes variables genómicas, empleando para ello programas computacionales apropiados.
Utilizando las herramientas bioinformáticas disponibles en la página Web del National Center for Biotechnology Information (NCBI) de los Estados Unidos y con la utilización de software de uso libre en el Genome Browser de la Universidad de California en Santa Cruz (http://genome.ucsc.edu/), se identificaron las diferentes clases de secuencias repetidas, los genes anotados (Refseq genes), su función, el proceso biológico asociado y la localización celular, además de la ubicación cromosómica y otras características de la cromatina adyacentes al provirus VIH-1. Además, se buscó información más detallada sobre genes, modificaciones de cromatina y de otro tipo, en las bases de datos GenCard (version 2.39) (http://www.genecards.org/cgi-bin/carddisp.pl), GeneEntrez (http://www.ncbi.nlm.nih.gov/ncbi/geneentrez) (National Human Genome Research Institute, 2004) (29) y Gene Ontology (www.geneontology.org/index.shtml) (30).
Caracterización cromosómica de la integración del VIH-1
Tomando como base los resultados de las características del genoma asociado a la integración del VIH-1, se construyó un ideograma de la distribución cromosómica, usando como plantilla de referencia el patrón de bandas G propuesto en la conferencia de París (1971), con sus posteriores actualizaciones (31) de los sitios específicos de integración dentro de los cromosomas humanos para los tres tipos de células. A partir de esta información, se obtuvieron datos con relación a la banda G correspondiente, el FISH electrónico (fluorescent in situ hybridization) y la respectiva Contig (NCBI).
Utilizando los valores de la relación del tiempo medio de replicación de cada uno de los cromosomas humanos reportados previamente (32), se calculó el tiempo promedio de replicación de cada región de integración proviral. Para el análisis correspondiente, se definieron como cromosomas de replicación temprana aquéllos que tienen una relación de tiempo de replicación cercano a 2; mientras que los cromosomas que tenían valores cercanos a 1 fueron considerados de replicación tardía. Con base en estos datos, se realizó un análisis de asociación entre el tiempo de replicación de cada región cromosómica y algunas de las características genómicas de la cromatina asociada a estas zonas, que incluyeron islas CpG, genes y transcritos.
Análisis estadísticos
Para determinar las diferencias estadísticas en el patrón de integración del VIH-1 entre los tres tipos de células humanas analizadas, a partir de una prueba de Kolmogorov (p>0,02), se efectuó un análisis de varianza de factores principales, utilizando el programa Statistica 7 (StataSoft Inc.NJ). Posteriormente, mediante una prueba post-ANOVA (Fisher-LSD), se determinaron las diferencias del número de provirus dentro de los cromosomas en los tipos celulares.
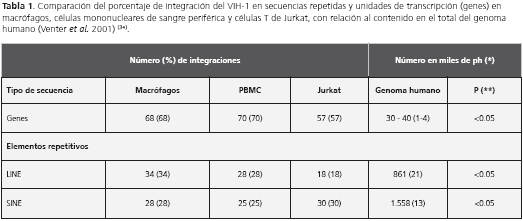
Las diferencias estadísticas entre cada uno de las células con el tipo de secuencia adyacente al ADN proviral (unidades de transcripción y elementos repetitivos) y la frecuencia de éstas en el genoma humano según el IHGSC-2001 (International Human Genome Sequencing Consortium) (33,34), se calcularon usando la prueba de χ2, considerando un valor de p<0,05 estadísticamente significante.
Mediante la prueba exacta de Fisher, se determinó si había diferencias significativas de las características genómicas analizadas (función génica, proceso biológico asociado y localización de los genes en la célula) entre macrófagos, células mononucleares de sangre periférica y células T de Jurkatt.
Resultados
Características generales de las secuencias adyacentes a provirus VIH-1
De las 300 secuencias analizadas, se obtuvieron 289 hits que coincidieron con secuencias del genoma humano, cuyas homologías fueron mayores o iguales a 95%. De ellas, 211 eran genes anotados en las bases GeneEntrez o GeneCard. De éstos, 186 correspondieron a genes humanos, mientras que el resto fueron genes ortólogos en otros organismos, como levaduras, Drosophila y Arabidopsis. Los análisis de las diferentes variables genómicas se realizaron a partir de 264 secuencias del genoma humano, que incluyeron 186 (70,5%) correspondientes a genes humanos anotados y 78 (29,6%) a secuencias no codificadoras y a elementos repetidos del tipo LINE, SINE, LTR y otros.
Localización cromosómica de los provirus VIH-1
En general, 53% de las integraciones se registraron en regiones cromosómicas correspondientes a bandas G. Con excepción del cromosoma Y, en todos los cromosomas humanos se observaron integraciones retrovirales (figura 1). El 31,4% de las integraciones (83/264) se localizó en los cromosomas 1 (22/264), 2 (21/264), 7 (20/264) y 12 (20/264); de otra parte, 7,19% (19/264) ocurrió en los cromosomas 14 (4/264), 20 (3/264), 21 (4/264), 22 (4/264) y X (4/264). Un análisis del grupo mostró que existen patrones de integración diferentes para cada tipo de célula (Fishers least significant difference – LSD - test, Cuadrado de la Media - Mean Square = 5,77, Grados de Libertad (GL) 46, p<0,05). Se determinó que el VIH-1 se integró con mayor frecuencia en regiones teloméricas y subteloméricas, mostrando diferencias significativas con la distribución observada alrededor de las zonas centroméricas (prueba χ2, GL =1, p<0,05).
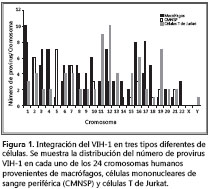
De acuerdo con lo que se muestra en la figura 2, los patrones de distribución cromosómica de provirus VIH-1, en los tres tipos celulares estudiados, fueron diferentes. En macrófagos se observó que los cromosomas 1, 3, 4, 16, y 12 tuvieron las mayores frecuencias de integración (18,18%), en comparación con las de las células mononucleares de sangre periférica, en donde los cromosomas blanco con mayor proporción de integraciones fueron el 1, 2, 4, 7, 8 y 17 (17%), y en las células T de Jurkatt, en que los cromosomas 2, 6, 7, 11, 12 y 16 incluyeron el 14% de total de integraciones registradas para este tipo celular (figura 2).

Distribución de la integración del VIH-1 en el genoma humano
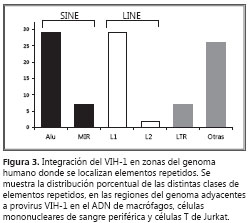
En su conjunto, se observaron diferencias estadísticamente significativas de los tipos de secuencias genómicas (secuencias codificadoras –genes anotados– y no codificadoras –LINE y SINE) localizadas en aquella fracción del genoma humano en donde ocurrieron integraciones, con relación a la distribución de las mismas en el total del genoma de los tres tipos de células (tabla 1). Una clasificación de los elementos repetitivos que no codifican asociados a los sitios de integración, mostró que predominaban en igual proporción las secuencias Alu (familia SINE) (29%) y L1 (familia LINE) (29%), mientras que el restante correspondió a elementos MIR, L2, LTR y otros (figura 3).

Distribución funcional y localización celular de los genes adyacentes a los provirus VIH-1
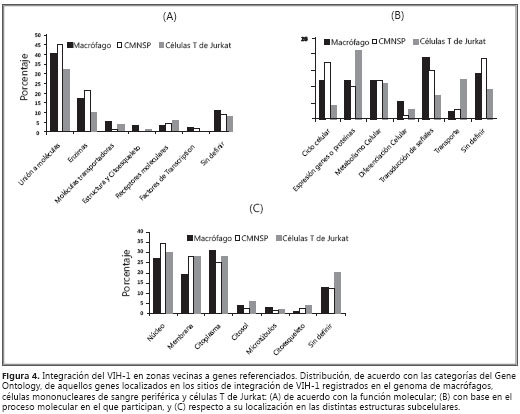
Del total de secuencias analizadas, el 70,45% correspondió a genes referenciados en el Gene Ontology. Respecto a su función molecular, no hubo diferencias entre los tres tipos celulares (p>0,05), y las categorías funcionales más representativas fueron las proteínas que se unen a otras moléculas (52,4%) y las enzimas que participan en varios procesos metabólicos (21,5%) (figura 4a).
Con relación al proceso biológico en el que intervienen, se determinó una mayor frecuencia de integración en zonas del genoma adyacentes a genes que participan en el ciclo celular, en la transducción de señales y en el transporte (figura 4b). Sin embargo, cuando se analizaron estos patrones de distribución entre macrófagos y células mononucleares de sangre periférica con el de células T de Jurkat, se encontraron diferencias significativas (p<0,05). En este sentido, se determinó que, para los macrófagos y las células mononucleares de sangre periférica, 62% de los genes participaba en la regulación del ciclo celular, en la transducción de señales y en el metabolismo, mientras que, en células T de Jurkat, 50% de sus productos génicos participaba tanto en la regulación de la expresión de genes como en el transporte de moléculas.
En cuanto a la localización celular de las proteínas codificadas por estos genes, se determinó que 80% se encuentran en el citoplasma, en el núcleo y en sistema endomembranoso. Las restantes se distribuyeron entre el citosol y el citoesqueleto.
Con relación a la distribución de la localización celular, no se observaron diferencias significantes entre los tres tipos celulares analizados (p>0,05) (figura 4c).
Un análisis comparativo entre la relación de tiempo medio de replicación y el número de provirus por cromosoma, mostró un patrón coincidente en la distribución para los cromosomas 1, 12 y 17 cuyas relaciones de tiempo medio de replicación (RTMR) variaban de 1,5 a 1,64. Sin embargo, 62,0% de los provirus analizados se localizó en cromosomas cuyas RTMR variaban de 1,3 a 1,5. Un aspecto interesante del análisis fue que los cromosomas de replicación más temprana (17,19,22) cuyos RTMR oscilaban entre 1,54 y 1,75, tuvieron sólo el 10% del total de provirus (figura 5). En los cromosomas grandes del grupo A con RTMR tiempo medio de replicación de 1,43 a 1,52, se registró 28,6% del total de integraciones, mientras que, en los cromosomas pequeños de los grupos F y G, se localizó el 6,7% del total de integraciones. Excepto el cromosoma 21, los otros de estos grupos presentan los valores de relaciones de tiempo de replicación medio más altos, por lo que se consideran de replicación muy temprana (figura 5).
Discusión
En este trabajo se llevó a cabo un análisis in silico sobre las características genómicas de extensas regiones de la cromatina celular que son blanco de integración de ADN viral del VIH-1, en tres tipos diferentes de células humanas. En su conjunto, las diferencias observadas entre la distribución de genes y las secuencias repetidas que no codifican, en aquellas zonas de la cromatina en donde ocurrieron integraciones retrovirales y su correspondiente distribución en todo el genoma celular, permiten confirmar que el VIH-1 no se integra al azar y que antes, por el contrario, existen ambientes genómicos de integración que varían según el tipo de célula.
Los datos sobre la distribución del complejo de preintegración del VIH-1 en el núcleo, sugieren que se encuentra preferencialmente en áreas de la cromatina menos condensadas situadas en la periferia nuclear (15,16). De acuerdo con lo obtenido en esta investigación, es posible postular que en los ciclos posteriores de replicación a la infección inicial, la distribución de los provirus sería favorecida hacia zonas de cromatina más abierta, en donde ésta exhibe una estructura más flexible, de curvatura más abierta o localizada en territorios cromosómicos periféricos caracterizados por tener una baja densidad de genes (35). Estas características aumentarían la probabilidad de integración viral, pues la cromatina estaría más accesible al contacto con el complejo de preintegración. El mecanismo que proponemos sería similar a lo reportado para el virus de la inmunodeficiencia de los simios y el de la inmunodeficiencia felina (35), pero diferente para la integración que ocurre en el virus de la leucemia de ratón (murine leukemia virus, VLM), el virus del sarcoma/leucosis aviar (avian sarcoma leukosis virus, ASLV) y el espumavirus (foamy virus) (21,36-39).
Las diferencias en la distribución cromosómica del provirus VIH-1 observadas en este estudio, se pueden explicar sobre la base que tanto los macrófagos como los células mononucleares de sangre periférica provenían directamente de la sangre de pacientes con VIH-1 y se pueden considerar como los blancos naturales de la infección; mientras que las células T de Jurkat son una línea celular linfocítica establecida que ha sido sometida a procesos de proliferación in vitro estimulándola con interleucinas (40). Estos resultados permiten postular que, durante el proceso de infección natural y en el de infección in vitro, ocurrirían eventos epigenéticos diferenciales de la cromatina asociada a la integración retroviral del VIH-1, que conformarían la distribución específica por células de las zonas de cromatina accesibles para la integración retroviral.
La distribución cromosómica del provirus registrada en los tres tipos de células analizadas, mostró la existencia de algunas zonas con gran frecuencia de integración, las cuales se localizaron preferencialmente en regiones cromosómicas teloméricas y subteloméricas. En este sentido, se ha reportado previamente que las poblaciones de linfocitos T CD4+ y CD8+ de pacientes infectados por VIH-1, sufren un acortamiento dinámico de sus telómeros, debido a una expansión celular continua y prolongada que puede potencialmente llevar a un agotamiento prematuro de la respuesta protectora antiviral y a una replicación senescente (41-42). Además, los telómeros son regiones dinámicas, compuestas por heterocromatina facultativa, que se activan o inactivan en fases definidas del desarrollo celular y que cambian activamente por recombinación e integración de elementos transponibles (43,44).
Con base en los resultados obtenidos en este estudio y con aquéllos provenientes de otros trabajos referidos previamente, es posible proponer que, en la fase temprana del ciclo de infección, la integración del ADN viral estaría topológicamente favorecida en aquellas regiones de cromatina interfásica que comprenden los telómeros; a medida que la infección progresa, se produciría un desplazamiento lateral del provirus dirigido hacia regiones pericentroméricas. Uno de los factores condicionantes de este proceso podría ser el acortamiento progresivo de los telómeros observado cuando la célula se torna senescente. Así, el proceso in toto se completaría por repetidas ondas integradoras hacia las zonas subteloméricas y pericentroméricas del mismo cromosoma; ello conllevaría a una irradiación proviral a lo largo de la región de cromatina.
Los resultados del análisis in silico realizado en este estudio coincidieron con aquéllos obtenidos experimentalmente con relación a la selección del sitio blanco de integración en regiones del genoma que poseen una elevada densidad génica (21,23,26,27) y en las que se generan modificaciones epigenéticas locales (28). De igual forma, concuerdan con los hallazgos para macrófagos, células mononucleares de sangre periférica y células T de Jurkat, en donde no se encontraron diferencias entre las repeticiones de tipo SINE y LINE. Dentro de la familia de los SINE, las repeticiones Alu fueron los más prevalentes. Este tipo de secuencia constituye de 5% a 10% del genoma humano y es clave en los rearreglos cromosómicos y en la regulación génica, y se asocia con genes esenciales (housekeeping) y con alto contenido de islas CpG (45,46).
Por otra parte, las características funcionales de los genes localizados en la vecindad de provirus, coincidieron con las reportadas (47,48), en donde los genes de macrófagos y células mononucleares de sangre periférica participan en el ciclo celular, en la transducción de señales y en los procesos metabólicos de la célula, mientras que, para las células T de Jurkat, actúan en la regulación de la expresión génica y el transporte de moléculas.
De acuerdo con toda la información obtenida en éste y otros estudios referidos anteriormente, es posible proponer que, en una primoinfección viral, los primeros sitios blanco de integración estarían condicionados por aquellas regiones de cromatina interfásica localizadas en los territorios cromosómicos periféricos caracterizados por tener una baja densidad de genes. Éstos serían los primeros sitios de contacto con el complejo de preintegración y generarían la primera onda de integraciones en la cromatina de la célula huésped. Posteriormente, cuando ocurre el acortamiento de los telómeros, se produciría un desplazamiento de la cromatina. Sin embargo, éste no sería el único mecanismo que explicaría los patrones de distribución proviral observados; otros eventos, como los de remodelación de la cromatina, y aun eventos epigenético locales, tendrían efectos reguladores sobre la expansión por retrotransposición hacia las zonas de cromatina interfásica localizadas en el centro del núcleo que presentan una alta densidad génica.
Esta dinámica del proceso de integración sería el posible mecanismo por el cual las regiones ricas en provirus se distribuyen finalmente por todo el genoma de la célula. Sin embargo, faltan estudios que determinen cuáles, de todos los componentes que caracterizan los ambientes genómicos de la integración, serían los principales y cómo influenciarían la dinámica de este proceso.
Agradecimientos
El trabajo se culminó con recursos proporcionados por la Vicerrectoría de Investigaciones de la Universidad del Valle, bajo el acta de trabajo y compromiso 1576 del 2008.
Declaración de conflictos de interés
Los investigadores declaran que no existe ningún tipo de conflicto de intereses con respecto a los resultados de esta investigación.
Correspondencia:
Felipe García-Vallejo, Laboratorio de Biología Molecular y Patogénesis, Departamento de Ciencias Fisiológicas, Facultad de Salud, Universidad del Valle, Aparta do aéreo 25360, Cali, Colombia. labiomol@gmail.com
Referencias
1. Barre-Sinoussi F, Chermann JC, Rey F, Nugeyre MT, Chamaret S, Gruest J, et al. Isolation of a T-lymphotropic retrovirus from a patient at risk for acquired immune deficiency syndrome (AIDS). Science. 1983;220:868-71. [ Links ]
2. Rosenberg ZF, Fauci AS. The immunophatogenesis of HIV Infection. Adv Immunology. 1989;47:377-431. [ Links ]
3. Levy JA. Pathogenesis of human immunodeficiency virus infection. Microbiol Review. 1993;57:183-289. [ Links ]
4. Vink C, Lutzke R, Plasterk R. Formation of a stable complex between the human immunodeficiency virus integrase protein and viral DNA. Nucleic Acids Res. 1994;22:4103-10. [ Links ]
5. van Maele B, Busschots K, Vandekerckhove L, Christ F, Debyser Z. Cellular co-factors of HIV-1 integration. Trends Biochem Sci. 2006;31:98-105. [ Links ]
6. Cherepanov P, Maertens G, Proost P, Devreese B, Beeumen J. HIV-1 integrase forms stable tetramers and associates with LEDGF/ p75 protein in human cells. J Biol Chem. 2003;278:372-81. [ Links ]
7. Ciuffi A, Bushman FD. Retroviral DNA integration: HIV and the role of LEDGF/p75. Trends in Genetics. 2006;22:388-95. [ Links ]
8. Grandgenett D. Symmetrical recognition of cellular DNA target sequences during retroviral integration. Proc Natl Acad Sci USA. 2005;102:5903-4. [ Links ]
9. Karczewski M, Strebel K. Cytoskeletal association and virion incorporation of the human immunodeficiency virus type 1 Vif protein. J Virol. 1996;70:494-507. [ Links ]
10. Le Rouzic E, David A, Mazzolini J, Bouchet J, Bouaziz S, Niedergang F, et al. Localization of HIV-1 Vpr to the nuclear envelope: impact on Vpr functions and virus replication in macrophages. Retrovirology. 2007;4:84-98. [ Links ]
11. Bukrinsky MI, Sharova N, Dempsey MP, Stanwick TL, Bukrinskaya AG, Haggerty S, et al. Active nuclear import of human immunodeficiency virus type 1 ipreintegration complexes. Proc Natl Acad Sci USA. 1992;89:6580-4. [ Links ]
12. von Schwedler U, Kornbluth R, Trono D. The nuclear localization signal of the matrix protein of human immunodeficiency virus type 1 allows the establishment of infection in macrophages and quiescent T lymphocytes. Proc Natl Acad Sci USA. 1994;91:6992-6. [ Links ]
13. Gallay P, Swingler S, Song J, Bushman F, Trono D. HIV-1 infection of nondividing cells; C-terminal tyrosine phosphorylation of the viral matrix protein is a key regulator. Cell. 1995;83:569-76. [ Links ]
14. Pemberton L, Blobel G, Rosenblum J. Transport routes through the nuclear pore complex. Curr Opin Cell Biol. 1998;10:392-9. [ Links ]
15. Bukrinsky M, Haffar O. HIV-1 nuclear import: in search of a leader. Front Biosci. 1997;2:d578. [ Links ]
16. Levin A, Armor-Omer A, Rosenbluh J, Melamed-Book N, Graessmann A, Waigman E, et al. Inhibition of HIV-1 integrase nuclear import and replication by a peptide bearing integrase putative nuclear localization signal. Retrovirology. 2009;6:112. [ Links ]
17. Lewinski MK, Bisgrove D, Shinn P, Chen H, Hoffmann C, Hannenhalli S, et al. Genome-wide analysis of chromosomal features repressing human immunodeficiency virus transcription. J Virol. 2005;79:6610-9. [ Links ]
18. Bisgrove D, Lewinski M, Bushman FD, Verdin E. Molecular mechanisms of HIV-1 proviral latency. Expert Rev Anti Infect Ther. 2005;3:805-14. [ Links ]
19. Rick SM, Beitzel BF, Schroder AR, Shinn P, Chen H, Berry CC, et al. Retroviral DNA integration: ASLV, HIV, and MLV show distinct target site preferences. Plos Biology. 2004;2:1127-37. [ Links ]
20. Arendt CW, Littman DR. HIV: master of the host cell. Genome Biol. 2001;2:1030.1-4. [ Links ]
21. Derse D, Crise B, Li Y, Princler G, Stewart C, Connor F, et al. HTLV-1 integration target sites in the human genome: comparison with other retroviruses. J Virol. 2007;81:6731-41. [ Links ]
22. Albanese A, Arosio D, Terreni M, Cereseto A. HIV-1 pre-integration complexes selectively target decondensed chromatin in the nuclear periphery. Plos One. 2008;3:1-9. [ Links ]
23. Schroder AR, Shinn P, Chen H, Berry C, Ecker JR, Bushman F. HIV-1 integration in the human genome favors active genes and local hotspots. Cell. 2002;110:521-9. [ Links ]
24. Meaburn KJ, Misteli T. Cell biology: chromosome territories. Nature. 2007;445:379-81. [ Links ]
25. Rosa A, Everaers R. Structure and dynamics of interphase chromosomes. PLoS Comput Biol. 2008;4:e1000153. [ Links ]
26. Barr S, Ciuffi A, Leipzig J, Shinn P, Ecker J, Bushman F. HIV Integration site selection: targeting in macrophages and the effects of different routes of viral entry. Molecular Therapy. 2006;14:218-25. [ Links ]
27. Berry CC, Ecker JR, Bushman FD. Retroviral DNA integration: ASLV, HIV, and MLV show distinct target site preferences. PLoS Biol. 2004;2:E234. [ Links ]
28. Wang GP, Ciuffi A, Leipzig J, Berry CC, Bushman FD. HIV integration site selection: Analysis by massively parallel pyrosequencing reveals association with epigenetic modifications. Genome Res. 2007;17:1186-94. [ Links ]
29. Pruitt KD, Tatusova T, Maglott DR. NCBI Reference Sequence (RefSeq): a curated non-redundant sequence database of genomes, transcripts and proteins. Nucleic Acids Res. 2005;33(database issue):D501-4. [ Links ]
30. Wheeler DL, Barrett T, Benson DA, Bryant SH, Canese K, Chetvernin V, et al. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res. 2006;34 (database issue):D173-80. [ Links ]
31. Paris Conference 1971. Supplement standardization in human cytogenetics. Cytogenetic Cell Genet. 1975;15:203-38. [ Links ]
32. Woodfine K, Fiegler H, Beare D, Collins J, Mccann O, Young B, et al. Replication timing of the human genome. Hum Mol Genet. 2004;13:191-202. [ Links ]
33. Collins FS, Morgan M, Patrinos A. The Human Genome Project: lessons from large-scale biology. Science. 2003;300:286-90. [ Links ]
34. Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, et al. The sequence of the human genome. Science. 2001;291:1304-51. [ Links ]
35. Liu H, Dow EC, Reetakshi A, Kimata JT, Bull LM, Arduino RC, et al. Integration of human immunodeficiency virus type 1 in untreated infection occurs preferentially within genes. J Virol. 2006;80:7765-68. [ Links ]
36. Crise B, Li Y, Yuan C, Morcock DR, Whitby D, Munroe DJ, et al. Simian immunodeficiency virus integration preference is similar to that of human immunodeficiency virus type 1. J Virol. 2005;79:12199-204. [ Links ]
37. Wu X, Li Y, Crise B, Burgess SM. Transcription start regions in the human genome are favored targets for MLV integration. Science. 2003;300:1749-51. [ Links ]
38. Trobridge GD, Miller DG, Jacobs MA, Allen JM, Kiem HP, Kaul R, et al. Foamy virus vector integration sites in normal human cells. Proc Natl Academy Science USA. 2006;103:1498-503. [ Links ]
39. Kang Y, Moressi C, Scheetz T, Xie L, Thi-Tran D, Casavant T, et al. Integration site choice of a feline immunodeficiency virus vector. J Virol. 2003;80:8820-3. [ Links ]
40. Nakada H, Inoue M, Tanaka N, Wakamiya N, Yamashina I. Expression of the T antigen on a T-lymphoid cell line, supT1. Glycoconj J. 1995;12:356-9. [ Links ]
41. Effros RB, Allsopp R, Chiu CP, Hausner MA, Hirji K, Wang L, et al. Shortened telomeres in the expanded CD28- CD8- cell subset in HIV disease implicate replicative senescence in HIV pathogenesis. AIDS. 1996;10:F17. [ Links ]
42. Dagarag M, Evazyan T, Rao N, Effros RB. Genetic manipulation of telomerase in HIV-specific CD8+ T cells: enhanced antiviral functions accompany the increased proliferative potential and telomere length stabilization. J Immunol. 2004;15: 6303-11. [ Links ]
43. Flint J, Bates GP, Clark K, Dorman A, Willingham D, Roe BA, et al. Sequence comparison of human and yeast telomeres identities structurally distinct subtelomeric domains. Hum Mol Genet. 1997;6:1305-13. [ Links ]
44. Riethman H. Human telomere structure and biology. Annu Rev Genomics Hum Genet. 2008;9:1-19. [ Links ]
45. Kumar PP, Mehta S, Purbey PK, Ranveer DN, Jayani S, Purohit HJ, et al. ATB1-binding sequences and Alu-like motifs define a unique chromatin context in the vicinity of human immunodeficiency virus type 1 integration sites. J Virol. 2007;81:5617-27. [ Links ]
46. Batzer MA, Deininger PL. Alu repeats and human genomic diversity. Nat Rev Genet. 2002;3:370-9. [ Links ]
47. Eller CD, Regelson M, Merriman B, Nelson S, Horvath S, Marahrens Y. Repetitive sequence environment distinguishes housekeeping genes.Gene. 2007;390:153-65. [ Links ]
48. Giri MS, Nebozhyn M, Showe L, Montaner LJ. Microarray data on gene modulation by HIV-1 in immune cells: 2000-2006. J Leukoc Biol. 2006;80:1031-43. [ Links ]














