Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Infectio
Print version ISSN 0123-9392
Infect. vol.14 no.4 Bogotá Oct./Dec. 2010
ARTÍCULO ORIGINAL
1Microbióloga, maestra en ciencias, Laboratorio de Ecología Microbiana (LEMA), Departamento de Ciencias Biológicas, Universidad de Los Andes, Bogotá D.C., Colombia
2Microbióloga, Laboratorio de Ecología Microbiana (LEMA), Departamento de Ciencias Biológicas, Universidad de Los Andes, Bogotá D.C., Colombia
Recibido: 22/07/2010; Aceptado: 22/10/2010
Resumen
Introducción. La microbiota del tubo digestivo humano contiene bacterias benéficas para la salud que regulan el funcionamiento del colon e inhiben algunos microorganismos patógenos intestinales. Las bifidobacterias aisladas de neonatos y de leche materna se usan como microorganismos probióticos para prevenir enfermedades infecciosas, incluidas las transmitidas por alimentos.
Objetivo. Aislar e identificar Bifidobacterium sp. en humanos y determinar su capacidad bactericida frente a patógenos causantes de enfermedades transmitidas por alimentos, importantes en Colombia y en el mundo.
Materiales y métodos. Se recolectaron 17 muestras de leche materna, y 19 muestras de meconio y heces de neonatos, en diferentes hospitales de Bogotá. Los 26 aislamientos sospechosos se identificaron a nivel de género mediante PCR 16-23S; para la identificación de especie, se secuenciaron algunos de los aislamientos. La capacidad antagonista de las 26 cepas de Bifidobacterium sp. fue evaluada contra Staphylococcus aureus ATCC 25923, Escherichia coli ATCC 25922, Salmonella enteritidis ATCC 13076, Listeria monocytogenes ATCC 7644 y E. coli O157:H7 ATCC 35150. Para las cepas que presentaron mayor actividad antagonista, se analizó el extracto inhibidor con ensayos de difusión en placa.
Resultados. Todas las cepas amplificaron la banda esperada para la confirmación de género; asimismo, las cepas Bif 013 y Bif 023 se identificaron por secuenciación como Bifidobacterium breve, con una homología del 97%. Del total de cepas, 17 mostraron capacidad de inhibir, al menos, uno de los patógenos evaluados. E. coli ATCC 25922 fue el patógeno más inhibido. Se determinó que la cepa Bif 023 es eficiente como antagonista, ya que inhibió todos los patógenos evaluados. Los halos de inhibición presentaron diámetros mayores a lo esperado, lo que indica una muy buena capacidad antagonista de las cepas nativas. Se aisló Bifidobacterium sp. de leche materna, meconio y heces de neonatos, por lo cual se confirmó que este microorganismo es microbiota humana (2).
Conclusión. Se concluye que, debido a la gran capacidad antagonista de la mayoría de las bacterias aisladas, éstas pueden estar cumpliendo una importante función protectora en el recién nacido, en particular las cepas Bif 013 y Bif 023 aisladas de materia fecal. Estos microorganismos deben continuar siendo estudiados para definir su potencial probiótico. También, se pueden evaluar para bioconservación en la industria y contra patógenos transmitidos por alimentos.
Palabras clave: leche materna, antimicrobiano, Bifidobacteria spp., probióticos, bactericida.
Abstract
Introduction. The microbiota in the human gastrointestinal tract contains beneficial microorganisms for human health, which contribute to the regulation of colonic function and inhibition of some intestinal pathogens growth. Bifidobacterium sp. isolated from newborns and breast milk are used as probiotic microorganisms, which are useful in the prevention of infectious diseases including foodborne illnesses.
Objective: To isolate and identify human Bifidobacterium sp. and to determine its antibiotic activity against important pathogens which cause foodborne illnesses in Colombia and the world.
Materials and methods. 17 breast milk samples and 19 meconium and newborn faeces samples were collected from different hospitals in Bogotá. 26 presumptive Bifidobacterium strains were identified at genus level by PCR 16-23S; some strains were identified at species level by nucleic acid sequencing. The antagonistic activity of 26 Bifidobacterium sp. strains was tested against Staphylococcus aureus ATCC 25923, Escherichia coli ATCC 25922, Salmonella enteritidis ATCC 13076, Listeria monocytogenes ATCC 7644 and E. coli O157:H7 ATCC 35150. For the strains that showed a greater antibiotic activity, the inhibitory compound was analyzed using disk diffusion tests.
Results. All strains amplified the expected band for genus confirmation. Strains Bif 023 and Bif 013 were identified by DNA-sequencing as Bifidobacterium breve, with 97% homology. 17 strains were able to inhibit at least one of the pathogens tested. Escherichia coli ATCC 25922 was the most inhibited. It was determined that the strain Bif 023 is highly efficient as an antagonist strain due its ability to inhibit all the evaluated pathogens. Inhibition areas showed higher diameters than expected, suggesting an enhanced antagonist capacity of native strains. Bifidobacterium sp. was isolated from breast milk, meconium and newborn faeces which confirmed that this microorganism is human microbiota.
Conclusion. Due to the high antagonist activity of most isolated bacteria, they could be playing an important protective function in the newborn, in particular strains Bif 013 and Bif 023, isolated from faeces. Other studies must be performed with these organisms to determine their probiotic potential as well as their use in the biocontrol industry due their activity against foodborne pathogens.
Key words. Breast milk, antimicrobial, Bifidobacterium, probiotics, antibiotic.
Introducción
La microbiota del tubo digestivo humano está compuesta por poblaciones bacterianas diversas con diferentes funciones: modifican la fisiología del colon, modulan mecanismos de defensa contra patógenos e influyen sobre la capacidad metabólica de los seres humanos (1,2,3). Dentro de este grupo de microorganismos se encuentran las bacterias ácido lácticas y Bifidobacterium sp. (4). Estas bacterias se caracterizan por ser Gram positivas, no forman esporas, anaerobias, sin movilidad y negativas para la catalasa.
Bifidobacterium fue aislada por primera vez de las heces de neonatos lactantes por Tissier et al. (1990) (5), quienes la describieron con forma de bastón, no productora de gas, anaerobia y con morfología bífida. La nombraron Bacillus bifidus (6).
Bifidobacterium sp. hace parte de la microbiota intestinal humana, donde genera efectos benéficos para la salud. Normalmente, hace parte de la flora intestinal de los neonatos (4) y está presente en el humano durante la mayor parte de su vida (1,7). La prevalencia depende de la edad y principalmente del tipo de alimentación. El efecto que tiene en la salud humana se debe a diferentes funciones, por ejemplo, colabora en la degradación de micronutrientes a nivel intestinal, puede modular el sistema inmunitario, influye en el equilibrio de la microbiota gastrointestinal y funciona como antagonista de microorganismos patógenos (7,8). Entre los mecanismos empleados por esta bacteria para actuar contra los microorganismos patógenos entéricos (4) se encuentra la síntesis de sustancias antimicrobianas, como las bacteriocinas, y la competencia por los nutrientes y por los sitios de adhesión a las células epiteliales (9,10).
Bifidobacterium sp. es un microorganismo "generalmente reconocido como seguro" (Generally Recognized as Safe, GRAS). Es un microorganismo probiótico debido a que tiene características que favorecen la salud humana, no tiene efectos negativos, es decir, es inocuo (9,13). Los probióticos colaboran en la prevención de diferentes enfermedades humanas, así como en las enfermedades transmitidas por alimentos (ETA). En la actualidad se prefiere que los nuevos probióticos que se utilicen tengan origen humano. Por todo lo anterior, se han venido utilizando ampliamente en la industria alimentaria (10,11).
Las bifidobacterias aisladas de neonatos y de leche materna se utilizan ampliamente en la industria, y para su identificación de género y de especie se dispone de diversos métodos de microbiología tradicional y de biología molecular. Entre ellos, uno de los más aplicados es la técnica basada en la reacción en cadena de la polimerasa (PCR) para la región 16S del ADN y su secuenciación. Esta ultima técnica permite discriminar características específicas dentro de las distintas especies (4,13). La combinación de estos métodos produce resultados rápidos y confiables que se utilizan tanto en la caracterización inicial de las cepas, como en la industria de alimentos, en el proceso de verificación de viabilidad de las cepas probióticas (14).
Autores interesados en la identificación y caracterización de Bifidobacterium sp. han reportado sus efectos, o los de sus metabolitos, en la inhibición del crecimiento de importantes agentes etiológicos de enfermedades transmitidas por alimentos, como Staphylococcus aureus, Salmonella sp., Escherichia coli y Listeria monocytogenes (15). Estas bacterias pueden ser aisladas de diferentes alimentos tanto en Colombia como a nivel mundial y son de gran importancia por su prevalencia e impacto en la salud pública (16). También se reporta la capacidad antagonista de estas bacterias contra Salmonella spp., Proteus spp., Shigella spp. y Candida spp. (17).
Actualmente, en la industria alimentaria se emplean bacterias del género Bifidobacterium como una nueva estrategia natural para la prevención de las enfermedades transmitidas por alimentos. Debido al incremento de estas enfermedades a nivel mundial, y al aumento de la resistencia a los antibióticos por parte de las bacterias patógenas, se ha hecho necesario encontrar alternativas naturales, tales como el uso de probióticos, para su prevención y control. En Colombia existen productos comerciales a los que se han añadido cultivos probióticos importados, como Bifidobacterium Bb12 y Bifidobacterium infantis. En nuestro país hay pocos estudios enfocados en la determinación de la capacidad bactericida de cepas humanas, como característica importante del potencial probiótico.
El objetivo de este estudio fue aislar e identificar Bifidobacterium sp. de leche materna, meconio y heces de neonato, para determinar su capacidad bactericida frente a agentes causantes de enfermedades transmitidas por alimentos.
Materiales y métodos
Mantenimiento de cepas
Las 26 cepas de Bifidobacterium sp. (BIF 01 - BIF 026) humanas se cultivaron en caldo MRS (3 ml) (Scharlau 02-135, España) y se incubaron a 35±2°C en anaerobiosis, durante 48 horas. Las cepas se mantuvieron en agar MRS inclinado (Scharlau 01-135, España) y se conservaron en MRS más glicerol al 10% a -20°. Se utilizó como control B. brevis ATCC 15700.
Aislamiento e identificación
Un total de 36 muestras, 10 de leche materna y 16 de materia fecal de neonato (6 de meconio y 10 de heces), se analizaron para el aislamiento e identificación de Bifidobacterium sp. El muestreo se realizó con el consentimiento de los padres y la participación voluntaria de las madres, y de acuerdo con los requisitos del Comité de ética de la Universidad de los Andes.
Para el aislamiento de Bifidobacterium sp. se tomaron 3 g de materia fecal directamente del pañal, se homogenizaron en 10 ml de solución salina y se sembraron en cajas de agar MRS (Scharlau 01-135, España). Para el aislamiento a partir de la leche materna, se sembró 0,1 ml de la muestra en agar MRS (Scharlau 01-135, España).
La confirmación del género de los aislamientos, presuntamente positivos, se realizó por medio de PCR uniplex 16-23S, utilizando los cebadores (Bif164-F 5'GGGTGGTAATGCCGGATG3' y Bif662-R 3'CCACCGTTACACCGGGAA5'), y el protocolo estandarizado en el Laboratorio de Ecología Microbiana, con base en el reporte de Kaufman et al. (14).
Para la identificación de especie de las bacterias seleccionadas por su gran capacidad antagonista, se amplificó el gen 16S con oligonucleótidos universales específicos para el dominio bacteria (27F y 1492), y se empleó un PCR uniplex de acuerdo con lo descrito por Michael (18). El ciclo de amplificación se hizo a 94°C por 2 minutos, seguido de 35 ciclos de 94°C por 45 segundos, 53°C por 45 segundos y 72°C por 1 minuto; la extensión final se realizó a 72°C por 10 minutos.
Los productos de amplificación (1.500 pb) fueron secuenciados por Macrogen Inc. y las secuencias, editadas en BioEdit Sequence Alignment Editor, versión 7.0.5 (http://www.mbio.ncsu.edu/BioEdit), se compararon contra secuencias de ADN del National Center for Biotechnology Information (NCBI); se usaron alineaciones locales y, con la herramienta BLAST (http://blast.ncbi. nlm.nih.gov/BLAST), se estableció la homología entre nucleótidos.
Pruebas de inhibición
Los ensayos de inhibición se hicieron con cada una de las bifidobacterias identificadas, y con las cepas patógenas de S. aureus ATCC 2592, E. coli ATCC 25922, Salmonella enteritidis ATCC 13076, L. monocytogenes ATCC 7644 y E. coli O157:H7 ATCC 35150. Se sigió el protocolo de doble capa (19).
Sobre el agar MRS se fijaron gotas del cultivo de cada Bifidobacterium sp hasta el día siguiente. Posteriormente, se adicionó la segunda capa de agar MRS y se inoculó con 1 ml de cada patógeno previamente crecido en caldo nutritivo (Oxoid Ltd. CM1, Basingstoke, Inglaterra). Se utilizó como control positivo la cepa de Bifidobacterium breve ATCC 15700. Los ensayos realizados por duplicado fueron incubados durante 72 horas a 35±2°C. El resultado de la prueba se consideró positivo cuando apareció un halo de inhibición superior a 1 cm de diámetro (19).
Determinación de un metabolito termorresistente similar a las bacteriocinas
Alícuotas de los sobrenadantes de Bif 013 y Bif 023 (seleccionadas por su actividad antagonista) fueron calentadas a 80°C (19) por 5 minutos. Posteriormente, a cada una de las muestras se les realizó el protocolo de doble capa. Se utilizaron como cepa blanco las siguientes bacterias: (L. monocytogenes ATCC 1119, S. aureus ATCC 25923, E. coli ATCC 25922 y S. Enteritidis ATCC13076). Los ensayos se incubaron a 30±1°C por 24 horas. Como control positivo se utilizó Lactococcus lactis sub lactis ATCC11454.
Resultados y discusión
De las 36 muestras procesadas se aislaron 26 cepas de Bifidobacterium sp. (10 de leche materna, 6 de meconio y 10 de heces de neonato). Las 16 cepas aisladas de recién nacido indican que este microorganismo es un miembro importante de la microbiota intestinal de los recién nacidos. Estos resultados coinciden con los reportados por otros autores que han demostrado la presencia de bacterias ácido lácticas en la leche materna, y la predominancia de Bifidobacterium en la microbiota intestinal de neonatos, en donde estas bacterias tienen un papel protector para el recién nacido respecto a las enfermedades infecciosas intestinales (4). Se aislaron 10 cepas en leche materna, lo cual ratifica la importancia de ésta para la protección del bebé frente a diferentes patógenos.
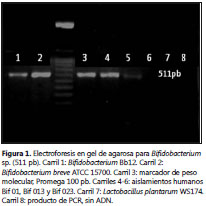
Los 26 aislamientos amplificaron con el PCR uniplex 16-23S y generaron una banda de 511 pb específica para Bifidobacterium sp. (figura 1). Las cepas Bif 013 y Bif 023, que fueron seleccionadas por su gran capacidad antagonista, se secuenciaron e identificaron como B. breve, debido a que las secuencias analizadas mostraron una homología de 97% (E value: 0.0; score: 1604/1604) con B. breve. Estos resultados concuerdan con reportes disponibles sobre la microbiota intestinal de neonatos, ya que B. breve y B. infantis se han reportado con frecuencia en los aislamientos de las primeras semanas de vida (1).
De las 26 cepas aisladas, 17 (65,4%) inhibieron, al menos, uno de los patógenos evaluados (tabla 1), y 9 (34,6%) no inhibieron a ninguno de ellos. Escherichia coli ATCC 25922 fue el patógeno más inhibido: por 13 cepas de Bifidobacterium sp. (50%). La sigue L. monocytogenes ATCC 7644. S. enteritidis ATCC 13076 sufrió inhibición por la acción de 10 cepas (38,5%), el crecimiento de S. aureus ATCC 2593 resultó afectado por 8 cepas (30,8%), Â y la cepa menos inhibida fue E. coli O157:H7 ATCC 35150, ya que solamente 3 cepas (11,5%) de Bifidobacterium sp. tuvieron efecto antagonista contra ese patógeno. Las cepas de Bifidobacterium con capacidad bactericida pueden ser productoras de bacteriocinas o de otros metabolitos antimicrobianos, como ácidos, peróxidos, etc. (6)
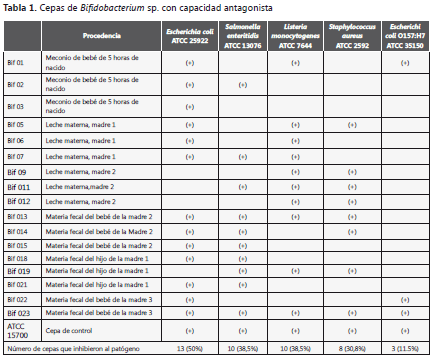
Las cepas Bif 013 y Bif 023 aisladas de heces de neonato mostraron gran capacidad antagonista, ya que Bif 023 inhibió a los cinco patógenos evaluados en este estudio, y Bif 013 inhibió a cuatro. Las cepas Bif 05, Bif 07, Bif 011, aisladas de leche materna, la cepa Bif 01, aislada de meconio de bebé, y las cepas Bif 014 y Bif 019, de heces de neonatos, inhibieron, cada una, al menos, tres de los cinco patógenos.
Las cepas Bif 013 y Bif 023 producen un metabolito termorresistente, similar a las bacteriocinas, el cual tiene actividad antagonista contra L. monocytogenes (figura 2). Este es un compuesto soluble en el medio de cultivo y se obtuvo en el sobrenadante después de la precipitación de las células (extracto crudo); además no fue desnaturalizado por la exposición a 80° por 10 minutos, lo que podría sugerir que es una proteína termorresistente (17). Otros compuestos, como el peróxido de hidrgeno o el ácido acético, se ven afectados por la temperatura y pierden su capacidad bactericida. En este caso, el compuesto continuó siendo activo después del tratamiento térmico, lo que descarta la posibilidad de que la actividad listericida fuera por alguno de ellos. Este resultado es coherente con lo reportado en la literatura por otros autores (6,16,20), quienes han demostrado la producción de metabolitos bactericidas producidos por las bacterias ácido lácticas, y el efecto inhibitorio que tienen sobre patógenos o bacterias que deterioran los alimentos. Las bacterias ácido lácticas son excelentes productoras de estos péptidos antimicrobianos (bacteriocinas), compuestos que deben continuar en estudio para caracterizar y confirmar su naturaleza proteica.
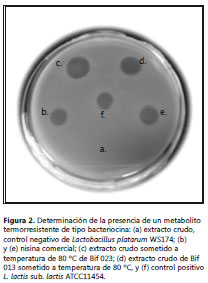
El metabolito termorresistente, producido por las cepas evaluadas, sólo fue efectivo en contra de L. monocytogenes, lo que concuerda con lo reportado por Trias, quien describió la actividad antagonista de compuestos secretados por las bacterias ácido lácticas para controlar bacterias patógenas, incluida L. monocytogenes (21). Además, se puede proponer que el compuesto inhibidor corresponde a una bacteriocina de clase II, debido a que su espectro de acción se restringe a especies del género Listeria, tal como sucede en este estudio (8) confirmación que se debe hacer bioquímicamente.
Por otro lado, los resultados sugieren que Bif 013 y Bif 023 pueden producir otra clase de compuestos, que pueden ser responsables de la inhibición de los demás patógenos evaluados (S. aureus ATCC25923, S. enteritidis ATCC 13076, E. coli ATCC25922 y E. coli O157:H7).
En este estudio, las cepas de Bifidobacterium que tuvieron mayor capacidad antimicrobiana fueron las Bif 023 y Bif 013, por lo cual se proponen como potenciales probióticos o agentes biocontroladores. Estas cepas podrían ser investigadas para su uso en el control de patógenos y microorganismos “deteriorantes” en la industria. Además, es necesario continuar los ensayos preclínicos y clínicos para demostrar los beneficios de estos microorganismos en la salud humana, para que puedan ser utilizados como probióticos.
La disponibilidad de cepas probióticas nativas en el mercado nacional puede estimular la producción de alimentos con valor agregado, menor costo mayor cobertura de la población, y con un papel profiláctico; por supuesto, si la determinación de sus propiedades clínicas, cumple con la reglamentación exigida por la FAO y la OMS (11).
Correspondencia:
María Consuelo Vanegas, Laboratorio de Ecología Microbiana (LEMA), Departamento de Ciencias Biológicas, Universidad de Los Andes, Cra 1 No 18ª -, J 209 Bogotá D.C, Colombia. Tel: 339-4949, extensión 3339. Dirección electrónica: mvanegas@uniandes.edu.co
Referencias
1. Liévin V, Peiffer I, Hudault S, Rochat F, Brassart D, Neeser JR, et al. Bifidobacterium strains from resident infant human gastrointestinal microflora exert antimicrobial activity. Gut. 2000;47:646-52. [ Links ]
2. Fernández M, Boris S, Barbés C. Probiotic properties of human lactobacilli strains to be used in the gastrointestinal tract. J Appl Microbiol. 2003;94:499-55. [ Links ]
3. Gibson GR, Wang X. Regulatory effects of Bifidobacteria on the growth of other colonic bacteria. J Appl Bacteriol. 1994;77:412-20. [ Links ]
4. Martín R, Langa S, Reviriego C, Jiménez E, Marín ML, Xaus J, et al. Human milk is a source of lactic acid bacteria for the infant gut. J Pediatr. 2003;143:754-58. [ Links ]
5. Biavati B, Vescovo M, Torriani S, Bottazzi V. Bifidobacteria: History, ecology, physiology and applications. Ann Microbiol. 2000;50:117-31. [ Links ]
6. Cheikhyoussef A, Pogori N, Chen W, Zhang H. Antimicrobial proteinaceous compounds obtained from bifidobacteria: From production to their application. Int J Food Microbiol. 2008;125:215-22. [ Links ]
7. Neish A. Microbes in gastrointestinal health and disease. Gastroenterology. 2009;136:65-80. [ Links ]
8. Ouwehand AC. Antimicrobial components from lactic acid bacteria. In: Salminen S, von Wright A, editors. Lactic acid bacteria. New York: Marcel Dekker; 1998. p. 139-59. [ Links ]
9. Gueimonde M, Margolles A, De los Reyes-Gavilán C, Salminen S. Competitive exclusion of enteropathogenes from human Intestinal mucus by Bifidobacterium strains with acquired resistance to bile. Int J Food Microbiol. 2007;113(2):228-32. [ Links ]
10. Guglielmetti S, Tamagnini I, Minuzzo M, Arioli S, Parini C, Comelli E. et al. Study of the adhesion of Bifidobacterium bifidum MIMBb75 to human intestinal cell lines. Curr Microbiol. 2004;59:167-72. [ Links ]
11. FAO/OMS. Informe del Grupo de Trabajo Conjunto FAO/OMS sobre borrador de directrices para la evaluación de los probióticos en alimentos. 30 de abril a 1 de mayo de 2002. (Internet). Consultado en septiembre de 2010. Disponible en: ftp://ftp.fao.org/docrep/fao/009/a0512s/a0512s00.pdf [ Links ]
12. Wall R, Gerard-Hussy S, Ryan A, O´Neill M, Fitzgerald G, Stanton C. Presence of two Lactobacillus and Bifidobacterium probiotic strains in the neonate ileum. ISME J. 2008;29:83-91. [ Links ]
13. Naidu AS, Bidlack WR, Clemens RA. Probiotics spectra of lactic acid bacteria (LAB). Crit Rev Food Sci Nutr. 1999;38:113-26. [ Links ]
14. Kaufmann P, Pfefferkorn A, Teuber M, Meile L. Identification and quantification of Bifidobacterium species isolated from food with genus-specific 16S rRNA-targeted probes by colony hybridization and PCR. Appl Environ Microbiol. 1997;63:1268-73. [ Links ]
15. Tissier H. Traitement des infections intestinales par la méthode de la flore bactérienne de l´intestin. Critical Reviews of the Society for Biology. 1906;60:359-61. [ Links ]
16. Vanegas MC. Detección de patógenos en alimentos. Revista Hipótesis. 2004;1:34-40. [ Links ]
17. Ammor S, Tauveron G, Dufour E, Chevallier I. Antibacterial activity of lactic acid bacteria against spoilage and pathogenic bacteria isolated from the same meat small-scale facility 1-screening and characterization of the antibacterial compounds. Food Control. 2006;17:454-61. [ Links ]
18. Dojka MA, Harris JK, Pace NR.. Expanding the known diversity and environmental distribution of an uncultured phylogenetic division of bacteria. Appl Environ Microbiol. 2000;66(4):1617-21. [ Links ]
19. Botina B, Zimmermann B, Vanegas MC, González L. Caracterización preliminar de compuestos antilisteriales producidos por bacterias ácido lácticas nativas. Revista MVZ Córdoba. 2008;13:1476-85. [ Links ]
20. Kaur IP, Chopra K, Saini A. Probiotics: potential pharmaceutical applications. Eur J Pharm Sci. 2002;15(1):1-9. [ Links ]
21. Trias R, Bañeras L. Bioprotection of golden delicious apples and iceberg lettuce against foodborne bacterial pathogens by lactic acid bacteria. Int J Food Microbiol. 2008;123:50-60. [ Links ]














