Introducción
Las infecciones asociadas a la atención en salud (IAAS) representan un reto constante en la práctica clínica actual, no solo en términos económicos sino en la morbimortalidad1,2. Sumado a esto, la emergencia y diseminación de la resistencia bacteriana, secundarias al uso inapropiado de antimicrobianos, comprende un problema de salud pública3,4. En el ámbito hospitalario, en especial en las Unidades de Cuidado Intensivo (UCI), la generación de resistencia se ha visto favorecida por la presencia de pacientes altamente susceptibles, el uso cada vez mayor de dispositivos médicos invasivos, la transmisión horizontal por gérmenes resistentes y la utilización intensiva y prolongada de antibióticos5. Desde 2004, la Organización Mundial de la Salud (OMS) lidera la alianza mundial para el cuidado seguro del paciente, en la que por años ha desarrollado diversas estrategias como lavado de manos, cirugía segura y cuidado limpio y seguro. En esta última se definen las estrategias para la reducción de las IAAS6, en las que el pilar fundamental está dado por la vigilancia tanto en la resistencia bacteriana como en las principales IAAS (infecciones asociadas a dispositivos intravasculares, neumonía asociada a ventilador, infección de sitio operatorio y estudio de brotes).
Según el Centro para el Control de Enfermedades (CDC) se estima que las infecciones por brotes corresponden al 5% de las IAAS, en tanto que en Colombia el 3,5% es atribuido a esta causa.
La transmisión horizontal a través del personal de la salud es un problema constante en el que las medidas de aislamiento y el lavado de manos desempeñan un papel importante para su control. La identificación de brotes en resistencia bacteriana se da por vigilancia pasiva o activa; esta última, que se aplica de manera frecuente y rutinaria en nuestro medio, demanda una gran inversión de tiempo y recursos humanos en la revisión de tableros de pacientes y una comunicación constante con los laboratorios de microbiología para poder realizar una notificación oportuna7; sin contar con que el factor humano es altamente susceptible de error.
Desde 1991, Glenister et al. evaluaron el rendimiento diagnóstico de los diversos procesos para la detección de brotes y encontraron que la vigilancia basada en los reportes de laboratorio clínico enlazada con los servicios (vigilancia activa) era efectiva hasta en un 71%, y que consumía 7,75 h por semana del personal de enfermería y del laboratorio clínico8.
En Colombia, la vigilancia activa en las instituciones de atención en salud y en las redes y sistemas de vigilancia como el Grupo para el Control de la Resistencia Bacteriana de Bogotá (GREBO), desde 2001, el de la Secretaría Distrital de Salud (SIVIGILA), desde 2007 y la Red Nacional de vigilancia de Resistencia Bacteriana (Instituto Nacional de Salud)9 desde 2010, cuentan con la implementación del programa Whonet, software creado en 1983 (WHO Collaborating Centre for Surveillance of Antimicrobial Resistance) para el manejo y análisis de los datos de los laboratorios de microbiología, centrado en los análisis de las pruebas de susceptibilidad antimicrobiana. En la actualidad su uso está difundido en más de 155 hospitales en el país.
Por todo esto, tratando de optimizar y sistematizar los procesos, se han planteado modelos matemáticos (vigilancia virtual) con el fin de empoderar aún más el laboratorio clínico, utilizando las bases de datos de los microorganismos aislados de muestras clínicas7,10. Por esta razón, desde 2010 el programa Whonet integró entre sus aplicaciones de análisis el SaTScan, un paquete estadístico de permutaciones de espacio-tiempo que permite la detección de brotes con un rendimiento muy aproximado a la vigilancia activa11.
Estas pruebas estadísticas de espacio-tiempo han sido usadas previamente en la detección de alertas para muchas enfermedades incluyendo cáncer, enfermedad de Creutzfeldt-Jakob, esclerosis y diabetes mellitus, entre otras, en las que han mostrado gran utilidad. En principio, este análisis estadístico sirvió para escanear una ventana que se movía a través de un espacio o tiempo determinados. Para cada espacio y tamaño de cada ventana se contó el número de casos observados y esperados, con un exceso inusual en los primeros. La significancia estadística es evaluada teniendo en cuenta múltiples pruebas derivadas del tamaño y del espacio de potenciales clústeres (alertas de brotes)12.
Huang et al. plantearon el uso de permutaciones espacio tiempo para la detección prospectiva de brotes en enfermedades infecciosas con las que evidenciaron una buena correlación entre la identificación de alertas por el sistema convencional (vigilancia activa) y las permutaciones espacio-tiempo11. Se evidenció que dicha aplicación caracterizó de manera precisa la dinámica del comportamiento del brote. Sin embargo, dicho estudio no hace referencia a qué sistema detecta primero las alertas de los brotes. Y, en el contexto de oportunidad, donde la detección precoz permite instaurar medidas orientadas a su contención y evitar su diseminación, esto se traduce en menos casos y, por ende, en menos costos para el sistema de salud por disminución de morbimortalidad, estancia hospitalaria, cohortización de pacientes, medidas de aislamiento, entre otros.
En nuestro medio, la utilización de Whonet es muy aceptada, ya que todo el sistema de notificación a los entes reguladores exige que sea presentado en este formato. Sin embargo, dicha aplicación para la vigilancia virtual (SaTScan-Whonet) no es utilizada, posiblemente por desconocimiento de sus funciones o de su utilidad.
El objetivo de este estudio es determinar la utilidad de esta herramienta para la detección precoz de brotes de infecciones bacterianas, comparada con la vigilancia tradicional, en una institución de alta complejidad de Colombia.
Metodología
Población de estudio y base de datos
Estudio retrospectivo ecológico en una institución de tercer nivel de atención. De la información de pacientes hospitalizados, se seleccionaron los microorganismos aislados entre 2010 y 2011. El Hospital Universitario San Ignacio es una institución de alta complejidad y centro académico que cuenta con Servicio de Oncología y UCI, tiene una disponibilidad de 356 camas, con un porcentaje de ocupación promedio superior al 90%.
Se obtuvo la información de la base de datos electrónica de los aislamientos microbiológicos del laboratorio clínico del sistema automatizado (MicroScan(r)-Rochem Biocare), que incluye la identificación de los pacientes, fecha de muestra de los aislamientos, tipo de localización por servicio (internado UCI/no UCI), tipo de muestra y pruebas de susceptibilidad antimicrobiana.
Vigilancia activa: metodología de detección convencional
Se realizó vigilancia activa por la Unidad de Infectología con comunicación constante con el laboratorio clínico, llevada a cabo para microorganismos de importancia clínica (Staphylococcus sp., resistente a meticilina; Enterococcus sp., resistente a vancomicina [ERV]; enterobacterias productoras de betalactamasas de espectro extendido y producción de carbapenemasas; microorganismos no fermentadores multirresistentes; cualquier microorganismo aislado con perfil de resistencia inusual). Se definió el caso como "paciente hospitalizado entre los meses de marzo y mayo de 2011 con aislamiento microbiológico de cualquier muestra, con reporte de Enterococcus faecium resistente a vancomicina (ERV)".
La base de datos obtenida en el laboratorio se convirtió a formato Whonet mediante BackLink 2.0(r) (OMS). El análisis se realizó por paciente, seleccionando el primer aislamiento positivo. Se utilizó el software Whonet 5.6(r) (OMS) para los análisis descriptivos, según el Clinical and Laboratory Standards Institute (CLSI)13.
Se obtuvo la identificación y caracterización de E. faecium resistente a vancomicina de la Unidad de Infectología, junto con el reporte final de la notificación del brote a la SDS. Adicionalmente, se obtuvo la secuenciación del gen Van A de las muestras clínicas y se estudió la relación genética del brote mediante Pfge (UGRA, Universidad El Bosque).
Vigilancia virtual: detección automática de brotes Se utilizó la herramienta integrada de SaTScan del software Whonet 5.6(r), gratuito desde 200914, que utiliza las permutaciones de espacio-tiempo para la detección de brotes mediante los modelos de Poisson y Bernoulli. También se emplearon simulaciones de Montecarlo (9.999 replicaciones) para definir la significación estadística, estableciendo un valor de p y definiendo un intervalo de recurrencias (1/p). Se tomaron como espacio las localizaciones de los aislamientos o los servicios (por ejemplo hospitalización de cirugía, especialidades quirúrgicas, salas de cirugía). El perfil de resistencia también fue usado como espacio para detección de brotes con perfil fenotípico de resistencia similar para el E. faecium. Para el tiempo se utilizó la fecha de toma de la muestra.
Se les realizó un análisis retrospectivo a todos los microorganismos para evaluar la detección del brote previamente caracterizado. Las simulaciones se corrieron de manera retrospectiva hasta identificar el primer día en que el modelo predecía alerta para el clúster.
Posteriormente, se ejecutó la aplicación de manera prospectiva día a día para todos los microorganismos durante el primer semestre de 2011, simulando la búsqueda rutinaria en tiempo real para determinar en qué momento se identificó la alerta del brote para E. faecium por esta metodología. Las simulaciones se realizaron en todos los microorganismos, en servicios y localizaciones, para determinar el día de detección de la alerta del brote.
Parámetros para Whonet-SaTScan
Se creó un diccionario de aislamientos desde 2010 hasta 2011; también se consideró un número máximo de días en los que los aislamientos podrían contribuir en un brote de hasta 60 días, basado principalmente en la plausibilidad biológica. El intervalo de recurrencia se seleccionó de 365 días según la metodología propuesta en estudios similares 11. Se evaluó la utilidad de la implementación de la detección automática de brotes en comparación con la detección convencional con la oportunidad en la detección de la alerta.
Se considera una investigación sin riesgo para los pacientes según el artículo 11 de la Resolución 8430 de 1993, del Ministerio de Salud y respeta íntegramente las disposiciones del artículo 8.
Resultados
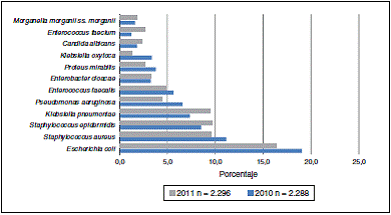
Se aislaron 4.584 microorganismos en los servicios de hospitalización tanto en UCI como en no UCI entre 2010 y 2011 (2.288 y 2.296 respectivamente). La frecuencia de microorganismos se describe en la figura 1.
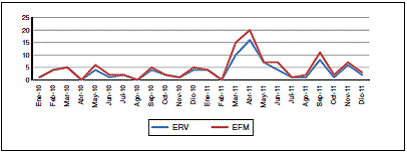
Se encontró que el aislamiento de E. faecium aumentó en 2011 en comparación con el año previo (de 11 a 36 microorganismos en UCI, de 17 a 24 en los servicios no UCI). De la misma manera, se evidenció un incremento en la cantidad de aislamientos para los meses de marzo y abril de 2011 en relación con la tendencia de los periodos anteriores (fig. 2).
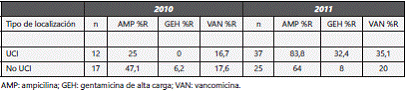
El porcentaje de resistencia según los marcadores se describe en la tabla 1.
Reporte de brote por metodología de detección convencional
El brote se reconoció y notificó la alerta de E. faecium a la SDS el día 28 de marzo de 2011. Se identificaron 6 casos en la UCI de adultos que cumplían criterios para ser considerados dentro del brote según la definición; todos los pacientes presentaron un antecedente quirúrgico previo al aislamiento (100%).
El 100% de los aislamientos fue documentado en UCI; sin embargo, 3 casos identificados tuvieron en común su estancia en el piso de especialidades quirúrgicas. Todos los pacientes presentaron estancias hospitalarias prolongadas mayores a un mes, con un promedio de 40 días. Según el tipo de muestra, 3 aislamientos fueron provenientes del torrente sanguíneo (50%); uno proveniente de secreción mediastinal (16,6%); un aislamiento de hueso (16,6%) y otro de trombo arterial (16,6%). Todos los pacientes habían recibido antibióticos previos al aislamiento microbiológico y 5 de ellos, vancomicina (83,3%). Se reportaron 4 casos de mortalidad asociada a la infección por ERV, sin considerar mortalidad atribuible. Dentro de los factores de riesgo relacionados con la emergencia de dicho germen solo se anota la presencia de uno de los casos con enfermedad neoplásica de base y 3 casos (50%) presentaban en común reingresos a la UCI relacionados con su enfermedad quirúrgica de base.
Se iniciaron medidas para la contención del brote en las que destacan la cohortización de pacientes, 9 reuniones extraordinarias del Comité de Infecciones, una auxiliar exclusiva por paciente, vigilancia estricta de lavado de manos al ingreso y salida de la UCI, cultivo de hisopados rectales a todos los pacientes hospitalizados en UCI y especialidades quirúrgicas, cultivo de superficies y de personal de salud, reentrenamiento en lavado de manos y medidas de aislamiento entre el 30 de marzo y el 15 de abril para el personal tanto del turno diurno como del nocturno. Se logró la contención del brote el día 24 de mayo de 2011.
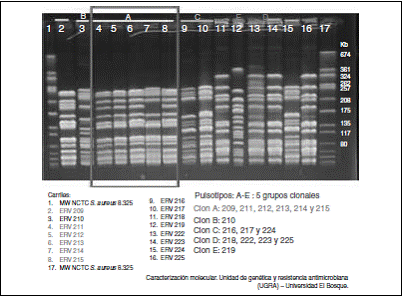
Se realizó caracterización molecular de las muestras obtenidas tanto de los aislamientos de los pacientes caso como de las muestras positivas para ERV de la búsqueda activa. Se repicaron y enviaron en medios de transporte al laboratorio UGRA (Universidad El Bosque), donde se realizó tipificación para el gen Van A: se encontró positividad en todas las muestras, con diferentes isotipos. Los pacientes caso correspondían al isotipo A; los otros, a muestras de cultivos de hisopados rectales y superficies (fig. 3).
Reporte de brote por detección automática
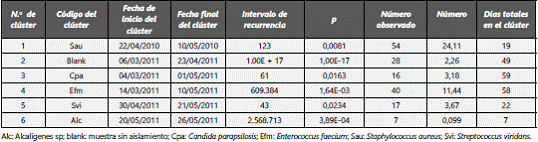
Se realizó análisis con la herramienta SaTScan-Whonet de manera retrospectiva: se hicieron 34 simulaciones para todos los microorganismos en todas las localizaciones, y se encontró la identificación de 6 alertas de brotes el día 28 de mayo de 2011, que se describen en la tabla 2.
Se llevaron a cabo análisis por localizaciones y servicios quirúrgicos sin evidenciar alerta para brote de E. faecium. Adicionalmente, se realizó una alerta por perfil fenotípico de resistencia. El brote de esta bacteria se documentó el 28 de mayo de 2011.
Se realizaron análisis de manera prospectiva diariamente para el primer semestre de 2011 para todos los microorganismos, con la intención de identificar el brote de E. faecium previamente caracterizado. Se obtuvo la primera alerta el día 13 de abril de 2011 con 103 simulaciones (tabla 3).
Se corrió el modelo de manera prospectiva día a día para la UCI: se encontró la detección el 13 de abril de 2011. Al realizar la simulación por localización no se encontró alerta para el brote (180 simulaciones).
Discusión
Los estudios para la detección de brotes en enfermedades infecciosas por vigilancia virtual con permutaciones espacio- tiempo por SaTScan-Whonet son limitados. Hasta el momento no existe una herramienta considerada como patrón de oro para la detección de brotes, lo que plantea una gran dificultad para las instituciones de salud al momento de identificarlos.
La frecuencia de microorganismos fue muy similar entre 2010 y 2011 en cuanto a número de aislamientos y gérmenes aislados. Los datos encontrados fueron similares a la epidemiología local comparados con otras instituciones de igual complejidad15. El incremento de aislamientos del E. faecium se correlacionó con el brote identificado; se observó un aumento en el porcentaje de resistencia general a la vancomicina para el periodo 2011 en la UCI.
La utilización de detección convencional permitió identificar y caracterizar el brote de E. faecium desde el 28 de marzo de 2011, 2 semanas después de que se instaurara el primer caso. Se puede considerar este tiempo como periodo de incubación, en el que no se constituyó el brote hasta superar la endemia, considerada de uno a 2 casos por mes, y se realizó la investigación para notificar el brote. La vigilancia activa en este estudio pudo ser la alerta más precoz por diversas razones, como la presencia de un germen multirresistente que encendería las alarmas de cualquier programa de control de infecciones y el seguimiento que se realizó de manera rutinaria. La vigilancia manual de los tableros de pacientes en los distintos servicios se vio reforzada con la utilización del Whonet para el análisis de la base de datos de microbiología de la institución. Sumado a esto, la calidad de la base de datos que se mantiene día a día permite analizar comportamientos inusuales de los aislamientos, sin mencionar la política institucional del cuidado seguro del paciente.
En la vigilancia virtual, fue posible identificar las alertas de manera posterior a la vigilancia activa de los análisis retrospectivos. Para generar la primera alerta se requirió de información de 2 meses adicionales para reconocer la presencia del brote (28 de mayo de 2011), en tanto que para los análisis prospectivos simulados se requirió de información de 15 días adicionales para generar la primera alerta.
Aunque la información es limitada, en la revisión de la literatura se encuentran 2 estudios en los que se propone evaluar el rendimiento diagnóstico de los modelos matemáticos para la detección prospectiva de brotes.
Mellman et al. realizaron un estudio en 2006, en el que buscaron brotes de Staphylococcus aureus (S. aureus) resistente a la meticilina entre 1998 y 2003, en un hospital alemán de tercer nivel de atención, con secuenciación por biología molecular de la proteína A (spa) del Staphylococcus para poder detectar la manifestación del mismo germen utilizando vigilancia virtual con un sistema de alertas incluido en el programa Ridom StaphType (permutaciones temporales). Este estudio fue comparado con la metodología tradicional de detección, considerando como definición de alerta el aislamiento de 2 casos de S. aureus resistente a la meticilina en menos de 2 semanas en la institución. De los casos detectados se encontraron 13 brotes significativos con la tipificación del spa, que se correlacionaron con la metodología convencional. Las alertas clonales por permutación temporal fueron 100% sensibles y 95% específicas. Para la detección por metodología convencional (vigilancia activa) fueron 62,1% sensibles y 97,3% específicas. La diferencia en especificidad entre la detección clonal y por método convencional no fue estadísticamente significativa. Ambos métodos tuvieron VPP del 80%16.
A pesar de que el rendimiento diagnóstico planteado con esta herramienta en dicho estudio es muy bueno, la metodología virtual se plantea únicamente con permutaciones temporales y no evaluó la oportunidad. Esto quiere decir que, si bien la herramienta captura las alertas de brotes tipificados por biología molecular, no especifica el tiempo en el que los detecta en comparación con la metodología convencional, y este dato representa información sensible para disminuir los costos en la detección. Nuestro estudio mostró que la metodología convencional identificó más precozmente el brote en comparación con la utilización del SaTScan-Whonet. Esto, en términos prácticos, significa que identificar estas alertas de brotes a la mayor brevedad de tiempo implica disminución de la transmisión, de la morbi-mortalidad y del número de casos observados.
El estudio realizado por Huang et al. en 2010 tomó los datos de alertas de brotes obtenidos en la vigilancia activa por 2 epidemiólogos altamente entrenados entre 2002 y 2006 en un hospital universitario de Estados Unidos y los comparó con la implementación de los análisis de alertas de manera prospectiva. Encontró que la vigilancia virtual fue superior en la identificación de brotes de microorganismos inusuales (Pseudomonas aeruginosa y Acinetobacter baumannii multirresistentes) y detectó una mayor cantidad de alertas, de las cuales aproximadamente el 25% requirió seguimiento. En nuestro estudio, la identificación de alerta de brote para ERV fue posible en la vigilancia virtual, sin embargo, de una manera menos oportuna que la vigilancia activa. Es un dato que podría ser explicado por el porcentaje de prevalencia de este germen en la institución, lo que indica que la vigilancia activa podría ser útil en la identificación de alertas para casos muy inusuales, sin reemplazar la vigilancia activa.
La metodología de la detección automática de brotes retrospectiva ha mostrado previamente que la herramienta podría llegar a ser muy precisa, al caracterizar de manera fiel la dinámica del comportamiento del brote. En 2012 se presentaron los resultados de un estudio realizado por el GREBO en el que se corrió de manera retrospectiva el SaTScan-Whonet en un sistema de vigilancia de 37 instituciones de alta complejidad en UCI y se comparó con la notificación de brotes reportada por dichas instituciones a la SDS: encontraron una correlación de alertas, estadísticamente significativas, en 3 instituciones entre 2010 y 201117. En nuestro estudio, la relación al ejecutar los análisis de manera retrospectiva en la totalidad de los datos permitió identificar el brote de E. faecium con un rango de fechas para los casos observados más aproximado a la realidad. Por esta razón, podría ser de gran utilidad para los sistemas de vigilancia de los entes reguladores, ya que permitiría hacer seguimiento a las instituciones que no realizan una vigilancia o presentan deficiencias en sus procesos, con el fin de armonizar con la política de cuidado seguro del paciente. Sin embargo, se debe tener en cuenta que para que la herramienta pudiera identificar el brote requirió de los datos de los 2 meses posteriores al evento para su caracterización.
Según nuestro estudio no consideramos que la implementación de la herramienta para análisis prospectivos sea superior a la detección convencional realizada habitualmente para ERV, en cuanto a la oportunidad de alertas de brotes, puesto que la notificación por el sistema convencional se realizó el 28 de marzo de 2011, mientras que en las simulaciones prospectivas (por detección automática de brotes) se identificó la primera alerta el 13 de abril de 2011. Sin embargo, se debe aclarar que la búsqueda activa en la institución de estudio cuenta con un grupo altamente calificado y con vasta experiencia; la herramienta prospectiva podría ser de utilidad en establecimientos que no cuenten con una vigilancia activa tan rigurosa y para la identificación de alertas muy inusuales que puedan pasar desapercibidas. Otro aspecto importante hace referencia al tipo de microorganismo que compone el brote: el ERV es un germen de importancia clínica.
La utilización del SaTScan ha mostrado una gran utilidad en la detección de alertas en varias enfermedades; en 2005 Kulldorff, el creador de dicha aplicación, demostró la utilidad del software para detectar, de manera precoz, alertas de los brotes infecciosos. Al tomar los registros de las bases de datos por motivo de consulta (diarrea y síntomas respiratorios) de centros de atención médica de urgencias en Nueva York pudo detectar en algunos sectores de la ciudad la diseminación de un brote de rotavirus y del virus de la influenza12.
Para nuestro estudio no se discuten la utilidad del software ni las características de los análisis estadísticos que permiten hacer el estudio de muchas enfermedades basados en el número de observaciones, sin necesidad de conocer la prevalencia de la enfermedad en la población. Sin embargo, existe una gran limitación para la implementación de la metodología de SaTScan en el seguimiento de brotes infecciosos con resistencia bacteriana en instituciones de salud, teniendo en cuenta que, si se genera un aumento en el número de aislamientos en varios servicios o localizaciones con fechas de aislamientos similares, los análisis de permutaciones espacio-tiempo podrían no reconocer la alerta. Esta dificultad presenta un riesgo, dado que los pacientes hospitalizados se mueven a través de los servicios conforme a su evolución clínica y no se restringen a una sola localización en la institución, lo que podría mostrarle una limitación al software. Adicionalmente, si un clon cambia rápidamente, la agrupación temporal podría fallar y resultar con falsos positivos o grupos de falsos negativos que podrían no ser registrados16.
No existe en la actualidad una prueba de oro para la detección de brotes, por lo que la aplicación de varias estrategias para la detección, como proponen Donna et al., aumenta la sensibilidad para la detección10. Es imprescindible que cada institución de salud cuente con un sistema de vigilancia en lo posible multimodal (activa y virtual), ya que la vigilancia e identificación de brotes en centros de atención en salud, sumada a las medidas de aislamiento, disminuye los costos en la atención y va en concordancia con la política de cuidado seguro del paciente18.
Existen algunas limitaciones en la realización de este estudio. Primero, se realizó en una sola institución; segundo, la vigilancia activa en dicho centro es una prioridad dentro de la política de cuidado seguro del paciente y esta puede no reflejar la situación general en el país, por lo que se requiere de un estudio multicéntrico para dar mayor validez.
Las alertas para los clústeres en la vigilancia virtual no representan la importancia biológica de los aislamientos, dadas las características del análisis estadístico, en el que se tiene en cuenta únicamente el número de casos.
Conclusión
La implementación del SaTScan-Whonet puede ser una herramienta de gran utilidad para los sistemas de vigilancia en la búsqueda retrospectiva de brotes. Sin embargo, la utilización de dicha herramienta de manera prospectiva no demostró ser superior en estos momentos a la vigilancia activa en cuanto a oportunidad en la detección para gérmenes con cierto grado de endemia como el ERV. Sin desconocer que la herramienta tiene un alto rendimiento diagnóstico y gran utilidad para detectar la diseminación de brotes de bacterias en instituciones de salud en pacientes críticos, esa diferencia de días en la detección representa un costo elevado en morbimortalidad, número de casos, días de estancia y aumento en el costo de las medidas de implementación para la contención de los brotes. De implementar dicha herramienta, su uso debe ser adicional a la vigilancia activa y no se propone reemplazarla por la virtual.
Responsabilidades éticas
Protección de personas y animales. Los autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datos. Los autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informado. Los autores declaran que en este artículo no aparecen datos de
pacientes.