Introducción
La tuberculosis (TB) en la actualidad es considerada como una de las principales causas de mortalidad a nivel mundial, generando cada vez más preocupación por el incremento en la prevalencia de variantes genéticas con resistencia a los fármacos antituberculosos1. La Organización Mundial de la Salud (OMS) reportó que, en el año 2015 cerca de 10,4 millones de personas contrajeron la enfermedad a nivel mundial, de las cuales 1,8 millones de personas fallecieron, y el 20% eran TB-MDR (multidrogorresistencia) y de estos un 9,7% de las personas con TB-MDR presentan XDR-TB2. Tratamientos subóptimos y la mala adherencia a las terapias de drogas antituberculosas contribuyen al aumento de la resistencia a fármacos; mientras que tratamientos inadecuados aumentan el riesgo de transmisión directa de Mycobacterium tuberculosis resistente a los medicamentos3-5. Ecuador se encuentra entre los países priorizados dada la gran carga de la enfermedad y el elevado índice de TB-MDR, reportándose un 4,6% de TB-MDR en 20156.
La isoniacida (INH: Isonicotinic Acid Hydrazide) una de las drogas de primera línea junto a la Rifampicina (RFM), son consideradas las drogas de mayor eficacia frente a una infección por Mycobacterium tuberculosis7. La INH actúa inhibiendo la biosíntesis de los ácidos micólicos de la pared celular, e ingresa en el Mycobacterium tuberculosis como un profármaco por difusión pasiva y es activada por la enzima catalasaperoxidasa codificada por katG, para generar radicales libres, que atacan a continuación múltiples objetivos en las células8. Se ha reportado en varios estudios que la resistencia a isoniacida está relacionada con mutaciones específicas en varios genes importantes de Mycobacterium tuberculosis, existiendo variantes genéticas tales como: katG (S315T, S315N, S315D, S315I, S315R, V61G, Q247H, A62T, G383A, C701G, G1388T), inhA (S94A, I21V, I21T, I47T, I194T, 3, 258, 190) y su región promotora (-15 C-T, -8 T-C, T-A ó T-G,-17 G-T), ahpC, nhd, kasA, oxyR, furA, fabG19-21, de igual manera, se ha podido mostrar que la aplicación de PCR-RFLP del gen katG permite la identificación eficiente de aislados resistentes a isoniacida pudiendo emplearse como una prueba rápida de detección22-25.
Aún existiendo evidencia de la aplicación de pruebas moleculares en búsqueda de la resistencia en Mycobacterium tuberculosis, en el Ecuador no existen mayores estudios relacionados a la identificación de mutaciones que interfieren en la acción de las drogas antituberculosas, ni las mutaciones presentes en los genes relacionados a la resistencia a fármacos, limitando la detección de la resistencia a análisis microbiológicos, o escasamente se emplea métodos genotípicos comerciales centrándose solo a la detección de resistencia a Rifampicina (GeneXpert MTB/RIF), excluyendo otras tecnologías que pueden detectar genotipicamente resistencias a otros medicamentos y que se emplean actualmente en varios países2. Por lo antes expuesto, el presente estudio propone determinar la mutación S315T del gen katG en aislados de Mycobacterium tuberculosis resistentes a isoniacida mediante la técnica PCR-RFLP utilizando las enzimas de restricción MspI y SatI, estrategia que permitiría caracterizar en menor tiempo la resistencia a Isoniacida comparado con el método de las proporciones, sugiriendo su aplicación como método rápido de tamizaje, complementario de las pruebas fenotípicas, que permitan mejorar la vigilancia epidemiológica de esta enfermedad a nivel país por el conocimiento de variantes genéticas circulantes, personalizando el tratamiento y disminuyendo los índices de TB-MDR.
Materiales y métodos
Un total de 68 aislados de Mycobacterium tuberculosis a los que se les había determinado previamente la resistencia mediante el método de las proporciones de Canetti, Rist y Grosset23 fueron obtenidos del banco de cepas del Centro de Referencia Nacional de Micobacterias del Instituto Nacional de Investigación en Salud Pública INSPI “Dr Leopoldo Izquieta Pérez”. El protocolo de investigación fue aprobado por el Comité de Ética de Investigación de Seres Humanos de la Universidad San Francisco de Quito bajo código 2017-018E.
Extracción de ADN
La extracción del ADN se realizó de acuerdo al protocolo descrito por van Soolingen et al.24, con ligeras modificaciones. A partir de 100 μL de aislado de Mycobacterium tuberculosis se adicionó 50 μL de lisozima (10mg/ml), incubando a 37oC por una hora, luego se adicionó 80 μL de SDS 10% y 5 μL de proteinasa K (10mg/ml), e incubó a 65oC por 10 minutos; seguido se adicionó 100 μL de NaCl 5M y 100 μL CTAB/NaCl, se mezcló e incubo a 65oC por 10 minutos; posteriormente se agregó 100 μL de Cloroformo: Alcohol Isoamilico (24:1), se agitó y centrifugó por 20 minutos a 14.000 r.p.m., se transfirió la fase acuosa y adicionó 60 μL de isopropanol al 90% y 2 μL de RNAsa, se mezcló e incubó a -20oC hasta el siguiente día. Se centrifugó 20 minutos a 14.000 r.p.m a 4oC. Se descartó el sobrenadante, se realizaron dos lavados con etanol al 70% centrifugando cada vez por 20 minutos a 14.000 r.p.m., a 4oC. El ADN se resuspendió en 200 μL de buffer TE 1X y se almacenó a -20oC hasta su utilización.
Amplificación del gen katG
Para determinar el polimorfismo de la región 315 del gen katG, dos productos fueron amplificados por reacción en cadena de la polimerasa (PCR) con los iniciadores katG500fUT1 5´ACCCAACTGAATGGAGCATGGGCGGAC3´ (1054 pb), katG904: 5-AGCTCGTATGGCACCGGAAC-3 (630 pb), respectivamente y katG1533rUT2: 5-ACGCACTTGACTTGTCTTCGTCGGGGTCGTGACCTCCCA-3 (modificado de Herrera-León et al.20) los cuales reconocen una región circundante del codón 315. Las amplificaciones se realizaron en un volumen de reacción final de 50μL conteniendo 1X de GoTaq Colorless Master Mix (Promega, Madison, WI 53711 USA) el cual contiene Taq DNA Polimerasa, 400μM dNTPs y 3mM MgCl2, 0,25 μM de cada iniciador y 150 ng de ADN extraído. Las amplificaciones fueron llevadas a cabo en un termociclador C1000 (Bio-Rad Laboratories, CA,USA) y las condiciones de PCR fueron desnaturalización inicial a 94°C por 4 min, seguido de 40 ciclos a 94°C por 45 s, hibridación a 65°C por 45 s, extensión a 72°C por 1 min y una extensión final a 72°C por 5 min. Los productos de PCR fueron analizados por electroforesis en gel de agarosa al 2% con SYBR safe bajo luz ultravioleta.
Resultados del PCR-RFLP del gen katG con MspI y SatI
Para la generación de los perfiles de digestión del fragmento amplificado del gen katG se utilizó el procedimiento descrito por Leung et al.19. Los perfiles teóricos fueron analizados previamente “in silico” utilizando el programa online Webcutter 2.0. Los productos amplificados fueron digeridos por separado con las enzimas de restricción MspI (Promega, Madison, WI 53711 USA) y SatI (Thermo Fisher Scientific, Waltham, MA USA). La enzima MspI reconoce la secuencia de corte C↑CGG, y SatI GC↑NGC, permitiendo detectar la mutación en la posición nucleotídica 945, codón 315 del gen katG (AGC →ACC).
Las reacciones de digestión fueron preparadas para un volumen final de 20 μL, conteniendo 3,0 U de enzima MspI, 1×del Buffer 10X (suplementado con la enzima), 2 μg de BSA acetilado, 10μg μL-1, y 2,0 U de enzima Sat I, 1X Buffer Anza™ 10X acorde a las instrucciones del proveedor. Las mezclas de reacciones fueron incubadas a 37◦C por 4 horas en un equipo de PCR C1000 (Bio-Rad Laboratories, CA, USA). Los productos fueron separados por electroforesis en gel de agarosa al 3% en buffer TAE 1X durante 3 horas a 100 voltios, y se visualizó los fragmentos obtenidos bajo luz ultravioleta y digitalizados en un foto documentador GelDoc XR (Bio-Rad Laboratories, CA, USA). Los resultados de los perfiles PCR-RFLP de cada enzima fueron interpretados acordes a estudios publicados por Leung et al.19 y Herrera-León et al.21.
Análisis de datos
Los datos fueron sistematizados en una hoja de cálculos de Excel diseñada para el estudio la cual incluyó datos epidemiológicos como edad, sexo, procedencia, tipo de muestra, fase de tratamiento, susceptibilidad a isoniacida proporcionados por el Centro de Referencia Nacional de Micobacterias del Instituto Nacional de Investigación en Salud Pública INSPI “Dr Leopoldo Izquieta Pérez”. Los análisis de sensibilidad (S), especificidad (E), valor predictivo positivo (VPP) y negativo (VPN), razón probabilística positiva (LR+) y negativa (LR-) fueron realizados mediante tablas cruzadas de 2 x 2 considerando como prueba estándar los resultados del método de las proporciones empleando el programa para Windows SPSS versión 22 (SPSS Inc, Chicago, IL, USA).
Resultados
De los 68 aislados, 69,1% (47) correspondió a aislados fenotípicamente resistentes y 30,9% (21) a aislados fenotípicamente sensibles de acuerdo al método de las proporciones. 77,9% (53) provenía de hombres y 22,1% (15) de mujeres. 48,0% eran de pacientes vírgenes al tratamiento (VT), 41,0% habían abandonado el tratamiento (AT) y 11,0% se encontraba en control del tratamiento (CT). La mayoría de los aislados provenían de Guayaquil (70,6%), seguidos por los obtenidos en Babahoyo (8,8%), Quito (7,4%), Portoviejo (5,9%), Machala (4,4%), Esmeraldas (1,5%) y Santa Elena (1,5%) (Figura 1).
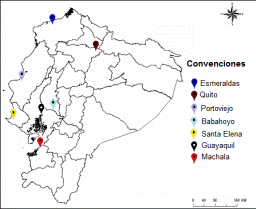
Figura 1 Distribución geográfica de los aislados resistentes y sensibles a Isoniacida (INH) de Mycobacterium tuberculosis. Convenciones presentan la procedencia de los aislados en cada una de las provincias: negro: Guayaquil; marrón: Quito; rojo: Machala; celeste: Babahoyo; amarillo: Santa Elena; lila claro: Portoviejo; azul oscuro: Esmeraldas.
Se observó mediante electroforesis que al emplear PCR-RFLP en el producto de 1054 pb con la enzima MspI y SatI los aislados resistentes y sensibles fenotípicamente presentaron similar perfil de restricción. A partir del producto de 630 pb se pudo evidenciar la presencia de un fragmento de aproximadamente 132 pb al utilizar MspI y 205 pb con SatI, en los aislados que presentaban la mutación en el codón 315. Los productos que presentaron bandas diferenciables fueron confirmados por secuenciación (datos no mostrados).
Al emplear las enzimas MspI y SatI se determinó que un 52,9% de los aislados (35 resistente y 1 sensible) presentó el codón mutado en la posición S315T; un 38,2% y 33,8% no presentaron la mutación; mientras un 8,8% y 13,2% presentaron un perfil no diferenciable para MspI y SatI, respectivamente.
Los aislados resistentes presentaron similar porcentaje del perfil con mutación (74,5%) con la enzimas MspI y SatI, mientras que en los aislados sensibles el 4,8% presentó el perfil con mutación para ambas enzimas (Tabla 1).
Tabla 1 Resultado comparativo de la susceptibilidad de Isoniacida (INH) en aislados de Mycobacterium tuberculosis analizados mediante métodos genotípicos y fenotípicos.
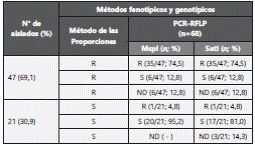
R, resistente; S, sensible; ND, patrones no diferenciado.
Al analizar la incidencia de los perfiles con mutación S315T y su relación con los tratamientos antifímicos recibidos, se puede observar que los aislados con mutación presentan igual porcentaje de incidencia entre los obtenidos de personas antes tratadas (AT) y vírgenes al tratamiento (VT) con 23,5% respectivamente, para ambas enzimas utilizadas. En los aislados resistentes fenotípicamente, aquellos que provienen de personas vírgenes al tratamiento, un 34,0% de estos aislados presentó la mutación, seguido de los antes tratados con 31,9%, para ambas enzimas. En el caso de los aislados sensibles fenotípicamente, un 4,8% de los aislados con la mutación provienen de personas antes tratados, para ambas enzimas utilizadas (Tabla 2).
Tabla 2 Resultado comparativo de la susceptibilidad de Isoniacida (INH) en aislados de Mycobacterium tuberculosis analizados mediante PCR-RFLP MspI y SatI entre los diferentes tratamientos recibidos.
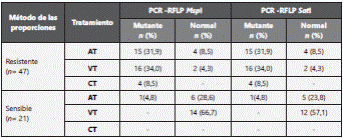
VT, virgen al tratamiento; AT, antes tratados; CT, con tratamiento; ND, no perfil determinado.
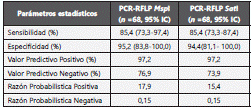
Para determinar la especificidad y sensibilidad de la PCRRFLP, el método de las proporciones fue utilizado como la prueba de referencia considerando que es el método estándar empleado en la actualidad para evaluar la resistencia en Ecuador. Se obtuvo 85,4% de sensibilidad y 95,2% de especificidad con MspI y 85,4% de sensibilidad y 94,4% de especificidad con SatI para la detección de la resistencia a Isoniacida en Mycobacterium tuberculosis (Tabla 3).
Discusión
La detección de la resistencia a drogas en pacientes con TB es necesaria para combatir la creciente prevalencia de TB-MDR.En los últimos años se han realizado estudios relacionados con la resistencia a medicamentos que han permitido el desarrollo de métodos fenotípicos y genotípicos25.
La secuenciación del genoma de Mycobacterium tuberculosis y la identificación de genes asociados a drogo-resistencia, ha permitido la aplicación de métodos de diagnóstico genotípicos tales como PCR-RFLP que ofrecen la posibilidad de identificar mutaciones relacionadas a resistencia26. Las mutaciones en los genes de Mycobacterium tuberculosis katG e inhA, han sido reportados como los responsables del 60 - 80% de los aislados resistentes a isoniacida27-30.
En nuestro estudio se observó que la PCR-RFLP tiene una aceptable capacidad resolutiva para la detección de resistencia a isoniacida en aislados de Mycobacterium tuberculosis, pero se debe tener presente que existen aislados que no presentan la mutación y tienen un patrón fenotípico resistente el cual podría estar relacionado a otros genes que confieren resistencia a isoniacida, de igual manera, dependiendo de las variantes geneticas de la mutación 315 que sean más frecuentes, la capacidad resolutiva dependerá de la enzima empleada (MspI: 95,2 (83,8-100,0), SatI: 94,4 (81,1- 100,0). Similares resultados han sido descritos por Herrera-León et al.,16, y Mathuria et al.,18, esto sería debido a que MspI solo detecta la sustitución de Ser→Thr (AGC→ACC), mientras que SatI puede detectar las otras sustituciones (AGC→ACC, ACA, ACT, ACG) que ocurren en el codón 315 que afectan los nucleótidos GC en esta posición.
Nuestros resultados mostraron que al emplear las enzimas MspI y SatI, el 74,5% de los aislados resistentes presentaron perfil con la mutación S315T, mientras que en los aislados sensibles el 4,8% la presentó. Estos resultados difieren de trabajos previos publicados en los cuales solo se reporta la presencia de perfiles de mutación en la posición S315T en aislados resistentes7,19,20,22,31, el 25,5% de aislados fenotípicamente resistentes que no presentaron la mutación estudiada, pudo deberse a la presencia de polimorfismos en regiones diferente a la posición S315T15-20en el gen katG u otros genes que aporten resistencia a Isoniacida 9-14 indiciendo a la sobre expresión que superen el poder inhibitorio de INH26,31; otra causa puede ser la presencia de mecanismos de resistencia diferente a las mutaciones tales como mecanismos de barrera (reducción de la permeabilidad32-33 y bomba de eflujo34-37). En cuanto a la presencia de un aislado sensible con la mutación S315T, posiblemente se deba a que el límite de la detección por PCR-RFLP empleada se encuentre cercano al 1% de resistentes que basado en el método de las proporciones pasa imperceptible y se considera como sensible, también podría deberse a una inadecuada identificación del perfil de resistencia mediante la prueba de las proporciones en el aislado sensible, y/o contaminación del aislado después de su conservación.
Al analizar los diferentes casos de pacientes con TB, se puedo observar que, 46,05% de muestras con el perfil mutado S315T mediante PCR-RFLP correspondió a pacientes antes tratados (AT) y vírgenes al tratamiento (VT), esto sugiere que posiblemente estos pacientes no cumplieron el tratamiento correctamente y/o abandonaron el mismo generando cepas resistentes a esta droga, adicionalmente, que existan paciente sin antecedentes de tratamiento (VT) y que presenten la mutación S315T sugiere que están circulando dentro de la población, aislados de M. tuberculosis que presentan resistencia previa a la isoniacida, haciendo poco efectiva la medicación recibida.
La sensibilidad general fue del 85,4% (IC95%:73,3-97,4), en contraste a otros estudios que mostraron 88,2%23, 91,5%38, 95,0%39, sin embargo, presenta un alto valor predictivo positivo (0,972) cuando se lo compara con el método de las proporciones, lo que aseguraría que un 97,2% de los aislados en los cuales se ha detectado mediante PCR-RFLP la mutación S315T tendrían la resistencia al medicamento, siendo un porcentaje útil para la toma de decisiones terapéuticas mientras se obtiene el resultado por cultivo microbiológico, de igual manera, es bien conocido que los valores predictivos de las pruebas son directamente dependientes de la prevalencia de la característica de la población particular44, lo cual al considerar la prevalencia de resistentes a Isoniacida de nuestro estudio (69,12%) impacta en la disminución del VPP del test empleado, lo que se mejoraría ampliando la población de estudio.
En relación a las muestras que presentaron patrones no diferenciables (8,8% para MspI y 13,2% para SatI ), es probable que se deba a causas como: la presencia de mecanismos de resistencia diferente a las mutaciones que afectan la permeabilidad de la membrana y bomba de eflujo, la calidad de la muestra, errores técnicos y presencia de inhibidores los cuales pueden afectar la calidad de amplificación y posterior generación del perfil de restricción.
El contar con buenas prácticas de laboratorio, que garanticen condiciones pre-analíticas adecuadas, es primordial con la finalidad de evitar contaminaciones y/o falsos positivos.
Una desventaja de cualquier prueba genotípica es la incapacidad de detectar mutaciones silenciosas (mutaciones que no conducen a ningún cambio de aminoácido). Mutaciones en otras regiones diferentes a la S315T dentro del mismo gen15-20 u otros genes parte del genoma de Mycobacterium tuberculosis pueden contribuir al desarrollo de la resistencia a isoniacida9-14, por lo cual realizar estudios con poblaciones mayores en las que se involucre la identificación de mutaciones de otros genes relacionados a la resistencia en isoniacida u otros medicamentos mejoraría la caracterización de las variantes genéticas presentes en los linajes de Mycobacterium tuberculosis, adicionalmente, al conocer las nuevas variantes presentes en los diferentes genes relacionados a la resistencia se aportaría al mejoramiento de los sistemas de detección comerciales actuales, considerando que los aislados presentan variaciones genéticas propias de su distribución geográfica13-18,40-43.
En conclusión, las pruebas moleculares no reemplazan los métodos convencionales, sin embargo, son un eficiente método de tamizaje cuando se combinan con datos clínicos y epidemiológicos, impactando significativamente en una diagnosis temprana de la resistencia, lo que contribuye a estrategias de control de la resistencia de TB en los países44. De igual manera, investigaciones dirigidas al estudio de la resistencia por secuenciamiento del genoma completo ayudarían en el futuro a dilucidar los mecanismos moleculares de resistencia a isoniacida u otros medicamentos en aislados de Mycobacterium tuberculosis circulantes en el Ecuador45-47. La PCR-RFLP resulta una alternativa confiable y rápida para el diagnóstico de la resistencia a isoniacida, mediante la detección de mutación S315T del gen katG, otros codones mutados del mismo gen y/o demás genes que producen resistencia, en comparación con el método convencional de las proporciones.