Introducción
En 2015 la Asamblea Mundial de la Salud aprobó el plan de acción global para enfrentar la resistencia a los antimicrobianos (RAM), que tiene entre sus objetivos fortalecer los conocimientos y la base de evidencia científica a través de la vigilancia y la investigación.1 Según la Organización Mundial de Salud (OMS), la resistencia a antibióticos es un problema en aumento; y reporta que la resistencia de enterobacterias al tratamiento con carbapenémicos se ha diseminado mundialmente; lo que ha llevado a que en algunos países este grupo de antibióticos ya no sean eficaces en más de la mitad de los pacientes con infecciones por Klebsiella pneumoniae.2 Por ello, las infecciones bacterianas a causa de enterobacterias resistentes a carbapenémicos, son de interés tanto desde el punto de vista clínico como en salud pública, debido a que actualmente los carbapenémicos hacen parte de los antibióticos de primera línea de tratamiento para infecciones severas por enterobacterias productoras de betalactamasas de espectro extendido (BLEES).3
Dada la relevancia del problema para la salud pública, la vigilancia de la RAM es una prioridad a nivel mundial, puesto que permite establecer las medidas de intervención de manera oportuna, así como orientar el tratamiento de los pacientes y definir estrategias para el control de la resistencia; por lo tanto el aumento de infecciones por microorganismos multirresistentes conlleva un reto en la rápida identificación de cepas resistentes, mediante métodos costo efectivos, como las pruebas fenotípicas; que permitan tomar decisiones seguras para los pacientes4 y que sean una alternativa frente al mayor costo que demandan las pruebas de biología molecular.
Por ello, Colombia en el Plan Decenal de Salud Pública (PDSP), propone entre sus metas la disminución de la morbilidad, mortalidad y discapacidad general por enfermedades transmisibles, y entre sus objetivos plantea la vigilancia de las mismas, así como reducir la carga de Infecciones Asociadas a la Atención en Salud (IAAS), y contener la resistencia a los antimicrobianos.5
En la Vigilancia de la RAM, el Instituto Nacional de Salud (INS) reportó que en Colombia en el primer semestre de 2016 en los servicios de hospitalización diferentes a cuidados intensivos (no UCI), hubo aumento en la resistencia a carbapenémicos para Klebsiella pneumoniae en comparación con 2015; mientras que para Escherichia coli y Enterobacter cloacae la resistencia aumentó tanto en los servicios no UCI como en los UCI. Con base en estos hallazgos, el INS recomienda realizar pruebas de tamizaje como el test de Hodge Modificado (MHT por su sigla en inglés), así como las pruebas de sinergismo con ácido etilen-diamino-tetra-acético (EDTA) y ácido fenil-borónico (PBA), para fortalecer la capacidad de los laboratorios en la detección de mecanismos de resistencia.6
Aunque las pruebas de biología molecular son los métodos de referencia para la detección de genes de resistencia, las pruebas fenótípicas para el tamizaje como el Test de Hodge Modificado (MHT)7 pueden ser útiles en lugares de recursos limitados, para la identificación de microorganismos productores de carbepenemasas (CPM), los cuales tienen capacidad de hidrolizar dichos antibióticos y casi todos los betalactámicos de uso clínico.8 Entre las pruebas fenotípicas, se encuentra el THT, que permite la detección de bacterias Gram negativas (Enterobacterias y/o bacilos Gram negativos no fermentadores - BGNNF) productoras de carbapenemasas, y se ha utilizado en Colombia, por su bajo costo y facilidad de implementación.9,10
La prueba THT, es una variante del Test de Hodge, propuesta para la confirmación de betalactamasas de tipo AmpC en enterobacterias, con 2 variantes según la forma de obtención de las enzimas: En el método directo se aplica la suspensión de bacterias mientras que en el método indirecto se hace previamente la extracción de las betalactamasas a partir de la cepa, mediante 5 a 7 ciclos de congelación-descongelación, seguido de centrifugación para obtener el sedimento.11,12 En ambos casos se puede aplicar la preparación de las colonias o el extracto enzimático sobre la superficie del agar o en una zona de incisión a partir del disco con antibiótico.13,14,15
Con el tiempo se han incorporado otras variaciones para la detección de CPM, tales como la utilización de un carbapenémico en lugar de Cefoxitina y utilización de otros métodos de lisis bacteriana de tipo mecánico o físico, tales como sonicación, ciclos de congelación - descongelación o el uso de buffer de lisis. Diferentes autores han utilizado la prueba THT para la detección de CPM en BGNNF, con resultados de sensibildad en Acinetobacter baumannii del 99,1% (IC 95% 94,6%-100,0%) en la detección de enzimas tipo OXA-23-like. Del mismo modo, en Pseudomonas aeruginosa se han descrito resultados de sensibilidad del 75,0 al 100,0% y de especificidad desde 71,4% a 98,4%, además en este germen se ha reportado buena concordancia en comparación con PCR múltiple, para la detección de blaVIM.10
El objetivo de este trabajo fue comparar la prueba THT para detección de carbapenemasas, con la prueba PCR múltiple (prueba de referencia), en cepas de bacterias Gram negativas, del programa de vigilancia de multirresistencia en un hospital de tercer nivel, con el propósito de aportar evidencia de la posible utilidad de este método como prueba de tamizaje, en la detección de resistencia a carbapenémicos.
Materiales y métodos
Se hizo un estudio descriptivo para comparar la pruebas THT y PCR múltiple en la detección de resistencia a antibióticos carbapenémicos, en bacterias Gram negativas de la colección de cepas del programa de vigilancia de multiresistencia bacteriana de un Hospital de tercel nivel de la cuidad de Medellín, aisladas de pacientes infectados entre 2012 y 2014. El estudio fue considerado sin riesgo, con base en lo establecido en la resolución 8430/1993 del Ministerio de salud.
Las cepas de bacterias fueron previamente caracterizadas por PCR múltiple, para detección de genes de resistencia bla KPC , bla NDM , bla VIM , bla OXA-48 , bla OXA-23 y bla OXA-51 ; según la técnica descrita por Poirel.16 En la reacción de amplificación se utilizó un termociclador C1000TouchTM (BIO-RAD Laboratories, Inc., Francia). Los productos de PCR fueron visualizados en gel de agarosa al 1,5%, teñidos con bromuro de etidio. El patrón de Bandas fue analizado con Fotodocumentador (BIO-RAD Laboratories, Inc., Francia).
Procedimientos: A partir de las cepas previamente caracterizadas por PCR múltiple, para detección de genes de resistencia a carbapenémicos (método de referencia), el personal del laboratorio sin conocimiento de dichos resultados y previamente estandarizados en la prueba THT (prueba a evaluar), procesó las cepas como se describe a continuación: Se preparó una suspensión 0,5 McFarland de la cepa indicadora (Escherichia coli ATCC® 25922™) y se sembró en una caja de Muller-Hinton Agar™(Becton Dickinson®), se adicionó un disco de Imipenem con concentración de 10μg (bioMérieux Clinical DiagnosticsMR - Marcy L´Etoile France) en el centro de la caja. A continuación se hizo una incisión de 5mm de profundidad a partir del disco, hasta el borde del medio de cultivo. De cada cepa en estudio, se tomaron 2 porciones de la colonia con asa calibrada de 10μL y re-suspendieron en un vial con 200 μL de buffer de lisis (B-PERTM Bacterial Protein Extraction Reagent -Thermo Scientific Rockford, Il USA), la mezcla se agitó con vortex por 1 minuto y luego se incubó a temperatura ambiente durante 30 minutos y se centrifugó a 13.000 g por cuatro minutos. A partir de la suspensión se dispensaron 15 a 20 μL en la incisión del agar y se incubó la caja a 37oC por 18 a 22 horas. Se utilizaron como controles de la prueba las siguientes cepas de referencia de la “American Type Culture Collection” (ATCC): Klebsiella pneumoniae ATCC® BAA-1705™ (expresa gen bla KPC), como control positivo, y Klebsiella pneumoniae subsp. pneumoniae ATCC® 700603™ (expresa bla SHV-18 no productora de carbapenemasas), como control negativo.
En la identificación de los aislamientos y las pruebas de sensibilidad a carbapenémicos, se utilizó el Sistema Automatizado Vitek® Compact 2 (bioMérieux Clinical DiagnosticsTM - Marcy L´Etoile France), según las recomendaciones del fabricante y acorde con estándares internacionales de control de calidad, del Instituto de estándares clínicos y de laboratorio - CLSI M100-S24.17 En la interpretación de los resultados para el método de referencia, se consideraron positivos los aislamientos con presencia de al menos uno de los genes de resistencia evaluados; mientras que en la prueba THT, los resultados fueron positivos cuando se observó deformación del halo de inhibición en la intersección con la cepa en estudio y la observación del halo de inhibición sin deformidad fue interpretado como negativo.
En el análsis de los datos se utilizaron el programa SPSS® Stadistics 19,0 para el analísis univariado, y el Programa EPIDAT 3.1 para cálculos de sensibilidad y especificidad, el índice de Youden y los valores predictivos.
Resultados
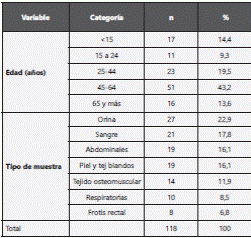
Las cepas evaluadas provenían de aislamientos de pacientes infectados o colonizados, identificados como sospechosos de resistencia a antibióticos betalactámicos del grupo de los carbapenémicos, con base en los criterios CLSI M100-S24. La mayoría de los aislamientos provenían de hombres (66,1%), y por tipo de ubicación del paciente al momento de toma de la muestra, el 44,8% estaban en servicios de hospitalización diferentes de UCI/UCE, el 28,4% en UCI/UCE y el 26,7% en urgencias. El tipo de muestra y la edad de los pacientes se presentan en la tabla 1.
Las cepas aisladas correspondieron a Klebsiella pneumoniae en 53 casos (44,9%); Pseudomonas aeruginosa, en 46 (39,0%); Enterobacter cloacae complex, en 12 (10,2%); Acinetobacter baumannii, en 5 (4,2%) y de otros aislamientos se presentaron 2 (1,7%).
En la evaluación de resultados de sensibilidad antibiótica de los aislamientos de bacterias portadoras de genes de resistencia para carbapenémicos, se observó sensibilidad reducida en comparación con aquellas sin producción de CPM.
En general los porcentajes de sensibilidad fueron más altos en los aislamientos sin CPM, excepto en Ceftazidima (CAZ), cuyo comportamiento fue igual en aquellos con y sin CPM. La mayor diferencia en sensibilidad se observó en trimetroprim sulfametoxazol, con alta sensibilidad en enterobacterias no productoras de CPM; mientras que la menor diferencia se encontró en colistina, figura 1 .
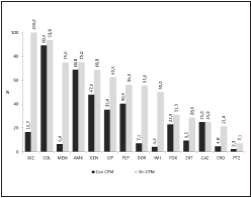
Figura 1 Distribución de la sensibilidad a los antibióticos, en los aislamientos de enterobacterias con y sin carbapenemasas (CPM) CPM: carbapenemasa, SZX: Trimetoprim Sulfametoxazol, COL: Colistina, MEM: Meropenem, AMK: Amikacina, GEN: Gentamicina, CIP: Ciprofloxacina, FEP:Cefepime, DOR: Doripenen, IMI: Imipenem, FOX: Cefoxitina, ERT: Ertapenen,CAZ: Ceftazidima, CRO: Ceftriaxona, PTZ: Piperacilina-Tazobactam
Las cepas de Pseudomonas aeruginosa productoras de CPM no mostraron sensibilidad a ninguno de los betalactámicos evaluados, además en las otras clases de antibióticos la sensibilidad fue baja; excepto en el caso de colistina, figura 2.
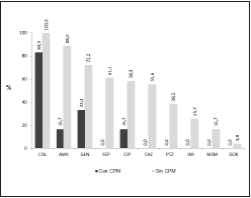
Figura 2 Distribución de la sensibilidad a los antibióticos, en los aislamientos de Pseudomonas aeruginosa con y sin carbapenemasas CPM: carbapenemasa, COL: Colistina, AMK: Amikacina, GEN: Gentamicina,FEP: Cefepime, CIP: Ciprofloxacina, CAZ: Ceftazidima, PTZ: Piperacilina- Tazobactam, IMI: Imipenen, MEM: Meropenen, DOR: Doripenen
En la prueba PCR múltiple, se observó que en 55 de los aislamientos (46,6%) no estaban presentes los genes de resistencia evaluados y en 63 (53,4%) si se detectaron; de estos últimos, la mayoría fue bla KPC (79,4%), seguido de bla VIM (11,1%) y bla OXA- 23/+OXA-51 (7,9%). Se presentó un caso de Pseudomonas aeruginosa con genes tipo bla KPC y bla VIM simultáneamente (1,5%). No se detectaron genes de resistencia tipo bla NDM ni bla OXA-48 .
Por otro lado, los valores de concentración inhibitoria mínima (MIC) más altos para imipenem y meropenem se encontraron en Klebsiella pneumoniae y en Enterobacter cloacae, en los portadores de genes bla KPC ; mientras que en los negativos para carbapenemasas se observaron MICs más bajos, tabla 2. De los 118 aislamientos, 63 fueron positivos para los genes de resistencia evaluados con la prueba PCR múltiple, de los cuales 62 también fueron positivos por la prueba THT y uno resultó negativo (falso negativo), que correspondía a un aislamiento de hemocultivo identificado como Pseudomonas aeruginosa con carbapenemasa tipo VIM. Además, 9 casos que fueron negativos con prueba de biología molecular, presentaron resultado positivo con la prueba THT (probables falsos positivos), todos enterobacterias (6 Enterobacter cloacae complex y 3 d e Klebsiella pneumoniae), figuras 3, 4 y tabla 2.
Tabla 2 Resultados de pruebas y concentración inhibitoria mínima (MIC) para Imipenem y Meropenem , según germen
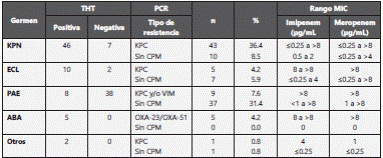
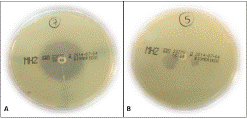
Figura 3 Hallazgos de la prueba Tridimensional en procedimientos de laboratorio en hospital de tercer nivel A) Prueba THT con Resultado negativo: sin producción de carbapenemasas, no se observa deformación del halo de inhibición B) Prueba THT con Resultado positivo: con producción de carbapenemasas, se observa deformación del halo de inhibición
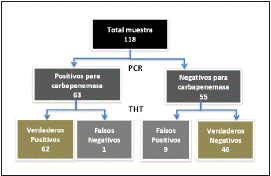
Figura 4 Resultados de la prueba de biología molecular (PCR múltiple) y la prueba Tridimensional (THT)
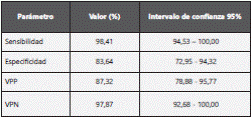
Los hallazgos de la prueba THT, en comparación con la prueba PCR múltiple, mostraron una sensibilidad de 98,41% (IC 95% 94,53% - 100,00%), y la especificidad fue del 83,64 % (IC 95 %: 72,95% - 94,32%). Adicionalmente el índice de validez fue 91,53 (IC 95 %: 86,08 - 96,97), el índice de Youden 0,82 (IC 95 %: 0,72 - 0,92) y el coeficiente de Kappa de Cohen 0,828 (IC 95 %: 0,727 - 0,929), tabla 3.
Discusión
El mecanismo de acción de los carbapenémicos es similar al de otros betalactámicos, con algunas particularidades que les permiten un espectro más amplio y mayor estabilidad a la actividad hidrolítica en bacterias Gram negativas. En enterobacterias la resistencia se debe con más frecuencia a la producción de carbapenemasas y en menor proporción a la modificación en la afinidad de la PBPs, disminución de la permeabilidad de la membrana externa y/o la presencia de bombas de expulsión.18 En Pseudomonas aeruginosa son más frecuentes los mecanismos intrínsecos por disminución de la permeabilidad, presencia de bombas de expulsión y sobre-expresión de enzimas modificadoras. La resistencia adquirida ocurre por carbapenemasas y/o betalactamasas de espectro extendido.19 En Acinetobacter baumannii presenta con mayor frecuenta betalactamasas de la Clase D (oxacilinasas), seguido de bombas de expulsión y modificación de porinas.20
Los métodos fenotípicos para detección de resistencia son los más utilizados de rutina en los laboratorios, entre ellos el test de Hodge modificado (THM), debido a su bajo costo y fácil montaje, además posee buena concordancia en enterobacterias, aunque presenta falsos positivos en aislamientos con enzimas tipo BLEES y AmpC; sin embargo no ha sido recomendado para BGNNF.17 En los últimos años la atención se ha centrado en las pruebas de hidrólisis de carbapenémicos tales como Carba NP y su versión comercial más reciente: Rapidec Carba NPTM (bioMérieux Clinical Diagnostics - Marcy L´Etoile France), cuyos resultados dependientes del cambio de color de la reacción, pueden generar dificultades en la interpretación, además se han reportado limitaciones para detección de enzimas tipo OXA y GES.21
El mayor potencial para pruebas como THT puede estar en la detección de carbapenemasas en BGNNF. Este tipo de bacterias representan una creciente amenaza en la región, en especial Acinetobacter baumannii y Pseudomonas aeruginosa.
En el primero el INS reportó una alta proporción de aislamientos no sensibles a carbapenémicos, de los cuales en 185 de 189 (98,4%) se detectaron genes de resistencia, identificados en su mayoría: 155 (82,8%) como productores de enzimas del grupo D de Ambler, y en alta proporción (75,5%) mediadas por genes tipo bla OXA-23+OXA-51 22 En este estudio el 100% de los aislamientos de Acinetobacter baumanii presentó resistencia a carbapenémicos, mediada por carbapenamasas tipo bla OXA-23+OXA-51 . Desde el 2000 se ha reportado la producción de oxacilinasas (enzimas del grupo D de Ambler) como principal responsable de la resistencia a los carbapenémicos en Acinetobacter baumanii, desafortunadamente una limitación de diferentes métodos fenotípicos es su baja sensibilidad para la detección de enzimas de este tipo,23 lo cual aumenta el riesgo de diseminación y presentación de brotes. En el presente estudio en método THT detectó todos los aislamientos de Acinetobacter baumanii que eran portadores de genes tipo bla OXA-23+OXA-51 .
La presencia de carbapenemasas en Colombia, en Pseudomonas aeruginosa, según los resultados de diferentes estudios se debe en primer lugar a genes tipo bla VIM . El INS reportó este mecanismo en 527 de 1274 (41,4%), seguido de KPC en 276 (21,7%) y en tercer lugar la combinación de VIM+KPC en 81 (6,4%) y en un estudio de Ocampo y col encontraron bla VIM en 14 aislamientos de Pseudomonas aeruginosa de los cuales 9 fueron positivas por THT, con 5 resultados falsos negativos. En este estudio, la prueba THT presentó concordancia del 0,91 (IC95% 0,78 a 1,00) frente a PCR múltiple en BGNNF al detectar 13 de 14 cepas productoras de CPM y un resultado falso negativo. En contraste en el estudio de Ocampo-Rios, realizado en aislamientos de Pseudomonas aeruginosa: de 20 productoras de CPM confirmadas por PCR múltiple, la prueba THT fue positiva en 15, con resultados de concordancia moderada. En otro estudio realizado en aislamientos clínicos provenientes de 14 hospitales de Colombia, Correa A y colaboradores, de un total de 151 aislamientos de Pseudomonas aeruginosa detectaron genes de resistencia bla KPC o bla VIM en 28, de los cuales 26 fueron positivos con la prueba THT, con resultados de Sensibilidad del 100% (IC95% 84-100), y Especificidad del 98,4 (IC95%: 93,8 a 99,7). 9,10,22
Aunque en la literatura se han evaluado diferentes métodos fenotípicos en comparación con la detección de genes de resistencia y técnicas de secuenciación (métodos moleculares) para BGN, tanto enterobacterias como BGNNF, con el propósito de formular recomendaciones; los resultados sugieren que no existe un método fenotípico con potencial para detectar todos los tipos de CPM documentadas hasta el momento.24,25
En el caso particular de las enterobacterias el reto más importante es el riesgo de presencia de carbapenemasas en cepas con MICs en los rangos de sensibilidad con los puntos de corte actuales; en este estudio en 6 de 42 (9,5%) los MICs para meropenem en Klebsiella pneumoniae fueron menores o iguales a 2, este tipo de germen podría pasar inadvertido en los programas de vigilancia y ser la fuente de diseminación, incluso si se contara con pruebas moleculares en formato de PCR múltiple, cuya limitación es la incapacidad de detectar bacterias con nuevas mutaciones.26 Este hallazgo también se ha observado en Escherichia coli productora de KPC y OXA-48, según lo reportado por investigadores del Laboratorio de Salud Pública de Ontario (Canadá), entre 14 a 20% de enterobacterias productoras de carbapenemasas se observan resultados con MIC clínico en el rango sensible.27
El panorama de la detección de resistencia en los bacilos Gram negativos no fermentadores presenta dificultades aún no resueltas, en primer lugar porque la mayoría de métodos fenotípicos disponibles no han sido recomendados en forma explícita en las guías CLSI para detección de carbapenemasas, en segundo lugar porque incluso métodos comerciales más recientes, tales como Rapidec-Carba pueden presentar falsos negativos en aislamientos de Acinetobacter baumannii con enzimas tipo OXA-48, OXA-23 y OXA-24,28 o el método Carba NP, en el que también se reportan falsos negativos en carbapenemasas tipo VIM y KPC en cepas con fenotipo hipermucoide de Pseudomonas aeruginosa.29 En este último germen se ha documentado también la amplia diversidad de mecanismos de resistencia diferentes a la producción de carbapenemasas como causa de resultados discrepantes frente a pruebas moleculares.
Lo antes expuesto, sugiere falta de acuerdo en los métodos de rutina a implementar en los laboratorios, especialmente en países como Colombia, en los que se dispone de pruebas moleculares para confirmación de carbapenemasas solo en laboratorios especializados, debido a su mayor costo y a las dificultades del sistema de salud vigente. En este contexto las pruebas fenotípicas con buen desempeño frente a los métodos moleculares, y con características como el costo menor e implementación simple, pueden ser una opción para la contención de la diseminación de resistencia.
En conclusión, dado que la prueba THT presentó buen desempeño para detección de CPM, tanto en enterobacterias como en BGNNF, en comparación con la prueba PCR múltiple; puede ser una alternativa en laboratorios de microbiología no especializados, dado su bajo costo y fácil implementación por la similitud técnica con el test de Hodge modificado utilizado de rutina por la mayoría de laboratorios, con la diferencia de la inoculación del extracto bacteriano en vez de la siembra directa de las colonias. En el presente estudio se preparó el extracto con el mismo buffer de extracción recomendado por CLSI para la prueba Carba NP, pero se pueden utilizar otros métodos, de tipo mecánico y/o físicos seguidos de centrifugación, utilizados en otras pruebas de rutina en microbiología. Los medios de cultivo, los materiales e instrumentos necesarios para esta prueba hacen parte de los elementos mínimos requeridos para implementación de Laboratorios de Microbiología en Colombia y como todo procedimiento de laboratorio, se requiere entrenamiento y estandarización de la técnica.