Introducción
Los métodos recomendados para establecer el diagnóstico etiológico de las infecciones del torrente sanguíneo (ITS) y de las infecciones del tracto urinario (ITU), son el hemocultivo y el urocultivo, respectivamente. La identificación del agente etiológico cultivado se basa en pruebas fenotípicas, incluyendo la coloración de Gram, el crecimiento en medios selectivos y las pruebas bioquímicas (manuales o automatizadas), las cuales pueden requerir hasta 48 horas en generar un resultado final de identificación1.
Con el fin de mejorar la oportunidad en los resultados, se han desarrollado sistemas de detección más rápidos basados en la identificación directa del microorganismo presente en las muestras clínicas, como la reacción en cadena de la polimerasa (PCR), la hibridación fluorescente in situ (FISH) y la espectrometría de masas MALDI-TOF. La elección de una u otra depende de los requerimientos particulares de cada laboratorio2,3,4.
El desarrollo de la tecnología MALDI-TOF (matrix-assisted laser desorption ionization time-of-flight, en español: Desorción/Ionización láser asistida por una matriz con detección de masas por tiempo de vuelo) permite el análisis de proteínas, principalmente de tipo ribosomal, para la identificación exacta de bacterias (incluyendo micobacterias), mohos y levaduras a través de la creación de un espectro de masas específico para cada microorganismo. Esta tecnología genera identificaciones rápidas y confiables, y tiene la capacidad de analizar un gran número de aislamientos simultáneamente. Además, tiene una aplicación futura en la detección de mecanismos de resistencia a los antimicrobianos4,5.
Recientemente, diferentes estudios han evaluado el rendimiento y la utilidad del MALDI-TOF MS para la identificación rápida de microorganismos directamente de muestras biológicas (orina, sangre y líquido cefalorraquídeo) utilizando diferentes métodos de preparación de la muestra. Para la identificación directa desde hemocultivos se han descrito métodos que incluyen pasos de centrifugaciones diferenciales6,7, el uso de detergentes como dodecilsulfato sódico (SDS) o saponina8,9, la utilización de tubos con gel separador de suero10, la filtración a través de membranas11 y la utilización de un kit comercial MALDI Sepsityper12,13,14,15; algunos autores adicionan el paso de extracción de proteínas con etanol/ácido fórmico o ácido trifluoroacético para obtener una mejor identificación10,1). En las orinas se han evaluado métodos de identificación directa por MALDI-TOF utilizando centrifugación diferencial16,17, pre-tratamiento con Tween-80 o SDS18,19 y extracción de proteínas20,21.
Este estudio tuvo por objetivo evaluar la utilidad de la identificación directa de microorganismos a partir de muestras de orina y hemocultivos utilizando MALDI- TOF MS mediante el análisis de concordancia en la identificación, y la comparación del tiempo promedio del resultado y los costos asociados a cada prueba.
Material y método
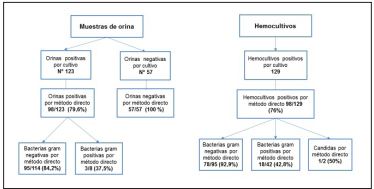
Se realizó un estudio descriptivo de febrero de 2017 a octubre de 2017, se seleccionaron a conveniencia 180 muestras de orina (123 positivas y 57 negativas) y 129 hemocultivos detectados como positivos por el sistema automatizado BacT/ALERT® 3D (bioMérieux, Marcy l’Etoile, France), provenientes de pacientes que consultaron o fueron hospitalizados en alguna de las dos sedes de la Clínica El Rosario en Medellín, y se analizaron directamente y a partir del cultivo por MALDI- TOF MS (Vitek® MS, bioMérieux).
Identificación a partir del cultivo utilizando MALDI-TOF MS
Orina Se realizó coloración de Gram, recuento de leucocitos y cultivo cuantitativo en agar sangre y agar MacConkey (bioMé-rieux, Marcy l’Etoile, France), incubados a 35 °C con CO2 al 5% durante 24 horas. Las colonias aisladas fueron analizadas por MALDI-TOF MS de acuerdo con las instrucciones del fabricante (Matriz VITEK® MS-CHCA, bioMérieux).
Hemocultivos
Los hemocultivos fueron previamente incubados en el sistema automatizado BacT/ALERT® 3D (bioMérieux, Marcy l’Etoile, France). Después de la señal de positividad, se les realizó coloración de Gram, y fueron sub-cultivados en agar chocolate (bioMérieux, Marcy l’Etoile, France) e incubados a 35 °C con CO2 al 5% durante 24 horas. Los cultivos positivos con colonias puras fueron identificados por MALDI-TOF MS.
Identificación directa por MALDI-TOF MS
Orina
4 mL de orina fueron centrifugados a 3400 rpm por 5 minutos. El sobrenadante fue transferido a otro tubo estéril y centrifugado a 3400 rpm por 15 minutos. El sobrenadante fue descartado y el sedimento fue lavado dos veces con 1 mL de agua desionizada, seguido de una agitación con vórtex y centrifugación a 3400 rpm por 5 minutos, de acuerdo a una adaptación del protocolo previamente descrito por L. Ferreira et al 16. Se transfirió 5 uL del sedimento a un pozo de la placa de montaje y se dejó secar para finalmente adicionar 1 uL de la solución de matriz CHCA (ácido α-ciano-4-hidroxicinámico), y se procesó en el MALDI-TOF.
Hemocultivos
Se utilizaron 2 tubos con gel separador de suero (BD Vacutainer®) y a cada uno se le adicionó 4 mL del hemocultivo positivo y se centrifugaron a 3400 rpm durante 10 minutos. El sobrenadante fue descartado, y se adicionó a cada tubo 500 uL de agua desionizada, se resuspendió el sedimento contenido en la superficie de la capa del gel, sin dañarlo. La suspensión obtenida de ambos tubos fue transferida a un tubo Eppendorf de 1,5 mL, y se centrifugó a 1000 rpm por 1 minuto. El sobrenadante fue transferido a otro tubo Eppendorf y centrifugado a 13000 rpm durante 1 minuto. Se realizaron dos lavados del sedimento obtenido con 1 mL de agua desionizada a 13000 rpm durante 1 minuto.
Extracción de proteínas
El sedimento fue resuspendido en 1 mL de Etanol al 70% y centrifugado a 13000 rpm por 2 minutos. Se descartó el sobrenadante y se adicionó 50 uL de ácido fórmico al 70%, se mezcló rápidamente con agitación en vórtex, y se adicionaron 50 uL de acetonitrilo, mezclando con vórtex seguido de centrifugación a 13000 rpm por 2 minutos, de acuerdo a una adaptación del protocolo de Stevenson et al 10. Se adicionó 2 uL del sobrenadante a un pozo de la placa de montaje, y se dejó secar, se adicionó 1 uL de la solución de matriz CHCA (ácido α-ciano-4- hidroxicinámico) y se procesó por MALDITOF MS.
Para el análisis de las muestras se utilizó el sistema VITEK® MS de bioMérieux, y la base de conocimientos V3.0. Las placas para el montaje en el MALDI-TOF se calibraron y controlaron utilizando la cepa de referencia Escherichia coli ATCC8739.
Análisis estadístico
Se calculó el porcentaje de identificación obtenida por el método directo comparado con la identificación obtenida desde el cultivo para muestras de orina y hemocultivos, respectivamente. El porcentaje de concordancia se calculó para los aislamientos positivos de acuerdo con la identificación obtenida a nivel de especie. Además, se determinó la concordancia de lo observado en la coloración de Gram con la identificación del microorganismo utilizando el método directo.
Análisis de tiempo y costos
El tiempo requerido para obtener un resultado de identificación de microorganismos por el método directo y por el cultivo, fue medido en horas y se calculó desde la fecha y hora en que se ingresó la muestra de orina al laboratorio, hasta el reporte de la identificación directa y a partir del cultivo, y en los hemocultivos positivos se calculó desde la fecha y hora en que el sistema BacT/ALERT® 3D detectó el hemocultivo como positivo hasta el reporte de la identificación directa y por cultivo.
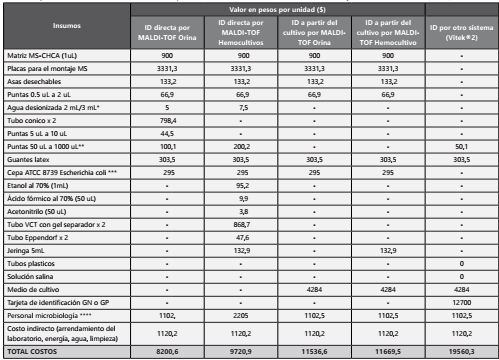
Para el análisis de costos asociados a la identificación de microorganismos por el método directo y a partir del cultivo utilizando MALDI-TOF MS y el sistema automatizado Vitek®2, se calcularon los costos directos (costo de insumos, reactivos, personal), y los costos indirectos (costo del arrendamiento del laboratorio y servicios generales: electricidad, agua, limpieza). El costo directo del equipo MALDI-TOF MS fue calculado de acuerdo con el valor del arrendamiento mensual (incluido el mantenimiento) sobre el promedio histórico del número de aislamientos identificados por mes en el laboratorio, sin incluir aislamientos no identificados, ni repeticiones. Los costos aplicados para este análisis se basaron en una estimación real de los costos en el laboratorio, y fueron analizados en pesos colombianos y en dólares (fecha del cálculo a diciembre de 2018).
Resultados
Orina
En las muestras de orina positivas, el 79,6% (98/123) de los aislamientos fueron identificados por el método directo. Analizando los urocultivos negativos, el 100% (57/57) fueron negativos por el método directo utilizando MALDI-TOF MS. Los aislamientos identificados por el método directo tuvieron una concordancia a nivel de especie del 100% al compararlo con los resultados obtenidos desde el cultivo. El 96,9% de las orinas positivas identificadas directamente tuvieron una coloración de Gram de orina sin centrifugar positiva y el 76,5% presentaron leucocituria significativa.
Del total de bacterias gram negativas el 84,2% fueron identificadas por el método directo, siendo las más comunes Escherichia coli, Klebsiella pneumoniae y Proteus mirabilis. No se identificaron por el método directo: 13 aislamientos de Escherichia coli, uno de Klebsiella pneumoniae, uno de Klebsiella oxytoca y un aislamiento de Pseudomonas aeruginosa. Del total de bacterias gram positivas el 37,5% se identificaron de forma directa por MALDI-TOF MS, una levadura (Candida tropicalis) identificada a partir del cultivo, no fue identificada por el método directo (Tabla 1). Del total de orinas positivas identificadas por cultivo, el 92,7% presentaban un recuento de más de 100.000 UFC/mL, de los cuales el 85,1% se identificaron directamente. De las muestras de orina que no se identificaron directamente, 8 presentaban un recuento en el cultivo de 1.000 a 10.000 UFC/mL y 17 orinas un recuento de más de 100.000 UFC/mL.
Tabla 1 Identificación directa de microorganismos en muestras de orina por MALDI-TOF comparada con la identificación a partir del cultivo puro por MALDI-TOF.
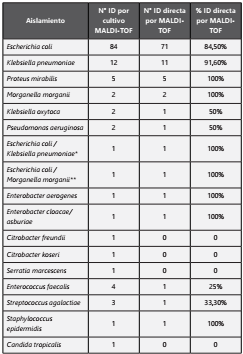
* Por el método directo se identificaron ambos microorganismos
** Por el método directo se identificó solo Escherichia coli
Hemocultivo
El 76% (98/129) de los aislamientos en hemocultivos positivos se identificaron utilizando el método directo. La concordancia de los aislamientos identificados por el método directo fue del 98,9% a nivel de especie al compararlo con la identificación obtenida desde el cultivo. Además, se obtuvo una concordancia del 100% al compararlo con la coloración de Gram.
El 92,9% de las bacterias gram negativas fueron identificadas al utilizar el MALDI- TOF MS directamente desde la botella de hemocultivo positivo, y las especies más comunes fueron Escherichia coli y Klebsiella pneumoniae. No se identificaron por el método directo cuatro aislamientos de Escherichia coli, uno de Pseudomonas aeruginosa y uno de Burkholderia arboris. En las bacterias gram positivas se obtuvo una identificación del 42,8% por el método directo, y se evidenció una menor identificación directa en los Staphylococcus coagulasa negativo y Streptococcus spp. Uno de los dos aislamientos de Candida albicans obtenidos desde el cultivo se identificó por el método directo (Tabla 2).
Tabla 2 Identificación directa de microorganismos en hemocultivos positivos por MALDI-TOF comparada con la identificación a partir del cultivo puro por MALDI-TOF
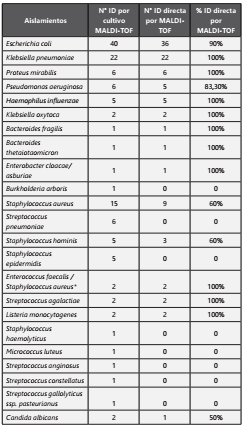
* Por el método directo se identificó solo Enterococcus faecalis
El tiempo de identificación directa fue evaluado en 70 muestras de orina y en 70 hemocultivos y se obtuvo tuvo una media de 6 horas con desviación estándar (DS±) de 2,2 en orinas y DS±2,5 en hemocultivos. Para la identificación por cultivo la media fue de 29 horas, DS±8,9 en orinas y DS±4,3 en hemocultivo. La diferencia en los tiempos para el reporte de resultados fue de 23 horas.
Sin considerar el valor de la adquisición o arrendamiento del equipo (MALDI-TOF), el costo total por aislamiento identificado de forma directa fue de $8.200 (2,58 USD) en muestras de orina y de $9.720 (3,06 USD) en hemocultivos positivos. El costo total de identificar un aislamiento a partir del cultivo por MALDI-TOF fue $11.536 (3,63 USD), y por el Vitek®2 $19.560 (6,16 USD) (Tabla 3).
Tabla 3 Costo total por aislamiento identificado mediante MALDI-TOF y otro método alternativo*
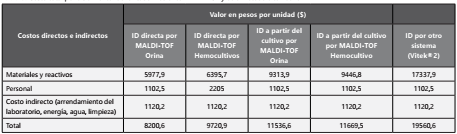
*No se tiene en cuenta el costo del equipo considerado como un costo directo variable de acuerdo con el número de identificaciones procesadas.
Al considerar el valor del arrendamiento mensual del equipo MALDI-TOF MS (bajo las condiciones que la compañía aplica en Colombia) y el número promedio de aislamientos identificados por mes (n=485) en el laboratorio donde se realizó el estudio, el costo de utilización del equipo le agrega $23.917 (7,54 USD) a cada identificación realizada.
Discusión
En este estudio se identificaron microorganismos en el 79,7% de las muestras de orina y en el 76,7% de los hemocultivos utilizando MALDI-TOF MS directamente en las muestras. La concordancia a nivel de especie de los aislamientos identifcados por el método directo al compararlo con las identificaciones obtenidas desde el cultivo fue del 100% en orinas y del 98,9% en hemocultivos. El porcentaje de identificación directa fue mayor para la identificación de bacterias gram negativas (84,2% en orinas y 92,9% en hemocultivos) comparado con la identificación de cocos gram positivos (37,5% en orinas y 42,8% en hemocultivos). Lo anterior concuerda con estudios previos que han obtenido porcentajes de identificación directa más altos para bacterias gram negativas comparado con las bacterias gram positivas. Sin embargo, algunos autores han obtenido porcentajes de identificación más altos de 64,8%22 y 67%1) para bacterias gram positivas en hemocultivos.
Una importante diferencia entre las bacterias gram negativas y las bacterias gram positivas, que podría estar relacionada con la dificultad que presenta esta metodología para la identificación de esta últimas, es la composición diferente y el grosor de la pared celular en las bacterias gram positivas. Para mejorar el desempeño de la identificación directa en bacterias gram positivas se ha recomendado la utilización de pre-tratamientos en la muestra que mejoren la disrupción de la pared y faciliten la extracción de proteínas, como por ejemplo la adición de ácido fluroacético (FA), buffer de lisis o el uso de detergentes como dodecilsulfato sódico (SDS) o saponina5,8,9,23.
En la identificación directa las muestras con crecimiento bacteriano mixto no son identificadas, o el MALDI-TOF MS solo genera un espectro de masas para un aislamiento, lo cual dificulta el diagnostico de coinfecciones. En este estudio dos muestras de orinas y un hemocultivo presentaron crecimiento mixto identificado a partir del cultivo, pero por identificación directa solo se identificó uno de los dos aislamientos que tenía la muestra.
La morfología observada en la coloración de Gram y la identificación directa del microorganismo mostraron una buena concordancia (96,9% en orinas y 100% en hemocultivos). Esto sugiere que la integración de ambos métodos puede proporcionar información complementaria que ayude a mejorar la oportunidad en el diagnóstico. Estos resultados son muy similares a los obtenidos por Burillo y colaboradores, que evaluaron un algoritmo secuencial que incluía la coloración de gram con la identificación directa por MALDI-TOF MS en muestras de orina y encontraron que el uso de ambos métodos aportó información anticipada en el 82,7% de los casos18.
Los resultados de este estudio confirman la utilidad del sistema MALDI-TOF MS para generar identificaciones más rápidas cuando se utiliza la identificación directa, demostrando una reducción promedio en el tiempo de identificación de 23 horas, comparado con el proceso de identificación a partir del cultivo. Estos datos son similares al estudio realizado por Lagacé-Wiens y colaboradores en el que obtuvieron una reducción del tiempo de respuesta de 34,3 horas al utilizar la identificación directa en una situación ideal en la que todos los microorganismos pudieron ser identificados y de 26,5 horas en una situación en la que se necesitó de métodos adicionales para su identificación final15. Lo anterior, evidencia que la incorporación del MALDI-TOF MS en la identificación directa de microorganismos en los laboratorios de microbiología mejora la oportunidad en el diagnóstico y tiene el potencial de generar un impacto positivo en el manejo adecuado del paciente.
En este estudio se observó una disminución en los costos por procedimiento de identificación cuando se utilizó la identificación directa desde la muestra, sin incluir la adquisición o el arrendamiento del equipo. El costo directo del equipo incrementó el costo agregándole $23.970 a cada aislamiento identificado. Este último costo es variable y depende del número de identificaciones que realice el laboratorio, a un mayor número de identificaciones disminuye el costo directo del equipo que se adiciona a cada identificación.
En un estudio realizado por Seng y colaboradores se hizo un análisis de costos, en el cual incluyeron el costo de consumibles específicos, el costo del salario del personal y el costo del equipo con una depreciación de 5 años sobre la base de 20,000 aislamientos analizados por año. Los resultados mostraron una reducción en los costos por aislamiento al utilizar MALDI-TOF MS como método de identificación comparado con otros sistemas de identificación (2,44 € vs 4,60-8,23 €)24. Tran y colaboradores, realizaron un análisis de costos que incluía el costo de reactivo, técnico, y costos de mantenimiento del equipo, y encontraron un ahorro de costos anual en el laboratorio del 51,7% al compararlo con la identificación tradicional25.
Actualmente se dispone de un kit comercial Sepsityper™ (Bruker Daltonics, Bremen, Germany) que es utilizado en el sistema MALDI Biotyper de Bruker Daltonics, sin embargo, tiene un alto costo debido a los reactivos que utiliza, por tanto, una gran alternativa es el uso de protocolos caseros, donde la principal ventaja es que estos protocolos se pueden adecuar según las necesidades de cada laboratorio, además, diferentes autores han documentado que los protocolos caseros tienen un mejor desempeño al compararlo con el kit comercial13,14.
Una de las limitaciones de este estudio es que no se evaluó el impacto clínico de la identificación directa por MALDI-TOF, siendo el impacto más importante del uso de MALDI TOF la disminución de los costos de atención de los pacientes al generar resultados más rápidos. Diferentes estudios han demostrado el impacto de su uso a nivel hospitalario, Huang y colaboradores, mostraron que la integración del MALDITOF MS con la intervención del equipo de administración antimicrobiano mejoró el tiempo de instauración de la terapia antibiótica efectiva (30,1 h frente a 20,4 h) y el tiempo de terapia óptima (90,3 h frente a 47,3 h) y se asoció con la disminución de la mortalidad, la disminución de la estancia en la unidad de cuidados intensivos y la disminución de bacteriemias recurrentes26. Similarmente, Pérez y colaboradores demostraron la disminución de la estancia hospitalaria y una reducción del 43% en costos hospitalarios al utilizar la tecnología MALDI-TOF MS27. En conclusión, la introducción del sistema MALDI-TOF MS en los laboratorios de microbiología genera identificaciones más rápidas cuando se utiliza directamente de muestras clínicas, lo cual mejora el tiempo en el diagnóstico etiológico por el laboratorio y tiene el potencial de generar un impacto positivo en el manejo adecuado del paciente. Sin embargo, tiene un bajo desempeño en la identificación directa de bacterias gram positivas, y por tanto es necesario evaluar la utilización de otros protocolos que mejoren la identificación directa. Su integración en el flujo de trabajo del laboratorio genera una disminución en los costos de los consumibles; sin embargo, la elección de esta tecnología conlleva costos importantes relacionados con la adquisición o arrendamiento del equipo. Este último costo es variable y depende del volumen de muestras identificadas en el laboratorio que puede influir en los costos finales de cada aislamiento identificado.