Introducción
Klebsiella pneumoniae es una de las enterobacterias más comúnmente asociada con infecciones adquiridas en la comunidad y en el hospital. Es el bacilo gram negativo clínicamente más importante caracterizado por su alto grado de multirresistencia. Es un patógeno oportunista ya que afecta, en su mayoría, a pacientes debilitados. Klebsiella pneumoniae es responsable de infecciones al tracto urinario, tracto respiratorio, infecciones intraabdominales, bacteriemia y neumonía1.
Los β-lactámicos son los antibióticos más utilizados en el tratamiento de infecciones causadas por bacterias, destacándose los carbapenémicos. Este grupo de antimicrobianos es comúnmente utilizado como último recurso en el tratamiento de infecciones en pacientes críticos. En los últimos años, las bacterias han desarrollado múltiples mecanismos de resistencia contra estos antimicrobianos, como: alteraciones en la permeabilidad de la membrana externa por pérdida de porinas, presencia de bombas de flujo, hiperproducción de β-lactamasas tipo Amp-C y producción de enzimas inactivadoras2.
En la producción enzimática, las enzimas más relevantes son las carbapenemasas tipo KPC, NDM, IMP, VIM y OXA. Estas enzimas son capaces de hidrolizar a los antibióticos carbapenémicos, inactivando su efecto contra los microorganismos. La producción de estas enzimas es considerada el principal mecanismo de resistencia contra este importante grupo de antibióticos y es producido principalmente por miembros de la familia Enterobacteriaceae3.
El reciente aumento de enterobacterias resistentes a carbapenémicos (ERC) ha dejado a los médicos con opciones limitadas de tratamiento para combatir infecciones bacterianas. La enzima KPC (Klebsiella pneumoniae carbapenemasa) producida principalmente por K. pneumoniae convierte a esta bacteria en uno de los patógenos ERC más ampliamente diseminado y asociado con altas tasas de morbilidad y mortalidad4.
La carbapenemasa KPC es una enzima presente en plásmidos y pertenece a la Clase A de la clasificación de Ambler. Fue reportada por primera vez en Estados Unidos en 1996 y en pocos años se diseminó por todo el mundo, observándose en varias especies de bacilos gram negativos2.
La diseminación de Klebsiella pneumoniae resistente a carbapenémicos es causado por clones epidémicos o por difusión horizontal de elementos genéticos móviles como plásmidos, transposones y secuencias de inserción (SI). Su rápida diseminación, la convierte en una verdadera amenaza a la salud pública5.
Materiales y Métodos
Población de estudio
De noviembre de 2012 a abril de 2013 fueron obtenidos 30 aislados no duplicados de Klebsiella pneumoniae resistentes a carbapenémicos. Los aislados clínicos fueron obtenidos de diferentes casas de salud de las ciudades de Quito y Guayaquil. Todos los aislados fueron determinados por identificación de la morfología de las colonias sobre placas de agar Eosina Azul de Metileno (BBLTM), pruebas bioquímicas y tarjetas VITEK® 2 GN ID (bioMérieux, Inc.). La identificación bacteriana por pruebas bioquímicas fue adicional a la identificación por el método automatizado VITEK 2. Los aislados bacterianos se conservan a temperatura ambiente y en congelación formando parte de la Colección Bacteriana-Quito Católica (CB-QCA). El comité de ética eximió a este estudio de revisión por cuanto se trabajó únicamente con aislados bacterianos.
Prueba de Susceptibilidad antimicrobiana
Las pruebas de susceptibilidad a los antibióticos de todos los aislados analizados empleó el sistema automatizado VITEK2® (bioMérieux, Inc.). Las tarjetas AST-N272 fueron empleadas en la determinación de la concentración inhibitoria mínima (CIM), siguiendo las instrucciones del fabricante.
Los antimicrobianos probados fueron: Ampicilina/Sulbactam (SAM); Piperacilina/Tazobactam (TZP); Cefoxitina (FOX); Ceftazidima (CAZ); Ceftriaxona (CRO); Cefepima (FEP); Doripenem (DOR); Ertapenem (ETP); Imipenem (IPM); Meropenem (MEM); Amicacina (AMK); Gentamicina (GEN); Ciprofloxacina (CIP); Tigeciclina (TGC) y Colistina (COL).
La susceptibilidad y puntos de corte de cada antibiótico probado fue interpretado con el manual del Instituto de Normas Clínicas y de Laboratorio (CLSI M100 2018) (6. Para la susceptibilidad antibiótica a la colistina y tigeciclina se emplearon los puntos de corte del Comité Europeo para Pruebas de Susceptibilidad Antimicrobiana (EUCAST) y de la Administración de Drogas y Alimentos de los Estados Unidos (FDA), respectivamente. Las cepas ATCC 25922 de Escherichia coli y ATCC 27853 de Pseudomonas aeruginosa se utilizaron como control interno de las pruebas de susceptibilidad.
Los aislados clínicos que mostraron una reducción en la susceptibilidad de alguno de los carbapenémico probado (Imipinem CIM 4 µg/mL, meropenem CIM 4 µg/mL y ertapenem CIM 2µg/mL) fueron considerados como posibles productores de carbapenemasas.
Detección de genes de resistencia a antibióticos
La reacción en cadena de la polimerasa (PCR) fue desarrollada para la detección de genes que codifican β-lactamasas de Espectro Extendido BLEE (blaCTX-M, blaSHV y blaTEM)7 y carbapenemasas (blaKPC, blaIMP, blaVIM, blaNDM and blaOXA)8,9,10.
Los productos de amplificación fueron enviados a purificar y secuenciar a la empresa Macrogen Inc., Corea del Sur. Los resultados del secuenciamiento fueron alineados y analizados en el programa Geneious Pro 5.6.4. y comparadas con secuencias obtenidas a partir de la base de datos del GenBank por Blast (http://www.ncbi.nlm.nhi.gov/blast).
(PFGE)
La preparación del ADN cromosomal de los aislados clínicos de Klebsiella pneumoniae siguió el protocolo estandarizado por la red internacional PulseNet de los Centros para el Control y Prevención de Enfermedades (CDC) (https://www.cdc.gov/pulsenet/pdf/ecoli-shigella-salmonella-pfge-protocol-508c.pdf). El protocolo empleado es el recomendado en la subtificación de E. coli 0157:H7, E. coli Non-0157, Salmonella, Shigella sonnei y Shigella flexneri. Para la normalización y comparación exacta de las imágenes del gel obtenido por Campo Pulsado se empleó como estándar la cepa sugerida por PulseNet Internacional: Salmonella serovar Braenderup (H9812). Los fragmentos obtenidos producto de la digestión con la enzima de restricción XbaI fueron comparados con la cepa estándar. Los patrones de restricción fueron analizados por el software BioNumerics Seven (Applied Math, Sint-Maten-Latem, Belgium).
Tipificación multilocus de secuencias (MLST)
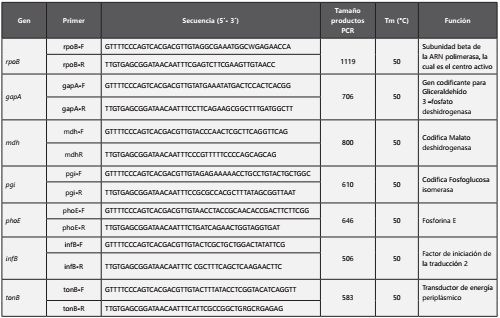
Los aislados fueron analizados siguiendo el protocolo descrito para la tipificación multilocus de secuencias (MLST) de K. pneumoniae de la página web (https://bigsdb.pasteur.fr/klebsiella/). Para el estudio fueron empleados siete genes conservados housekeeping: gapA, infB, mdh, pgi, phoE, rpoB y tonB. Las secuencias obtenidas fueron comparadas con los perfiles alélicos disponibles en la página web MLST de Klebsiella pneumoniae del Instituto Pasteur en Paris, Francia. El número alélico que confiere la secuencia tipo (ST) fue determinada para cada secuencia obtenida por PCR Tabla 1.
Resultados
Población de Estudio
Los 30 aislados bacterianos no duplicados de Klebsiella pneumoniae procedieron de: urocultivo (n=5), aspirado traqueal (n=5), secreción de herida (n=5), sonda vesical (n=4), hemocultivo (n=4), líquido peritoneal (n=2), punta de catéter (n=2), colección abdominal (n=1), trasplante renal (n=1) y secreción bronquial (n=1) Figura 1.
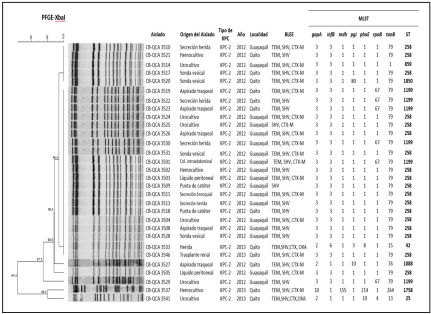
Figura 1 Dendrograma obtenido con el programa BioNumerics 7.0 donde se observa la relación clonal de cada uno de los aislados de Klebsiella pneumoniae productores de KPC-2, su origen, tipos de betalactamasas de espectro extendido (BLEE) y secuencia tipo (ST) obtenidas por tipificación multilocus de secuencias (MLST).
Pruebas de Susceptibilidad antimicrobiana
En la detección de la susceptibilidad antibiótica por el sistema automatizado VITEK2® el 100% de los aislados bacterianos fue resistente a los 3 carbapenémicos probados (ertapenem, meropem e imipenem). Todos los aislados mostraron resistencia a todos los antibióticos probados excepto a colistina y dos (6 %) aislados fueron resistentes a tigeciclina Tabla 2.
Tabla 2. Susceptibilidad antimicrobiana de aislados clínicos de Klebsiella pneumoniae.
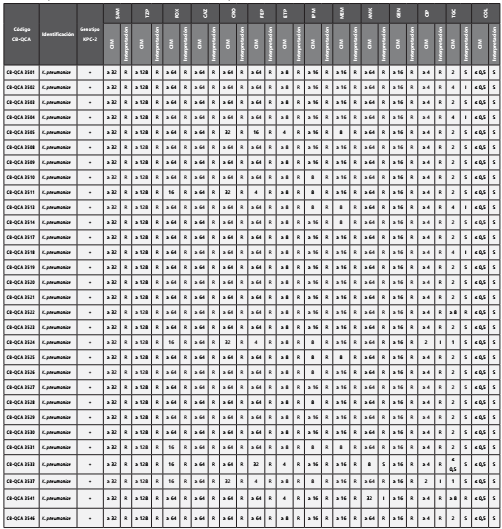
CB-QCA: Colección Bacteriana-Quito Católica; KPC: Klebsiella pneumoniae carbapenemasa; (+): positivo; (-): negativo; CIM: Concentración Inhibitoria Mínima; S: sensible; I: intermedio; R: Resistente; SAM: ampicilina/sulbactam; TZP: piperacilina/tazobactam; FOX: cefoxitina; CAZ: ceftazidima; CRO: ceftriaxona; FEP: cefepime; DOR: doripenem; ETP: ertapenem; IPM: imipenem; MEM: meropenem; AMK: amicacina; GEN: gentamicina; CIP: ciprofloxacina; TGC: tigeciclina; COL: colistina.
Detección de Genes de Resistencia a antibióticos
Todos los aislados bacterianos amplificaron el gen blaKPC. La variante alélica identificada por secuenciamiento fue KPC-2.
En la amplificación de genes que codifican carbapenemasas ningún aislado mostró la presencia de los genes blaIMP, blaVIM yblaNDM. En dos aislados (6 %, 2/30) se amplificó el gen blaOXA. La variante alélica fue OXA-1
De los 30 aislados analizados, (67 %, 20/30) fueron positivo para el gen blaCTX-M, (100%, 30/30) fueron positivos para el gen blaSHV y 28 (93 %, 28/30) fueron positivos para el gen blaTEM.
El 63% (19/30) de los aislados presentó amplificación simultánea de 3 genes que codifican para BLEE: bla(TEM/SHV/CTX); el 30% (9/30) de los aislados mostraron la amplificación simultánea de 2 genes codificantes de BLEE: bla(TEM/SHV); un aislado mostró la combinación de los genes BLEE bla(SHV/CTX) y un aislado mostró la presencia de un solo gen BLEE: bla(SHV) Figura 1.
Genotipificación por Electroforesis en Gel de Campo Pulsado (PFGE)
El dendrograma obtenido a partir de los 30 aislados clínicos de Klebsiella pneumoniae analizados mostró diseminación clonal. El dendrograma reveló 7 pulsotipos: PT1 (5 aislados), PT2 (20 aislados) y PT3 a PT7 (con un aislado cada uno). Los aislados que pertenecían al mismo grupo (PT2) clonal mostraron una relación entre ellos de más de 98.8% de similitud según el análisis obtenido con el Coeficiente de SorensenDice como indicador de similitud de los patrones de bandas Figura 1.
Tipificación multilocus de secuencias (MLST)
El estudio demostró que la secuencia tipo predominante fue ST258 encontrada en el 60% (18/30) de los aislados estudiados de Klebsiella pneumoniae productoras de KPC. La segunda secuencia tipo más abundantes fue ST1199 presente en el 20% (6/30) de los aislados. Se determinaron secuencias únicas ST1758, ST25, ST859, ST1088, ST42 y ST1850 Figura 1.
Discusión
Klebsiella pneumoniae es la principal enterobacteria productora de la enzima carbapenemasa KPC, el mecanismo más importante de resistencia a los carbapenémicos. El presente estudio trabajó con 30 aislados resistentes a carbapenémicos y los resultados revelaron que el mayor número de aislados resistentes fueron obtenidos a partir de muestras de urocultivo, aspirado traqueal y secreción de herida. Esto indicaría una mayor exposición a factores de riesgo de contraer bacterias resistentes como Klebsiella pneumoniae. A este patógeno se le atribuye una amplia gama de infecciones en el tracto urinario, respiratorio, en la sangre, asociadas a la atención en salud3.
Los aislamientos de Klebsiella pneumoniae resistentes a carbapenémicos por la producción de la enzima KPC están relacionados con altas tasas de morbilidad y mortalidad. Estudios demostraron que los aislados de K. pneumoniae que adquieren genes que codifican la producción de estas enzimas aumentaron la gravedad de las infecciones al complicar los tratamientos11.
La variante KPC-2 fue el alelo predominante del gen blaKPC.
Esta variante estuvo presente en los 30 aislados analizados en el estudio. Su importante presencia se puede explicar por la facilidad de transmisión intraespecífica que se presenta entre los miembros de la especie Klebsiella pneumoniae12. Los 20 aislados agrupados en el pulsotipo PT2, que se observa en el dendrograma obtenido por electroforesis en gel de campo pulsado, pusieron en evidencia diseminación clonal. Los aislados involucrados provenían de dos ciudades del país: Quito y Guayaquil, con la participación de diferentes instituciones de salud. Para la ciudad de Guayaquil, los aislados procedían de dos instituciones y para la ciudad de Quito de tres instituciones. A pesar de la separación geográfica que existe entre las dos ciudades de estudio, la dispersión del clonal fue eficiente.
En países como: Colombia, Brasil, Estados Unidos, Argentina, Puerto Rico, Grecia, Israel y China, KPC es considerada endémica, gracias a su habilidad de propagación. En Argentina, la emergencia de KPC que registró este país se debió a dos patrones importantes de dispersión de resistencia: la propagación del gen blaKPC a través del plásmido pK048 y la dispersión clonal de la secuencia tipo ST25813
La incidencia de enterobacterias resistentes a carbapenémicos (ERC) ha aumentado notablemente en todo el mundo durante la última década. Durante el año 2008 países como Colombia, Argentina, Uruguay y Brasil experimentaron incrementos considerables de microorganismos KPC positivos, alcanzando porcentajes de hasta 800%14. Los reportes demostraron que la variante enzimática producida por estos aislados fue KPC-2. La variante también es común en otros países de Latinoamérica, diseminándose entre aislamientos de la misma especie y especies diferentes. KPC ha sido identificada en diversas especies bacterianas como Enterobacter spp., Escherichi coli, Salmonella spp., Acinetobacter spp. y Pseduomonas spp. (22.
KPC-3 es la segunda variante mayormente reportada y se asocia también con aislados resistentes a carbapenémicos15. La variante KPC-3 y KPC-2 difieren en un solo aminoácido (H272Y). Así, el reporte de esta variante en estudios similares realizados en Latinoamérica puede deberse a eventos mutacionales y propagación clonal16.
A nivel mundial, la propagación de la carbapenemasa KPC, se explica a través de la asociación del gen blaKPC-2 a un elemento genético importante: el transposón Tn4401. El transposón Tn4401 se relaciona con una amplia variedad de plásmidos que varían en tamaño, naturaleza y estructura. En la mayoría de los casos, los plásmidos son autotransferibles, principalmente a miembros de la especie Escherichia coli2, contribuyendo, aún más, con la propagación de estas enzimas. En la actualidad, estas plataformas génicas se identifican en numerosas enterobacterias, incluyendo el género Salmonella y bacilos gram negativos no fermentadores del género Pseudomonas17. Los plásmidos identificados y asociados con el gen blaKPC y el transposón Tn4401 llevan a su vez genes que confieren resistencia a penicilinas, cefalosporinas, monobactámicos, inhibidores de betalactamasas, aminoglucósidos y fluoroquinolonas18.
El éxito epidemiológico del ST258 ha sido relacionado con factores cromosómicos y plasmídicos, más allá de la resistencia a antibióticos. Estos factores estarían incrementando la aptitud de la cepa bacteriana y proporcionando una ventaja que favorece su prevalencia. La propagación de K. pneumoniae productora de KPC se explicaría por su asociación a un tipo de secuencia multilocus ST258 y sus variantes relacionadas. Estos aislados están distribuidos alrededor del mundo por diseminación de clones, donde ST258 ha alcanzado un estado epidémico. A pesar de su baja virulencia, las altas tasas de mortalidad que exhibe este clon se relacionan con fracaso terapéutico debido a los altos niveles de resistencia a múltiples fármacos19.
La secuencia tipo ST258 circula en más de 20 países del mundo y es miembro del grupo clonal GC258. Este grupo clonal alberga varios tipos de secuencias vinculados con brotes. Esto sugiere que estas cepas pueden compartir características genéticas que las predispone a la patogenicidad o a un incremento en la transmisibilidad. Se presume que el surgimiento de este patógeno emergente es el resultado de eventos de recombinación homóloga que derivó en el clon híbrido ST258 de Klebsiella pneumoniae compuesto de un 80% del genoma de ST11 y 20% del genoma de ST44220.
La segunda secuencia tipo reportada con mayor frecuencia en la población de estudio fue ST1199. Estudios afirman que ST512 y ST1199 son el resultado de mutaciones puntuales de ST258. ST1199 forma parte del linaje de ST258 y se encuentran estrechamente relacionadas formando parte de un mismo grupo ancestral20.
Un solo aislado presentó el ST25. Esta secuencia tipo se caracteriza por formar parte de la población de ST que cuentan con aislados bacterianos hipervirulentos e hipermucosos y se diferencia de las poblaciones conformadas por ST11 y ST258 asociadas a multirresistencia. Otros miembros de la población hipervirulenta comprenden ST23, ST86, ST65 y ST375. El estudio no reportó la presencia de este tipo de secuencias, como ST23, cuyos aislados exhiben fenotipos hipermucoviscosos19.
Informes recientes revelaron la convergencia de virulencia y resistencia antimicrobiana. Este fenómeno da como resultado de transferencia de genes mediada por plásmidos y la aparición de cepas hipervirulentas y multirresistentes de Klebsiella pneumoniae. Estas bacterias son identificadas como ST11 y han sido reportadas en China, lo que plantea un peligro en la aparición de nuevos organismos híbridos21.
El estudio también reportó cinco casos únicos de ST: 1850, 1088, 42, 1758 y 859. Estudios relacionados con estas secuencias tipo no fueron encontrados.
Las opciones en el tratamiento de infecciones causadas por microorganismos ERC son limitadas porque la resistencia a múltiples familias de antibióticos es mediada por una variedad de mecanismos de resistencia. En el Ecuador, el tratamiento de microorganismos resistentes como K. pneumoniae se basa en aspectos como: el tipo de resistencia que presenta la bacteria, la aplicación de dosis máximas de cada fármaco utilizado y el tiempo requerido de tratamiento (10 - 14 días), según la evolución clínica del paciente. En el Ecuador, la terapia antibiótica combinada es empleada con los siguientes fármacos: carbapenémicos (imipenem, meropenem), polimixinas (colistina), glicilciclinas (tigeciclina), aminoglucósidos (amikacina), fosfomicina y/o rifampicina23. La combinación de tratamientos entre polimixina, glicilciclinas y carbapenémicos es sugerida por autores, gracias a los resultaron favorables observados contra aislados resistentes13,28.
Las polimixinas son importantes agentes antimicrobianos para el tratamiento de infecciones por bacterias Gram negativas resistentes. La microdilución es el método de referencia empleado en la determinación de la susceptibilidad antibiótica. Métodos automatizados como Vitek-2 han demostrado errores importantes en la obtención de la susceptibilidad de este antibiótico. Sin embargo, para análisis de aislados de Klebsiella pneumoniae, Escherichia coli y otras enterobacterias, el sistema Vitek-2 mostró un mejor rendimiento en la obtención de la CIM comparado con los resultados obtenidos para bacilos Gram negativos no fermentadores como Pseduomonas aeruginosa. Estudios sugieren que todos los aislados resistentes a colistina por Vitek 2 deben ser confirmados a través del método de referencia: por microdilución. La precisión en la obtención de la susceptibilidad de los métodos automatizados aún no está clara24.
Ante la prevalencia de patógenos productores de carbapenemasas, el uso de tratamientos alternativos como: ceftolozano/tazobactam, ceftazidima/avibactam o meropenem/vaborbactam promueven el ahorro de carbapenémicos empleados en el tratamiento de infecciones resistentes. Estas nuevas alternativas terapéuticas no han sido implementadas en el país25. Estos medicamentos han demostrado una eficiente acción contra la síntesis de la pared celular bacteriana y una alta estabilidad contra la hidrólisis producida por betalactamasas26.
A pesar de la autorización de uso y reconocida eficacia de estos antibióticos en otros países de la región, en Ecuador, el posicionamiento terapéutico de estos nuevos antimicrobianos es nulo27. La terapia combinada sigue siendo el principal sistema de control de infecciones resistentes.
La producción de carbapenemasas puede ser identificada a través de pruebas fenotípicas implementadas en los laboratorios de microbiología. Debido a la versatilidad de estas enzimas, hasta la fecha, no existe una sola prueba fenotípica con la suficiente capacidad de detección y diferenciación de todos los tipos de enzimas que existen. En la actualidad, el uso de plataformas moleculares basados en reacción en cadena de la polimerasa constituye la técnica más confiable de detección de este tipo de enzimas. La elevada especificidad y sensibilidad, y el poco tiempo requerido, hacen de las técnicas moleculares las más apropiadas28,29.
El estudio del funcionamiento de los factores de resistencia y su diseminación permitirá una mejor comprensión de la epidemiología molecular de Klebsiella pneumoniae productora de KPC. Esto ayudará en la implementación de medidas efectivas para su control y prevención. Los resultados obtenidos demuestran la importancia de implementar y combinar estudios clínicos, moleculares y epidemiológicos que permitan entender de manera precisa la distribución de la resistencia entre las bacterias.
La salud pública requiere herramientas de detección rápidas, completas y eficaces que ayuden a rastrear y generar información oportuna para el control de las infecciones. Un adecuado conocimiento de la epidemiología molecular y los principales mecanismos de resistencia que exhiben las bacterias proporcionan adecuadas guías en el tratamiento de las infecciones.