Introducción
Las especies de Candida spp. se consideran los hongos más encontrados en entornos hospitalarios y son uno de los agentes etiológicos más frecuentes de infecciones fúngicas invasivas1. Candida auris es uno de los patógenos emergentes más destacados y se ha convertido en un importante hongo oportunista relacionado con infecciones asociadas a la atención en salud2. Generalmente, esta levadura se caracteriza por su perfil de resistencia a múltiples fármacos, fácil transmisibilidad, persistencia en diferentes superficies y sobre todo discrepancias en la identificación de este hongo con varias plataformas diagnósticas empleadas en los laboratorios3,4.
La identificación adecuada y rápida de C. auris es crítica en el tratamiento del paciente y en la implementación oportuna de medidas de salud pública para controlar la propagación de la infección5. Existen sistemas de identificación para levaduras, tanto estándares como automatizados que incluyen paneles de asimilación de carbohidratos y otros compuestos, pero algunas bases de datos de estos sistemas aún no incluyen el perfil de C. auris, de manera que varias plataformas son incapaces de diferenciarla de otras especies de levadura, resultando en una confusa identificación.
Los análisis genéticos han mostrado una estrecha relación entre C. auris y el complejo C. haemulonii, principalmente6. Por lo anterior, esta levadura puede ser identificada erróneamente como C. haemulonii, C. duobushaemulonii y otras especies de Candida spp. como C. catenulata, C. famata, C. guilliermondii, C. lusitaniae y C. parapsilosis, entre otras7.
Algunos métodos de identificación, comúnmente encontrados en los laboratorios de diagnóstico, incluyen VITEK 2, Phoenix BD y MicroScan. Por ejemplo, VITEK 2, recientemente incluyó a C. auris en su base de datos, junto con otros tres taxones: C. haemulonii var. vulnera, C. doubushaemulonii y Cryptococcus gattii (versión 8.01)8.
En la actualidad, las técnicas más eficientes son las basadas en espectrometría de masas MALDI-TOF MS y métodos moleculares como PCR (amplificación de la región ITS1, ITS2) o secuenciación de la región D1/D2 o ITS9.
Los Centros para el Control y la Prevención de Enfermedades (CDC) han recibido reportes de la levadura emergente en más de cuatro continentes desde el año 2009. Dada la ocurrencia de infección por C. auris en una gran variedad de países, el CDC ha emitido alertas para la vigilancia de casos10 .
Teniendo en cuenta la importancia a nivel de salud pública sobre la circulación de C. auris en Colombia, se generó la alerta nacional mediante la Circular Externa 0025 de 2017 donde se emitieron los criterios de envío de aislamientos presuntivos de C. auris al Grupo de Microbiología del Instituto Nacional de Salud (INS), recuperados en los laboratorios clínicos de los establecimientos hospitalarios y en los laboratorios de los centros de investigación que procesan muestras procedentes de Unidades de Cuidados Intensivos, con el fin de confirmar la identificación11.
En Colombia, la disponibilidad de tecnologías robustas que permitan una correcta identificación de C. auris aún no es muy amplia. Se tienen implementados algunos sistemas de diagnóstico, sin embargo, éstos no identifican correctamente todos los aislamientos de C. auris, de manera que se restringe el diagnóstico preciso y rápido del patógeno, el tratamiento oportuno del paciente y la instauración de medidas de control adecuadas.
Por tanto, en este estudio se compararon los resultados de identificación obtenidos por diferentes sistemas de identificación de aislamientos de C. auris provenientes de 22 departamentos de Colombia desde el año 2016, fecha en la cual se inició la vigilancia de este patógeno en el país.
Materiales y métodos
Se evaluaron los resultados emitidos por los sistemas de identificación MicroScan, Phoenix BD, VITEK 2 y MALDI-TOF de acuerdo con las identificaciones de 843 aislamientos clínicos sospechosos de C. auris remitidos al Grupo de Microbiología del INS a través de la vigilancia nacional por laboratorio. Los aislamientos provenían de 114 laboratorios clínicos y establecimientos hospitalarios, distribuidos en 22 departamentos de Colombia recolectados desde el 2016 al 2019. Se tuvo en cuenta que la casa comercial realizó la actualización del sistema VITEK 2 a la versión 8.01 desde septiembre de 2018 en los laboratorios.
Cada aislamiento se confirmó mediante MALDI-TOF MS Biotyper (Bruker Daltonics) MBT versión 4.1.80 y/o PCR en el INS, métodos considerados como el “gold standard” para la identificación de C. auris.
Procesamiento de los aislamientos para la confirmación mediante MALDI-TOF MS Biotyper (Bruker Daltonics)
El procedimiento de extracción de proteínas con ácido fórmico en tubo de 1.5 ml (Eppendorf) se siguió de acuerdo con las instrucciones del fabricante para la identificación de levaduras. Se transfirió 1 µl del sobrenadante a una placa de acero y se cubrió con 1 µl de matriz. Una vez que se secó al aire, la placa se colocó en el instrumento y se analizó. La identificación de la levadura se logró mediante el análisis de los espectros con los de la base de datos MALDI-TOF MS. Los picos de estos espectros se compararon con el patrón característico de la especie o género de levadura, lo que condujo a la identificación12.
Procesamiento de los aislamientos para la confirmación mediante PCR convencional
La extracción de ADN de los aislamientos fúngicos se realizó mediante el protocolo de extracción de ADN para cultivos fúngicos por choque térmico13.
Se emplearon los iniciadores específicos para C. auris: CauF 5´-CGCACATTGCGCCTTGGGGTA-3´ y CauR 5´-GTAGTCCTACCTGATTTGAGGCGAC-3´. Para la realización de esta PCR, se siguió el protocolo propuesto por Kordalewska M y colaboradores en 201714.
Resultados
De los 843 aislamientos clínicos remitidos al INS, 689 (81,7%) fueron confirmados como C. auris mediante MALDI- TOF MS o PCR; solamente el 0,6% (4/689) fueron confirmados por los dos métodos y la identificación fue 100% concordante entre éstos. Por otra parte, los aislamientos restantes (154/843 aislamientos 18,3%) fueron identificados como otras especies de Candida spp u otras levaduras.
En general, las identificaciones discrepantes enviadas por los laboratorios principalmente fueron C. haemulonii, C. guilliermondii, C. albicans y otras levaduras como Rhodotorula spp y especies de Candida spp. sin identificación de especie (Figura 1); éstas representaron el 57,6% (397/689) de las cepas confirmadas como C. auris en el INS.
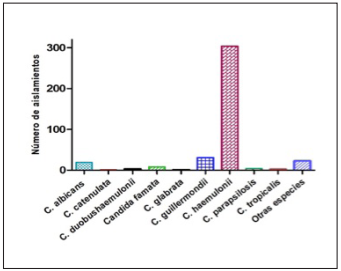
Figura 1 Distribución de la frecuencia de especies enviadas para análisis al laboratorio del INS (Bogota).
MicroScan identificó C. auris principalmente como C. haemulonii, C. guilliermondii, C. albicans y C. famata; Phoenix BD, VITEK 2 y MALDI-TOF MS identificó C. auris como C. haemulonii (Figura 2). Resultados discrepantes como C. haemulonii se obtuvieron en su mayoría con el sistema Phoenix BD 89,6% (26/29).
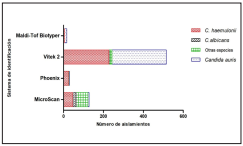
Figura 2 Comparación de los sistemas utilizados por los laboratorios de los establecimientos hospitalarios con respecto a la identificación de la levadura Candida auris (todos los aislamientos mostrados en la figura fueron confirmados como C. auris en el INS). El sistema MALDI-TOF MS de BioMérieux se excluyó de la gráfica debido a que presentó solo un dato y este no fue representativo para incluirlo en la gráfica.
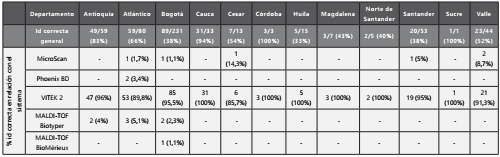
Por otro lado, las identificaciones correctas con el sistema VITEK 2 representaron el 29,3% (77/263 aislamientos identificados con VITEK 7.01) y 77,4% (195/252 aislamientos identificados con VITEK 8.01) (Figura 3).
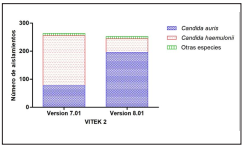
Figura 3 Comparación de las dos versiones de VITEK 2 en relación con el número de aislamientos identificados correctamente como Candida auris y la identificación discrepante Candida haemulonii y otras especies de levaduras (todos los aislamientos mostrados en la figura fueron confirmados como C. auris en el INS).
El 48,2% de los laboratorios (55/114 laboratorios) obtuvieron identificaciones correctas de C. auris en la mayoría con VITEK 2 y MALDI-TOF. En este estudio las identificaciones correctas enviadas por los laboratorios en general representaron el 42,4% (292/689).
Por otro lado, se observaron 7 aislamientos falsos positivos procesados con VITEK 2 en los laboratorios de los establecimientos hospitalarios, enviados como C. auris y al confirmar por MALDI-TOF y/o PCR en el INS dieron otros tipos de Candida como C. haemulonii var. vulnera, C. albicans, C. parapsilosis, C. krusei, C. tropicalis y C. glabrata.
En general, la identificación correcta según el departamento remitente osciló entre 33-100%, dependiente del sistema de identificación disponible en cada región. (Tabla 1).
Discusión
En las últimas décadas, la infección por hongos se ha convertido en un problema de salud pública, especialmente para los pacientes inmunocomprometidos y aquellos hospitalizados con enfermedades graves de base. Candida auris es uno de los patógenos más importantes, responsables de las infecciones fúngicas invasivas y de brotes intrahospitalarios7.
Nuestros resultados muestran que las identificaciones discrepantes representan un 57,6%, esto indica que aún sigue siendo un desafío el diagnóstico adecuado de este patógeno emergente no solo en Colombia sino en el mundo, influyendo directamente en brindar al paciente un tratamiento adecuado y oportuno.
El estudio de Ambaraghassi G y colaboradores publicado en el 2019 reportó que el VITEK 2 versión 8.01 identificó correctamente el 52% (18/35 del total de los aislamientos del estudio), el 27% fueron aislamientos indistinguibles entre C. auris, C. duobushaemulonii y C. famata y las identificaciones erradas representaron el 21%8. En nuestro trabajo, las identificaciones correctas correspondieron al 42,4% del total de aislamientos confirmados como C. auris por MALDI-TOF MS y/o PCR.
Los primeros tres casos de infección fúngica invasiva en un hospital por C. auris reportados en 2009 en Corea del Sur mencionan que esta levadura es generalmente identificada como C. haemulonii y R. glutinis por los sistemas de identificación comercial VITEK 215. Estos casos se asemejan a los resultados de este estudio, donde VITEK 2 presentó la mayoría de las identificaciones como C. haemulonii.
Nuestros análisis muestran que el sistema VITEK 2 versión 8.01 tiene más capacidad de identificación correcta de C. auris en comparación a la versión 7.01 que tiene menos identificaciones acertadas; pese a la actualización desde el 2018, el diagnostico sigue siendo un reto debido a que esta base de datos aún no tiene la completa capacidad de discriminar entre C. auris y otras especies de Candida spp. estrechamente relacionadas.
Los sistemas comerciales de identificación como MicroScan, Phoenix BD, VITEK 2, entre otros, usan un panel que se basa en pruebas de asimilación y crecimiento, utilizando compuestos de carbono y nitrógeno y se emplean de rutina en los laboratorios para la identificación de levaduras16. Un estudio realizado en el 2016 valida y compara varias plataformas y encontraron que los aislamientos de C. auris fueron identificados por Phoenix BD y VITEK 2 como C. haemulonii y por MicroScan como C. famata, C. lusitaniae, C. guilliermondii o C. parapsilosis17. Así como se observa en el estudio mencionado, la plataforma MicroScan identifica erróneamente a C. auris como una variedad de especies de Candida spp; por el contrario, Phoenix y VITEK 2 generalmente identifican C. auris como C. haemulonii.
Varias investigaciones se han concentrado en comprender la razón de la asociación entre C. auris y C. haemulonii. Estudios señalan que C. auris exhibe una estrecha relación filogenética con C. haemulonii y se diferencia con base al análisis de secuencia del dominio D1/D2 de la subunidad 26S ARNr y las regiones espaciadoras transcritas internas (ITS)18.
Sin duda, MALDI-TOF MS se considera una técnica de diagnóstico rápida y confiable para la identificación de C. auris. Actualmente, el enfoque MALDI-TOF MS es comercializado por 2 fabricantes: Bruker-Daltonics y BioMérieux. Estudios mencionan que el sistema de Bruker Biotyper tiene una biblioteca más amplia que la de BioMérieux19. En Colombia son pocas las instituciones que tienen MALDI-TOF MS debido a la restricción de costos; en este estudio, los resultados extrañamente arrojaron que el MALDI-TOF MS Biotyper solo tuvo el 69% de identificaciones correctas enviadas por los laboratorios de los establecimientos hospitalarios. Probablemente, esto se debió a que este equipo en su momento tenía una base de datos limitada. En general MALDI-TOF MS proporciona resultados comparando los espectros de la biblioteca con la cepa de estudio, de manera que puede generar resultados de especies filogenéticamente relacionadas.
Debido al hecho de que esta levadura puede ser multi- resistente a antifúngicos, productos de limpieza y a diferentes ambientes y además es de fácil transmisibilidad, es importante identificarla correctamente para proporcionar una atención efectiva y pronta al paciente. Así que, las deficiencias de los sistemas comerciales para la identificación de C. auris deben ser complementados con otros sistemas como MALDI-TOF MS o pruebas moleculares; debido a los costos y disponibilidad de estas herramientas, el envío al laboratorio de referencia es esencial para hacer una identificación adecuada del microorganismo, determinar la circulación de este patógeno a nivel local y nacional con el fin de prevenir la transmisibilidad y realizar control de propagación.