Introducción
La antibiótico-resistencia es un fenómeno por el cual las bacterias son capaces de sobrevivir al tratamiento con antimicrobianos a los que previamente eran sensibles; esta resistencia puede ser intrínseca dada la naturaleza de sus estructuras (sucede en bacterias de la misma especie, no presenta cambios y no se determina por dosis de antimicrobianos), o adquirida (dada por cambios a nivel genético sean cromosómicos o extra cromosómicos)1,2.
Este fenómeno se puede presentar en microorganismos en todo el mundo lo que ha llevado a declararlo problema de salud pública3. Entre las causas que han potenciado la dispersión de bacterias con resistencia a antimicrobianos, se encuentran; la poca regulación para la adquisición de antibióticos (permitiendo el consumo indiscriminado por parte de la comunidad); la prescripción inadecuada por parte de los profesionales de la salud; el empleo sobre animales de engorda para promover su crecimiento y la circulación sobre fómites contaminados en la comunidad4-6.
Se denomina fómite, a todo elemento inanimado que aloja y sirve de superficie de crecimiento de microorganismos patógenos por medio del contacto con fluidos o aerosoles6. Los agentes infecciosos sobre estos fómites pueden diseminarse con facilidad entre personas ya que estos objetos son de uso común (billetes, monedas, puertas, cerraduras, pasamanos, entre otros) 6.
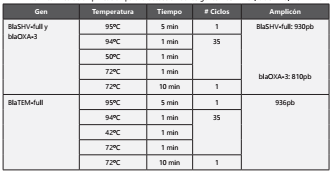
Dada su alta circulación y manipulación, se considera que los fómites facilitan la transmisión de infecciones en determinadas poblaciones. En el caso particular de las bacterias se ha visto que además de diseminarse con facilidad, cuentan con una carga genética natural o adquirida como resultado de la constante exposición agentes desinfectantes, por lo que se ha descrito en estudios previos cepas de bacterias con resistencia a diferentes antibióticos, dentro de los que se destaca la resistencia a betalactámicos, dado principalmente por inactivación enzimática, siendo las betalactamasas (Bla) tipo TEM-full, OXA-3 y SHV-full, enzimas involucradas en la resistencia de gramnegativas y grampositivas3,6.
De acuerdo a lo anterior este estudio tuvo por objetivo describir bacterias presentes en fómites de alta circulación en el municipio de Armenia-Quindío Colombia junto a su perfil de sensibilidad fenotípica y presencia de genes de betalactamasas tipo TEM-full, OXA-3 y SHV-full.
Materiales y métodos
Obtención de muestras
Se realizó un estudio descriptivo de corte transversal, a partir de tres tipos de fómites con manipulación constante en la comunidad (pasamanos de escaleras eléctricas de 3 centros comerciales, botones de 3 cajeros automáticos y 4 billetes de baja denominación circulando en diferentes puntos de la ciudad elegidos de forma aleatoria).
La toma de todas las muestras se realizó mediante frotis con un hisopo estéril previamente humedecidos en caldo cerebro-corazón (BHI OXOID®), se frotaron las superficies y se depositaron dentro de un recipiente con caldo BHI. Posteriormente las muestras se incubaron a 37º C, durante 16 horas en agitación de 200 revoluciones por minuto.
Al evidenciar crecimiento por turbidez en el medio BHI, se cultivó en agar sangre (OXOID® Agar de sangre Base2, suplementado con 10 ml de sangre) y agar MacConkey (OXOID® MacConkey No3) por técnica de agotamiento, los cuales se incubaron por 24 horas a 37°C. Para asegurar la pureza de las colonias bacterianas se realizó resiembra y se identificó género y especie; empleando técnicas automatizadas mediante el equipo Vitek 2®.
Análisis de perfil de susceptibilidad antimicrobiana
Se realizó por concentración mínima inhibitoria mediante tarjetas VITEK® 2 AST de bioMérieux® con puntos de corte actualizados de normativa CLSI M100-S297 y EUCAST versión 9.08. Los antibióticos analizados en gramnegativos (tarjeta vitek AST-NO86, AST-NO82) incluyeron Ampicilina/Sulbactam, Piperacilina/Tazobactam, Ceftazidima, Ceftriaxona, Cefepima, Doripenem, Imipenem, Gentamicina, Ciprofloxacino, Tigeciclina, Meropenem, Amicacina, Colistina, Cefoxitina, Ertapenem.
Los antibióticos analizados en grampositivos (tarjeta Vitek AST-P577) incluyeron: Ciprofloxacino, Ampicilina, Gentamicina, Estreptomicina, Levofloxacino, Eritromicina, Quinupristina/Dalfopristina, Linezolid, Teicoplanina, Vancomicina, Minociclina, Tetraciclina, Nitrofurantoína, Cefoxitina, Oxacilina, Moxifloxacino, Rifampicina, Trimetropina-sulfametoxazol, Clindamicina.
Detección genes para betalactamasas tipo TEM-full, OXA-3 y SHV-full
Se realizó PCR en colonia; las colonias bacterianas fueron disueltas en 450µL de agua destilada estéril y calentadas a 95°C 10 minutos. De esta preparación se tomaron 10µL como plantilla para el PCR, se empleó el master mix 2X MyFi™ Mix de Bioline Cat. BIO-25049 y los primers descritos por Jamali, et al 20179. Los productos de PCR fueron corridos en gel de agarosa al 1% teñido con bromuro de etidio al 10%, buffer TBE-1X a 80 voltios durante 45min y visualizados en tras iluminador majorScience. Como marcador de peso molecular se empleó HyperLadder™I cat.No.BIO-33025.
Las condiciones de amplificación para cada gen se muestran en la Tabla 1 (todas las amplificaciones se realizaron en un termociclador BioRad T100) .
Resultados
Distribución de microorganismos aislados
De los fómites ambientales estudiados el 30% fueron pasamanos de escaleras, 30% botones de cajeros y 40% billetes; recolectados de diferentes puntos concurridos de la ciudad de Armenia. Todos los fómites revelaron ser portadores de bacterias grampositivas y gramnegativas; siendo de mayor prevalencia las gramnegativas como se observa en la figura 1.
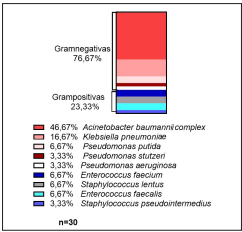
Figura 1 Distribución bacterias aisladas. En la figura se indica la distribución de los microorganismos aislados a partir de los fómites estudiados. En este estudio se encontró un predominio de gramnegativas sobre grampositivas.
En la figura 2, se muestra la distribución de las cepas aisladas y su distribución en cada fómite.
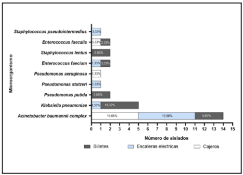
Figura 2 Distribución de microorganismos en fómites. A. baumannii, estuvo presente en todos los fómites analizados, K. pneumoniae en escaleras y billetes, P. putida en billetes, P. stutzeri en pasamanos de escaleras eléctricas, P. aeruginosa en cajeros, E. faecium en billetes y pasamanos de escaleras eléctricas, S. lentus en billetes, E. faecalis en billetes y cajeros y S. pseudointermedius en pasamanos de escaleras eléctricas.
Identificación genotípica de betalactamasas tipo SHVfull, OXA-3 y TEM-full
Después de realizar la identificación genotípica (SHV, OXA-3 y TEM) de los 30 aislados, se encontró que 16(53%) portaron al menos uno de los genes y los 14(47%) restantes no portaron ninguno de los genes incluidos en este estudio. De los aislados portadores de los genes incluidos en este estudio, 4(25%) portaron un solo gen, mientras que 12(75%) portaron más de uno (Tabla 2).
Tabla 2 Genes de betalactamasas SHV-full, OXA-3 Y TEM-full en bacterias gramnegativas, identificados en fómites
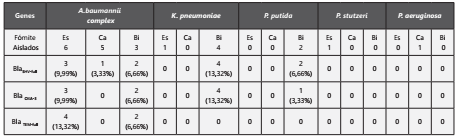
Es: Pasamanos de escaleras eléctricas. Ca: Botones de cajeros automáticos. Bi: Billetes
El gen con mayor prevalencia fue BlaSHV-full, presente en el 50% de los aislamientos positivos para los genes de estudio. Las combinaciones observadas fueron las siguientes: BlaSHVfull+BlaOXA-3+ Bla TEM-full, BlaSHV-full+BlaOXA-3, BlaSHVfull, BlaSHV-full+Bla TEM-full y BlaOXA-3+ Bla TEM-full observadas en el 31,25%, 31,25%, 25%, 6,25% y 6,25% de los aislados, respectivamente. La bacteria con mayor prevalencia y positiva para resistencia fue A. baumannii. Los fómites con mayor colonización fueron los billetes (43,33%), y en las bacterias aisladas a partir de estos se encontró mayor resistencia. El análisis genético de los microorganismos encontrados se describe en las tablas 2 y 3. En la figura 3 se observan los productos de PCR obtenidos para los genes SHV-full, OXA-3 y TEM-full.
Tabla 3 Genes de betalactamasas SHV-full, OXA-3 Y TEM-full en bacterias grampositivas, identificados en fómites
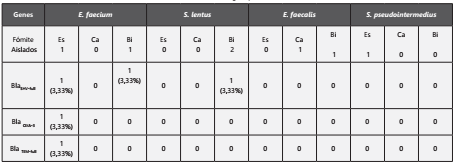
Es: Pasamanos de escaleras eléctricas. Ca: Botones de cajeros automáticos. Bi: Billetes

Figura 3 Genes asociados a betalactamasas identificados. A Gen BlaSHV-full. Pozo 1. Marcador de peso HyperLadder TM I. Cat. No. BIO-33025 BIOLINE. Pozo 2 a Pozo 10. Acinetobacter baumannii Pozo 16. Marcador de peso HyperLadder TM I. Cat. No. BIO-33025 BIOLINE. Pozo 19. Enterococcus faecium Pozo 20. Acinetobacter baumannni Pozo 21. Acinetobacter baumannii Pozo 22. Staphylococcus lentus Pozo 23. Enterococcus faecium Pozo 24. Pseudomonas putida Pozo 26. Pseudomonas putida Pozo 29 a 32. Klebsiella pneumoniae Pozo 33. Escherichia coli- control Pozo 34. Control positivo Pozo 35. Control negativo. B. Gen BlaOXA-3. Pozo 1. Marcador de peso HyperLadder TM I. Cat. No. BIO-33025 BIOLINE. Pozo 4 y 5. Acinetobacter baumannii. Pozo 16. Marcador de peso HyperLadder TM I. Cat. No. BIO-33025 BIOLINE. Pozo 19. Enterococcus faecium Pozo 20 y 21. Acinetobacter baumannni Pozo 30. Klebsiella pneumoniae Pozo 33. Escherichia coli- control Pozo 34. Control positivo Pozo 35. Control negativo. C. Gen BlaTEM-full. Pozo 1. Marcador de peso HyperLadder TM I. Cat. No. BIO 33025 BIOLINE. Pozo 2 a 5. Acinetobacter baumannii Pozo 6. Enterococcus faecium Pozo 7. Acinetobacter baumannii Pozo 8. Control positivo Pozo 9. Control negativo
Perfil de resistencia fenotípica de los microorganismos aislados
En las tabla 4 y 5 se muestran únicamente el perfil de resistencia presentado por los microorganismos estudiados gramnegativos y grampositivos respectivamente.
Tabla 4 Resistencia fenotípica en gramnegativas
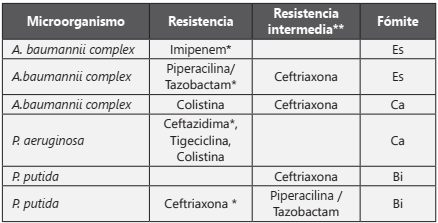
Es: Pasamanos de escaleras eléctricas. Ca: Botones de cajeros automáticos. Bi: Billetes. *la resistencia fenotípica a betalactámicos en estos microorganismos coincide con el hallazgo de BlaSHV-full, BlaTEM-full y BlaOXA-3 **Se adopta el término de resistencia intermedia de acuerdo a los puntos de corte de normativa CLSI- M100 Ed. 29(7) y EUCAST Versión 9.08, Categoría resistencia intermedia.
Tabla 5 Resistencia fenotípica en grampositivas
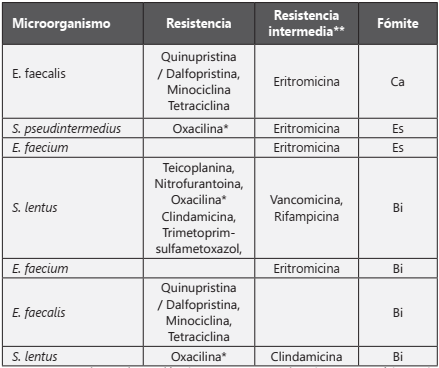
Es: Pasamanos de escaleras eléctricas. Ca: Botones de cajeros automáticos. Bi: Billetes. *la resistencia fenotípica a betalactámicos en estos microorganismos coincide con el hallazgo de BlaSHV-full, BlaOXA-3. **Se adopta el término de resistencia intermedia de acuerdo a los puntos de corte de normativa CLSIM100 Ed. 297 y EUCAST Versión 9.08, Categoría resistencia intermedia.
Discusión
Pese a que la presencia bacteriana en diversos fómites ha sido descrita con un predominio principalmente por bacterias grampositivas, como Bacillus sp., y S. aureus10,11, en este trabajo se encontró un predominio de bacterias gramnegativas con un 76,67% dado principalmente por A. baumannii frente a un 23,33% de grampositivas; este fenómeno puede explicarse de acuerdo a lo reportado por Angelakis, et al, 201412, quienes indican que este grupo de patógenos gramnegativos tienen mayor capacidad de supervivencia en superficies inertes, trayendo como consecuencia una alta diseminación; en especial si se tiene en cuenta su capacidad de formación de biopelículas y transmisión de información genética por medio de plásmidos con genes de resistencia asociados a betalactamasas13; demostrando a su vez, que los elementos inanimados se comportan como un factor determinante en la diseminación de bacterias con resistencia en la comunidad. Adicionalmente cabe recordar que: Acinetobacter sp., E. faecalis, K. pneumoniae y P. aeruginosa, hacen parte de la lista de prioridad de la OMS, en donde se consideran como patógenos de difícil manejo, por lo que encontrarlos en circulación en elementos de alta circulación en la comunidad debería ser considerado por las autoridades de salud pública de cada región14.
Es importante resaltar que en estudios multidisciplinarios el enfoque de detección de enterobacterias y betalactamasas se ha dirigido a animales de consumo humano, demostrando que los productos de dicha industria también contribuyen a la diseminación de este tipo de microorganismos13. Este tipo de hallazgos junto al comportamiento de la microbiota en fómites en la comunidad, contribuyen a complementar la comprensión relacionada con la circulación y prevalencia de microorganismos con resistencia antimicrobiana, fuera de los sitios de atención hospitalaria; debido a que se ha reconocido que existen grandes lagunas en el conocimiento de la dinámica de transmisión de enfermedades infecciosas; dispersión de microorganismos y agentes infecciosos en la población, por lo demás también invita a la comunidad científica al estudio interdisciplinar, no sólo de elementos inanimados sino de aerosoles de largo y corto alcance; con la finalidad de controlar este tipo de exposiciones y disminuir los potenciales riesgos para la salud humana15.
La presencia de bacilos gramnegativos pertenecientes a la familia Enterobacteriaceae, en los aislamientos obtenidos a partir de billetes de este estudio coincide con los resultados obtenidos del mismo fómite en el estudio realizado por Betancur et. al, 2010 en Medellín-Colombia16. En este estudio se describe la presencia de Klebsiella sp., en los aislamientos obtenidos a partir de cajeros automáticos, lo que difiere de lo descrito en un estudio realizado en la ciudad de HamadanIrán, donde además se describió la presencia de P. aeruginosa en el mismo sitio de aislamiento17.
Por otra parte, se identificó que A. baumannii complex expresó SHV-full, TEM-full y OXA-3 hallazgo relevante si se tiene presente que son microorganismos que han mostrado tener una variedad amplia de resistencia y se han asociado a infecciones severas18. A su vez se ha determinado que K. pneumoniae presenta resistencia intrínseca a ampicilina por SHV codificada en el cromosoma de la bacteria, lo que sustenta estos hallazgos, también se encontró expresión de OXA-3, correspondientes a las β-lactamasas de las clases D, sin embargo, fenotípicamente no se observó resistencia por parte de estas cepas a antibióticos como cefalosporinas que pudiera indicar la relación genotípica y fenotípica.
En el presente trabajo no se encontró resistencia a antibióticos por parte de P. stutzeri, característica que se ha mantenido en varios reportes19; mientras que las cepas de P. aeruginosa (usualmente con múltiples mecanismos de resistencia19), presentaron resistencia fenotípica frente a ceftazidima (cefalosporina de 3 generación), polipéptidos y glicilciclinas17,19,20 aunque no se identificaron los genes asociados a betalactamasas (TEM, OXA-3, SHV).
La presencia de genes asociados a betalactamasas en grampositivos como E. faecium y S. lentus puede tomarse como un indicador de la dispersión de resistencia bacteriana inter especie, dada principalmente por plásmidos, lo cual contribuye a la problemática emergente de antibioticorresistencia21).
La resistencia fenotípica encontrada en este estudio indica que los aislados obtenidos, no solamente presentan genes de resistencia asociados a inactivación enzimática de betalactámicos (las cepas aisladas presentan resistencia desde cefalosporinas hasta carbapenémicos e inhibidores de betalactamasas); sino que también se encontró resistencia fenotípica frente a otros grupos terapéuticos como los polipéptidos(colistina), glicilciclinas(tigeciclina); en el grupo de los grampositivos frente a glucopéptidos(vancomicina), rifamicinas(rifampicina), macrólidos (eritromicina) y agentes que actúan en sinergia frente a la resistencia del último grupo (quinupristina y dalfopristina)22-24.
Cepas gramnegativas y positivas aisladas en este estudio, presentaron resistencia frente a uno o dos grupos de antimicrobianos, lo cual debe considerarse como un hallazgo significativo para intervención en la comunidad; en especial si se tiene en cuenta que son microorganismos oportunistas con alta capacidad de supervivencia en superficies y formación de biopelículas de difícil manejo terapéutico24,25.
Conclusión
Los fómites de amplio contacto con la comunidad presentan colonizaciones de grupos bacterianos que han sufrido presión selectiva por el uso constante de geles anti-bacteriales o exposición a desinfectantes sin rotación cíclica; por lo que la circulación de estos grupos bacterianos con resistencias no solamente debe estudiarse en el entorno intrahospitalario sino que también deben tomarse medidas de salud pública que busquen educar a la comunidad (sin generar alarma), del potencial peligro de contraer infecciones bacterianas con resistencia a fármacos, y reforzar medidas de prevención como el adecuado lavado de manos. Es importante reconocer la circulación de las cepas bacterianas con su perfil de resistencia en nuestra región y las tomar medidas sanitarias pertinentes.