Introducción
Escherichia coli productora de toxinas Shiga (STEC) es un agente zoonótico transmitido por alimentos, que puede causar enfermedades severas en humanos, como colitis hemorrágica y Síndrome Urémico Hemolítico (SUH)1. Estudios de prevalencia realizados en distintos países confirman el rol del ganado bovino como el principal reservorio de cepas STEC, que han sido transmitidas al hombre a través de alimentos derivados del bovino contaminados con sus heces, causando brotes de infección en todo el mundo)2,3.
La patogénesis de STEC involucra diferentes factores de virulencia)4, algunas cepas poseen el gen eae, que codifica la intimina, una proteína de membrana externa de 97 KDa responsable de la adherencia íntima de STEC a las células del enterocito y de causar el borrado de las microvellosidades intestinales (lesión attaching/effacing). En cepas que carecen de la proteína intimina, se ha identificado una proteína autoaglutinante de membrana externa (Saa) que permite la adherencia de la bacteria a las células del intestino)5, lo que sugiere que las cepas STEC presentan diferentes mecanismos para la adherencia de la bacteria a la célula blanco. Además, se describe una hemolisina enterohemorrágica, codificada por el gen hlyA, ubicado en el megaplásmido pO157)6, esta hemolisina se expresa durante la infección por STEC y permite captar el hierro de los eritrocitos, necesario para la supervivencia bacteriana)7.
Las complicaciones severas de la infección por STEC se deben a la producción de toxinas, denominadas toxinas Shiga (Stxs), por su similaridad con la toxina Shiga sintetizada por la bacteria Shigella dysenteriae)8. Las Stxs son catalogadas como el principal factor de virulencia de STEC ya que tienen un rol crítico en la patogenia en humanos, causando daños en órganos esenciales como el riñón, el páncreas y el Sistema Nervioso Central)9. Estas Stxs se encuentran codificadas por bacteriófagos insertos en el cromosoma bacteriano, lo que facilita la transferencia horizontal a bacterias no patógenas convirtiéndolas en patógenas)5. La producción de Stxs está ligada a la inducción del ciclo lítico del fago que ocurre cuando la bacteria es expuesta a condiciones de estrés y agentes mutagénicos como luz UV y antibióticos)10. Antigénicamente, las Stxs se han clasificado en Stx1 y Stx2, siendo estas últimas encontradas con mayor frecuencia en cepas aisladas de humanos con SUH)11. Aunque Stx1 y Stx2 poseen el mismo modo de acción sobre la célula blanco, sus efectos in vitro e in vivo difieren considerablemente. Stx1 se une de manera más eficiente al receptor celular y es más potente en células Vero. Sin embargo, Stx2 es 400 veces más letal para ratones que Stx1 inyectado por vía intravenosa, y es 1000 veces más tóxica en células de la microvasculatura renal humana)12,13.
En estudios previos se ha reportado la presencia de STEC en Colombia en poblaciones porcinas y productos cárnicos de origen bovino y porcino)14,15,16, sin embargo, estas investigaciones son escasas y se han centrado en el contexto de la detección de cepas del serotipo O157:H7, a través de métodos antigénicos y de microbiología clásica. Actualmente, no se cuenta con reportes de la presencia y caracterización a nivel molecular de cepas STEC en heces de bovinos, y teniendo en cuenta que estas bacterias continúan representando una amenaza para las poblaciones de todo el mundo17 y que en Colombia no son incluidas en los sistemas de vigilancia epidemiológica, el objetivo de este estudio fue aislar STEC en las heces del ganado bovino en el municipio de Ulloa, Valle del Cauca y detectar factores de virulencia asociados con la patogénesis.
Materiales y Métodos
Tipo de muestreo y toma de muestras
Se realizó un estudio descriptivo de corte transversal con un muestreo a conveniencia; con previa autorización del propietario del predio, se colectaron muestras de heces por hisopado rectal en 21 bovinos en la vereda el Cofre, ubicada en el km 4 vía Ulloa-Valle del Cauca, Colombia. Los animales incluidos en el estudio correspondieron a los bovinos que se encontraban disponibles en el predio al momento del muestro. Cada muestra fue transportada en tubos de ensayo con agua peptonada a temperatura ambiente, hasta el Centro de Investigaciones Biomédicas (CIBM) de la Universidad del Quindío para su procesamiento.
Procesamiento de las muestras
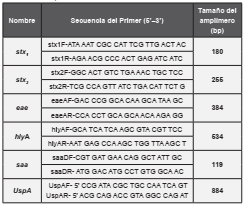
Una vez en el CIBM, cada muestra se sembró en agar MacConkey y se incubó por 24 h a 37 ºC. Posteriormente, una muestra de la zona de crecimiento confluente se cultivó en medio Brain Heart Infusion (BHI) por 4 h a 37 ºC con agitación a 180 rpm. Luego se tomaron 400 μL de H2O ultrapura y 100 μL de cultivo bacteriano, y se llevó a ebullición por 10 minutos para extracción de ADN (por lisis celular en caliente) y posteriormente se centrifugó a 11500 rpm para eliminar los restos celulares en suspensión)18. El ADN recuperado se evaluó para detectar los genes stx1 y stx2, mediante PCR Múltiplex en un equipo Applied Biosystems Veriti 96 Wells Thermal Cycler, con cebadores específicos según lo propuesto por Paton & Paton)4 los cuales se muestran en la Tabla 1. Se utilizó como control positivo la cepa de referencia de E. coli EDL933, proveniente del Laboratorio de Referencia de Escherichia coli (LREC), Lugo-España, y como control negativo, se empleó la cepa E. coli ATCC 25922, que no posee factores de virulencia. Las muestras positivas para uno o ambos genes stx se sembraron en agar MacConkey y se cultivaron por 24 h a 37 ºC. Se realizaron “pooles” de 5 colonias cada uno para detectar por PCR Múltiplex los genes codificantes de Stx1 o Stx2. Las colonias de los “pooles” positivos se cultivaron individualmente en BHI durante 18 h a 37 ºC y se analizaron entre 5 y 50 colonias por muestra dependiendo del número de colonias individuales que crecieran en el agar, para detectar por PCR Múltiplex, los genes eae, saa y hlyA)4,19.
Las condiciones para la PCR fueron las siguientes: el volumen final de reacción fue de 25 μL, conteniendo 2 μL de ADN genómico, 2,5 μL de buffer para PCR (10X), 2 μL de MgCl2 50 mM, 0,2 μL de dNTP a 10 mM, 0,1 μL de Taq polimerasa Recombinante de Invitrogen, 16,2 μL de agua estéril desionizada y 1 μL de cada cebador a una concentración de 10 μM. Las muestras se sometieron a 35 ciclos de PCR, cada uno consistió en 1 min de desnaturalización a 95 °C; 2 min de anillamiento a 65 °C por los 10 primeros ciclos, disminuyendo a 60 °C para el ciclo 15; y 1,5 min de elongación a 72 °C, incrementando a 2,5 min desde el ciclo 25 a 35)4. Los productos de PCR se analizaron por electroforesis en gel de agarosa al 2% (p/v) en buffer Tris-borato-EDTA (TBE), teñido con SYBR Safe marca Invitrogen, separado a 100V por 40 minutos y fotografiado a través del transiluminador GelDoc-It Imager, software UVP. Los tamaños de los productos se estimaron por comparación con el marcador de peso molecular HyperLadder II 50pb de Bioline. Posteriormente, para confirmar que los aislados correspondían a bacterias E. coli, se realizó una PCR convencional propuesta por Chen & Griffiths20, para detectar la presencia del gen UspA que codifica una proteína de estrés universal de E. coli.
Actividad citotóxica en células Vero
Para determinar si los aislamientos positivos para los genes stx 1 y stx2 expresaban las Stxs, se evaluó el efecto citotóxico de 14 aislamientos STEC, sobre la línea celular Vero ATCC-CLL 81.4 siguiendo la metodología propuesta por Kruger et al., )21 con algunas modificaciones. Para esto se cultivaron los aislamientos STEC en medio Luria Bertani Broth (LB) a 37 °C durante toda la noche en agitación a 100 rpm, luego, se subcultivaron en LB a 37 °C hasta alcanzar una densidad óptica a 600 nm (DO600) de ≈ 0,2 - 0,3; en este punto se dividió el cultivo en dos, uno de los cultivos fue inducido con mitomicina C a 0,5 μg/mL y el otro no. Ambos se cultivaron en agitación a 100 rpm por 24 h a 37 °C. Luego, se tomó una alícuota de cada cultivo y se centrifugó a 11500 rpm por 10 minutos a 4 °C en una centrífuga Thermo Scientific Heraeus Megafuge 11 y se filtraron los sobrenadantes con filtros Milipore Millex-GV de 0,22 µm.
Las células Vero se cultivaron en medio D-MEM (dulbecco modified Eagles minimal essential medium) marca Gibco, suplementado con suero fetal bovino al 10%, antibiótico antimicótico 1X (contiene 10000 unidades/mL de penicilina, 10000 µg/mL de estreptomicina y 25 µg/mL de anfotericina B de Gibco) e incubadas a 37 ºC en una atmósfera de 5% de CO2. Se realizaron diluciones seriadas en base 2 en medio DMEM de los sobrenadantes filtrados anteriormente. En placas de 96 pocillos marca Nest se añadieron 150 μl de una suspensión de células Vero de 2 x 104 cel/mL en cada pozo, y una vez establecida la monocapa celular, se agregaron 40 μl de cada dilución. Las placas se incubaron a 37 °C y 5% de CO2 por 48 h. Las monocapas celulares se fijaron con formaldehído al 10% y se tiñeron con cristal violeta al 0,2% (p/v) en buffer fosfato salino (PBS, phosphate buffered saline) por 30 minutos. Por último, se tomó como CC50 la dilución donde se observó el 50% de destrucción de la monocapa celular utilizando el Microscopio invertido Olympus CK2. Para evaluar el efecto de la mitomicina C en los cultivos STEC frente a la CC50, se realizó la prueba de Kruskal Wallis.
Resultados
Aislamiento de STEC y detección de factores de virulencia
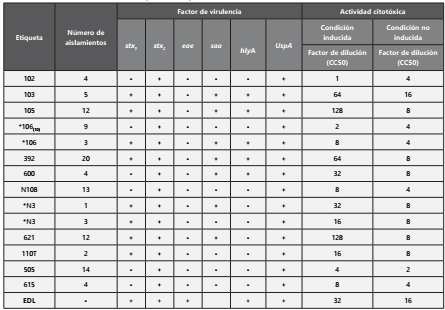
De los 21 hisopados rectales, nueve (42,85%) fueron negativos y 12 (57,14%) fueron positivos para uno o ambos genes codificantes de toxinas Shiga. De todas las muestras analizadas se obtuvo un total de 106 aislamientos STEC. Se observaron diferencias en cuanto a la presencia y ausencia de los genes de virulencia evaluados en los aislamientos de cada bovino, obteniendo cinco combinaciones de genes (1. stx2; 2. stx1/stx2; 3. stx1/stx2/saa/hlyA; 4. stx2 /saa/hlyA y 5. stx1/stx2/saa). En dos de los bovinos STEC positivos, fue posible obtener aislamientos con diferentes combinaciones de genes dentro de la misma muestra (Tabla 2). De los 106 aislamientos STEC, 48 presentaron únicamente el gen stx2 y 58 aislamientos presentaron tanto el gen stx1 como el gen stx2. En cuanto a los otros factores de virulencia, se detectaron 44 muestras que portaban el gen hlyA y 57 muestras el gen saa. Se verificó que todos los aislamientos evaluados fueran de la especie Escherichia coli por medio de la detección del gen UspA que codifica para una proteína de estrés universal y el cual estuvo presente en todos los aislados (Tabla 2).
Actividad citotóxica en células Vero
De un total de 106 aislamientos se seleccionó un aislamiento representante de cada bovino STEC positivo, obteniendo así, 14 aislamientos (Figura 1) a los cuales se les evaluó el efecto citotóxico sobre la línea celular Vero. Se observó que todos los sobrenadantes de los 14 aislamientos STEC evaluados, mostraron efecto citotóxico sobre las células Vero; las células de la monocapa, originalmente alargadas, se tornaron redondeadas y posteriormente, sufrieron destrucción celular pasadas 48 horas de incubación (Figura 2A). Por otra parte, en los pozos donde no se agregó sobrenadante de cultivo bacteriano (control celular), así cómo donde se agregó el sobrenadante puro de la cepa E. coli ATCC 25922 (control negativo), no se observó cambio en la morfología de la monocapa ni se observó destrucción celular (Figura 2B).
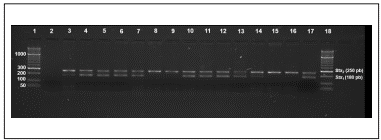
Figura 1 Amplificación por PCR de genes de toxina Shiga. Se amplificó un producto de 180 y 250 pb usando las secuencias de oligonucleótidos que flanquean los genes stx1 y stx2. Carril 1 y 18, marcador de peso molecular HypperLader II 50 pb. Carril 2, cepas de E. coli ATCC 25922 como control negativo. Los carriles 3 a 16 muestran los productos de PCR amplificados a partir de muestras de ganado. Carril 17, cepa de E. coli EDL933 como control positivo.
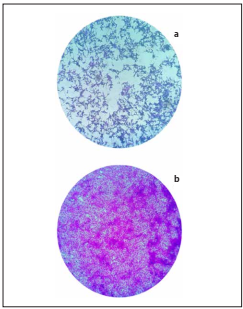
Figura 2 Efecto citotóxico en las células Vero después de 48 horas de incubación con sobrenadantes de cultivo STEC, tinción con cristal violeta al 0,2%. Vista en Microscopio invertido Olympus CK2 a objetivo 10X. a. Destrucción celular. b. Monocapa de células.
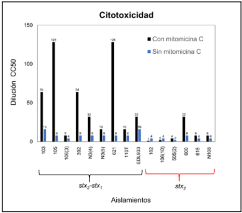
Al realizar la prueba de Kruskal Wallis se observaron diferencias en cuanto a la CC50 (P = 0,024), evidenciando valores más altos de citotoxicidad en los cultivos inducidos con mitomicina C. Se destacan cuatro aislamientos STEC portadores de los genes stx1 y stx2 que según los ensayos in vitro mostraron un efecto citotóxico mayor a la EDL933 (control positivo) en condición inducida; a diferencia de dos aislamientos portadores del gen stx2, los cuales evidenciaron una mayor CC50 en condiciones basales (Figura 3).
Discusión
STEC se encuentra asociado a brotes y casos esporádicos de enfermedad en el hombre como colitis hemorrágica y SUH. Esta bacteria produce dos tipos de toxinas, Stx1 y Stx2 o ambos, codificadas por los genes stx1 y stx2, respectivamente, responsables de muchas de las características patológicas y de las complicaciones severas de la infección por STEC1. Las muestras estudiadas en este trabajo se evaluaron con el fin de detectar la presencia de los genes codificantes de toxina Shiga y de otros genes de virulencia que portan los aislamientos STEC y evaluar el efecto citotóxico sobre la línea celular Vero. Esta investigación aporta los primeros datos en Colombia sobre la presencia de STEC en muestras de hisopado rectal en ganado bovino, y su relevancia está, dado que en Colombia son escasos los estudios acerca de la presencia de esta bacteria en animales bovinos, los cuales son el principal reservorio de esta bacteria2.
En los aislamientos STEC evaluados se encontraron cinco perfiles de genes de virulencia (Tabla 2.), siendo más frecuentes los perfiles stx2 (41,5%) y stx1/stx2/saa/hlyA (37,7%). Estos mismos perfiles fueron encontrados en heces de bovinos en Argentina con porcentajes del 35,6% y 25,5%22; en Brasil con 6,6% y 50,5% en bovinos de lechería23 y en la región metropolitana de Chile en heces de bovinos con 9,8% y 15,8% respectivamente24.
Se ha reportado que la presencia del gen stx2 aumentan la patogenicidad con relación a los aislamientos que portan únicamente el gen stx1. Según la literatura, las muestras positivas para el gen stx2 son un importante factor de riesgo debido a la asociación con enfermedades humanas más graves en comparación con las cepas STEC portadoras de stx125. Louise & Obri13 demostraron que la toxina Stx2 es mil veces más potente que Stx1 en las células endoteliales microvasculares renales humanas y es más probable que se encuentre relacionada con la producción de SUH26.
Es importante destacar que ninguno de los aislamientos presentó el gen eae que codifica la proteína intimina, involucrada en la unión de la bacteria al enterocito. Se ha reportado que el gen eae no está presente en todas las STEC aisladas de bovinos y no parece ser esencial para la patogénesis19, sin embargo, algunas cepas STEC presentan el gen saa como otro mecanismo de adhesión a las células epiteliales4, influyendo en la colonización de la bacteria al intestino bovino27. Por otra parte, el gen saa ha sido encontrado en mayor proporción en aislados bovinos que en aislados humanos28, lo que explicaría que en este estudio se encontraran aislamientos saa positivos y eae negativos. Lo anterior sugiere que la población bovina se encuentra expuesta a STEC en su tracto gastrointestinal, es excretada en las heces y se transmite al hombre a través de la ingestión de alimentos o agua contaminados con heces o por medio del contacto directo con estos animales y con su medio ambiente2,3,29.
Los ensayos de citotoxicidad mostraron que todos los sobrenadantes ensayados tuvieron efecto citotóxico sobre las células Vero. Al igual que Kruger et al., (21 se encontró que, en la mayoría de los aislamientos, al agregar mitomicina C a los cultivos bacterianos presentaban un aumento en la citotoxicidad, no obstante, en este estudio se observó mayor grado de citotoxicidad entre los aislamientos que portan ambos genes de toxina (stx2 /stx1), a diferencia de lo reportado por Kruger et al., (21, donde los aislamientos portadores del gen stx 2 mostraban mayor citotoxicidad. Por otro parte, en este estudio se evidenció que los aislamientos 10610 y 102 presentaron mayor citotoxicidad basal; quizás este comportamiento estaría asociado a que estos dos podrían portar un subtipo de Stx2 diferente al de los demás aislamientos.
En este estudio se destaca que todos los aislamientos STEC evaluados tuvieron la capacidad de producir toxina, debido a que se evidenció la destrucción de las monocapas celulares.
En contraste, Koitobashi et al., (30 reporta que la presencia de los genes stx en las cepas STEC no implica que las bacterias los expresen, como se han documentado algunos casos de aislamientos STEC del continente asiático que portan genes stx pero al ser inducidos con mitomicina C, no hay expresión y producción de toxinas.
En este estudio se demostró que en los aislamientos STEC predominó la presencia del gen stx2, el cual ha sido asociado a enfermedades graves en humanos y se comprobó la actividad citotóxica de las Stx a través del efecto que producen sobre las células Vero. Finalmente, y como perspectiva, se recomienda continuar con esta investigación aumentando el muestreo y realizando una caracterización más amplia de los aislamientos STEC que permita identificar el serotipo y la prevalencia de STEC en esta región.