Introducción
El cáncer de cuello uterino (CaCU) representa la cuarta cau sa de incidencia y mortalidad por cáncer en mujeres a nivel mundial. En Colombia, esta enfermedad representa la quinta y cuarta causa en incidencia y mortalidad, respectivamente1. La carcinogénesis del CaCU comienza con la infección de células epiteliales por el virus del papiloma humano (VPH) en 99,9 % de los casos, generando lesiones intra-epiteliales de bajo (LIEBG) y alto grado (LIEAG), hasta continuar con la aparición del CaCU infiltrante2. Sin embargo, este proceso no siempre es continuo y, por el contrario, aproximadamente un 60 % de LIEBG regresan y sólo un 10 % de ellos progresan a LIEAG3. Teniendo como antecedente los resultados previos obtenidos por García et al. (4, que detectaron 132 genes tumo res-supresores hipermetilados y subexpresados en muestras de cuello uterino normal, displásico y tumoral infiltrante, siete de ellos presentaban evidencia científica de su hipermetila ción en cáncer humano y fueron escogidos como posibles biomarcadores: SFRP1, PTPRN, CDO1, EDNRB, CDX2, EPB41L3 y HAND1. El propósito del presente estudio fue evaluar si la cuantificación de la expresión de mRNA y la valoración cua litativa por estudio de inmunohistoquímica en este grupo de genes candidatos pudieran predecir la progresión o regresión de una lesión intra-epitelial escamosa del cuello uterino.
Materiales y métodos
Obtención de muestras
Se diseñó un estudio longitudinal con biopsias en parafina de pacientes con lesión intra-epitelial escamosa cervico-ute rina de bajo y alto grado, quienes tuvieron posteriormente progresión o regresión de la misma. El estudio contó con aprobación del comité de ética de la Facultad de Odontolo gía de la Pontificia Universidad Javeriana (acta 004 de 2013). Los tejidos en parafina fueron suministrados por “Estudios Anatomopatológicos Ltda” quien actuó como custodio. A cada bloque de parafina, se les realizaron dos cortes (inicial y final) de 3μm para estudio histológico y verificación de la lesión, dos cortes de 5μm para extracción del ARN y 5 cortes de 3μm para análisis de inmunohistoquímica.
Extracción de ARN y RT-PCR
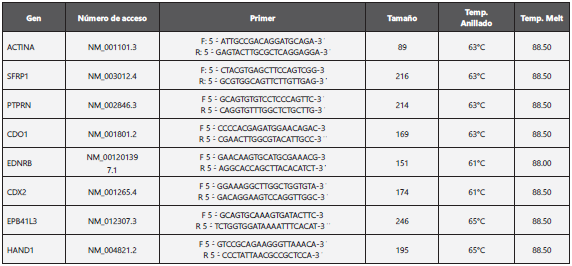
Las biopsias se incluyeron en parafina; para la extracción del ARN, los bloques se desparafinaron y para la extracción se utilizó el kit AllPrep DNA/RNA/ Protein Quigen®, seguido del proceso de digestión con DNAse I Invitrogen® y retro-transcripción con el kit Super Script VILO de Invitrogen®. La cuantificación de concentración de cDNA se realizó con Na nodrop 1000®, manteniendo conservadas las muestras a -70 °C. Los genes incluidos en el estudio fueron SFRP1, PTPRN, CDO1, EDNRB, CDX2, EPB41L3 y HAND1, de quienes se les realizó RT-PCR (Tabla 1), en el equipo CFX96-Biorad® utili zando 1X de Fast SybrGreen de Kappa, 200 nM de cada ce bador y 10 ng de cDNA para un volumen final de 10 uL para cada uno de ellos. Como control, se utilizó el gen ß-Actina. Finalmente, se obtuvo el Fold Change (2 Ct actina - Ct gen blanco) para los genes en estudio, analizados con el progra ma estadístico GraphPad Prism versión 6.0 (GRAPH PAD soft ware Inc., California, USA).
Inmunohistoquímica
Los cortes fueron colocados en láminas Slidetech®, se des parafinaron a 60°C por 2 horas, se deshidrataron con xilol y alcohol, se aplicó peróxido de hidrógeno y solución lavadora CINtech para ser tratados con calor por 40 minutos en re cuperador novocastra en steamer OSTER. Posteriormente, se lavaron y bloquearon con Power Block por 10 min; luego se aplicó el anticuerpo primario por 1 hora así: SFRP1 (1:50, Sigma-Aldrich), CDO1 (1:30, Sigma-Aldrich); CDX2 (1:150, Sigma-Aldrich), EPBL41L3 (1:1000, Sigma-Aldrich) y HAND1 (1:150, Sigma-Aldrich). Finalmente, se utilizó el kit Super sen sitiveTM y Liquid DAB Substrato-Cromógeno, como contras te se usó hematoxilina.
Las muestras fueron analizadas independientemente por dos médicos patólogos y luego se confrontaron sus resultados. En casos donde no existió concordancia, las láminas fueron revisadas nuevamente de manera conjunta por los patólogos hasta llegar a un diagnóstico definitivo sobre la expresión del marcador de inmunohistoquímica (IHQ). La tinción de las láminas para estudio de IHQ fue graduada como “0” (au sencia de expresión en células neoplásicas); “1+” (presencia de expresión entre 1 % y 25 % entre células neoplásicas); “2+” (presencia de expresión entre 26 % y 50 % entre células neoplásicas); “3+” (presencia de expresión mayor a 50 % en tre células neoplásicas) (5.
Análisis de datos
Mediante el software GraphPad Prism v. 6.0, se realizaron las pruebas no paramétricas Wilcoxon, coeficiente de correlación de Spearman y el índice de concordancia (Kappa) para los dos patólogos fue de 0,82 (Muy buena concordancia). Para el análisis estadístico, se compararon los resultados en dos mo mentos diferentes del grado histológico de la lesión de cada uno de los pacientes; por lo tanto, estas muestras pareadas tuvieron un “momento 1” (diagnóstico inicial de la paciente) y un “momento 2” (diagnóstico histológico de seguimiento).
Resultados
Descripción de las muestras del estudio
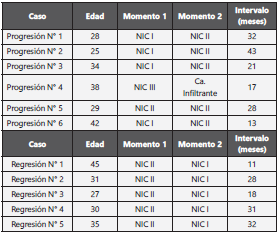
Se incluyeron las muestras correspondientes a seis pacientes con progresión y cinco con regresión de la lesión intra-epite lial escamosa, con promedios de edades de 32,7 y 36,6 años para los grupos de progresión y regresión, respectivamente. La mayoría de las muestras con progresión (66,6 %) indicaron un cambio de NIC I hacia NIC II; uno de los casos progresó de NIC II a NIC III (16,7 %) y el restante de NIC III a carcinoma infiltrante (16,7 %). Con respecto al intervalo en meses entre el diagnóstico del “momento 1” Vs. “momento 2”, éste fue en promedio de 25,6 y 22 meses para los grupos de progresión y regresión, respectivamente. Las muestras incluidas fueron ob tenidas a través de biopsias y producto de conización (Tabla 2).
Expresión de mRNA y proteína de genes en estudio
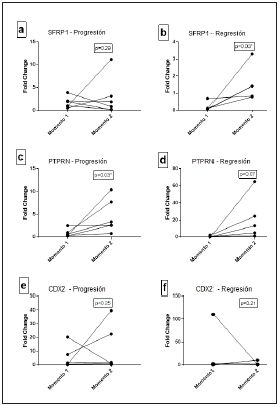
SFRP1 no mostró una tendencia ni cambios significativos en la expresión de mRNA en los casos de progresión (Fig. 1a). Sin embargo, en los casos de regresión, hubo una tendencia de mayor expresión en muestras de LIEBG (momento 2) Vs. LIEAG (momento 1). Esta diferencia de expresiones de mRNA fue estadísticamente significativa (p = 0,030*), (Tabla 2 y Fig. 1b). En cuanto a la expresión de SFRP1 por IHQ en los ca sos de progresión, se observó que al comparar el grado de tinción evidenciado en las muestras del momento 2 Vs. mo mento 1, hubo cinco de seis casos (83,3 %) que disminuyeron en su graduación (p = 0,0313). En los casos de regresión no hubo variación estadísticamente significativa (p = 0,2500) en la expresión de SFRP1 (Tabla 3, Fig. 2a - 2b).
Tabla 3 Graduación de la expresión por IHQ de los genes candidatos en los casos de progresión y regresión.
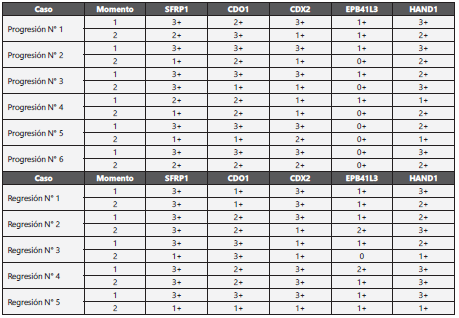
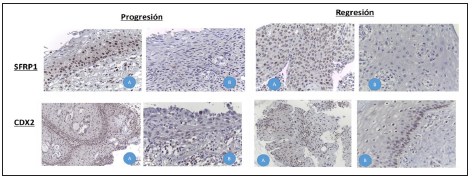
Figura 2 a. Expresión de SFRP1 y CDX2 a través de IHQ en tejido cervical (10x); caso de progresión LIEBG (A), LIEAG (B). b. Expresión de SFRP1 y CDX2 a través de IHQ en tejido cervical; caso de regresión LIEAG (A), LIEBG (B).
PTPRN mostró mayor expresión en muestras con displasia severa o carcinoma infiltrante (momento 2) en compara ción con las muestras del diagnóstico inicial (momento 1), las cuales fueron predominantemente de displasia leve (Ta bla 2 y Fig. 1c). Este incremento fue observado en cinco de seis pacientes (83,3 %) con una diferencia estadísticamente significativa (p = 0,03*). En los casos de regresión, PTPRN también mostró la tendencia mayor expresión de mRNA en las muestras del momento 2, las cuales todas corresponden a NIC I. No obstante, esta diferencia de expresiones no fue estadísticamente significativa (p = 0,07) (Fig. 1d).
CDX2 no mostró una tendencia definida ni cambios signifi cativos con respecto a la expresión de mRNA, tanto en casos de progresión como de regresión (Fig. 1e-1f). En cuanto a su expresión por IHQ en los casos de progresión, hubo cinco de seis casos (83,3 %) que mostraron disminución en su gradua ción (p = 0,0313). Por otra parte, con respecto a los casos con regresión, no existió variación de su expresión estadística mente significativa (p = 0,2500) (Tabla 3, Fig. 2a - 2b).
Para el caso de los genes CDO1, EDNRB, EPB41L3 y HAND1, estos no mostraron ningún tipo de tendencia definida ni cam bios significativos en sus expresiones de mRNA, tanto para los casos de progresión como de regresión. Caso similar fue ob servado con relación a la variación en la graduación de sus ex presiones para aquellos también analizados por IHQ (Tabla 3).
Correlación de expresiones por RT-PCR - Inmunohistoquímica
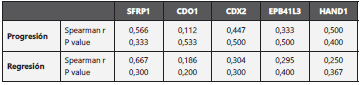
Para obtener el coeficiente de correlación de Spearman, se consideraron las siguientes categorías: 1= aumento expre sión génica y proteíca momento 2 vs momento 1, 2= dismi nución expresión génica y proteíca momento 2 vs momento 1, y 3= sin cambios momento 2 vs momento 1. Todos los genes mostraron un coeficiente de correlación positivo; sin embargo, SFRP1 mostró el mayor coeficiente tanto para pro gresión como regresión con valores de 0,6 y 0,7, respectiva mente, aunque sin significancia estadística (Tabla 4).
Discusión
El CaCU es una entidad de interés creciente en Colombia de bido al alto costo económico, personal y social que implica su tratamiento, y a las cifras epidemiológicas que indican un incremento de la incidencia y mortalidad en el país6. La aso ciación con la infección por VPH está ampliamente justificada en la literatura, así como la posibilidad de prevenirlo en esta dios tempranos7; sin embargo, las relaciones de progresión y regresión han sido difíciles de establecer debido a dificulta des de recolección de muestras; pese a lo anterior, los hallaz gos del presente estudio muestran importantes hallazgos en relación con SFRP1.
SFRP1 es un regulador negativo de la vía de señalización WNT. El presente estudio mostró un aumento de la expresión de RNA en todos los casos de regresión, con disminución de la expresión por IHQ en 83 % de los casos de progresión. Este comportamiento biológico de SFRP1 se ajusta a resul tados publicados en estudios previos donde se encontró hi permetilada la región promotora del gen, en tejido cervical con LIEAG8-11. Adicionalmente, la subexpresión de SFRP1 debido a hipermetilación (regulación negativa) fue reportada en varios canceres, como carcinoma hepatocelular12, carcino ma del cuello uterino9 y carcinoma de mama13,14, lo cual colabora con su silenciamiento15 y el proceso de transición epitelial-mesenquimatosa del CaCU16.
Recientemente, se mostró la relación de SFRP1 en la tran sición epitelial a mesenquimatosa, estaminalidad, prolifera ción y metástasis en un modelo animal de cáncer epitelial17; otros estudios en modelos animales expusieron que la res tauración en la expresión de SFRP1 conllevó a disminución en la acumulación anormal de beta-catenina intranuclear, así como de proliferación tumoral. Adicionalmente, SFRP1 favo reció la expresión de E-cadherina a través de la inhibición de SLUG, TWIST y SNAIL, que son factores de transcripción invo lucrados en el proceso de transición epitelio-mesénquima9.
PTPRN es un gen que participa en varios procesos biológi cos, tales como proliferación, diferenciación y ciclo celular, además de procesos fisiológicos, como la función autóno ma, equilibrio agua-sal, reproducción y conducta alimentaria, mediante la secreción de hormonas y neurotransmisores18. Por otra parte, también fue reportado como hipermetilado en ciertos tipos de cáncer, como carcinoma urotelial19, ova rio20 y está estrechamente relacionado con la tumurogénesis hepática21. En nuestro estudio, PTPRN mostró aumento en los niveles de expresión de mRNA en cinco de los seis casos que presentaron progresión de la lesión, lo cual fue observa do en el cáncer de pulmón de células pequeñas, asociándose con crecimiento y proliferación tumoral. Adicionalmente, se encontró que una expresión alta de PTPRN está fuertemente asociada con un mal pronóstico en pacientes con Glioblasto ma Multiforme22; pese a esto, los resultados no son conclu yentes debido a que un resultado esperado podría ser una disminución en la expresión de este gen.
CDX2 es un biomarcador de maduración y diferenciación del epitelio gastrointestinal. La baja expresión de CDX2 en los tumores de colon ha sido asociado con peor pronóstico de sobrevida23,24. Con respecto al cuello uterino, CDX2 ha sido detectado con mayor frecuencia por IHQ en tumores del ca nal endocervical con morfología usual de tipo endocervical y endometrioide25; no obstante, un estudio de adenocarci nomas cervicales invasivos encontró una rara expresión de CDX2, no pudiendo concluir su papel en este tipo de lesio nes. A pesar de que nuestro estudio no mostró diferencia significativa en los niveles de expresión de mRNA para los casos de progresión ni regresión, el análisis con inmuno histoquímica sí evidenció disminución en la graduación de la expresión de CDX2 en las muestras de progresión. Por lo tanto, podría plantearse la posibilidad que CDX2 en LIEAG, también le pudiera brindar cierto factor pronóstico favorable, que estaría representado en la regresión de la lesión. Es de anotar, que algunos biomarcadores no presentan correlación directa entre la expresión de mRNA y proteína, lo cual puede ser explicado por limitaciones técnicas o a procesos post-transcripcionales26.
Como limitaciones del estudio se encuentran el análisis de metilación de los biomarcadores evaluados, adicionalmente, realizar un estudio con un tamaño muestral de tipo proba bilístico y finalmente, lograr obtener muestras frescas de ce pillados cervicales con el fin de minimizar el riesgo en el de terioro de la muestra por el uso de fijadores como el formol.
Conclusión
En conclusión, nuestros resultados mostraron que SFRP1 dis minuye significativamente por IHQ en casos de progresión y aumento significativo en la expresión de ARN en casos de regresión, pudiendo ser considerado, por lo tanto, como un potencial biomarcador en las lesiones preneoplásicas del cuello uterino asociadas al VPH. Además, en el estudio de inmunohistoquímica, SFRP1 mostró adecuada correlación al comparar la expresión obtenida por RT-PCR.