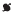Las enfermedades transmitidas por los alimentos (ETA) representan un problema para la salud pública mundial, siendo la causa más común la infección bacteriana; se estima que aproximadamente 76 millones de casos ocurren en Estados Unidos cada año 1 y generan un considerable costo económico 2. Se ha descrito a Salmonella spp., Listeria monocytogenes, y Yersinia enterocolitica entre los principales agentes etiológicos de las ETA. Salmonella se clasifica en más de 2 500 serotipos patógenos, de los cuales los serotipos Enteritidis y Typhimurium se constituyen como los más comunes a nivel mundial y la infección resulta de la ingestión de carne, cerdo, huevos o leche contaminados con esta bacteria 3. L. monocytogenes es transmitida principalmente por alimentos contaminados; asociados al consumo de leche, alimentos procesados y carnes, entre otros. Clínicamente la infección por Listeria incluye gastroenteritis febril, meningitis, bacteriemia y puede llegar a ser fatal 4,5. La infección por Yersinia, se ha hecho más frecuente en los últimos años debido a la transmisión al humano por el consumo de animales de granja que pueden ser fuente de infección. La patogénesis es una infección aguda que se inicia en el intestino y continúa con la infección en los nódulos linfáticos 6.
La rápida identificación del agente causal de brotes de ETA es importante en la disminución de la morbimortalidad, y reduce los costos económicos asociados a las enfermedades transmitidas por los alimentos. Los métodos convencionales de cultivo son laboriosos y demandan de mucho tiempo para emitir un resultado (aproximadamente tres a cinco días), además el transporte de muestras afecta la conservación de microorganismos viables en la muestra, lo cual puede verse reflejado en una baja sensibilidad. El desarrollo de técnicas diagnósticas moleculares como la reacción en cadena de la polimerasa en tiempo real (PCR-TR) permite mayor eficiencia, sensibilidad y rapidez en el tiempo de detección de microorganismos en muestras clínicas y de alimentos frente a los métodos convencionales, superando la detección de casos positivos obtenidos por cultivo 7.
En Colombia, el sistema de vigilancia ha notificado que la enfermedad transmitida por alimentos incrementó de 2 983 a 11 836 casos, desde el año 2000 al 2012, respectivamente 8. Sin embargo, estos datos no reflejan la incidencia real de las enfermedades transmitidas por los alimentos en Colombia debido a las limitaciones del sistema de información epidemiológica, en donde no todos los casos son notificados, y entre los notificados, algunos no son investigados. Considerando que la investigación de los brotes de enfermedades transmitidas por los alimentos es necesaria para identificar las posibles causas y factores de riesgo, para prevención y control de brotes, como objetivo este estudio aplicó una técnica de Reacción en Cadena de la Polimerasa múltiple en tiempo real (PCR-TR) para la detección de Salmonella spp., Listeria monocytogenes y Yersinia enterocolitica, como herramienta de apoyo diagnóstico en la vigilancia de brotes de enfermedad transmitida por alimentos.
MATERIALES Y MÉTODOS
Estandarización de la PCR-TR múltiple
Se emplearon como controles positivos cepas de L. monocytogenes ATCC 19115, S. Typhimurium ATCC 14028 y Y. enterocolitica ATCC 33114. El ADN genómico de cada bacteria fue extraído empleando el estuche Wizard (Promega, USA) de acuerdo a las especificaciones de casa matriz. El ADN fue cuantificado usando el espectrofotómetro NanoDroop ND-1000 (NanoDrop Technologies, Rockland, DE).
Se emplearon iniciadores y sondas Taqman para amplificar los blancos genéticos utilizados en la detección de Salmonella spp., gen invA9, L. monocytogenes (hly)10, y Y. enterocolitica (ail)11. Para la reacción de amplificación se empleó el estuche LightCycler 480 Probes Master (Roche Diagnostics, Mannheim, Germany) y se utilizó el equipo LightCycler 480 (Roche Diagnostics, Mannheim, Germany). Los volúmenes y concentraciones estandarizadas fueron: 10µl de Probes Master (Roche Diagnostics, Mannheim, Germany); 600 nM de iniciadores para Salmonella spp., 300 nM para L. monocytogenes y Y. enterocolitica; 400 ПМ de cada sonda; 0,2 µl de agua y 7,4 μl del extracto purificado del ADN de la muestra, para un volumen final de 20 μl por reacción. Los parámetros de termociclado fueron: un ciclo de preincubación a 95°C por 10 minutos, seguido de 42 ciclos de 95°C por 10 segundos, 58°C por 20 segundos, extensión de 72°C por 20 segundos y un ciclo final de enfriamiento de 3 minutos. La sensibilidad analítica se determinó mediante diluciones seriadas del ADN de las cepas utilizadas a partir de suspensiones de concentración conocida de 20 pg.
La especificidad analítica se realizó a partir de cepas bacterianas presentes en el tracto gastrointestinal, relacionadas filogenéticamente a nivel de género o especie, o productoras de enfermedades transmitidas por los alimentos.
Aplicación de la PCR-TR múltiple en estudio de brotes de ETA
La PCR-TR múltiple se empleó para la identificación del agente causal en cuatro brotes de enfermedad transmitida por alimentos, identificados en las ciudades de Cali (Valle del Cauca) y Pasto (Nariño) ciudades situadas a una distancia de 440 y 798 Km, respectivamente de Bogotá.
Las muestras clínicas se colectaron de las personas que cumplían con la definición de brote de ETA, que se define como un incidente por el que dos o más personas experimentan una enfermedad similar que resulta de la ingestión de un alimento común al inicio de los síntomas 12, posteriormente las muestras fueron enviadas a los respectivos Laboratorios de Salud Pública Departamentales (LSPD) en los medios de transporte Cary Blair (Salmonella spp., Y. enterocolitica) y Amies (L. monocytogenes), para la detección e identificación de bacterias por la metodología convencional de cultivo 13,14. Las muestras clínicas recolectadas se sembraron en caldos de enriquecimiento Selenito (Becton Dickinson) y Listeria Enrichment Broth Base (BLEB) (Becton Dickinson) y se incubaron a 37°c durante 18 y 24 horas respectivamente. Se tomaron alícuotas de los caldos y se procesaron siguiendo el Manual de Procedimientos para el Diagnóstico Bacteriológico de Enfermedad Diarreica Aguda e identificación de Salmonella spp., Shigella spp., Vibrio cholerae13. El volumen restante de cada caldo de enriquecimiento (Selenito y LEB) se centrifugó a 3500 rpm durante 10 minutos, el sedimento bacteriano se transfirió a un nuevo tubo de 1,5 ml y se almacenaron a -20°c para procesamiento por la metodología molecular. Posteriormente, los LSPD remitieron al Grupo de Microbiología del Instituto Nacional de Salud (INS) los sedimentos de las muestras de materia fecal enriquecidas para procesamiento por la metodología molecular y los aislamientos recuperados de las muestras clínicas para confirmación y serotipificación de acuerdo al esquema establecido de Kauffmann-White - Le Minor 13,14. En los casos en los cuales fue posible tomar muestras de alimentos, estos se procesaron en el LSPD y los aislamientos obtenidos fueron confirmados y serotipificados en el Laboratorio de Alimentos del Instituto Nacional de Vigilancia de Medicamentos y Alimentos (INVIMA) de acuerdo al Manual de Técnicas para el Control de Calidad Microbiológico de Alimentos para Consumo Humano 15.
En el grupo de Microbiología del INS, los sedimentos (Selenito y LEB) de cada paciente se mezclaron y procesaron como una sola muestra para la extracción y purificación de ADN empleando el estuche DNeasy blood and tissue (Qiagen Gmbh, Hilden, Germany) con modificaciones menores: ebullición a 95°c durante 10 minutos y volumen final de elución de la columna en 50µl. Estos aislamientos fueron testeados por la PCR-TR múltiple. En cada prueba se incluyeron controles negativos de extracción de ADN, reactivos, adición de ADN (uno por cada cinco muestras procesadas) y dos controles positivos.
Electroforesis en campo pulsado (PFGE)
A los aislamientos de Salmonella spp. obtenidos se les evaluó relación genética mediante la técnica de electroforesis en campo pulsado (PFGE) de acuerdo con la metodología estandarizada por la Red PulseNet, usando la enzima de restricción XbaI (Promega, USA) y la cepa S. Braenderup H9812 como marcador de peso molecular 16. En el análisis se utilizó el coeficiente de Dice, y el algoritmo UPGMA mediante el programa Gel Compare 2® V.4.0 (Applied Maths, Sint Martens-Latem, Belgium).
RESULTADOS
La sensibilidad analítica de la técnica de PCR-TR múltiple para L. monocytogenes fue de 20 fg (4 equivalentes genómicos), de 200 fg (40 eg) para Salmonella spp., y 200 fg (39 eg) para Y. enterocolitica. En el ensayo de especificidad analítica no se observó amplificación ni reacciones cruzadas con ninguna de las bacterias evaluadas.
Un total de 123 casos cumplieron con la definición de brotes de ETA, de los cuales 35 fueron confirmados por laboratorio y 88 fueron confirmados por nexo epidemiológico (Tabla 1). El primer brote se presentó en el mes de octubre de 2012 en el área urbana del municipio de Pasto (Nariño), el cual fue asociado al consumo de emparedado de pollo en un restaurante comercial. La tasa de ataque fue de 53 % (32/60) y ninguno de los casos requirió hospitalización. El 100 % de los casos presentaron: vómito, diarrea, fiebre, dolor abdominal, cefalea, malestar general. El grupo de edad más afectado fue el de 20 a 24 años (n=10). El período de incubación más corto y más largo fue 7 y 27 horas, respectivamente. Se procesaron 14 muestras de materia fecal por cultivo de las cuales se obtuvo un aislamiento (1/14) de Salmonella spp. En contraste, la metodología molecular identificó seis muestras positivas (6/14) para Salmonella spp. (Tabla 1). De los alimentos implicados en este brote se tomaron muestras de los ingredientes y se identificó Salmonella spp., en la muestra de salsa casera a base de huevo. El aislamiento clínico (INS-S.En2012) y el del alimento fueron serotipificados como S. Enteritidis. Por la técnica de PFGE, ambos aislamientos presentaron el mismo patrón electroforético identificado como COIN12JEGX01.0038, estableciendo una relación clonal entre los dos aislamientos y confirmando el origen del brote (Figura 1).
Tabla 1 Resultados de la aplicación de la PCR-TR múltiple frente al cultivo
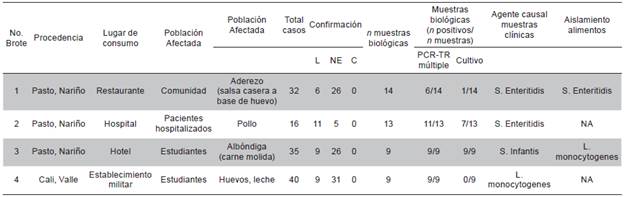
L: Confirmado por laboratorio; NE: confirmado por nexo epidemiológico; C: confirmado por clínica
El segundo brote ocurrió en el mes de agosto de 2013 en un hospital del área urbana del municipio de Pasto (Nariño), se asoció al consumo de dieta corriente y blanda. La tasa de ataque fue de 10 % (16/158). Los signos y síntomas presentados por los casos fueron: diarrea (100 %), fiebre (63 %), vómito (44 %) y deshidratación (31 %). Los grupos de edad más afectados fueron de 15 a 44 años (n=6) y de 45 a 64 años (n=6). El período de incubación más corto y más largo fue 9 y 25 horas, respectivamente. Se procesaron 13 muestras clínicas de materia fecal de las cuales por cultivo y serotipificación se identificó S. Enteritidis en 7 muestras (7/13), mientras que por PCR-TR múltiple se detectaron 11 muestras positivas (11/13) para Salmonella spp., incluyendo las identificadas por la metodología convencional (Tabla 1). No se recuperaron muestras de alimentos, sin embargo, de acuerdo a la encuesta a los consumidores la tasa de ataque por alimento consumido más alta fue para el pollo. Los aislamientos clínicos fueron analizados por la técnica de PFGE arrojando como resultado el patrón COIN13. JEG.X01.0038 (Figura 1).
El tercer brote sucedió en el mes de septiembre de 2013 y fue asociado al consumo de albóndigas en un hotel del municipio de Pasto (Nariño). La tasa de ataque fue de 22.4 % (35/156), ninguno de los casos requirió hospitalización. Los signos y síntomas presentados por los casos fueron: diarrea (71 %), dolor abdominal (54 %), nauseas (46 %), vómito (29 %) y fiebre (23 %). El grupo de edad más afectado fue el de 5 a 14 años (n=25). El período de incubación más corto y más largo fue 2 y 12 horas, respectivamente. Se procesaron 9 muestras que fueron positivas por ambas metodologías (9/9) para Salmonella spp., y serotipificadas como S. Infantis. Los aislamientos clínicos se evaluaron por la técnica de PFGE obteniendo como resultado un único patrón electroforético COIN13JFXX01.0001 (Figura 2). Se procesaron cuatro muestras de alimentos que contenían arroz, papa, pasta, carne (albóndiga) y pollo, identifican do en todas las muestras L. monocytogenes por ambas metodologías y en la muestra que contenía albóndiga se identificó Salmonella spp. por PCR-TR múltiple.
El último brote ocurrió en el mes de octubre de 2013, en el área urbana del municipio de Cali (Valle) en un establecimiento militar. La tasa de ataque fue de 9.6 % (55/570) y ninguno de los casos requirió hospitalización. Los signos y síntomas presentados por los casos fueron: diarrea (78 %), nauseas (75 %), vómito (69 %), deshidratación (67 %), cefalea (56 %), escalofrío (40 %) y fiebre (33 %). El grupo de edad más afectado fue el de 15 a 19 años (n=31). El periodo de incubación más corto y más largo fue 1 y 23 horas, respectivamente. No se procesaron muestras de alimentos, sin embargo, la tasa de ataque por alimento consumido más alta fue para huevos y leche. Se procesaron 9 muestras clínicas de materia fecal, por cultivo no se obtuvo ningún aislamiento (0/9) mientras que por la metodología molecular todas fueron positivas para L monocytogenes (9/9).
De acuerdo con los resultados del estudio, la metodología molecular empleada como herramienta de apoyo diagnóstico en brotes de ETA tuvo una sensibilidad del 77,7 % al identificar el agente causal en 35 muestras (35/45) frente a 37 % de sensibilidad de la metodología convencional donde solo en 17 muestras (17/45) se obtuvo aislamiento del agente bacteriano.
DISCUSIÓN
La aplicación de la PCR-TR múltiple para la detección de Salmonella spp., L. monocytogenes y Y. enterocolitica demostró ser una herramienta útil de apoyo diagnóstico, en la caracterización de brotes de enfermedades transmitidas por los alimentos, presentando mayor sensibilidad frente a la metodología convencional. También se logró evidenciar casos donde se obtuvo aislamientos de las muestras clínicas y de alimentos, la técnica de PFGE permitió confirmar la fuente del brote.
S. Enteritidis fue el principal agente causal identificado en los brotes estudiados. Esto concuerda con lo reportado en la literatura donde a nivel mundial, con excepción de Oceanía y Norte América, donde es clasificado como el serotipo más común en brotes de ETA con una proporción global del 43 % 17. El huevo y el pollo son fuentes frecuentes de infección y multiplicación de Salmonella spp. 17. Alimentos complejos que incluían huevo en su preparación, fueron relacionados como fuente de S. Enteritidis en brotes de ETA en Estados Unidos 18 y en Latinoamérica, información epidemiológica de veinte países obtenida del Regional Information System on foodborne diseases surveillance, implicaron a la carne, el pollo, el cerdo y el huevo como las principales fuentes de brotes por Salmonella spp. 19.
La tipificación serológica y molecular de los microorganismos es importante en la investigación de brotes de ETA porque permite determinar la fuente de infección que facilita la toma de medidas de control y prevención de futuros brotes. En Colombia, los aislamientos de Salmonella spp. recuperados de pacientes, se remiten al Laboratorio de Nacional de Referencia en el INS para serotipificación y tipificación molecular por PFGE como parte del programa PulseNet de Latino América y el Caribe. El patrón electroforético COINII. JEG.X01.0038 se identificó en dos brotes por S. Enteritidis, el cual se ha identificado como el segundo patrón más frecuente (23,5 %) en el país. (Datos no publicados, Grupo de Microbiología, INS).
S. Infantis co1N12jEGX01.0001 recuperada de albóndigas fue la causa de uno de los brotes incluidos en el estudio. S. Infantis es un serotipo común en la producción ganadera y pollos de engorde, que se ha asociado en baja frecuencia con brotes y casos esporádicos de salmonelosis humana 20,21,22. A nivel mundial, la proporción de aislamientos de S. Infantis en humanos oscila entre 1,5 % a 2,2 % y representa el quinto serotipo más común en la Comunidad Europea 17. En Israel, aumento de 3.8 % en el 2004 a 44.3 % en 2009, convirtiéndose en el serotipo más frecuente y recuperado principalmente de brotes 23 y es el sexto serotipo en Colombia con una frecuencia del 1.8 % 24.
Los aislamientos de S. Infantis estudiados fueron genéticamente idénticos estableciendo un origen común. Los análisis de PFGE sugieren la dispersión de S. Infantis por un reducido número de clones identificados en humanos y animales. En Serbia se identificó un clon resistente a ácido nalidíxico y tetraciclina 25 y en Alemania, se determinó la existencia de clones epidémicos con una alta estabilidad en el tiempo 20. Esto sugiere la aparición y dispersión de clones específicos en cada país, sin embargo, por MLST se han identificado aislamientos con mismo tipo de secuencia en diferentes países lo que evidencia la diseminación internacional de algunos grupos clonales 22,26. Por lo tanto, la tipificación molecular no solo facilita la caracterización del brote, sino que permite la identificación y difusión de clones epidémicos de Salmonella spp. a nivel nacional e internacional.
En las muestras de alimentos asociadas al brote causado por S. Infantis, también se identificó L. monocytogenes, la cual se tomó como contaminante debido a que no fue recuperada de pacientes y a que este microorganismo es persistente en muestras ambientales de cocinas públicas y privadas 27. En Colombia, un estudio en manipuladores de alimentos cárnicos y lácteos, indicó una prevalencia de L. monocytogenes de 10,4 % (138/1322) e identificó como factor de riesgo el hecho de no practicar procedimientos de limpieza y desinfección adecuados (OR (1C 95 %)=1,292; p=0,005) 28. Otro factor que contribuye a la permanencia de L. monocytogenes en ambientes que entran o están en contacto con los alimentos, es la formación de biopelículas tanto en los equipos como en pisos, paredes, drenajes y tuberías, lo que dificulta su eliminación favoreciendo la re-contaminación del alimento 29,30.
L. monocytogenes se identificó como el agente causal de uno de los brotes. Debido a que la prevalencia de portadores en materia fecal de L. monocytogenes es de 1 % a 5 % 31, la identificación de este microorganismo en la materia fecal de nueve pacientes demuestra su implicación como agente causal del brote. Aunque con menor frecuencia y menor severidad la listeriosis también se puede presentar como gastroenteritis febril que normalmente es leve y autolimitada, no obstante, en algunas ocasiones la enfermedad invasiva puede ocurrir como una complicación de la enfermedad 31,32. Si bien no siempre se confirma, se ha informado en personas saludables con la aparición de fiebre, dolor muscular, dolor de cabeza, y diarrea que ocurren de 9-48 horas después de la exposición, pero hay reportes del periodo de incubación hasta de i0 días 31,33,34. L. monocytogenes se encuentra comúnmente en los alimentos, especialmente en los productos lácteos y cárnicos, y la ingestión de un gran inoculó de la bacteria se ha postulado como uno de los factores en la patogénesis de la enfermedad 31,34. Debido a que muchas personas no buscan atención médica y no es un microorganismo considerado en el diagnóstico de eta, es probable que se presente un subregistro de casos de enfermedad.
En cuanto a las limitaciones del estudio, no en todos los brotes se procesaron muestras de alimentos ni de superficies. De acuerdo con el protocolo de enfermedad transmitida por alimentos, se deben procesar junto a las muestras biológicas, muestras de alimentos y de las superficies que están en contacto con los alimentos, esto como parte de las acciones colectivas que se deben llevar a cabo para realizar una adecuada caracterización del brote 35.
Aunque en los brotes no se identificó a Y. enterocolitica, es importante contar con una herramienta molecular como la PCR para la detección de este patógeno, ya que es considerada como una de las causas frecuentes en ocasionar enfermedades transmitida por alimentos, principalmente en países de bajos y medianos ingresos, y las bajas tasas de recuperación de Y. enterocolitica pueden deberse a la sensibilidad limitada de métodos de cultivo 36. Adicionalmente, en nuestro país la vigilancia para L. monocytogenes y Y. enterocolitica no está establecida como obligatoria.
Se han desarrollado varios métodos basados en PCR a partir del cultivo de enriquecimiento para detectar bacterias causantes de ETA 37,38. Una revisión sistemática sugiere que ensayos de diagnóstico rápido, especialmente con pruebas de PCR múltiple para detectar varios patógenos simultáneamente, puede resultar una medida costo efectiva para el estudio de brotes de ETA 39. El impacto clínico de la identificación del patógeno en menor tiempo permite ofrecer un tratamiento oportuno al paciente, reduce el gasto de recursos de control de infecciones y es útil en el manejo comunitario de los brotes. Nuestros resultados aportan evidencia de la aplicabilidad de las técnicas moleculares en la caracterización de brotes de enfermedades transmitidas por alimentos. La introducción de PCR proporciona una herramienta útil para la detección rápida de los agentes patógenos en muestras clínicas y de los alimentos, que permite dar una respuesta más precisa y oportuna para la delimitación, tratamiento y prevención de los brotes por enfermedades transmitidas por alimentos