Introducción
La tortuga matamata, mata-matá o caripatúa, Chelus fimbriata (Schneider, 1783) es la especie de mayor tamaño de la familia Chelidae. Se distribuye en la cuenca amazónica (Bolivia, Brasil, Colombia, Ecuador, Perú y Venezuela), en el Orinoco (Colombia, Venezuela), las Guayanas (Guayana Francesa, Guyana, Surinam) y en la isla de Trinidad. En Colombia ha sido registrada para los departamentos del Amazonas, Arauca, Caquetá, Casanare, Guainía, Guaviare, Meta, Putumayo, Vaupés y Vichada (MoralesBetancourt y Lasso, 2012). Recientemente, estudios genéticos han demostrado que existe una marcada diferenciación genética entre individuos de matamata de la cuenca del río Orinoco y los que se distribuyen en la cuenca del Amazonas, siendo consideradas como unidades de manejo diferenciales (Amaya et al., 2016).
Esta especie está categorizada a nivel global y nacional como Preocupación Menor (LC). En Colombia se justifica esta categoría porque es una especie con amplia distribución y abundante, aunque es objeto de tráfico de neonatos y juveniles para la venta como mascotas (MoralesBetancourt et al., 2015). Su consumo es de subsistencia y ocasional por algunas comunidades indígenas, además de ser objeto de usos medicinales (Morales-Betancourt y Lasso, 2012). Al ser una especie que no tiene categoría de amenaza a nivel global, el comercio internacional no está regulado por la Convención sobre el Comercio Internacional de Especies Amenazadas de Fauna y Flora Silvestres-Cites, sino por la normativa de cada país. El comercio y tráfico de esta especie se sustenta en que es una de las tortugas más apetecidas a nivel internacional, como se observa en las ofertas de las redes virtuales y páginas web.
Colombia es objeto de tráfico de especies, y entre 2005-2009 se decomisaron unos 224000 animales vivos, donde los reptiles fueron la clase dominante. Dentro de estos, las especies más frecuentes fueron la tortuga hicotea (Trachemys callirostris), la babilla (Caiman crocodilus) y la iguana verde (Iguana iguana) (MADS, 2012). En este mismo estudio se menciona que el decomiso de matamatas proviene del departamento del Meta. Esta especie de acuerdo con la información disponible en los últimos años y basada principalmente en los decomisos de Corpoamazonía (2013, 2014, 2015, 2016, 2018), parece mover volúmenes importantes de tráfico.
Los decomisos de 2015 y 2016 realizados por Corpoamazonía correspondieron a tortugas incautadas en Leticia, departamento del Amazonas. En ambas circunstancias, las tortugas fueron incautadas a personas que las transportaban en moto después de recogerlas en el aeropuerto. Las tortugas estaban en cajas de cartón, hacinadas y en muy mala condición. En la incautación de 2016 el transportador ilegal aseguró que los animales provenían del vuelo de Villavicencio (Meta), en la cuenca del Orinoco.
Con estos antecedentes, el objeto del presente artículo es presentar el reporte del proceso desde el decomiso de las tortugas hasta su liberación, utilizando herramientas moleculares para determinar el lugar de origen de las tortugas y las posibles áreas de liberación. También se muestra la necesidad del trabajo articulado entre las diferentes instituciones del Sistema Nacional Ambiental de Colombia (Sina), las ONG y la Academia para atender estas situaciones.
Materiales y métodos
Muestras
Las tortugas matamata decomisadas fueron neonatos y en la mayoría de los casos los tortuguillos aún mostraban la cicatriz umbilical, los caparazones estaban blandos y presentaban manchas negras en las cúspides de los escudos costales, con plastrón de color rojizo. Las muestras genéticas se obtuvieron de los animales que fallecieron (Figura 1).
Estudio genético
Toma de muestras. A un grupo de 30 individuos decomisados en 2015 y 2016 se les tomó una muestra de piel para realizar los análisis genéticos. Estos ejemplares correspondían a los individuos que habían muerto y permanecían congelados en las instalaciones de Corpoamazonía. A cada individuo se le cortó una de las carnosidades o verrugas de la piel del cuello para obtener una muestra de tejido de aproximadamente 1cm3, el cual fue sumergido en etanol al 70 %, y etiquetado con un código único asignado. Los tubos con cada una de las muestras fueron trasladados al Laboratorio de Ecología Molecular de Vertebrados (LEMVA) de la Universidad de Los Andes. Las colecciones de referencia se encuentran depositadas en el Banco de Tejidos del Instituto Humboldt y en el LEMVA.
Extracción de ADN, reacciones de PCR y secuenciación. De las 30 muestras, 15 fueron analizadas en el 2015 y 15 en el 2016. Estas fueron procesadas en el laboratorio para extraer y purificar su ADN total por medio del kit comercial Invisorb® Spin Tissue Mini Kit (Stratec). Una vez hecho esto, se verificó la calidad de los productos por medio de electroforesis en geles de agarosa al 1 % y por cuantificación con espectrofotometría, con el dispositivo Nanodrop 2000 (Thermo-Scientific).
Algunos meses antes de realizar el primer decomiso, se inició la construcción de una base de datos genéticos para esta especie a partir de muestras provenientes de localidades conocidas del Amazonas y Orinoco colombiano. Debido a que no se han publicado estudios de genética poblacional sobre esta especie, fue necesario diseñar primers específicos para amplificar dos regiones del genoma mitocondrial. Para esto, se tomó como referencia el genoma mitocondrial completo de la especie, publicado en GenBank bajo el número de acceso HQ172156.1 (Wang et al., 2012) y se diseñaron dos juegos de primers o cebadores utilizando el software Primer3Plus (Untergasser et al., 2012). La especificidad de cada pareja de primers se verificó mediante la herramienta Primer-BLAST de NCBI (Ye et al., 2012).
Una vez se diseñó y se comprobó la especificidad y el éxito en la amplificación utilizando los primers diseñados, se llevaron a cabo las reacciones de PCR en volúmenes de 32 uL con las siguientes concentraciones de reactivos: 2 uL de ADN genómico, buffer de reacción 1X, BSA 0,4 ug/uL, MgCl2 (Bioline) 5 mM, dNTP´s (Bioline) 0,1 uM; 0,3uM de cada uno de los primers F y R y 0,5 U de BiolaseTM DNA Plymerase (Bioline). En el termociclador MyCyler Thermal Cycler (BioRad), el perfil térmico utilizado para las 2 regiones del ADN fue el siguiente: desnaturalización inicial a 94°C por 2 minutos, 35 ciclos de 94°C por 30 segundos, 55°C por 45 segundos y 72°C por 40 segundos y una extensión final a 72°C por 10 minutos. El éxito de las amplificaciones se verificó por medio de electroforesis en geles de agarosa al 1,5 %. Aquellos productos que tuvieron una amplificación de buena calidad se enviaron al Laboratorio de Secuenciación de la Universidad de Los Andes, donde fueron purificados con el kit comercial Exo-SAP-IT® (Affymetrix) y secuenciados por el método de Sanger en el analizador genético ABI PRISM 3500® (Applied Biosystems) (Tabla 1).
Análisis molecular. Todas las secuencias obtenidas (60, 2 en cada dirección, “forward y reverse”), se editaron en el software Geneious versión 8.1.8 (Kearse et al., 2012), con el fin de remover las secciones de baja calidad y determinar con certeza los nucleótidos correspondientes a cada una de las posiciones. Las secuencias editadas fueron luego alineadas entre sí con el programa MacClade (Maddison y Maddison, 2005), el cual permitió, además, eliminar todas las secuencias redundantes para establecer los haplotipos existentes en el set de datos. Las secuencias obtenidas de las muestras de los decomisos fueron comparadas con una base de datos de secuencias para estos fragmentos de genes de tortugas matamata, colectadas en diversas localidades del Orinoco y Amazonas colombiano, así como con secuencias de muestras de Perú y Guayana Francesa (localidad tipo de la especie). Esta base de datos fue construida con información colectada por los autores, como parte de una tesis de maestría (Amaya et al., 2016).
Liberación de individuos decomisados
Una vez identificado el sitio de origen de los individuos decomisados, se procedió a su liberación. Los individuos fueron transportados a Puerto Carreño, departamento de Vichada, para posteriormente ser liberados en las áreas seleccionadas.
Los individuos a liberar y los fallecidos se pesaron previamente. Las variables morfométricas medidas (cm): (a) longitud recta del caparazón (LRC), medida con calibrador desde la margen anterior del escudo nucal hasta la margen posterior del caparazón en la unión entre los escudos marginales 12 y (b) ancho del caparazón (AC), medido entre las suturas de los marginales número 6 y 7 a cada lado del caparazón (RuedaAlmonacid et al., 2007).
Para poder hacer seguimiento (sobrevivencia, crecimiento, entre otros) a las tortugas, la mitad de los animales liberados se marcaron mediante muescas en las escamas marginales del caparazón, utilizando la modificación del código de Cagle (1939) propuesto por Rueda-Almonacid et al. (2007), donde las escamas del caparazón tienen valores asignados que al ser sumados igualan el número del ID del individuo; las muescas se realizaron utilizando unas tijeras, ya que el caparazón estaba blando (Figura 2).
Resultados
Análisis moleculares
En la tabla 1 se muestran los primers seleccionados para la amplificación.
Tabla 1 Diseño de primers: secuencias de primers o cebadores diseñados para amplificar fragmentos mitocondriales de Citocromo Oxidasa I (COI) y Región Control (RC) en Chelus fumbriata.
| Gen mitocondrial | Longitud del fragmento (pares de bases, pb) | Secuencia (5’-3’) |
|---|---|---|
| Citocromo Oxidasa I (COI) | 597 pb | F 5’-ACCTAACCATCTTTTCCCTACACC-3’ R 5’-CTAGGGCTCATAGTATTGGAGCAT-3’ |
| Región Control (RC) | 559 pb | F 5’-AACTACCTTCTGATCCTCGACTTC-3’ R: 5’-GTATGTTCAGCTTTAAAACGAATGGGT 3’ |
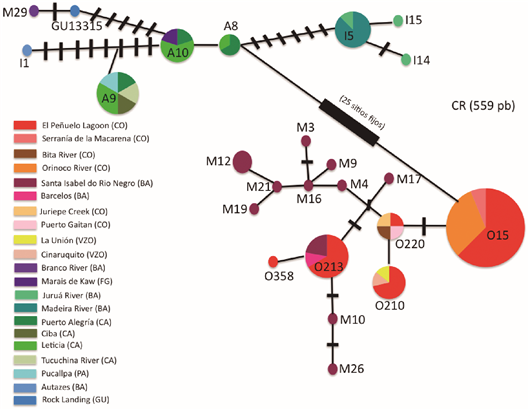
Asignación poblacional. En el análisis de 2 fragmentos, 572 pares de bases (pb) de COI y 521 pb de Región Control del ADN mitocondrial, se estableció que las primeras 15 muestras analizadas en 2015 presentaban un único haplotipo tanto para el gen COI como para la región control. Este haplotipo, identificado como O15 fue el más común identificado en la cuenca del Orinoco, con alta frecuencia alrededor de la zona de Puerto Carreño. Dicho haplotipo no se encontró en la cuenca del Amazonas y se diferencia por 11 sitios fijos en el COI y por 42 sitios fijos de todos los haplotipos encontrados en la cuenca del Amazonas. Posteriormente, al analizar las 15 muestras colectadas del decomiso del 2016, se observó que todas compartían este haplotipo O15, lo cual indica nuevamente que el origen de los animales decomisados era la cuenca del Orinoco. En la base de datos con la que se realizaron las comparaciones (Amaya et al., 2016), se identificaron varios haplotipos diferentes en la cuenca del Orinoco, seis para COI y cinco para RC. Sin embargo, el haplotipo O15, identificado en todos los individuos muestreados de los decomisos, es un haplotipo común (Figuras 3-4), presentando alta frecuencia en muestras de la Laguna el Pañuelo (Reserva Natural Privada Bojonawi) y en el río Orinoco en las cercanías a Puerto Carreño.
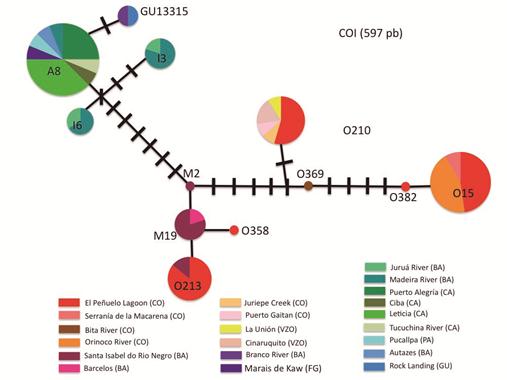
Figura 3 Red haplotípica para haplotipos identificados en Chelus fimbriata al analizar un fragmento de 597 pares de bases (pb) del gen mitocondrial Citocromo Oxidasa 1.
Liberación de tortugas
Una vez confirmado el origen de las tortugas, se realizó un trabajo articulado entre las diferentes instituciones involucradas, con el propósito de liberar los individuos que aún se encontraban en Leticia. Los individuos fueron transportados a Puerto Carreño, donde se mantuvieron cautivas por tres semanas para su aclimatación en la Reserva Natural Privada Bojonawi (RNP Bojonawi). Estos se ubicaron en estanques construidos en cemento y ladrillo, ubicados en el exterior, pero con protección del sol. Dichos estanques habían sido construidos por la Fundación Omacha para el mantenimiento de tortuguillos de Podocnemis expansa. Los estanques tenían las siguientes medidas: diámetro 3 m y profundidad 1,2 m; estanque cuadrado con 1,2 m por lado y 0,7 m profundidad (Figura 5). Ambos estanques fueron llenados hasta una altura de 30 cm de agua, la cual se cambió dos veces por semana.

Figura 5 Estanques donde se mantuvieron las tortugas matamata durante la aclimatación en la Reserva Natural Privada Bojonawi, Puerto Carreño. Foto: Carlos A. Lasso.
Los tortuguillos fueron alimentados con carnada viva (peces de la familia Characidae, fundamentalmente).
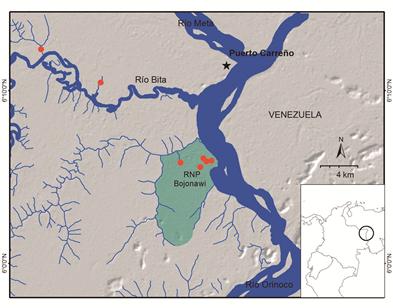
Posteriormente, fueron liberados 358 tortuguillos en los sitios seleccionados: el río Bita y la RNP Bojonawi, donde a partir de reconocimientos previos, se identificaron hábitats idóneos para esta especie (Tabla 2, Figuras 6 y 7). Los individuos liberados tuvieron como medidas promedio: 6 cm (intervalo 4-7,8 cm) de longitud recta del caparazón, 4,5 cm ancho de caparazón (intervalo: 3,2-6,4 cm) y un peso de 25,2 g (intervalo: 15-55 g).
Se marcaron el 50 % de los individuos liberados.
Tabla 2 Áreas y número de tortugas matamata liberadas en el río Bita y la Reserva Natural Privada Bojonawi.
| Fecha | Número de individuos | Nombre del sitio | Coordenadas |
| Río Bita | |||
| 03-mayo-16 | 100 | Caño Mosco | 6°5’32,9’’N / 67°30’7,5’’O |
| 03-mayo-16 | 90 | Caño Tigre | 6°10’29,8’’N / 67°35’55,7’’O |
| Reserva Natural Bojonawi | |||
| 05-mayo-16 | 34 | Laguna El Pañuelo (Puente del Babo) | 6°6’8,3’’N / 67°29’12,8’’O |
| 05-mayo-16 | 25 | Caño Temblón | 6°5’48,9’’ N / 67°29’51,3’’O |
| 05-mayo-16 | 34 | Caño Charapa | 6°5’32,9’’ N / 67°30’7,5’’O |
| 05-mayo-16 | 25 | Laguna El Pañuelo (extremo sur) | 6°5’31,6 N / 67°29’58,7’’O |
| 05-mayo-16 | 50 | Caño Verde | 6°3’57,3’’ N / 67°29’49,8’’O |
| Total | 358 |
Discusión
Estudios previos realizados en el Laboratorio de Ecología Molecular de Vertebrados Acuáticos (LEMVA) de la Universidad de Los Andes, en conjunto con el Instituto Humboldt en 2015 y usando la metodología ya descrita, demostraron que existe una marcada diferenciación genética entre individuos de matamata de la cuenca del río Orinoco y los de la cuenca del Amazonas (Amaya et al., 2016). Dichos estudios encontraron que en un fragmento de 572 pb del gen COI, 11 de los 29 sitios variables presentes, correspondían a diferencias fijas entre las 2 cuencas, mientras que en un fragmento de 512 pb de la región control, de 53 sitios variables, 42 son diferencias fijas. Al comparar la secuencia del haplotipo correspondiente a las tortugas decomisadas (2016) con los haplotipos establecidos en este estudio previo (2015), se encontró que era idéntica a uno de los ya muestreados y detectados en la cuenca del Orinoco. El haplotipo de las secuencias decomisadas es un haplotipo común encontrado con alta frecuencia en muestras de la Laguna El Pañuelo en la RNP Bojonawi y en el río Orinoco en las zonas cercanas a Puerto Carreño. Este haplotipo también fue encontrado en menor frecuencia en individuos colectados en la subcuenca del río Guayabero, afluente del río Guaviare, sierra de la Macarena (Meta), lo cual sugiere que es un haplotipo común en la cuenca del Orinoco. Esta evidencia molecular, a la luz del resultado de una diferenciación clara entre cuencas establecido en el estudio de Amaya et al. (op. cit.), permitió determinar la procedencia real de los neonatos decomisados y, a su vez, dirigir esfuerzos para su traslado y liberación a la vida silvestre.
El proceso de liberación de individuos mantenidos en cautiverio a poblaciones silvestres es un tema objeto de mucho debate en la biología de la conservación. Se considera que es importante tener en cuenta varios aspectos biológicos, ecológicos y moleculares, al momento de tomar decisiones con el fin de evitar afectar tanto a las poblaciones locales, como a los individuos que serán liberados (Lindenmayer y Burgman, 2005). En particular, a nivel molecular la cercanía genética entre ambos grupos debe ser una prioridad, pues existe el riesgo de inducir lo que se conoce como depresión por exogamia, que consiste en la dilución de adaptaciones locales como consecuencia de cruces con individuos que carecen de ellas (Edwards y Berry, 2013; Lindenmayer y Burgman, 2005). Sería importante a futuro realizar análisis genéticos complementarios para estudiar posibles adaptaciones locales en estas tortugas a cuencas particulares. Sin embargo, este estudio, así como la tesis de Amaya et al. (2016), son pioneros en intentar contestar preguntas complejas sobre la estructura genética poblacional de esta especie. Todo esto podría indicar que es necesaria más información de la matamata, no solo a nivel biológico y ecológico, sino también molecular, para asegurar que la zona seleccionada para la liberación sea la más idónea. No obstante, los resultados mostrados por estudios previos (Amaya et al. op. cit.), respecto a la diferenciación genética entre las matamatas del Orinoco y las del Amazonas, además del hecho de que el haplotipo mostrado por las tortugas decomisadas correspondió al encontrado con mayor frecuencia y con la distribución más amplia en el Orinoco en dicha investigación, determinó que la asignación de estas a una población en la cuenca del Orinoco fuera altamente confiable. El hecho de que este haplotipo O15 fuera el más común en la RNP Bojonawi y en alrededores del río Orinoco cerca de Puerto Carreño, permitió, de manera conservadora, utilizar esta información para proceder a la liberación en este lugar, donde además se cuenta con un área protegida y donde se puede hacer el monitoreo de los animales liberados. Para otros grupos taxonómicos que presenten una menor diferenciación a nivel genético entre sus poblaciones y para los cuales hay disponible más información de tipo molecular, es recomendable implementar metodologías más especializadas para realizar la asignación de los individuos a poblaciones locales, tales como las que ofrece el software Structure para marcadores moleculares microsatélites (Pritchard et al., 2000).
Existen antecedentes del tráfico y comercio nacional con extracción de individuos provenientes de la Orinoquia, en los ríos Inírida, bajo Guaviare y Orinoco (Castaño-M., 2002; Morales-Betancourt y Lasso, 2012) y en el departamento de Casanare (Alfaro et al., 2011). En todos los casos como en la Amazonia, el modus operandi fue transportarlas de manera ilegal en los embarques de peces ornamentales. Sumado a esto, está el factor de la demanda internacional y el hecho de que en Perú es legal la comercialización de fauna silvestre (Ley forestal y de fauna silvestre N° 29763). Este aprovechamiento se puede dar siempre y cuando la actividad esté enmarcada en un plan de manejo aprobado por la autoridad regional forestal y de fauna silvestre correspondiente. Así, con en el fin de beneficiar a las comunidades ribereñas de la Reserva Pacaya Samiria, se construyó un plan de conservación de tortugas (Águila-Chávez et al., 2005), que en principio estaba dirigido a Podocnemis expansa y P. unifilis, pero se incluyó igualmente a Chelus fimbriata. Para 2013 se reportaron ingresos para el Estado de Loreto en Perú por un valor de un millón de dólares como objeto de la comercialización de tortugas.
La posibilidad de comercialización legal de tortugas desde Perú puede influir en que el tráfico ilegal en otros países movilice esta especie hacia ese país, y de allí a mercados internacionales.
Por lo enunciado anteriormente, no sorprende que las tortugas matamata capturadas en la Orinoquia colombiana sean transportadas a la Amazonia para hacerlas entrar al Perú. Esta situación hace que sea necesario que todas las autoridades ambientales regionales (corporaciones) donde se distribuye esta especie, tengan en la mira el corredor de tráfico hacia Perú, y que se cuente con un protocolo claro con herramientas moleculares para identificar la región de donde proceden los individuos, y facilitar posteriormente su liberación.
El tráfico de esta especie en Colombia continúa. Así, muy recientemente (principios de abril 2018), se decomisaron nuevamente en la ciudad de Leticia por parte de Corpoamazonia más de 1000 neonatos de matamata (C. Tamayo com. pers.).
Por último, es importante mencionar que en el protocolo para el manejo posdecomiso (Resolución N° 2064 de 2010, anexo 9), quedó establecido que, en el caso de realizarse una liberación de individuos decomisados al medio natural, es necesaria la aplicación de herramientas genéticas para determinar su origen, y, con esto, poder asignar el lugar de liberación. Esto solo es necesario según dicha Resolución para el caso de las especies amenazadas. Sin embargo, a la luz de los resultados de este trabajo se recomienda que el requisito debería extenderse a todas las especies, estén amenazadas o no, a fin de evitar los efectos negativos genéticos de introducciones (trasplantes) de haplotipos únicos o Unidades Evolutivas Independientes, fuera de su área de distribución natural.
Conclusiones y recomendaciones
La comercialización global de tortugas como mascotas está soportada por criaderos y programas de rancheo en muchos países. Sin embargo, el tráfico también se ha consolidado como una actividad lucrativa, especialmente en países del Neotrópico. Esta situación ha sido pobremente documentada en Suramérica a pesar del número importante de decomisos en zonas de frontera, particularmente en la Amazonia. En este sentido, este trabajo constituye uno de los primeros esfuerzos en analizar el tráfico de tortugas matamata abordando el origen de los especímenes a partir de herramientas de biología molecular.
Es evidente que hay tráfico de matamatas como parte de una red lucrativa, a tal punto de que se capturen tortugas en la Orinoquia y se hayan generado canales de transporte hacia el sur del país (departamento del Amazonas).
El trabajo cooperativo entre las corporaciones ambientales, la policía, ONG, los institutos de investigación del Sistema Nacional Ambiental y las universidades, ha sido fundamental para entender la dinámica del tráfico de esta especie en Colombia, y se consolida en la creación de una red de información con protocolos de manejo y análisis genético de procedencia geográfica de las especies que sean incautadas en el futuro.
Igualmente, se recomienda ampliar el análisis genético de tortugas matamata, especialmente con muestras de diferentes localidades del Amazonas y Orinoco, tal que permita posteriormente direccionar liberaciones en sitios de origen más específicos. Para esto es necesario desarrollar marcadores nucleares, tipo microsatélites o SNPs, que actualmente no se encuentran disponibles para esta especie. Finalmente, a la luz de los decomisos recientes, es prioritario contar con un protocolo, procedimientos adecuados y coordinar las acciones entre las instancias correspondientes, especialmente las corporaciones, para el manejo de decomisos de esta especie y plantear protocolos efectivos de manejo y de ser posible la liberación.