Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Universidad y Salud
Print version ISSN 0124-7107On-line version ISSN 2389-7066
Univ. Salud vol.18 no.2 Pasto May/Aug. 2016
MicroRNAs asociados al Cáncer de Cuello Uterino y sus lesiones precursoras: Una revisión sistemática
MicroRNAs associated with Cervical Cancer and its precursor lesions: A systematic review
Oliva Alexandra Guerrero-Gómez1, Milena Guerrero-Florez2
1 Tesista Biología Universidad de Nariño. Pasto, Colombia. e-mail: bioalexagg@udenar.edu.co
2 Magister Ciencias Microbiología. Estudiante Doctorado Biotecnología. Coordinadora Microbiología Proyecto Investigación - Universidad de Nariño. Grupo de Investigación en Salud Pública, Centro de Estudios en Salud de la Universidad de Nariño. Pasto, Colombia. e-mail: milenague@udenar.edu.co
Fecha de recepción: Noviembre 29 - 2015 / Fecha de aceptación: Junio 28 - 2016
Guerrero-Gómez OA, Guerrero-Florez M. MicroRNAs asociados al Cáncer de Cuello Uterino y sus lesiones precursoras: Una revisión sistemática. Univ. Salud 2016;18(1):345-363.
Resumen
Introducción: Los miRNAs tienen especial interés en oncología por su papel en el control de la expresión de genes reguladores del ciclo celular, alteración génica y su implicación en diferentes tipos de cáncer, tales como el cáncer de cuello uterino (CCU). Materiales y métodos: Se realizó una búsqueda sistemática de literatura científica, en bases de datos, donde establecieran asociación de miRNAs con CCU. Se analizó la localización genómica y cromosómica de miRNAs, la clasificación funcional, grupos de miRNAs al que pertenecen y su implicación en la progresión de este cáncer. Resultados: Se incluyeron 139 artículos científicos sobre miRNAs relacionados al CCU. Se identificó un total de 272 miRNAs, de los cuales 252 miRNAs con expresión diferencial en tejidos con CCU: 97 sobre-expresados, 88 infra-expresados y 67 miRNAs con perfiles de expresión variable. La mayoría de miRNAs asociados al CCU se encontraron en los cromosomas 1, 14, 19 y X, así como en regiones intrónicas e intergénicas. Se identificaron miRNAs en procesos asociados al control del ciclo celular. Conclusión: Con esta revisión se destaca la importancia de miRNAs como potenciales biomarcadores pronóstico y diagnóstico, se brinda una actualización sobre miRNAs asociados al CCU y sus lesiones precursoras y se genera un recurso de recopilación y consulta valioso para orientar futuras investigaciones de medicina molecular en este campo. Se recomiendan más estudios experimentales para esclarecer los mecanismos de los miRNAs en desarrollo del CCU.
Palabras clave: Cáncer de cuello uterino; microRNAs; biomarcadores; diagnóstico; biología molecular. (Fuente: DeCS, Bireme).
Abstract
Introduction: MicroRNAs (miRNAs) have special interest in oncology because of its role in the control of the expression of cell cycle regulatory genes, gene alterations and its involvement on different types of cancer such as cervical cancer (CC). Materials and methods: A systematic literature search was performed using different databases to establish relationship of miRNAs with UCC. Genomic and chromosomic location of miRNAs were analyzed along with its functional classification, miRNAs groups they belong to, and possible roles in progression of the disease. Results: 139 scientific articles about the role of miRNAs in CC were included. A total of 272 miRNA were identified, from which 252 had differential expression in cervical cancer tissue: 97 miRNAs were overexpressed, 88 miRNA infra-expressed and 67 miRNAs with variable expression profile. The majority of miRNAs associated with CC were found in chromosome 1, 14, 19 y X, and in intronic and intergenic regions. MiRNAs associated with process of cell cycle control were identified. Conclusion: This review emphasizes the importance of miRNAs as potential biomarkers of prognosis and diagnosis; it also provides an update on miRNAs associated to CC and its precursor lesions and a resource of compilation and consultation valuable to guide future research in molecular medicine in this field is generated. However, there is need to develop more studies in order to clear the mechanisms of miRNAs in cervical cancer.
Keywords: Cervical cancer; microRNAs; biomarkers; diagnosis; molecular biology. (Source: DeCS, Bireme).
Introducción
El CCU es un grave problema de salud pública a nivel mundial y constituye una de las neoplasias malignas más frecuentes en la población femenina1,2. Datos recientes muestran a este cáncer como uno de los más comunes en países de Centro y Sur América, África Oriental y el Sudeste Asiático3,4, y lo relacionan con factores socioculturales, el acceso a servicios de salud, programas de prevención, control y esquemas de detección temprana deficientes. Estudios clínicos, epidemiológicos y moleculares asocian la infección de los Virus del Papiloma Humano de alto riesgo oncogénico (VPH-AR) con el desarrollo de este cáncer5-8. Los tipos de VPH-AR más prevalentes en el mundo son 16, 18, 31, 33, 35, 45, 52 y 58, tales hallazgos han permitido formular recomendaciones para el diseño y uso de nuevas vacunas contra este virus.
La introducción de nuevas vacunas profilácticas contra VPH9, las nuevas técnicas moleculares para la detección de este virus y la utilización en tamizaje de rutina de la prueba de Papanicolaou10 han reducido notablemente la incidencia del CCU. A pesar de estos avances científicos y los esfuerzos por controlar su aparición, cada año miles de mujeres padecen y mueren a causa de esta neoplasia, forzando a reflexionar sobre la manera como se implementan los programas de control y se utilizan los esquemas de seguimiento para combatir esta enfermedad.
Nuevos enfoques de investigación buscan comprender los complejos mecanismos de alteración en cáncer a nivel molecular, creando la posibilidad de elaborar novedosas herramientas de caracterización y desarrollar terapias génicas dirigidas11 apoyando cada vez más el desarrollo de la medicina molecular personalizada. En este sentido, los microRNAs (miRNAs) pueden servir como potenciales blancos de pronóstico y diagnóstico de cualquier enfermedad, y poco a poco constituyen una de las áreas más novedosas y crecientes de investigación en la ciencia médica moderna.
Los miRNAs son un grupo de pequeños RNAs (19 a 25 nucleótidos de longitud), no codificantes, conservados evolutivamente, que participan en la regulación génica post-transcripcional12,13. Por tal razón, los miRNAs intervienen en procesos celulares importantes, como diferenciación celular, proliferación celular, apoptosis y el desarrollo del cáncer14. Alrededor del 50% de los miRNAs se localizan en regiones asociadas con cáncer, como sitios frágiles y en regiones de amplificación o perdida de heterocigosidad15. La importancia de los miRNAs en la oncogénesis obedece principalmente a su localización y a las alteraciones en los niveles de expresión de estos RNAs14,16,17.
La comparación de perfiles de expresión de miRNAs entre tejidos sanos y tejidos con cáncer, ha permitido establecer una asociación entre la expresión aberrante de miRNAs y el desarrollo del cáncer18-20. Se ha reportado alteración de los perfiles de expresión de miRNAs en diferentes tipos de cáncer tales como el de mama, cervical, de colon, pulmón, ovario y próstata21-23. Además, existen evidencias sobre el papel que tiene estos miRNAs en las etapas iniciales, de progresión y desarrollo del cáncer, lo que ha motivado estudiar el papel biológico de los miRNAs en el CCU, y sus lesiones precursoras conocidas como neoplasias intraepiteliales cervicales (NIC)24.
En la presente investigación se recopiló información científica publicada entre 2007 y 2015; con el objetivo de revisar aspectos celulares y moleculares de los miRNAs asociados al CCU. Se proporciona información sobre localización cromosómica y genómica, expresión diferencial y desregulación de miRNAs evidenciando el potencial papel de los miRNAs como biomarcadores y su potencial uso como herramienta diagnostica y pronostica en CCU.
Materiales y métodos
La presente revisión sistémica de literatura científica analiza miRNAs asociados al CCU y sus lesiones precursoras. La búsqueda de literatura se efectuó en las siguientes bases de datos: PubMed (www.ncbi.nlm.nih.gov/pubmed), PubMed Central (PMC) (www.ncbi.nlm.nih.gov/pmc), Springer Link (http://link.springer.com/) y Science Direct (http://www.sciencedirect.com/), empleando los siguientes términos de búsqueda: "microRNAs and Cervical Cancer", "miRNAs and Cervical Cancer", "microRNAs and Cervical Carcinoma" y "miRNAs and Cervical Carcinoma".
Se seleccionaron y analizaron artículos científicos publicados en inglés en el periodo comprendido entre los años 2007 y 2015. Como parámetros de calidad de cada publicación se tuvo en cuenta el factor de impacto de la revista, además de que cumplieran con alguna de las siguientes características: (1) perfiles de expresión de miRNAs en pacientes con CCU; (2) comparación entre especímenes con CCU, ya sea con el tejido normal adyacente cervical o tejidos cervicales normales de sujetos sanos; (3) miRNAs expresados diferencialmente entre estados cancerosos y normales; y (4) método para la validación de los resultados descriptivos. Se excluyeron los estudios de perfiles a partir de sueros de pacientes con CCU (dado que el objetivo fue analizar la progresión en CCU) y los artículos de revisión.
De cada estudio, se registró la siguiente información: autor, año de publicación, tipo y número de muestras, técnicas moleculares utilizadas, miRNAs presentes, localización cromosómica y genómica, clasificación funcional, expresión diferencial y desregulación de los miRNAs.
Resultados y Discusión
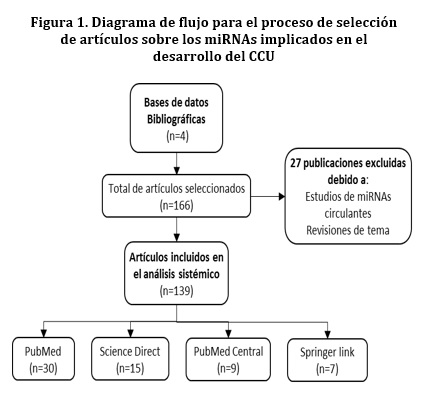
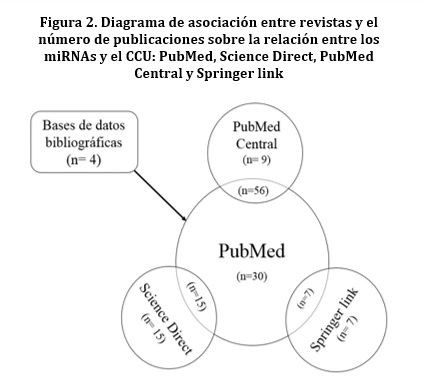
El proceso de selección de las publicaciones sobre la asociación de miRNAs con el desarrollo de CCU se resume en la figura 1; en la búsqueda sistémica de artículos, inicialmente se identificaron 166 publicaciones, sin embargo, 27 estudios fueron excluidos por tratarse de investigaciones sobre miRNAs circulantes, es decir, miRNAs que están presentes en diversos fluidos corporales como la sangre, y revisiones de tema.Los miRNAs asociados al CCU analizados en la presente investigación se obtuvieron de 139 publicaciones seleccionadas por cumplir con los requisitos de análisis, de las cuales el 21.59% de los artículos se obtuvieron de la Base de datos PubMed, seguido por Science Direct (10.79%), PubMed Central (6.47%) y Springer link (5.03%). En la figura 2 se indica la asociación entre el número de publicaciones y las bases de datos, así como también el número de divulgaciones científicas compartidas entre dos bases de datos: PubMed y PubMed Central compartieron el 40.28% de las publicaciones, PubMed y Science Direct el 10.79%, y finalmente, PubMed y Springer link comparten el 5.03%.
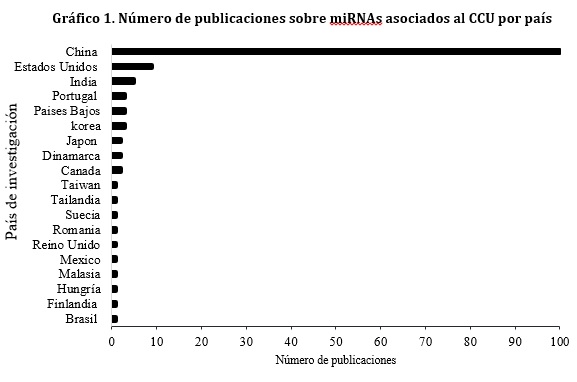
La literatura analizada ha sido publicada en diversas partes del mundo. En el gráfico 1, se muestra un 71.94% de los estudios provenientes de China, seguido de 6.47% en Estados Unidos y el 3.59% en India. Mientras tanto, hay 3 publicaciones de Corea, Países Bajos y Portugal y 2 publicaciones de Canadá, Dinamarca y Japón. Países como Brasil, Finlandia, Hungría, Malasia, México, Reino Unido, Romania, Suecia, Tailandia y Taiwán aportaron cada uno, una investigación sobre la asociación entre miRNAs y el desarrollo del CCU. En Latinoamérica son muy escasos los estudios sobre la implicación de los miRNAs con el desarrollo del CCU.
Para identificar miRNAs y determinar los perfiles de expresión de miRNAs entre tejidos con cáncer y tejidos normales del cuello uterino, los estudios analizados utilizaron una gran variedad de técnicas moleculares, tales como microarreglos, ensayo luciferasa, secuenciación, clonación y RT-qPCR (PCR cuantitativa con transcripción inversa).
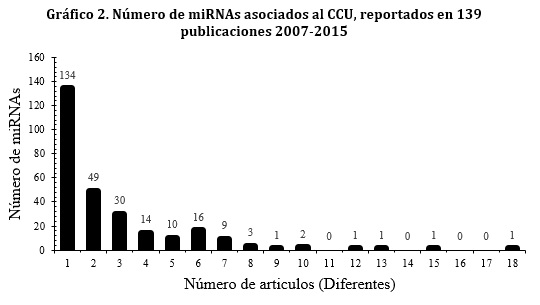
Los 139 artículos de investigación que cumplieron con los criterios de análisis describen 272 miRNAs implicados en el CCU, de los cuales, 134 miRNAs son reportados en un estudio cada uno, 49 miRNAs son reportados en dos estudios y 30 miRNAs son reportados en tres publicaciones.
Se encontró un miRNA (miR-21) como uno de los más frecuentes, reportado en 18 investigaciones diferentes, uno de los más importantes reportados en la literatura científica mundial por su relación con la aparición varios tipos de cáncer, entre ellos, cáncer de mama, colon y cuello uterino25-32 (Gráfico 2).
A continuación, se analizan ciertas características de los miRNAs asociados al CCU, entre ellas, localización genómica y cromosómica, grupos de miRNAs asociados al CCU y los perfiles de expresión en los tejidos cancerosos de cuello uterino.
Localización genómica: Los miRNAs pueden posicionarse en diversos lugares del genoma, incluyendo regiones intrónicas, exónicas e intergénicas33. La mayoría de miRNAs asociados al CCU se localizan en regiones intrónicas e intergénica: el 45.59% de estos miRNAs se encuentran en la región intrónica y el 44.85% en regiones intergénica, mientras tanto, 5.51% se localizan en exones (Tabla 1S); lo cual confirma lo reportado por Rodriguez et al. (2004), quienes encontraron que aproximadamente 40% de los miRNAs se encuentran dentro de intrones de genes codificantes de proteínas y menos del 10% en regiones de transcripción larga de ARN no codificante.
Localización cromosómica: Existen evidencias que establecen una relación entre la posición genómica de miRNAs y las regiones del genoma, cuya alteración o mutación se ha asociado con cáncer15. Reshmi et al. identificaron miRNAs en sitios frágiles del genoma relacionados con CCU, proporcionando un marco regulador sobre los miRNAs que puede tener un papel importante en el desarrollo de la oncogénesis de cuello uterino49, lo anterior permitió identificar la distribución de miRNAs sobre la base de la localización cromosómica en el CCU; los cromosomas 1, 14, 19 y X contienen más miRNAs que otros, en tanto, el cromosoma 18 contiene menos miRNAs lo cual obedece a su menor longitud (Tabla 1). Se identificó un gran número de miRNAs en el cromosoma 19, específicamente en las regiones 19p13 y 19q13.4. La región cromosómica 19q13.4 contiene el mayor grupo de miRNAs (conocida como el grupo de miRNAs en el cromosoma 19 "C19MC"). La región cromosómica 19q13 ha sido amplificada en varios tipos de cáncer, incluyendo el CCU50. Estudios previos han informado la asociación del cromosoma 1 con la transformación maligna en CCU y otros tipos de cáncer como de mama, colon e hígado51-53.
Grupos de miRNAs: Los miRNAs se distribuyen en los cromosomas, individualmente, o en grupos. Cuando se distribuyen en grupos, los miRNAs se localizan a corta distancia en el mismo cromosoma. Según la base de datos miRBase54,55, aproximadamente el 40% de los miRNAs se organizan en grupos. Los miembros de un grupo de miRNAs tienden a co-expresarse participando conjuntamente en diversos procesos fisiológicos y patológicos como el cáncer56.
Un caso típico, es el grupo miR-17-92, el cual ha sido reportado en varios tipos de neoplasias, como cáncer de colon, mama, pulmón, próstata, páncreas, tiroides, tracto gastrointestinal, leucemia, mieloma múltiple y linfomas de células B57-64.
Los miRNAs presentes en este grupo (miR-17, miR-18a, miR-19a/b, miR-20a, miR-92) se localizan en el cromosoma 13 y están sobre-expresados en el CCU, mostrando que el grupo miR-17-92 actúa como oncogén en este tipo de cáncer65-68.Hay pocos estudios que muestran el papel de los grupos de miRNAs en CCU. En 2013, Cai et al. identificaron el papel del grupo miR-302/367 en el bloqueo del crecimiento de las células cancerosas y la formación del tumor por la infra-regulación de las proteínas Ciclina-D1 y AKT1 y la sobre-regulación de las proteínas celulares inhibidoras p27 y p2169. En la tabla 2, se muestran 20 grupos de miRNAs implicados en el CCU e involucrados en varias vías de señalización celular, regulación de carcinogénesis, y que podrían ser útiles como blancos de diagnóstico, pronóstico o tratamiento de este tipo de cáncer70.
Hay evidencias sobre la asociación entre grupos de miRNAs y sus blancos de alteración, los cuales generalmente son genes que participan en funciones de crecimiento, migración e invasión de las células cancerosas51,65,67,68,71,72. No obstante, el mecanismo biológico de los grupos de miRNAs en el desarrollo y progresión del CCU es un campo de investigación emergente que requiere mayor estudio.
Perfiles de expresión de miRNAs: En esta revisión se identificaron 252 miRNAs expresados diferencialmente en cualquier etapa del tejido cervical normal y progresión a cáncer. De este total, 97 miRNAs están sobre-expresados y 88 miRNAs infra-expresados; 67 miRNAs mostraron perfiles de expresión variables probablemente en razón de las notorias diferencias en las muestras de la población analizada, condiciones y etapas del CCU seleccionados para cada estudio. (Figura 3)
Uno de los primeros estudios realizados sobre la implicación de miRNAs en CCU, se publicó en 2007, donde secuenciaron 166 miRNAs en tejido normal y tejido canceroso, encontraron seis miRNAs con perfiles de expresión diferencial; let-7b, let-7c, miR-23b, miR-143 y miR-196b con perfil infra-expresado en líneas celulares y tejidos cancerosos respecto a tejido normal y miR-21 se encontró sobre-expresado73. En otro estudio se analizaron 157 miRNAs obtenidos por RT-PCR e identificaron 68 miRNAs sobre-expresados y dos infra-expresados en células cancerosas, destacándose miR-9, miR-127, miR-133a, miR-133b, miR-145, miR-199a, miR-199b, miR-199s y miR-214 con un mayor incremento en sus perfiles de expresión, mientras miR-149 y miR-203 mostraron perfiles de expresión más bajos74.
Análisis de microarreglos, mostraron que miR-182, miR-183 y miR-210 sobre-expresados y miR-128, miR-143, miR-145 y miR-195 infra-expresados en el CCU, respecto a tejido normal44,75. Varios miRNAs se clonaron a partir de tejido normal, líneas celulares, tejidos infectados con VPH y tejidos con cáncer de los cuales, miR-15b, miR-16, miR-146a y miR-155 se encontraron sobre-expresados y miR-143, miR-145 y miR-128 infra-expresados22,76,77. Otro estudio mostró que miR-886-5p se sobre-expresa en los carcinomas de células escamosas de cuello uterino (SCC) en comparación con el tejido normal adyacente, mientras miR-302d, miR-346, miR-518b y miR-610 se infra-expresan78. Cuatro nuevos miRNAs (miR-1273f, miR-1273g, miR-5095 y MIR-5096) se han descubierto durante la búsqueda de los sitios frágiles relacionados con CCU, los cuales han sido observados en líneas celulares y tejidos cancerosos pero no en tejido normal49.
Conforme se han avanzado las investigaciones sobre miRNAs asociados al CCU, el número de miRNAs implicados en el desarrollo del CCU se ha incrementado considerablemente, donde se han descrito varios miRNAs sobre-expresados e infra-expresados en los tejidos cervicales cancerosos79-82; entre ellos, los miRNAs sobre-regulados: miR-10b, miR-15a, miR-16, miR-17, miR-20b, el miR-21, miR-93, miR-106a, miR-106b, miR-130b, miR-146b -5p, miR-155, miR-185, miR-195, miR-339-5p, miR-625, miR-941 y miR-1224-5p; y los infra-regulados: miR-99a, miR-100, miR-125, miR-139-5p, miR-139-3p, miR-145, miR-199a, miR-199b-5p, miR-149, miR-328, miR-375, miR-379, miR-381, miR-497, miR-574-3p, y miR-617, incluyendo los ARNm diana que tienen estos miRNAs y como contribuyen al desarrollo de este cáncer25,79,80,83-89.
El CCU se relaciona con la infección persistente de genotipos específicos de VPH, sin embargo, el conocimiento sobre todas las alteraciones genéticas y moleculares involucradas en esta patología requiere más estudios. Recientemente, se han identificado mecanismos moleculares para explicar el proceso tumoral y desarrollo del CCU90,91. La expresión aberrante de miRNAs ha sido reconocida como un mecanismo molecular importante que conduce a la carcinogénesis, dada la capacidad que tienen los miRNAs de regular la expresión de gran cantidad de genes implicados en procesos celulares importantes, e influir en la regulación de las vías de señalización relacionadas con el cáncer14,92,93.
En esta revisión, numerosos estudios muestran la correlación entre los miRNAs y la carcinogénesis de cuello uterino; mediante la comparación de los perfiles de expresión de miRNAs, entre tejidos cancerosos y tejidos cervicales normales, la identificación de miRNAs con perfiles de expresión alterados en los tejidos con cáncer de cuello uterino94-97, sugiriendo a la expresión aberrante de miRNAs como un factor importante en el desarrollo de este cáncer. No obstante, la función de la mayoría de miRNAs descritos en CCU continúa sin elucidarse. Lo anterior se podría atribuir en parte a inconsistencias en los perfiles de expresión de los miRNAs reportados en diferentes estudios (Figura 3)19, bien por el tipo de muestras analizado, antecedentes genéticos y ambientales de los donantes, características clínico-patológicas y las plataformas de análisis utilizadas para la interpretación de los perfiles de expresión de los miRNAs98. La expresión génica de cada miRNA es única de cada paciente, de cada tejido normal y canceroso de un mismo paciente y entre individuos, en este sentido, un estudio de SNPs (Polimorfismo de nucleótido único) podría servir de complemento a estos análisis.
Otra causa de variación en los perfiles de expresión de miRNAs se establece por la presencia de mutaciones cromosómicas tales como deleciones, inserciones, traslocaciones o la combinación de alteraciones genéticas y epigenéticas que conllevan a la sobre o infra-expresión de miRNAs99. Más del 50% de los miRNAs reportados en la literatura se localizan frecuentemente en regiones cromosómicas relacionadas con cáncer, tales como regiones no codificantes, regiones de pérdida de heterocigosidad, sitios frágiles comunes, regiones en o cerca de oncogenes o supresores tumorales, cerca de sitios de integración de VPH, los cuales son propensos a las aberraciones cromosómicas15. Por ello, la localización cromosómica de miRNAs es determinante en la patogénesis del cáncer.
De acuerdo con el patrón de expresión de miRNAs y el perfil global de expresión de miRNAs conocido como "miRNoma"16, se han identificado miRNAs que actúan como verdaderos oncogenes, o como supresores tumorales inhibiendo o estimulando la expresión de genes celulares relacionados con el desarrollo del cáncer y promoviendo o suprimiendo la oncogénesis, por lo que se han denominado oncomiRs100,101. Estas clases de miRNAs se han convertido en un objetivo atractivo para la terapia génica, pueden proporcionar potenciales biomarcadores para el diagnóstico o pronóstico de diferentes enfermedades humanas y representar nuevas dianas terapéuticas en cáncer96,102-104. En la carcinogénesis de cuello uterino, se han identificado miRNAs clasificados como oncomiRs100,101. Algunos miRNAs como miR-10a, miR-19a/b, miR-106b, miR-20, miR-21, miR,31, miR-133b, miR-135b, miR-141, miR-146, miR-148a, miR-182, miR-214 y miR-886-5p han sido considerados como oncogenes en CCU, pues regulan genes supresores de tumores y contribuyen a la progresión de esta neoplasia bien a través de la desregulación de productos génicos de proliferación celular, apoptosis o adhesión celular25,30,68,78,105-117. Entre tanto, miRNAs Let-7c, miR-124, miR-125b, miR-126, miR-138, miR-143 y miR-145 actúan como supresores de tumores en el desarrollo de este tipo de cáncer, regulan oncogenes y son infra-expresados en el CCU11,80,85,118-124. Lo anterior pone de relieve a la expresión aberrante de algunos miRNAs como un factor molecular activo en el desarrollo y la progresión de CCU.
Perfiles de expresión de los miRNAs en diferentes etapas del CCU (NIC I, II y III): Se han documentado las alteraciones en los patrones de expresión de algunos miRNAs durante la progresión de tejidos cervicales normales a lesiones intraepiteliales de alto grado (NIC II y III) o tejidos con cáncer invasivo90,125.
Li et al. (2011) mostraron miR-29a, miR-99a, miR-195 y miR-375 infra-expresados en NIC II y III, en contraste, miR-92a y miR-155 presentaron un patrón de sobre-expresión en NIC II y III19. Wang et al., demostraron una reducción constante y progresiva de miR-29a y miR-100 en NIC I - NIC III y CCU, mientras miR-16, miR-25, miR-27a, miR-92a y miR-378 tuvieron un perfil de expresión aumentado96. La participación de miR-375 en la progresión a cáncer aún no es clara dado que si bien disminuye su expresión en NIC II y III, no ocurre lo mismo en tejidos normales y en tejido con cáncer sugiriendo su participación diferencial en las etapas de progresión en CCU88,126,127.
Por tal razón, es necesario continuar estudiando los patrones de expresión de miRNAs que permitan dilucidar su participación en este tipo de cáncer. Otros estudios han demostrado perfiles de expresión diferencial entre tejido normal, lesiones precursoras (NIC) y tejidos con cáncer provenientes del cuello uterino19,20,87,93,102,128. Wilting et al. realizaron un análisis sobre los patrones de expresión de miRNAs entre tejidos cervicales normales, lesiones precursoras (NIC II, III) y tejidos cancerosos del cuello uterino, mostraron 33 miRNAs con perfiles de expresiones diferencial concordante; 18 miRNAs sobre-expresados (Let-7i, miR-19b, miR-21, miR-25, miR-28-5p, miR-30e, miR-34a, miR-34b*, miR-92a, miR-92b, miR-106b, miR-146a, miR-181d, miR-200a*, miR-206, miR-338-5p, miR-592 y miR-595) y 15 miRNAs infra-expresados (miR-23b, miR-134, miR-149, miR-193b, miR-203, miR-210, miR-296-5p, miR-365, miR-370, miR-493, miR-572, miR-575, miR-617, miR-622 y miR-638)51.
Durante esta revisión se identificó la asociación de varios miRNAs con etapas específicas del desarrollo del CCU. La tabla 3, muestra los perfiles de expresión de 59 miRNAs en diferentes lesiones intraepiteliales en la progresión del CCU. Se puede observar en la transición del tejido normal de cuello uterino a cualquier etapa del CCU (NIC I, II y III) se identificaron 10 miRNAs sobre-expresados (miR-10, -16, -21, -25, -92a, -93, -143, -196a, -378, -512), 11 infra-expresados (miR-26a, -29a, -99a, -100, -149, -199a, -210, -218, -342, -424, -497) y 3 miRNAs (miR-27a, -34a y -148a) con diferencias en los perfiles de expresión por cada lesión precursora del CCU. Por otro lado, se identificaron 35 miRNAs asociados a dos etapas precursoras de este cáncer. En las etapas NIC II y III, se identificaron 17 miRNAs sobre-expresados (miR-9, -15b, -18a, -19a, -19b, -20b, -34a, -106a, -106b, -125, -130a, -135, -146a, -155, 181b, -185, Let-7d) y 12 infra-expresados (miR-17, -29b, -99b, -124, -141, -181c, -192, -195, -223, -375, -449a, -494); mientras tanto, en las etapas NIC I y III se identificaron 2 miRNAs sobre-expresados (miR-142, -205) y 4 infra-expresados (miR-145, -203, -376a, -572).
Estos hallazgos sugieren que los miRNAs expresados diferencialmente en las etapas de desarrollo del CCU podrían jugar un papel importante en la transformación maligna de las células cervicales anormales, dado que la desregulación de estos miRNAs podría estar asociada con la progresión de esta enfermedad inducida por VPH de ato riesgo oncogénico de displasia leve a displasia severa o cáncer invasivo134. En razón de lo expuesto, los miRNAs específicos de cada etapa del CCU podrían ser utilizados en el seguimiento de la progresión a cáncer, bien para diferenciar entre las etapas del CCU, o el tejido cervical canceroso del tejido normal, por lo cual tendrían una nueva función como biomarcadores en el diagnóstico y la detección de este tipo de cáncer. Lo anterior nos proporciona un marco para futuros estudios en cohortes de pacientes independientes, y la posibilidad de validar los papeles funcionales de estos miRNAs en la carcinogénesis del cuello uterino.
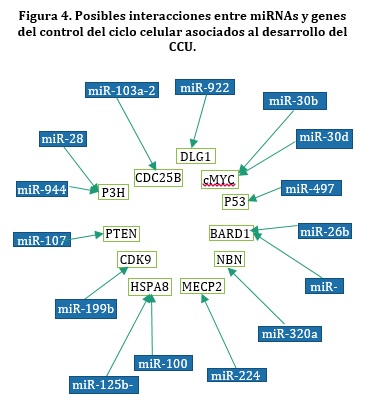
miRNAs durante el ciclo celular en el CCU: Se ha determinado la participación de algunos miRNAs en procesos celulares tan complejos como el control del ciclo celular; la modulación de los genes reguladores puede bloquear la progresión del ciclo celular87,93,135. La transición de la fase G1 a S en las líneas celulares de cuello uterino se ve afectada por miR-29a, miR-29b, miR-155, el grupo miR-302-367, miR-375 y miR-42452,69,126,136,137. La transición de fase S a G2 ha sido inhibida por la sobre-expresión de miR-372 en líneas de células del cuello del útero138 y la transición final de la fase G2 a M ha sido detenida por la infra-expresión de miR-100 en líneas de células del cuello del útero139. Se ha estimado que un 1-5% del genoma humano corresponde a miRNAs, reguladores de alrededor del 30% de los genes celulares codificantes. Cada miRNA puede regular la expresión de cientos de genes celulares y un mismo gen puede ser regulado por diferentes miRNAs140-142; si un miRNA especifico sufre algún tipo de alteración genética o epigenética podría causar variaciones significativas en la expresión de muchos genes celulares, provocando aberraciones en diversos procesos biológicos esenciales para la célula, conllevando al desarrollo y progresión de una amplia variedad de enfermedades humanas14.
En la figura 4, se indican las posibles interacciones entre 15 miRNAs asociados al CCU y 11 genes que participan en el control del ciclo celular. El conocimiento sobre los miRNAs implicados en la progresión del CCU, aquellos miRNAs que imparten efectos como supresores tumorales, reguladores de genes celulares y cuyo control depende de la interacción con genes celulares brinda un mejor entendimiento de los procesos biológicos que caracterizan el CCU y se convierten en potenciales herramientas para mejorar nuestra comprensión sobre los procesos celulares que intervienen en la carcinogénesis de cuello uterino. Así también permite nuevos enfoques para desarrollar sistemas de diagnóstico y pronostico en CCU o identificar blancos potenciales de terapia génica.
Conclusiones
En la presente revisión, se analizaron 139 artículos científicos que estudian la asociación de miRNAs con el CCU a través de diferentes técnicas moleculares, y los diferentes perfiles de expresión de miRNAs. Se identificaron 252 miRNAs con expresión diferencial en tejidos cancerosos de cuello uterino; de estos, 97 miRNAs están sobre-expresados y 88 miRNAs están infra-expresados. 67 miRNAs mostraron perfiles de expresión variables en diferentes estudios. La mayoría de los miRNAs asociados al CCU se encuentran en los cromosomas 1, 14, 19 y X, en regiones intrónicas e intergénicas.
Se han detectado ciertos perfiles de expresión de algunos miRNAs en líneas celulares del cáncer y las lesiones pre-malignas del cuello uterino, proporcionando información valiosa sobre el papel de los miRNAs en diferentes etapas de la carcinogénesis cervical, así como también podrían ser posibles marcadores para diferenciar entre las diferentes etapas de esta neoplasia.
En algunos estudios se ha evidenciado la expresión de moléculas importantes relacionadas con el cáncer, reguladas por la participación de miRNAs por lo cual, la identificación de tales genes celulares implicados y las vías de señalización involucradas, podría contribuir al desarrollo de productos terapéuticos basados en miRNAs, no obstante, se requieren más estudios destinados a esclarecer la función, objetivos transcripcionales y los mecanismos de regulación de eventos celulares. Así también analizar los miRNAs relacionados con eventos de apoptosis y necrosis y su posible interacción con secuencias del complejo CRISP (Clustered Regularly Interspaced Short Palindromic Repeats) tanto en tejidos sanos como tumorales del cuello uterino que podrían dar una mejor información sobre su papel como marcadores biológicos, así como en el pronóstico y la terapia del cáncer de cuello uterino.
Conflicto de interés: Las autoras declaran no tener ningún conflicto de interés.
Referencias
1. Ferlay J, Soerjomataram I, Dikshit R, Eser S, Mathers C, Rebelo M, et al. Cancer incidence and mortality worldwide: Sources, methods and major patterns in GLOBOCAN 2012. International Journal of Cancer. 2015;136:E359-86. [ Links ]
2. Torre L, Bray F, Siegel R, Ferlay J, Lortet-tieulent J, Jemal A. Global Cancer Statistics, 2012. Cancer journal clinical. 2015;65:87-108. [ Links ]
3. Sankaranarayanan R, Thara S, Esmy PO, Basu P. Cervical Cancer: Screening and therapeutic perspectives. Medical Principles and Practice. 2008;17(5):351-64. [ Links ]
4. Ferlay J, Shin HR, Bray F, Forman D, Mathers C, Parkin DM. Estimates of worldwide burden of cancer in 2008: GLOBOCAN 2008. International Journal of Cancer. 2010;127:2893-917. [ Links ]
5. Bosch FX, Lorincz A, Muñoz N, Meijer C, Shah KV. The causal relation between human papillomavirus and cervical cancer. Journal of clinical pathology. 2002;55(4):244-65. [ Links ]
6. Bosch FX, Qiao Y-L, Castellsagué X. CHAPTER 2. The epidemiology of human papillomavirus infection and its association with cervical cancer. International Journal of Gynecology & Obstetrics. 2006;94:S8-21. [ Links ]
7. Muñoz N, Bosch FX, de Sanjosé S, Herrero R, Castellsagué X, Shah KV., et al. Epidemiologic Classification of Human Papillomavirus Types Associated with Cervical Cancer. New England Journal of Medicine. 2003;348(6):518-27. [ Links ]
8. Muñoz N, Bosch FX, de Sanjosé S, Tafur L, Izarzugaza I, Gili M, et al. The causal link between human papillomavirus and invasive cervical cancer: a population-based case-control study in Colombia and Spain. International journal of cancer. 1992;52(5):743-9. [ Links ]
9. Joura E a, Giuliano AR, Iversen O-E, Bouchard C, Mao C, Mehlsen J, et al. A 9-Valent HPV Vaccine against Infection and Intraepithelial Neoplasia in Women. New England Journal of Medicine. 2015;372(8):711-23. [ Links ]
10. Nanda K, Mccrory DC, Myers ER, Bastian L a, Hasselblad V, Hickey JD, et al. Accuracy of the Papanicolaou Test in Screening for and Follow-up of Cervical Cytologic Abnormalities: A Systematic Review. Annals of Internal Medicine. 2000;132(10):810-9. [ Links ]
11. Phuah NH, In LL, Azmi MN, Ibrahim H, Awang K, Nagoor NH. Alterations of MicroRNA Expression Patterns in Human Cervical Carcinoma Cells (Ca Ski) toward 1'S-1'-Acetoxychavicol Acetate and Cisplatin. Reproductive Sciences. 2012;20(5):567-78. [ Links ]
12. Bartel DP. MicroRNAs: Genomics, Biogenesis, Mechanism, and Function. Cell. 2004;116:281-97. [ Links ]
13. Bartel DP. MicroRNA Target Recognition and Regulatory Functions. Cell. 2009;136(2):215-33. [ Links ]
14. Calin G, Croce C. MicroRNA signatures in human cancers. Nature reviews Cancer. 2006;6(11):857-66. [ Links ]
15. Calin G, Sevignani C, Dumitru C, Hyslop T, Noch E, Yendamuri S, et al. Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers. Proceedings of the National Academy of Sciences of the United States of America. 2004;101(9):2999-3004. [ Links ]
16. López-camarillo C, Marchat L a, Aréchaga-ocampo E, Azuara-liceaga E, Pérez-plasencia C, Fuentes-mera L, et al. Functional Roles of microRNAs in Cancer: microRNomes and oncomiRs Connection. Oncogenomic and Cancer Proteomics - Novel Approaches in Biomarkers Discovery and Therapeutic Targets in Cancer. 2013. p. 71-90. [ Links ]
17. Zhao X, Liu K, Zhu G, He F, Duval B, Richer J, et al. Original paper Identifying cancer-related microRNAs based on gene expression data. Bioinformatics. 2014;1-9. [ Links ]
18. Montufar M. Analisis de MiRNAs y sus genes blanco en lineas celulares del cancer de cervix. Universidad Autonoma de Nuevo León; 2013. [ Links ]
19. Li Y, Wang F, Xu J, Ye F, Shen Y, Zhou J, et al. Progressive miRNA expression profiles in cervical carcinogenesis and identification of HPV-related target genes for miR-29. Journal of Pathology. 2011;224(4):484-95. [ Links ]
20. Gocze K, Gombos K, Juhasz K, Kovacs K, Kajtar B, Benczik M, et al. Unique microRNA expression profiles in Cervical Cancer. Anticancer Research. 2013;33(6):2561-8. [ Links ]
21. Garzon R, Fabbri M, Cimmino A, Calin G a., Croce CM. MicroRNA expression and function in cancer. Trends in Molecular Medicine. 2006;12(12):580-7. [ Links ]
22. Wang X, Tang S, Le SY, Lu R, Rader JS, Meyers C, et al. Aberrant expression of oncogenic and tumor-suppressive microRNAs in cervical cancer is required for cancer cell growth. PLoS ONE. 2008;3(7):e2557. [ Links ]
23. How C. Characterization of Altered MicroRNA Expression in Cervical Cancer. Graduate Department of Medical Biophysics, University of Toronto; 2013. [ Links ]
24. Bosch FX, Castellsagué X, de Sanjosé S. HPV and Cervical Cancer: screening or vaccination? British journal of cancer. 2008;98:15-21. [ Links ]
25. Deftereos G, Corrie SR, Feng Q, Morihara J, Stern J, Hawes SE, et al. Expression of MIR-21 and Mir-143 in cervical specimens ranging from histologically normal through to invasive cervical cancer. PLoS ONE. 2011;6(12):e28423. [ Links ]
26. Liu S, Song L, Zhang L, Zeng S, Gao F. miR-21 modulates resistance of HR-HPV positive cervical cancer cells to radiation through targeting LATS1. Biochemical and Biophysical Research Communications. Elsevier Ltd; 2015;459(4):679-85. [ Links ]
27. Yao T, Lin Z. MiR-21 is involved in cervical squamous cell tumorigenesis and regulates CCL20. Biochimica et Biophysica Acta - Molecular Basis of Disease. Elsevier B.V.; 2012;1822(2):248-60. [ Links ]
28. Shishodia G, Shukla S, Srivastava Y, Masaldan S, Mehta S, Bhambhani S, et al. Alterations in microRNAs miR-21 and let-7a correlate with aberrant STAT3 signaling and downstream effects during cervical carcinogenesis. Molecular Cancer. 2015;14(1):116. [ Links ]
29. Bumrungthai S, Ekalaksananan T, Evans MF, Chopjitt P, Tangsiriwatthana T, Patarapadungkit N, et al. Up-Regulation of miR-21 Is Associated with Cervicitis and Human Papillomavirus Infection in Cervical Tissues. Plos One. 2015;10(5):e0127109. [ Links ]
30. Yao Q, Xu H, Zhang QQ, Zhou H, Qu LH. MicroRNA-21 promotes cell proliferation and down-regulates the expression of programmed cell death 4 (PDCD4) in HeLa cervical carcinoma cells. Biochemical and Biophysical Research Communications. Elsevier Inc.; 2009;388(3):539-42. [ Links ]
31. Han Y, Xu G-X, Lu H, Yu D-H, Ren Y, Wang L, et al. Dysregulation of miRNA-21 and their potential as biomarkers for the diagnosis of cervical cancer. International journal of clinical and experimental pathology. 2015;8(6):7131-9. [ Links ]
32. Shishodia G, Verma G, Srivastava Y, Mehrotra R, Das BC, Bharti AC. Deregulation of microRNAs Let-7a and miR-21 mediate aberrant STAT3 signaling during human papillomavirus-induced cervical carcinogenesis: role of E6 oncoprotein. BMC cancer. 2014;14(1):996. [ Links ]
33. Rodriguez A, Griffiths-Jones S, Ashurst JL, Bradley A. Identification of mammalian microRNA host genes and transcription units. Genome Research. 2004;14(10 A):1902-10. [ Links ]
34. Lin L, Cai Q, Zhang X, Zhang H, Zhong Y, Xu C, et al. Two less common human microRNAs miR-875 and miR-3144 target a conserved site of E6 oncogene in most high-risk human papillomavirus subtypes. Protein & Cell. Higher Education Press; 2015;1:1-14. [ Links ]
35. Xie H, Lee L, Scicluna P, Kavak E, Larsson C, Sandberg R, et al. Novel functions and targets of miR-944 in human cervical cancer cells. International Journal of Cancer. 2014;136(5):E230-41. [ Links ]
36. Shen Y, Li Y, Ye F, Wang F, Wan X, Lu W, et al. Identification of miR-23a as a novel microRNA normalizer for relative quantification in human uterine cervical tissues. Experimental & molecular medicine. 2011;43(6):358-66. [ Links ]
37. Chu Y, Ouyang Y, Wang F, Zheng A, Bai L, Han L, et al. MicroRNA-590 promotes cervical cancer cell growth and invasion by targeting CHL1. Journal of Cellular Biochemistry. 2014;115(5):847-53. [ Links ]
38. Botezatu A, Goia-Rusanu CD, Iancu IV, Huica I, Plesa A, Socolov D, et al. Quantitative analysis of the relationship between microRNA-124a, -34b and -203 gene methylation and cervical oncogenesis. Molecular Medicine Reports. 2011;4:121-8. [ Links ]
39. Shen S, Wang L, Jia Y, Hao Y, Zhang L, Wang H. Upregulation of microRNA-224 is associated with aggressive progression and poor prognosis in human cervical cancer. Diagnostic pathology. 2013;8(69):1-7. [ Links ]
40. Zhang B, Chen J, Ren Z, Chen Y, Li J, Miao X, et al. A specific miRNA signature promotes radioresistance of human cervical cancer cells. Cancer cell international. 2013;13(118):1-8. [ Links ]
41. Wang W-T, Zhao Y-N, Yan J-X, Weng M-Y, Wang Y, Chen Y-Q, et al. Differentially expressed microRNAs in the serum of cervical squamous cell carcinoma patients before and after surgery. Journal of hematology & oncology. 2014;7(1):1-10. [ Links ]
42. Liu W, Gao G, Hu X, Wang Y, Schwarz JK. Activation of miR-9 by human papillomavirus in cervical cancer. OncoTarget. 2014;5(22). [ Links ]
43. Li J, Ping Z, Ning H. MiR-218 impairs tumor growth and increases chemo-sensitivity to cisplatin in cervical cancer. International Journal of Molecular Sciences. 2012;13(12):16053-64. [ Links ]
44. Martinez I, Gardiner a S, Board KF, Monzon F a, Edwards RP, Khan S a. Human papillomavirus type 16 reduces the expression of microRNA-218 in cervical carcinoma cells. Oncogene. 2008;27(18):2575-82. [ Links ]
45. Kim HJ, Cho H, Choi CH, Chung J, Hewitt SM, Hewitt SM. MicroRNA as Biomarkers for Cervical Cancer. SM Journal of Gynecology and Obstetrics. 2015;1(2):1-8. [ Links ]
46. Chen J, Yao D, Zhao S, He C, Ding N, Li L, et al. MiR-1246 promotes SiHa cervical cancer cell proliferation, invasion, and migration through suppression of its target gene thrombospondin 2. Archives of Gynecology and Obstetrics. 2014;290(4):725-32. [ Links ]
47. Kaczkowski B, Morevati M, Rossing M, Cilius F, Norrild B. A Decade of Global mRNA and miRNA Profiling of HPV-Positive Cell Lines and Clinical Specimens. The open virology journal. 2012;6(Suppl 2: M7):216-31. [ Links ]
48. Yao T, Rao Q, Liu L, Zheng C, Xie Q, Liang J, et al. Exploration of tumor suppressive microRNAs silenced by DNA hypermethylation in cervical cancer. Virology journal. 2013;10(175):1-7. [ Links ]
49. Reshmi G, Chandra SSV, Babu VJM, Babu PSS, Santhi WS, Ramachandran S, et al. Identification and analysis of novel microRNAs from fragile sites of human cervical cancer: Computational and experimental approach. Genomics. Elsevier Inc.; 2011;97(6):333-40. [ Links ]
50. Rao PH, Arias-Pulido H, Lu X-Y, Harris CP, Vargas H, Zhang FF, et al. Chromosomal amplifications, 3q gain and deletions of 2q33-q37 are the frequent genetic changes in cervical carcinoma. BMC cancer. 2004;4(1):5. [ Links ]
51. Wilting SM, Snijders PJF, Verlaat W, Jaspers A, van de Wiel M a, van Wieringen WN, et al. Altered microRNA expression associated with chromosomal changes contributes to cervical carcinogenesis. Oncogene. 2013;32:106-16. [ Links ]
52. Yamamoto N, Kinoshita T, Nohata N, Yoshino H, Itesako T, Fujimura L, et al. Tumor-suppressive microRNA-29a inhibits cancer cell migration and invasion via targeting HSP47 in cervical squamous cell carcinoma. International Journal of Oncology. 2013;43(6):1855-63. [ Links ]
53. Ribeiro J, Marinho-Dias J, Monteiro P, Loureiro J, Baldaque I, Medeiros R, et al. miR-34a and miR-125b Expression in HPV Infection and Cervical Cancer Development. BioMed Research International. 2015;2015:1-6. [ Links ]
54. Kozomara A, Griffiths-Jones S. MiRBase: Integrating microRNA annotation and deep-sequencing data. Nucleic Acids Research. 2011;39(1):1-6. [ Links ]
55. Kozomara A, Griffiths-Jones S. MiRBase: Annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 2014;42(D1):68-73. [ Links ]
56. Wang X, Liu XS. Systematic curation of mirbase annotation using integrated small RNA high-throughput sequencing data for C. Elegans and Drosophila. Frontiers in Genetics. 2011;2(MAY). [ Links ]
57. Cioffi M, Trabulo S, Sanchez-Ripoll Y, Miranda-Lorenzo I, Lonardo E, Dorado J, et al. The miR-17-92 cluster counteracts quiescence and chemoresistance in a distinct subpopulation of pancreatic cancer stem cells. Gut. 2015;(0):1-13. [ Links ]
58. Concepcion C, Bonetti C, Ventura A. The miR-17-92 family of microRNA clusters in development and disease. Cancer journal. 2012;18(3):262-7. [ Links ]
59. Hayashita Y, Osada H, Tatematsu Y, Yamada H, Yanagisawa K, Tomida S, et al. A polycistronic MicroRNA cluster, miR-17-92, is overexpressed in human lung cancers and enhances cell proliferation. Cancer Research. 2005;65(21):9628-32. [ Links ]
60. Li Y, Vecchiarelli-Federico LM, Li YJ, Egan SE, Spaner D, Hough MR, et al. The miR-17-92 cluster expands multipotent hematopoietic progenitors whereas imbalanced expression of its individual oncogenic miRNAs promotes leukemia in mice. Blood. 2012;119(19):4486-98. [ Links ]
61. Lu Y, Thomson JM, Wong HYF, Hammond SM, Hogan BLM. Transgenic over-expression of the microRNA miR-17-92 cluster promotes proliferation and inhibits differentiation of lung epithelial progenitor cells. Developmental Biology. 2007;310(2):442-53. [ Links ]
62. Mestdagh P, Boström AK, Impens F, Fredlund E, Van Peer G, De Antonellis P, et al. The miR-17-92 MicroRNA Cluster Regulates Multiple Components of the TGF-B Pathway in Neuroblastoma. Molecular Cell. 2010;40(5):762-73. [ Links ]
63. Van Haaften G, Agami R. Tumorigenicity of the miR-17-92 cluster distilled. Genes and Development. 2010;24(1):1-4. [ Links ]
64. Wu T, Wieland A, Araki K, Davis CW, Ye L, Hale JS, et al. Temporal expression of microRNA cluster miR-17-92 regulates effector and memory CD8+ T-cell differentiation. Proceedings of the National Academy of Sciences of the United States of America 2012;109(25):9965-70. [ Links ]
65. Wei Q, Li Y-X, Liu M, Li X, Tang H. MiR-17-5p targets TP53INP1 and regulates cell proliferation and apoptosis of cervical cancer cells. IUBMB life. 2012;64(8):697-704. [ Links ]
66. Yu Y, Zhang Y, Zhang S. MicroRNA-92 regulates cervical tumorigenesis and its expression is upregulated by human papillomavirus-16 E6 in cervical cancer cells. Oncology Letters. 2013;6(2):468-74. [ Links ]
67. Kang HW, Wang F, Wei Q, Zhao YF, Liu M, Li X, et al. miR-20a promotes migration and invasion by regulating TNKS2 in human cervical cancer cells. FEBS Letters. Federation of European Biochemical Societies; 2012;586(6):897-904. [ Links ]
68. Xu XM, Wang XB, Chen MM, Liu T, Li YX, Jia WH, et al. MicroRNA-19a and -19b regulate cervical carcinoma cell proliferation and invasion by targeting CUL5. Cancer Letters. Elsevier Ireland Ltd; 2012;322(2):148-58. [ Links ]
69. Cai N, Wang Y-D, Zheng P-S. The microRNA-302-367 cluster suppresses the proliferation of cervical carcinoma cells through the novel target AKT1. RNA (New York, NY). 2013;19(1):85-95. [ Links ]
70. Servín-González L, Granados-López A, López J. Families of microRNAs Expressed in Clusters Regulate Cell Signaling in Cervical Cancer. International Journal of Molecular Sciences. 2015;16(6):12773-90. [ Links ]
71. Qiang R, Wang F, Shi LY, Liu M, Chen S, Wan HY, et al. Plexin-B1 is a target of miR-214 in cervical cancer and promotes the growth and invasion of HeLa cells. International Journal of Biochemistry and Cell Biology. Elsevier Ltd; 2011;43(4):632-41. [ Links ]
72. Kogo R, How C, Chaudary N, Bruce J, Shi W, Hill P, et al. The microRNA-218 ~ Survivin axis regulates migration, invasion, and lymph node metastasis in cervical cancer. OncoTarget. 2014;6(2). [ Links ]
73. Lui W-O, Pourmand N, Patterson BK, Fire A. Patterns of known and novel small RNAs in human cervical cancer. Cancer research. 2007;67(13):6031-43. [ Links ]
74. Lee JW, Choi CH, Choi JJ, Park YA, Kim SJ, Hwang SY, et al. Altered MicroRNA expression in cervical carcinomas. Clinical Cancer Research. 2008;14(9):2535-42. [ Links ]
75. Liang S, Tian T, Liu X, Shi H, Tang C, Yang S, et al. Microarray analysis revealed markedly differential miRNA expressionprofiles in cervical intraepithelial neoplasias and invasive squamous cellcarcinoma. Future Oncology. 2014;10(13):1-10. [ Links ]
76. Dreher A, Rossing M, Kaczkowski B, Andersen DK, Larsen TJ, Christophersen MK, et al. Differential expression of cellular microRNAs in HPV 11, -16, and -45 transfected cells. Biochemical and Biophysical Research Communications. Elsevier Inc.; 2011;412(1):20-5. [ Links ]
77. Lei C, Wang Y, Huang Y, Yu H, Huang Y, Wu L, et al. Up-regulated miR155 Reverses the Epithelial-mesenchymal Transition Induced by EGF and Increases Chemo-sensitivity to Cisplatin in Human Caski Cervical Cancer Cells. PLoS ONE. 2012;7(12):e52310. [ Links ]
78. Li JH, Xiao X, Zhang YN, Wang YM, Feng LM, Wu YM, et al. MicroRNA miR-886-5p inhibits apoptosis by down-regulating Bax expression in human cervical carcinoma cells. Gynecologic Oncology. Elsevier Inc.; 2011;120(1):145-51. [ Links ]
79. Wang L, Wang Q, Li HL, Han LY. Expression of MiR200a, miR93, metastasis-related gene RECK and MMP2/MMP9 in human cervical carcinoma-relationship with prognosis. Asian Pacific Journal of Cancer Prevention. 2013;14(3):2113-8. [ Links ]
80. Ye C, Sun N-X, Ma Y, Zhao Q, Zhang Q, Xu C, et al. MicroRNA-145 contributes to enhancing radiosensitivity of cervical cancer cells. FEBS letters. Federation of European Biochemical Societies; 2015;589(6):1-8. [ Links ]
81. Chen Y, Ma C, Zhang W, Chen Z, Ma L. Down regulation of miR-143 is related with tumor size, lymph node metastasis and HPV16 infection in cervical squamous cancer. Diagnostic Pathology. 2014;9(88):1-11. [ Links ]
82. Hu X, Schwarz JK, Lewis JS, Huettner PC, Rader JS, Deasy JO, et al. A microRNA expression signature for cervical cancer prognosis. Cancer Research. 2010;70(4):1441-8. [ Links ]
83. Lajer C, Garnæs E, Friis-Hansen L, Norrild B, Therkildsen M, Glud M, et al. The role of miRNAs in human papilloma virus (HPV)-associated cancers: bridging between HPV-related head and neck cancer and cervical cancer. British journal of cancer. 2012;106(9):1526-34. [ Links ]
84. Luo M, Shen D, Zhou X, Chen X, Wang W. MicroRNA-497 is a potential prognostic marker in human cervical cancer and functions as a tumor suppressor by targeting the insulin-like growth factor 1 receptor. Surgery. Mosby, Inc.; 2013;153(6):836-47. [ Links ]
85. Huang L, Lin J-X, Yu Y-H, Zhang M-Y, Wang H-Y, Zheng M. Downregulation of Six MicroRNAs Is Associated with Advanced Stage, Lymph Node Metastasis and Poor Prognosis in Small Cell Carcinoma of the Cervix. PLoS ONE. 2012;7(3):e33762. [ Links ]
86. Yoon S, De Micheli G. Prediction of regulatory modules comprising microRNAs and target genes. Bioinformatics. 2005;21(SUPPL. 2):93-100. [ Links ]
87. Cheung TH, Man KNM, Yu MY, Yim SF, Siu NSS, Lo KWK, et al. Dysregulated microRNAs in the pathogenesis and progression of cervical neoplasm. Cell Cycle. 2012;11(15):2876-84. [ Links ]
88. Shen Y, Zhou J, Li Y, Ye F, Wan X, Lu W, et al. miR-375 Mediated Acquired Chemo-Resistance in Cervical Cancer by Facilitating EMT. PLoS ONE. 2014;9(10):e109299. [ Links ]
89. Hummel R, Wang T, Watson DI, Michael MZ, Van Der Hoek M, Haier J, et al. Chemotherapy-induced modification of microRNA expression in esophageal cancer. Oncology Reports. 2011;26(4):1011-7. [ Links ]
90. González V, Palma L, Campos A, Lopez E, Peralta O, Vazquez R, et al. MicroRNAs are involved in cervical cancer development, progression, clinical outcome and improvement treatment response (Review). Oncology Reports. 2016;35:3-12. [ Links ]
91. He Y, Lin J, Ding Y, Liu G, Luo Y, Huang M, et al. A systematic study on dysregulated microRNAs in cervical cancer development. International journal of cancer. 2016;138(6):1312-27. [ Links ]
92. Pritchard CC, Cheng HH, Tewari M. MicroRNA profiling: approaches and considerations. Nature Reviews Genetics. Nature Publishing Group; 2012;13(5):358-69. [ Links ]
93. Pedroza A, López E, García V, Jacobo N, Herrera L, Peralta O, et al. MicroRNAs in Cervical Cancer: Evidences for a miRNA profile deregulated by HPV and its impact on radio-resistance. Molecules. 2014;19(5):6263-81. [ Links ]
94. Rao Q, Zhou H, Peng Y, Li J, Lin Z. Aberrant microRNA expression in human cervical carcinomas. Medical Oncology. 2012;29:1242-8. [ Links ]
95. Villegas V, Juárez S, Pérez O a., Arreola H, Paniagua L, Parra-Melquiadez M, et al. Heterogeneity of microRNAs expression in cervical cancer cells: Over-expression of miR-196a. International Journal of Clinical and Experimental Pathology. 2014;7(4):1389-401. [ Links ]
96. Wang X, Wang H-K, Li Y, Hafner M, Banerjee NS, Tang S, et al. MicroRNAs are biomarkers of oncogenic human papillomavirus infections. Proceedings of the National Academy of Sciences of the United States of America. 2014;111(11):4262-7. [ Links ]
97. Park H, Lee M, Jeong J, Choi MC, Jung SG, Joo WD, et al. Dysregulated microRNA expression in adenocarcinoma of the uterine cervix: Clinical impact of miR-363-3p. Gynecologic Oncology. Elsevier Inc.; 2014;135(3):565-72. [ Links ]
98. Ogechukwu OJ. Discordant Reports of miRNA Expression in Cervical Cancer: An Upshot of Overlapping Factors. Research in Cancer and Tumor. 2015;4(1):15-23. [ Links ]
99. Calin Ga, Croce CM. Chromosomal rearrangements and microRNAs: A new cancer link with clinical implications. Journal of Clinical Investigation. 2007;117(8):2059-66. [ Links ]
100. Reshmi G, Pillai MR. Beyond HPV: Oncomirs as new players in cervical cancer. FEBS Letters. Federation of European Biochemical Societies; 2008;582(30):4113-6. [ Links ]
101. Banno K, Iida M, Yanokura M, Kisu I, Iwata T, Tominaga E, et al. MicroRNA in Cervical Cancer: OncomiRs and tumor suppressor miRs in diagnosis and treatment. The Scientific World Journal. 2014;2014(ID 178075):1-8. [ Links ]
102. Pereira PM, Marques JP, Soares AR, Carreto L, Santos M a S. Microrna expression variability in human cervical tissues. PLoS ONE. 2010;5(7):e11780. [ Links ]
103. Hayes J, Peruzzi PP, Lawler S. MicroRNAs in cancer: Biomarkers, functions and therapy. Trends in Molecular Medicine. Elsevier Ltd; 2014;20(8):460-9. [ Links ]
104. Cho WCS. MicroRNAs: potential biomarkers for cancer diagnosis, prognosis and targets for therapy. The international journal of biochemistry & cell biology. 2010;42(8):1273-81. [ Links ]
105. Yang Z, Chen S, Luan X, Li Y, Liu M, Li X, et al. MicroRNA-214 is aberrantly expressed in cervical cancers and inhibits the growth of hela cells. IUBMB Life. 2009;61(11):1075-82. [ Links ]
106. Hou T, Ou J, Zhao X, Huang X, Huang Y, Zhang Y. MicroRNA-196a promotes cervical cancer proliferation through the regulation of FOXO1 and p27Kip1. British journal of cancer. 2014;110(5):1260-8. [ Links ]
107. Li X, Chu H, Lv T, Wang L, Kong S, Dai S. miR-342-3p suppresses proliferation, migration and invasion by targeting FOXM1 in human cervical cancer. FEBS Letters. 2014;588:3298-307. [ Links ]
108. Tang T, Wong HK, Gu W, Yu MY, To KF, Wang CC, et al. MicroRNA-182 plays an onco-miRNA role in cervical cancer. Gynecologic Oncology. Elsevier Inc.; 2013;129(1):199-208. [ Links ]
109. Wang L, Chang L, Li Z, Gao Q, Cai D, Tian Y, et al. miR-99a and -99b inhibit cervical cancer cell proliferation and invasion by targeting mTOR signaling pathway. Medical oncology. 2014;31(934):1-8. [ Links ]
110. Mlcochova J, Faltejskova-Vychytilova P, Ferracin M, Zagatti B, Radova L, Svoboda M, et al. MicroRNA expression profiling identifies miR-31-5p/3p as associated with time to progression in wild-type RAS metastatic colorectal cancer treated with cetuximab. Oncotarget. Impact Journals; 2015;6(36):38695-704. [ Links ]
111. Wang N, Zhou Y, Zheng L, Li H. MiR-31 is an independent prognostic factor and functions as an oncomir in cervical cancer via targeting ARID1A. Gynecologic Oncology. Elsevier Inc.; 2014;134(1):129-37. [ Links ]
112. Zheng W, Liu Z, Zhang W, Hu X. miR-31 functions as an oncogene in cervical cancer. Archives of Gynecology and Obstetrics. Springer Berlin Heidelberg; 2015;292(5):1-7. [ Links ]
113. Long M-J, Wu F-X, Li P, Liu M, Li X, Tang H. MicroRNA-10a targets CHL1 and promotes cell growth, migration and invasion in human cervical cancer cells. Cancer Letters. 2012;324(2):186-96. [ Links ]
114. Zhao S, Yao DS, Chen JY, Ding N. Aberrant Expression of miR-20a and miR-203 in Cervical Cancer. Asian Pacific Journal of Cancer Prevention. 2013;14(4):2289-93. [ Links ]
115. Qin W, Dong P, Ma C, Mitchelson K, Deng T, Zhang L, et al. MicroRNA-133b is a key promoter of cervical carcinoma development through the activation of the ERK and AKT1 pathways. Oncogene. 2012;31(36):4067-75. [ Links ]
116. Sun J, Ji J, Huo G, Song Q, Zhang X. miR-182 induces cervical cancer cell apoptosis through inhibiting the expression of DNMT3a. Int J Clin Exp Pathol. 2015;8(5):4755-63. [ Links ]
117. Wang F, Liu M, Li X, Tang H. MiR-214 reduces cell survival and enhances cisplatin-induced cytotoxicity via down-regulation of Bcl2l2 in cervical cancer cells. FEBS Letters. 2013;587(5):488-95. [ Links ]
118. Wan H-Y, Li Q-Q, Zhang Y, Tian W, Li Y-N, Liu M, et al. MiR-124 represses vasculogenic mimicry and cell motility by targeting amotL1 in cervical cancer cells. Cancer Letters. Elsevier Ireland Ltd; 2014;355(1):148-58. [ Links ]
119. Cui F, Li X, Zhu X, Huang L, Huang Y, Mao C, et al. MiR-125b inhibits tumor growth and promotes apoptosis of cervical cancer cells by targeting phosphoinositide 3-kinase catalytic subunit delta. Cellular Physiology and Biochemistry. 2012;30:1310-8. [ Links ]
120. Liu L, Yu X, Guo X, Tian Z, Su M, Long Y, et al. MiR-143 is downregulated in cervical cancer and promotes apoptosis and inhibits tumor formation by targeting Bcl-2. Molecular Medicine Reports. 2012;5(3):753-60. [ Links ]
121. Malta M, Ribeiro J, Monteiro P, Loureiro J, Medeiros R, Sousa H. Let-7c is a Candidate Biomarker for Cervical Intraepithelial Lesions: A Pilot Study. Molecular Diagnosis & Therapy. Springer International Publishing; 2015;19(3):191-6. [ Links ]
122. Wang Q, Qin J, Chen A, Zhou J, Liu J, Cheng J, et al. Downregulation of microRNA-145 is associated with aggressive progression and poor prognosis in human cervical cancer. Tumor Biology, 2015;36(5):3703-8. [ Links ]
123. Yu Q, Liu SL, Wang H, Shi G, Yang P, Chen XL. miR-126 suppresses the proliferation of cervical cancer cells and alters cell sensitivity to the chemotherapeutic drug bleomycin. Asian Pacific Journal of Cancer Prevention. 2013;14(11):6569-72. [ Links ]
124. Zhang J, Wang L, Li B, Huo M, Mu M, Liu J, et al. miR-145 downregulates the expression of cyclin-dependent kinase 6 in human cervical carcinoma cells. Experimental and Therapeutic Medicine. 2014;8(2):591-4. [ Links ]
125. Zeng K, Zheng W, Mo X, Liu F, Li M, Liu Z, et al. Dysregulated microRNAs involved in the progression of cervical neoplasm. Archives of Gynecology and Obstetrics. 2015;292(5):905-13. [ Links ]
126. Wang F, Li Y, Zhou J, Xu J, Peng C, Ye F, et al. MiR-375 is down-regulated in squamous cervical cancer and inhibits cell migration and invasion via targeting transcription factor SP1. American Journal of Pathology. 2011;179(5):2580-8. [ Links ]
127. Shen Y, Wang P, Li Y, Ye F, Wang F, Wan X, et al. miR-375 is upregulated in acquired paclitaxel resistance in cervical cancer. British journal of cancer. 2013;109(1):92-9. [ Links ]
128. Oh EK, Kim Y-W, Kim I-W, Liu H-B, Lee K-H, Chun HJ, et al. Differential DNA copy number aberrations in the progression of cervical lesions to invasive cervical carcinoma. International journal of oncology. 2012;41(6):2038-46. [ Links ]
129. Liu J, Sun H, Wang X, Yu Q, Li S, Yu X, et al. Increased exosomal microRNA-21 and microRNA-146a levels in the cervicovaginal lavage specimens of patients with cervical cancer. International Journal of Molecular Sciences. 2014;15(1):758-73. [ Links ]
130. Gardiner A, McBee W, Edwards R, Lesnock J, Bhargava R. MicroRNA Analysis in Human Papillomavirus (HPV)-Associated Cervical Neoplasia and Cancer. Journal of Carcinogenesis & Mutagenesis. 2011;2(1):1-9. [ Links ]
131. Mo W, Tong C, Zhang Y, Lu H. microRNAs' differential regulations mediate the progress of Human Papillomavirus (HPV)-induced Cervical Intraepithelial Neoplasia (CIN). BMC Systems Biology. 2015;9(4):1-17. [ Links ]
132. Tian Q, Li Y, Wang F, Li Y, Xu J, Shen Y, et al. MicroRNA Detection in Cervical Exfoliated Cells as a Triage for Human Papillomavirus-Positive Women. JNCI Journal of the National Cancer Institute. 2014;106(9):dju241-dju241. [ Links ]
133. Wilting S, Verlaat W, Jaspers A, Makazaji N a., Agami R, Meijer CJLM, et al. Methylation-mediated transcriptional repression of microRNAs during cervical carcinogenesis. Epigenetics. 2013;8(2):220-8. [ Links ]
134. Hao Z, Yang J, Wang C, Li Y, Zhang Y, Dong X, et al. MicroRNA-7 inhibits metastasis and invasion through targeting focal adhesion kinase in cervical cancer. Int J Clin Exp Med. 2015;8(1):480-7. [ Links ]
135. Ribeiro J, Sousa H. MicroRNAs as biomarkers of cervical cancer development: A literature review on miR-125b and miR-34a. Molecular Biology Reports. 2014;41(3):1525-31. [ Links ]
136. Lao G, Liu P, Wu Q, Zhang W, Liu Y, Yang L, et al. Mir-155 promotes cervical cancer cell proliferation through suppression of its target gene LKB1. Tumor Biology. 2014;35:11933-8. [ Links ]
137. Xu J, Li Y, Wang F, Wang X, Cheng B, Ye F, et al. Suppressed miR-424 expression via upregulation of target gene Chk1 contributes to the progression of cervical cancer. Oncogene. 2013;32:976-87. [ Links ]
138. Tian RQ, Wang XH, Hou LJ, Jia WH, Yang Q, Li YX, et al. MicroRNA-372 is down-regulated and targets cyclin-dependent kinase 2 (CDK2) and cyclin A1 in human cervical cancer, which may contribute to tumorigenesis. Journal of Biological Chemistry. 2011;286(29):25556-63. [ Links ]
139. Li BH, Zhou JS, Ye F, Cheng XD, Zhou CY, Lu WG, et al. Reduced miR-100 expression in cervical cancer and precursors and its carcinogenic effect through targeting PLK1 protein. European Journal of Cancer. Elsevier Ltd; 2011;47(14):2166-74. [ Links ]
140. Cai X, Li G, Laimins L a, Cullen BR. Human papillomavirus genotype 31 does not express detectable microRNA levels during latent or productive virus replication. Journal of virology. 2006;80(21):10890-3. [ Links ]
141. Wald AI. Role of MicroRNas-363- in HPV associated squamous cell carcinoma of the head and neck. University of Pittsburgh, Pensilvania, Estados Unidos; 2012. [ Links ]
142. Rothschild SI. microRNA therapies in cancer. Molecular and Cellular Therapies [Internet]. 2014;2(7):2-8. Available from: http://www.molcelltherapies.com/content/2/1/7 [ Links ]