Introducción
Los constantes brotes de enfermedades infecciosas emergentes y reemergentes como la gripe aviar y porcina, Ébola, MERS (Middle East Respiratory Syndrome) y SARS (Severe Acute Respiratory Syndrome), hicieron que hace más de 15 años la Organización Mundial de la Salud (OMS) pronosticara un nuevo ataque causado por un patógeno viral. La pregunta en ese momento no era ¿Cuál? ¿Cómo? o ¿Dónde? sino ¿Cuándo?1,2. De ahí que la explicación a estos fenómenos biológicos se debe buscar en trabajos investigativos que se someten a estrictos procesos de revisión por pares3,4. El tamaño microscópico y las características genotípicas de estos patógenos dificultan el reconocimiento del origen, la magnitud y el peligro que representan las enfermedades emergentes y reemergentes. La aparición de virus inéditos, así como las tasas de morbilidad y mortalidad asociadas con este tipo de agentes infecciosos, van a aumentar si no se establecen procedimientos clínicos claros para su diagnóstico, prevención y control1,2.
Los virus tienen una capacidad intrínseca para mutar y adquirir nuevas capacidades que les permiten alcanzar un nivel elevado de propagación en humanos, tal y como se está evidenciando con esta pandemia causada por la nueva enfermedad COVID-19 (Coronavirus Disease 2019)3. Esta enfermedad respiratoria es causada por el virus SARS-CoV-2 (Severe Acute Respiratory Syndrome Coronavirus 2) y fue inicialmente documentada como una neumonía viral atípica en la ciudad de Wuhan-China en diciembre de 20195,6.
Según la OMS, hasta el mes de junio, COVID-19 ya había afectado a más de ocho millones y cobrado la vida de más de 450,000 personas en todo el mundo7. Desafortunadamente, la alarmante capacidad de contagio que alcanzó SARS-CoV-2 hizo sinergia con la ausencia de inmunidad en la población humana, una pobre implementación de estrategias sanitarias y con la inexistencia de métodos terapéuticos eficientes para contener esta enfermedad, progresando así hasta convertirse en una peligrosa pandemia7.
Aunque todavía existen muchos interrogantes en relación con COVID-19, la secuenciación del genoma de SARS-CoV-2 ha representado un hito, ya que sentó las bases para un mejor entendimiento del virus y manejo de la epidemia8-11.
Con los avances en estudios moleculares, se logró descifrar rápidamente la estructura de la proteína S de SARS-CoV-2. Esta proteína superficial le permite al virus localizar y empezar su ciclo de replicación en células humanas susceptibles12,13. Igualmente, análisis genéticos, moleculares y estructurales fueron necesarios para caracterizar cómo la proteína S se adhiere al receptor celular ACE2 (Angiotensin Converting Enzyme 2)14,15. La interacción específica entre la proteína viral S y ACE2 es esencial para que SARS-CoV-2 reconozca e inicie su proceso infectivo en las células que expresan este receptor en su membrana citoplasmática15-17.
Debido a la expresión de ACE2 en las células epiteliales de las vías respiratorias, una de las manifestaciones clínicas más severas de COVID-19 ha sido la insuficiencia respiratoria18,19. Cabe resaltar que el descubrimiento de los mecanismos estructurales que median en el reconocimiento de ACE2 por la proteína S de SARS-CoV-2, aceleró el diseño de estrategias terapéuticas con potencial de combatir COVID-19, varias de ellas basadas en anticuerpos capaces de neutralizar SARS-CoV-220.
Durante los últimos meses se ha progresado en gran medida en el entendimiento de las razones que subyacen a los casos más severos de COVID-19. Muchos de los pacientes que desarrollan esta enfermedad con peor pronóstico y que requieren tratamiento en UCI padecen de condiciones médicas pre-existentes tales como obesidad, diabetes, enfermedades circulatorias y respiratorias, entre otras18,19,21. Ha llamado la atención que las altas tasas de mortalidad registradas para COVID-19 no están relacionadas únicamente con estas comorbilidades, sino también con factores demográficos tales como la edad avanzada y sexo masculino, así como también el consumo de cigarrillo18,19,21,22.
Una de las inmunopatologías más frecuentemente observada en los casos más críticos de COVID-19 consiste en una respuesta inmune exacerbada de tipo inflamatorio. Esta respuesta incontrolada puede desencadenar una insuficiencia respiratoria secundaria tan compleja que puede causar la muerte del paciente y que, por lo tanto, requiere intervención oportuna mediante soporte ventilatorio asistido y tratamiento con inmunosupresores23-25.
En este contexto, el objetivo de este artículo de revisión narrativa es presentar evidencias sobre el origen, evolución y efectos de esta enfermedad a nivel mundial, publicadas en su mayoría desde enero del 2020 en artículos científicos. Igualmente, se describen y analizan reportes de varias entidades internacionales. Se incluyen y discuten, además, diversos descubrimientos que contribuyen al entendimiento de esta nueva patología, tales como: (i) aspectos moleculares y replicativos de SARS-CoV-2, (ii) respuesta inmune que se ejecuta para su contención, (iii) sintomatología y patología, (iv) causas del origen y (v) facilidad de transmisión de COVID-19.
Aspectos Moleculares de SARS-CoV-2
1. Estructura viral
Durante diciembre del 2019 y enero del 2020, el incremento súbito de casos de una neumonía viral atípica en China, y sus manifestaciones clínicas, sugerían que el agente etiológico de este nuevo brote estaba relacionado con coronavirus que recientemente habían causado epidemias en Asia (SARS, 2002) y la península arábiga (MERS, 2012). Por este motivo se denominó a esta nueva enfermedad como COVID-19, y al virus causante se le asignó el nombre de SARS-CoV-27,26-28.
El SARS-CoV-2 pertenece a la familia Coronavirinae (género Betacoronavirus, o Beta-CoVs). Los Beta-CoVs causan trastornos respiratorios en humanos con sintomatología variada, desde resfriado común (HCoV-OC43) hasta síndrome respiratorio agudo severo (SARS-CoV, SARS-CoV-2 y MERS-CoV)29,30. Análisis microscópicos y estructurales del SARS-CoV-2 revelan una morfología esférica que está soportada principalmente por proteínas, glicoproteínas, ácidos nucleicos y fosfolípidos. Estos elementos estructurales de la partícula viral se distribuyen en sus dos regiones principales, (i) la envoltura (bicapa fosfolipídica y glucoproteínas E, M y S) y (ii) la nucleocápside (ARN genómico y proteína N)27,29 (Figura 1).
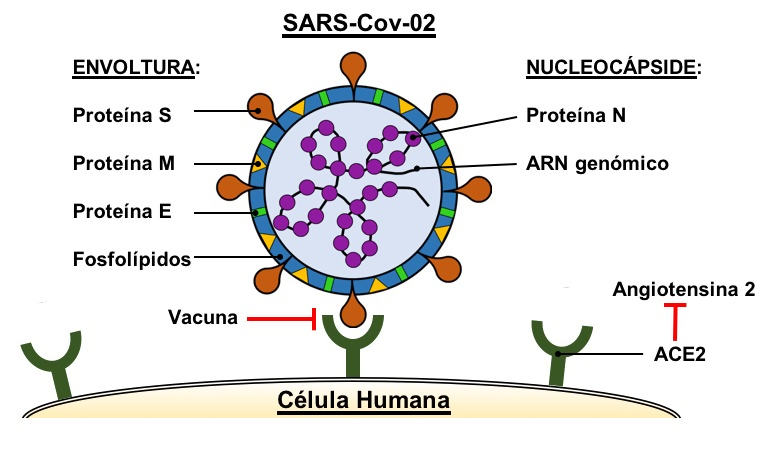
Figura 1 Organización estructural de SARS-CoV-2. La envoltura viral se compone de las proteínas S (Spike), M (Membrane), E (Envelope) y la bicapa fosfolipídica. La nucleocápside está formada por la proteína N (Nucleocapsid) que cubre al genoma viral (gRNA). Al comienzo de la infección, la proteína S se adhiere al receptor celular ACE2 (Angiotensin Converting Enzyme 2), enzima que también regula la presión sanguínea mediante la hidrólisis de la hormona peptídica angiotensina 2. La estrategia utilizada por varias vacunas en contra de COVID-19 se enfoca en impedir la interacción: proteína S - ACE2 (Tabla 2).
1.1. Nucleocápside
La cápside proteica de SARS-CoV-2 tiene morfología helicoidal, recubre al ARN genómico viral y se localiza en la parte más interna del virus. En los coronavirus la proteína N cumple esta función de “encapsulación” del material genético viral29. No obstante, N es una proteína multifuncional que desempeña papeles esenciales en otros procesos virales y celulares, tales como: (i) ensamblaje viral, (ii) replicación del ARN genómico, (iii) formación de la envoltura, (iv) liberación de la partícula viral, (v) regulación del ciclo celular, e (vi) inhibición de respuestas inmunes mediadas por el interferón29,31,32. El hecho que la proteína N cumpla con funciones tan fundamentales durante la multiplicación y patogénesis de SARS-CoV-2, la hace un excelente candidato para el desarrollo de antivirales efectivos en contra de este patógeno respiratorio31.
El genoma de SARS-CoV-2 (en inglés: gRNA) es de tipo ARN (Ácido Ribonucleico), no segmentado, monocatenario, de orientación positiva y de un tamaño aproximado de 30,000 nucleótidos. Este gRNA codifica la información genética necesaria para la síntesis de la totalidad de las proteínas virales, que incluyen: (i) nsp (del inglés: non-structural proteins), (ii) accesorias (ORFs, del inglés Open Reading Frame) y (iii) estructurales (E, M, N y S)8,10,27,29,33-35. Sin embargo, el mecanismo de expresión de estas proteínas es distinto, ya que las nsp (16 en total: nsp1 a nsp16) se originan a partir de un polipéptido precursor, el cual se somete a un proceso de fragmentación proteolítica ejecutado por proteasas de origen viral (nsp3 y nsp5). Por el contrario, las proteínas accesorias (seis en total: ORF3a, ORF6, ORF7a, ORF7b, ORF8 y ORF10) y estructurales de SARS-CoV-2 se originan a partir de la síntesis y posterior traducción de ARN subgenómicos (en inglés: subgenomic RNAs o sgRNAs)27,29,33.
En términos generales, se puede decir que las proteínas nsp son esenciales para la replicación viral dentro de la célula huésped. En este proceso, la proteína nsp12 (replicasa) cumple un papel esencial en la replicación del gRNA29. En contraste, las proteínas accesorias incrementan la patogenicidad de SARS-CoV-2, una vez nuevas partículas virales emergen de la célula infectada36. Sin embargo, se han identificado proteínas nsp y accesorias que SARS-CoV-2 no comparte con otros Beta-CoVs37. Entender las funciones de estas proteínas únicas de SARS-CoV-2 hace parte de las prioridades de los científicos en el área, ya que ellas podrían dar información sobre la patogenia de COVID-1929,37.
1.2. Envoltura viral
La envoltura viral es de carácter primordialmente lipídico y es la estructura más externa de SARS-CoV-2. Proteínas que tienen funciones netamente estructurales, tales como las glucoproteínas M y E, se encuentran intercaladas en esta envoltura. Mientras la proteína M es responsable de mantener la forma y promover el ensamblaje de SARS-CoV-2 a través de su interacción con la proteína N, la proteína E es necesaria para la producción y maduración de la partícula viral29,38.
La proteína S también se encuentra incrustada en la envoltura de SARS-C0V-02, pero adopta una disposición particular. En estudios basados en cromatografía, cristalografía de rayos X y microscopía electrónica, la proteína S se observa como filamentos que se proyectan hacia el exterior del virus, generando una morfología similar a la corona solar. Esta posición estratégica permite a la proteína S cumplir con funciones esenciales durante las etapas iniciales del ciclo multiplicativo del virus12,13.
El comienzo del proceso de infección por SARS-CoV-2 requiere la participación de la proteína S durante las etapas de reconocimiento, adhesión y penetración a la célula huésped (explicado con más detalle en la siguiente sección). Particularmente, la adhesión de SARS-CoV-2 a las células hospederas necesita la interacción específica entre receptores celulares y la proteína viral S. El receptor celular es la enzima ACE2 (Angiotensin Converting Enzyme 2), la cual está presente en grandes cantidades en las células epiteliales de los alvéolos pulmonares39,40.
La expresión elevada de ACE2 en células del tracto respiratorio inferior puede explicar la sintomatología respiratoria observada en los pacientes que desarrollan COVID-1912,13,15,17. Adicionalmente, esta enfermedad se ha visto asociada con daño multi-sistémico en pacientes susceptibles a desarrollar una enfermedad más severa, debido a que este receptor se expresa también en células de otros órganos tales como hígado, riñones, cerebro39,40.
Estudios genómicos han demostrado que una mutación en el gen que codifica para la proteína S (inserción de 12 nucleótidos) en un coronavirus de origen animal podría ser la causante del origen de SARS-CoV-2 y de esta pandemia. Dichos estudios sugieren que esta inserción confirió al virus la capacidad de saltar la “barrera de especies”, un mecanismo que normalmente restringe el rango de huéspedes que un virus puede parasitar12,13,15. Es posible que estas mutaciones aumentaran la capacidad de transmisión de SARS-CoV-2 desde animales tipo murciélago y/o pangolín a los humanos12,13,15,17,41.
La evidencia anteriormente presentada demuestra que los altos niveles de contagio y patogenicidad observados en pacientes con COVID-19 se deben al papel fundamental que la proteína S desempeña durante las fases iniciales de la infección. Por este motivo, laboratorios académicos/investigativos y compañías farmacéuticas/biotecnológicas están desarrollando anticuerpos que neutralicen eficientemente la función de la proteína S. La Tabla 1 presenta un resumen del estado actual de dichos emprendimientos, así como también describe otros antivirales con diferentes mecanismos de acción que tienen el potencial de ser utilizados en casos severos de COVID-1942-48. Es importante anotar que hasta la fecha no se ha diseñado o descubierto un terapéutico con la capacidad de contrarrestar SARS-CoV-2 única y específicamente.
Tabla 1 Potenciales estrategias terapéuticas en contra de SARS-CoV-2
| Nombre | Origen y mecanismo de acción | Estado actual | Aplicaciones anteriores | Referencias |
|---|---|---|---|---|
| 47D11 | Anticuerpos aislados de hibridomas obtenidos a partir de la fusión de linfocitos B de ratones (inmunizados con proteína S de SARS-CoV) con células de mieloma humano. 47D11 bloquea la adhesión de la proteína viral S al receptor celular ACE2, inhibiendo de este modo la infección de la célula huésped | Etapa preclínica | Ninguna | Wang et al., 202042 |
| VHH-2 | Anticuerpos aislados de llamas inmunizadas con proteína S de SARS-CoV. Mecanismo de acción similar a 47D11 | Etapa preclínica | Ninguna | Wrapp, De Vlieger, et al., 202043 |
| S309 | Anticuerpos aislados de pacientes infectados con SARS-CoV (2003). Mecanismo de acción similar a 47D11 | Etapa preclínica | Ninguna | Pinto et al., 202044 |
| B38 y H4 | Anticuerpos aislados de pacientes convalecientes de COVID-19 (2020). Mecanismo de acción similar a 47D11 | Etapa preclínica | Ninguna | Wu et al., 202045 |
| Arbidol | Antiviral que inhibe la fusión de la membrana de SARS-CoV-2 con la membrana del endosoma, bloqueando la etapa de desnudamiento del gRNA | Ensayos clínicos (humanos) | Tratamiento contra el virus de la influenza | Deng et al., 202046 |
| Remdesivir y Favipiravir | Antivirales que inhiben a la enzima replicasa de SARS-CoV-2 (nsp 12), evitando la replicación del gRNA | Ensayos clínicos (humanos) | Tratamiento contra los virus del Ébola, hepatitis C e influenza | Wang et al., 202047 |
| Lopinavir y Ritonavir | Antivirales que inhiben a la proteasa de SARS-CoV-2 (nsp3 y nsp5), impidiendo la producción de los productos derivados de la fragmentación del polipéptido | Ensayos clínicos (humanos) | Tratamiento contra el virus del VIH/SIDA | Chu et al., 200448 |
2. Ciclo multiplicativo de SARS-CoV-2
2.1. Adhesión y penetración
El proceso de adhesión y entrada de SARS-CoV-2 a una célula susceptible se conoce como la “entrada viral mediada por receptor”12,13,29,49. Este proceso involucra tres etapas que requieren la función de la proteína S: adhesión, entrada y fusión de membranas. Durante la etapa de adhesión se establece una asociación directa entre la proteína S y el receptor ACE2 presente en la membrana de una célula humana12,13,15,16 (Etapa 1, Figura 2). Esta adhesión estimula la entrada del virus al citoplasma a través de un mecanismo llamado endocitosis, que normalmente las células utilizan para la incorporación de nutrientes sólidos29.
La fase de penetración de los coronavirus genera una vesícula membranosa citoplasmática conocida como el endosoma (Etapa 2, figura 2). En el interior de este compartimiento, la membrana del virus se fusiona con la membrana del endosoma, liberando y exponiendo el material genético viral a la maquinaria bio-sintética presente en el citoplasma celular29 (Etapa 3, Figura 2). La fusión de membranas y posterior desnudamiento del gRNA es el producto de la fragmentación de la proteína S, la cual es orquestada por la proteasa de membrana TMPRSS212,49.
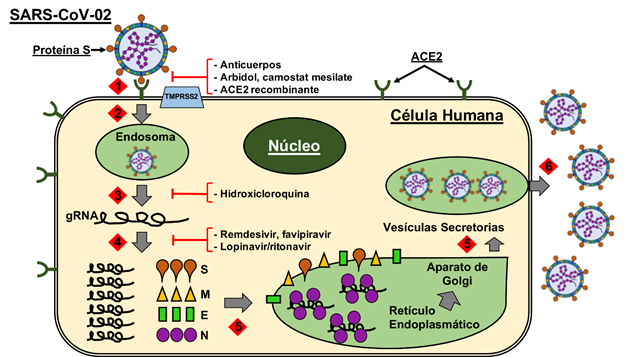
Figura 2 Multiplicación de SARS-CoV-2 en células humanas. El ciclo multiplicativo incluye las siguientes etapas: 1) Adhesión de SAR-CoV-2 al receptor celular ACE2; 2) Penetración al citoplasma celular; 3) Desnudamiento de gRNA a través de la fusión de membranas; 4) Síntesis de los componentes virales; 5) Ensamblaje de proteínas estructurales y genoma viral hasta formar una partícula viral completa; y 6) Liberación de SARS-CoV-2. Se incluyen algunos agentes antivirales con potencial terapéutico en contra de COVID-19. Ver texto para detalles.
2.2. Replicación
La siguiente fase del ciclo multiplicativo de SARS-CoV-2 consiste en la producción de todos los elementos estructurales, no estructurales y accesorios del virus. Para ello, el virus usurpa la maquinaria bio-sintética de la célula huésped, de ahí que se le catalogue como una entidad acelular y un parásito intracelular obligado27,29,33,38.
El gRNA viral tiene varias propiedades moleculares que lo asemejan al ARN mensajero eucariota. Por ejemplo, es de orientación positiva y tiene modificaciones terminales similares (caperuza 5’ y cola poli-A 3’). Estas características hacen que el gRNA viral sea utilizado como molde para la síntesis inicial de los transcriptos ORF1a y ORF1b, que a su vez son traducidos en las poliproteínas pp1a y pp1b. Gracias a la acción de proteasas virales (nsp 3 y 5), estas poliproteínas son procesadas en 16 nsp. También durante el proceso de síntesis, la replicasa viral (nsp12) sintetiza múltiples copias de gRNAs y sgRNAs, estos últimos siendo traducidos individualmente en las proteínas estructurales (E, M, N, S) y accesorias de SARS-CoV-227,29,33,38 (Etapa 4, figura 2). Esta descripción es en realidad una simplificación de un proceso extremadamente complejo, cuya explicación detallada va más allá del objetivo de este artículo.
2.3. Ensamblaje y liberación
El ciclo continúa con el ensamblaje de todos los elementos que hacen parte de la estructura del virión. Para ello, tanto el gRNA como la proteína N son sintetizados en el citoplasma y posteriormente transportados al retículo endoplasmático de la célula huésped. En contraste, las proteínas E, M y S se sintetizan primero en el retículo endoplasmático, para después ser transferidas al Aparato de Golgi. Durante su travesía a lo largo de estos dos organelos, todos estos elementos estructurales se ensamblan en una forma secuencial, de tal manera que un virus completamente formado emerge de la parte distal del Aparato de Golgi27,29,33,38 (Etapa 5, figura 2).
Los viriones maduros de SARS-CoV-2 que emergen del Aparato de Golgi son transportados en el interior de vesículas membranosas a la superficie de la célula. Finalmente, SARS-CoV-2 es expulsado de la célula por exocitosis, proceso en el cual la membrana de esta vesícula secretoria se fusiona con la membrana citoplasmática liberando de este modo su contenido al espacio extracelular 27,29,33,38) (Etapa 6 , Figura 2).
Aspectos Inmunológicos y Clínicos de COVID-19
1. Respuesta inmune contra SARS-CoV-2
Debido a que COVID-19 es una enfermedad reciente, ha sido difícil establecer un cuadro inmunológico y clínico claro que explique la progresión de esta enfermedad50. Sin embargo, la similitud de SARS-CoV-2 con SARS-CoV y MERS-CoV sugiere que estos virus pueden inducir los mismos mecanismos inmunes de defensa: respuestas innatas y respuestas adaptativas. En este contexto, la propagación inicial de SARS-CoV-2 es restringida por la acción rápida del sistema inmune innato, el cual se compone de células (neutrófilos, macrófagos y células dendríticas) y los moduladores proteicos que ellas liberan (interferón, cascada del complemento, quimioquinas, citoquinas)28,38,51.
Una función esencial de varios componentes del sistema del interferón (principalmente interferón tipo 1) consiste en activar sistemas de alerta en las células que todavía no han sido infectadas por un patógeno viral y prepararlas para su eventual infección. La adhesión del interferón a receptores celulares específicos induce mecanismos moleculares de inhibición de la replicación viral y/o apoptosis en caso que la infección sea abrumadora51. Por otra parte, las proteínas del complemento funcionan como mediadores proteicos que atraen a las células inmunes al sitio de infección viral y contribuyen con la respuesta inflamatoria localizada en esa área28.
Macrófagos y neutrófilos constituyen las celulas efectoras que se encargan de eliminar al patógeno mediante fagocitosis y liberación de citoquinas que intensifican la inflamación50. Sin embargo, la inmunopatología observada en COVID-19 indica que la descompensación respiratoria observada en los casos más severos no se debe a la infección viral en sí, sino que es el resultado de una respuesta inflamatoria desbordada52,53.
La ejecución casi inmediata de estos mecanismos innatos tiene un segundo objetivo y es el de proporcionar el tiempo necesario para la maduración de respuestas adaptativas más robustas, específicas y duraderas en contra de SARS-C0V-2. Estos componentes inmunes adaptativos incluyen: linfocitos T (cooperadores y citotóxicos), linfocitos B, células NKs (natural killers), citoquinas pro-inflamatorias y anticuerpos28,52.
El inicio de toda respuesta adaptativa requiere la función de las células dendríticas, las cuales cumplen el papel de “células presentadoras de antígenos”. Estas células, al estar presentes en la mayoría de tejidos, capturan y procesan antígenos de SARS-CoV-2 para presentarlos y activar células efectoras de tipo linfocitos T CD4+ (cluster of differentiation 4) y CD8+ (cluster of differentiation 8). Los linfocitos T cooperadores (CD4+) activan y amplifican las respuestas generadas por linfocitos B y macrófagos por medio de la liberación de interleuquinas (tales como la IL-12). Los linfocitos T citotóxicos (CD8+) activados y NKs destruyen células que están siendo infectadas por SARS-CoV-2 para detener la liberación de más viriones que agudicen el proceso patológico28,38,51.
Los linfocitos B, al ser activados por las células cooperadoras, empiezan a multiplicarse y a diferenciarse en células plasmáticas cuyo papel esencial es la producción de anticuerpos (también llamados inmunoglobulinas o Igs)54,55. Los anticuerpos son proteínas plasmáticas polifuncionales y son imprescindibles para el reconocimiento específico y posterior eliminación de SARS-CoV-2. Entre las funciones antivirales más importantes de las inmunoglobulinas están: (i) neutralización del reconocimiento y la adhesión del virus a ACE2, (ii) marcación de SARS-CoV-2 y de células infectadas para facilitar su reconocimiento y destrucción por células fagocíticas y NKs, y (iii) activación del sistema del complemento28,38,51.
Pero, ¿Cómo SARS-CoV-2 logra escapar de esta sofisticada red de respuestas innatas y adaptativas? La respuesta se encuentra en varias proteínas que este virus ha adaptado como estrategia para evadir los sistemas inmunes de defensa humanos. Por ejemplo, para escapar del sistema del interferón, SARS-CoV-2 “mimetiza” su ARN a través de modificaciones que lo asemejan al ARN mensajero celular (proteínas nsp10, nsp14 y nsp16)29. Otro mecanismo de escape es nsp1, proteína viral que cumple con las funciones de inactivar síntesis de proteínas celulares y degradar los ARN mensajeros del huésped, de tal forma que la síntesis del interferón es interrumpida29,38,51.
Evidencia reciente indica que existen otras proteínas de SARS-CoV-2, y de otros coronavirus, que contribuyen a que estos virus evadan los mecanismos inmunológicos de defensa, incrementando así su patogenicidad29,38. Entre estas proteínas se encuentran: nsp3 (interfiere con la producción de citoquinas e induce inflamación), nsp15 (corta ARN viral para evitar ser detectado por el sistema del interferón), ORF3a (facilita la liberación del virión al desestabilizar la membrana citoplasmática y activa producción de citoquinas proinflamatorias), ORF6 (bloquea comunicación intercelular mediada por el interferón) y ORF7a (facilita la liberación de SARS-CoV-2, aumenta producción de citoquinas proinflamatorias e induce apoptosis)29,38.
Es necesario identificar las funciones de otras proteínas y marcos de lectura exclusivos de SARS-CoV-2 (nsp2, nsp3, nsp11, ORF8, ORF9, ORF10) ya que ellos pueden ser los causantes de la infectividad, patogenia y mortalidad observados en los casos más graves de COVID-1929,38. El entendimiento de los factores de virulencia y mecanismos patogenicidad de SARS-CoV-2 nos permitirá desarrollar estrategias eficientes de prevención y tratamiento en contra de COVID-19.
La principal herramienta de prevención a corto y largo plazo en contra de COVID-19 es la inmunidad activa artificial (vacunación). En la actualidad, se han consolidado múltiples colaboraciones y consorcios internacionales en procura de obtener una vacuna que alcance niveles de eficiencia aceptables para su distribución y uso a corto plazo56,57. Muchos de estos proyectos involucran un amplio rango de plataformas biotecnológicas, entre las cuales se destacan: ácidos nucleicos, proteínas recombinantes, vectores virales, virus vivos atenuados y virus inactivados56,57. El papel central que la proteína S protagoniza en las etapas iniciales de multiplicación de SARS-C0V-02 ha hecho que la mayoría de compañías e institutos centren sus estrategias de desarrollo de la vacuna en esta proteína superficial56,57. FALTA 50
En la Tabla 2 se resumen las plataformas, fases de desarrollo y las compañías/instituciones a cargo de esta tarea56,57. Es importante mencionar que muchos de estos candidatos ya están siendo evaluados en humanos a través de ensayos clínicos (fase I/II/III), por lo cual se espera que la vacuna esté en camino de ser producida a escala industrial en los próximos meses. De este grupo, los candidatos de vacunas que están en la última etapa clínica en humanos (fase III) son las diseñadas por Moderna (USA), Universidad de Oxford (UK) y Sinovac (China). No obstante, las estimaciones más optimistas pronostican que la distribución a nivel mundial de una vacuna contra COVID-19 tardará entre 12-18 meses debido a las barreras involucradas en los procesos industriales de producción a gran escala56,57.
Tabla 2 Estrategias para desarrollar una vacuna contra de SARS-CoV-2
| Estrategia | Blanco | Estado actual | Compañía/Institución |
|---|---|---|---|
| ARN | Proteína S | Ensayos clínicos en humanos (fase I/II) | Pfizer/BNTECH, Moderna e Instituto Nacional de Salud (USA), CureVac |
| ADN | Proteína S | Ensayos clínicos en humanos (fase I) | Inovio/Beijing Advaccine Biotechnology y Applied DNA Sciences |
| Proteína recombinante | Proteína S | Ensayos clínicos en humanos (fase I/II) | Sanofi/GSK, ExpresS2ion, iBio, Novavax, Baylor College of Medicine, University of Queens-land y Schuan Clover Biopharmaceuticals |
| Vector viral | Proteína S | Ensayos clínicos en humanos (fase I/II) | Vaxart, Geovax, Universidad de Oxford AstraZeneca y Cansino Biologics |
| Vacuna viva atenuada | Virión completo | Etapa pre-clínica | Codagenix y Serum Institute of India |
| Vacuna inactivada | Virión completo | Ensayos clínicos en humanos (fase II) | Sinopharm/Chinese Academy of Sciences, Sinovac |
Fuentes: Amanat, F. y Krammer, F. J. immuni., 2020 56; https://www.genengnews.com/covid-19-candidates/covid-19-drug-and-vaccine-tracker/
2. Tormenta de citoquinas
Como se explicó anteriormente, la descompensación respiratoria y otras manifestaciones clínicas complejas de COVID-19 se presentan como resultado de una producción exacerbada de moduladores proteicos pro-inflamatorios tales como: interleuquinas (IL-1, IL-6 e IL-7), factor de necrosis tumoral alfa (TNF-ɑ), factor estimulante de colonias gránulo-monocíticas (GMCSF, por sus siglas en inglés) y proteínas del complemento, entre otros52,53. Este incremento súbito en la concentración sanguínea de citoquinas, entra en un ciclo incontrolable de auto-amplificación y daño, el cual continúa aún en la ausencia del agente infeccioso que indujo la respuesta inicial. Por lo tanto, el término “tormenta de citoquinas” hace referencia a este tipo de respuestas inflamatorias sistémicas que se ejecutan en el contexto de una enfermedad infecciosa y que ponen en riesgo la vida de los pacientes25,52.
COVID-19 induce una respuesta hiper-inflamatoria en los alvéolos de un porcentaje relativamente bajo de pacientes, quienes terminan desarrollando una insuficiencia respiratoria que requiere oxigenoterapia en ambiente hospitalario. En una proporción aún menor de los casos que se han reportado, esta enfermedad evoluciona en una neumonía severa que se trata con ventilación mecánica invasiva en UCI. Aunque los síntomas agudos causados por la tormenta de citoquinas en casos de COVID-19 son variados, algunos reportes de casos en China y USA indican que los pacientes generalmente presentan fiebre, tos, dificultad respiratoria, fatiga, síntomas gastrointestinales, dolor abdominal y pérdida del sentido del gusto y del olfato19,21,22,53. La manifestación clínica más severa de COVID-19 es la insuficiencia respiratoria, que puede ser menor, o evolucionar en un síndrome de dificultad respiratoria aguda que requiere manejo clínico por personal médico especializado en UCI19,21.
A nivel sistémico, la hiper-inflamación genera un descenso crítico de la presión arterial, lo cual deriva en fallo multi-orgánico que conlleva una alta mortalidad por COVID-1925. La presencia del receptor ACE2 en los túbulos renales puede provocar lesiones locales complejas que pueden conducir a una falla renal aguda y a fallo orgánico múltiple, en muchos casos causando la muerte del paciente58. Lesiones miocárdicas son también comunes en pacientes de COVID-19, que muy probablemente están asociadas con factores de riesgo cardiovascular. Al igual que en la falla renal, la disfunción cardiaca tiene un elevado índice de mortalidad19,21.
En la actualidad, se están utilizando múltiples estrategias de tratamiento con capacidad de contrarrestar las tormentas de citoquinas antes de que alcancen un estado incontrolable de auto-perpetuación25,59. El cuidado estándar de pacientes con COVID-19 severo, combina ventilación asistida con tratamiento con químicos inmunomoduladores que contrarresten la hiper-inflamación que desarrollan estos pacientes19,21. Respecto a inhibidores de hiper-inflamación, se han obtenido resultados prometedores con: interferón (IFN-λ), corticosteroides, inmunoglobulina intravenosa (IVIG), antagonistas de interleuquinas, inhibidores de TNF, substancias anti-inflamatorias (Ulinastatin), terapia con células madre, tratamientos de purificación sanguínea e inhibidores del reclutamiento y función de macrófagos mononucleares (mediante siRNAs)25,38. En el futuro, se espera que anticuerpos purificados de pacientes convalecientes de COVID-19 se incluyan en la lista de tratamientos20.
También, están en marcha ensayos pre-clínicos y clínicos con el propósito de evaluar si inhibidores específicos de interleuquinas, de proteínas del complemento y del factor estimulante de colonias gránulo-monocíticas pueden atenuar la respuesta inmune hiper-inflamatoria desencadenada en pacientes con COVID-19. De este modo, se podría prevenir que la enfermedad progrese en síndrome de insuficiencia respiratoria aguda60-62.
Asimismo, se ha demostrado que en ciertos casos de COVID-19, la respuesta inflamatoria inducida por la tormenta de citoquinas, es precedida por una reducción en la activación de varios genes del sistema inmune adquirido (CD4 y CD8). Esta evidencia soporta un posible mecanismo patológico que estaría operando en pacientes con COVID-19, en donde algunos elementos del sistema inmune adquirido previenen la amplificación de respuestas pro-inflamatorias mediadas por citoquinas24. Por ello, un mejor entendimiento de este mecanismo podría brindar nuevas herramientas para el diagnóstico, tratamiento y prevención de esta enfermedad.
3. Síndrome inflamatorio multi-sistémico en niños y adolescentes
Reportes clínicos recientes provenientes principalmente de Estados Unidos y Europa demuestran que COVID-19 puede desencadenar una enfermedad sistémica aguda, acompañada de una respuesta hiper-inflamatoria en niños y adolescentes63. Al comienzo, estos pacientes fueron diagnosticados con la enfermedad de Kawasaki, la cual es causada por una respuesta inmune frente a agentes infecciosos. Los niños que desarrollan esta condición autolimitante presentan signos y síntomas característicos que incluyen: fiebre por más de cinco días, eritema, inflamación y brotes cutáneos en manos y pies, vasculitis (inflamación de vasos sanguíneos) y mucositis. Sin embargo, tratamiento pronto y apropiado con inmunosupresores (inmunoglobulinas y esteroides) previene la aparición de patologías más severas64,65.
Aunque la mayoría de reportes de niños y adolescentes que se infectaron con SARS-CoV-2 exhibieron una sintomatología característica de Kawasaki, profesionales de salud observaron que estos pacientes también presentaban un cuadro clínico con síntomas inesperados y desconcertantes, tales como: trastornos gastrointestinales, daño cardiovascular y descompensación respiratoria que requieren tratamientos más agresivos en UCI66-69. Debido a la aparición cada vez más frecuente de casos de jóvenes y niños con COVID-19 que desarrollan este tipo excepcional de patología y sintomatología, la OMS oficialmente la empezó a catalogar como “Síndrome inflamatorio multi-sistémico en niños y adolescentes con COVID-19”63. Este nuevo trastorno puede provocar niveles críticamente bajos de presión arterial, desencadenando un fallo multi-orgánico complejo. Es por este motivo que dichos pacientes necesitan una pronta hospitalización en UCI63,66-69.
Aspectos epidemiológicos de COVID-19
1. Los murciélagos y el posible origen de COVID-19
La comunidad científica internacional ha aceptado que COVID-19 tiene un origen zoonótico3,27,30,35. En general, las zoonosis se caracterizan por tener un reservorio natural de tipo animal, en el cual los agentes infecciosos alcanzan la capacidad de ampliar su espectro de transmisión y replicar la enfermedad en nuevos huéspedes3. En su reservorio natural, generalmente animales silvestres, los virus no desencadenan patologías que pongan en riesgo la supervivencia del huésped. Sin embargo, el agente infeccioso puede generar signos, síntomas y condiciones clínicas muy severas en el nuevo organismo que lo hospeda, incluso causándole la muerte. Un ejemplo claro de este patrón viral de transmisión se empezó a registrar en diciembre de 2019 en Wuhan (China), cuando se piensa que SARS-CoV-2 adquirió las herramientas moleculares que le permitieron diversificar el espectro de organismos en los cuales pudo causar infección3,27,35.
Diferentes virus pertenecientes al género Betacoronavirus (Beta-CoVs) han desencadenado recientemente epidemias que causan síndromes respiratorios agudos en humanos y que han sido catalogados como zoonosis. Tanto el SARS (2002) como el MERS (2012) fueron enfermedades transmitidas a humanos desde un reservorio animal, y en ambos casos se ha demostrado que el huésped natural del agente etiológico es el murciélago. Los virus causantes de estas enfermedades, SARS-CoV y MERS-CoV, respectivamente, fueron transmitidos eventualmente al huésped final a través de animales intermediarios, tales como los civets (SARS) o camellos (MERS)8-10,27,28,30,34,35.
El hecho que SARS-CoV-2 sea un miembro de los Beta-CoVs y genere una enfermedad respiratoria similar en humanos, es una indicación que el virus causante del COVID-19 pudo haberse originado también en murciélagos y que esté siguiendo un patrón similar de transmisión. Estudios recientes demuestran que los gatos pueden contagiarse con SARS-C0V-02 sin desarrollar una enfermedad tan severa como el COVID-19 en humanos, lo que hace probable que estos animales estén cumpliendo el papel de huésped intermediario41,70.
Hallazgos recientes soportan la hipótesis que SARS-CoV-2 se originó en murciélagos. Estudios filogenéticos basados en análisis de genética evolutiva molecular revelan que SARS-CoV-2 es más cercano a Beta-CoVs de murciélagos (RaTG13) que al SARS-CoV de humanos. Es así, como el análisis de secuencias genómicas totales demuestra una similitud de nucleótidos cercana al 96% entre SARS-CoV-2 y RaTG133,34. De igual modo, estudios moleculares comparativos demuestran un 98% de identidad en la secuencia de aminoácidos de la proteína S de SARS-CoV-2 y RaTG1312,13. Finalmente, tanto SARS-CoV-2 como SARS-CoV y RaTG13 utilizan el mismo receptor celular (ACE2) para invadir las células epiteliales del aparato respiratorio41.
2. ¿Por qué SARS-CoV-2 es fácilmente transmisible?
La puerta de entrada que utiliza SARS-CoV-2 para ingresar al organismo humano y empezar el proceso infeccioso productivo son las membranas mucosas que recubren y protegen las vías respiratorias71. Debido a la cercanía física del sistema respiratorio superior con los ojos y la boca, estos dos órganos también representan rutas efectivas de contagio e infección. En consecuencia, la vía óptima de transmisión es el contacto directo o indirecto con secreciones respiratorias que emanan de un paciente con COVID-19 cuando esta persona respira, habla, grita, canta, estornuda o tose71-74.
Las secreciones respiratorias que contienen partículas virales infecciosas de SARS-CoV-2 incluyen “gotitas” de una amplia gama de tamaños, desde partículas visibles (> 1 mm) hasta aerosoles en el rango de 1-5 µm. Mientras los aerosoles pueden permanecer en el aire hasta por tres horas, las secreciones respiratorias de mayor tamaño pueden transportar más viriones que permanecen en estado viable e infeccioso hasta por tres días, si aterrizan sobre superficies de plástico y/o metal18,71-74. Estudios epidemiológicos de individuos pre-sintomáticos en Asia y Europa confirman que el máximo riesgo de contagio por secreciones respiratorias se presenta desde los tres días previos hasta la aparición de síntomas característicos de COVID-1975,76. Igualmente, estudios desarrollados en China indican que aerosoles (1-2.5 µm) pueden contener 10-40 partículas virales infecciosas de SARS-CoV-2 y permanecer en el aire de áreas críticas de hospitales (73) .
Aunque no existe un panorama claro para el contagio por SARS-CoV-2, los registros existentes sobre la velocidad de transmisión sugieren que no se requiere un número excesivamente alto de partículas virales infecciosas para desarrollar COVID-1950. Un simple examen visual de la cantidad de gotitas respiratorias producidas al toser o estornudar hace pensar que no se necesita un tiempo de exposición muy prolongado para estar expuesto a la dosis viral mínima requerida para desarrollar una infección productiva72-74. Estas variables, sumadas al tamaño de SARS-CoV-2 y a su alta afinidad por ACE2, dejan claro que en ausencia de tapabocas bastarían pocos minutos en contacto cercano con un paciente con COVID-19 para inhalar millones de viriones, que eventualmente llegarán a los alvéolos para empezar un proceso patogénico más severo.
En estos momentos es crítico analizar si SARS-CoV-2 puede transmitirse por otras rutas, lo que en parte ayudaría a explicar las elevadas tasas de transmisión observadas a nivel mundial. Hasta el día de hoy, hay evidencia experimental que demuestra la presencia del virus en secreciones corporales tales como semen y leche materna77,78. También, se ha identificado SARS-CoV-2 en células epiteliales del aparato digestivo79. Estos hallazgos sugieren una vía alterna de transmisión a las secreciones respiratorias.
Conclusiones y perspectivas
La aparición inesperada de casos de una neumonía viral causada por SARS-CoV-2 en la provincia de Wuhan (China) en diciembre del 2019 y su rápida diseminación en este país asiático deja muchos interrogantes relacionados con (i) la fecha exacta cuando empezó el brote; (ii) la existencia de otro reservorio natural aparte del murciélago; (iii) las mutaciones que le confirieron a SARS-CoV-2 la capacidad para cruzar la barrera de especies; y (iv) la transmisión a través de huéspedes intermediarios desconocidos. Encontrar las respuestas a estos interrogantes contribuirá a implementar mejores estrategias epidemiológicas para romper las cadenas de contagio y trasmisión no solamente de SARS-CoV-2, sino también de nuevos virus emergentes con potencial de causar enfermedades respiratorias similares a COVID-19.
SARS-CoV, SARS-CoV-2 y MERS-CoV empiezan su ciclo infectivo en células epiteliales del aparato respiratorio al adherirse a receptores superficiales característicos de estos tejidos, tales como ACE2 (SARS-CoV y SARS-CoV-2) y DPP4/CD26 (MERS). Esta adhesión específica es mediada por la proteína S de la envoltura viral que caracteriza a los coronavirus, proteína que además juega un papel esencial en las etapas subsiguientes de penetración y fusión de membranas. Por este motivo, la proteína S de SARS-CoV-2 se ha convertido en el candidato más apropiado para el desarrollo de estrategias terapéuticas para contener la pandemia generada por COVID-19.
Aunque ha habido avances significativos en las áreas descritas anteriormente, virólogos e inmunólogos deben colaborar para enfrentar retos futuros importantes, tales como: (i) caracterizar la estructura ACE2 y su función en la infección de órganos que son frecuentemente lesionados por SARS-CoV-2; (ii) identificar moléculas accesorias de origen celular y viral que contribuyen a la adhesión y penetración de SARS-CoV-2; (iii) descubrir posibles mutaciones del gen humano que codifica ACE2 que incrementan su susceptibilidad al reconocimiento por la proteína viral S; (iv) encontrar y/o diseñar un modelo animal apropiado donde estudiar la dinámica de la interacción entre la proteína S y ACE2; (v) culminar la fase III de pruebas clínicas en humanos donde se pueda cuantificar niveles de inmunogenicidad, de patogenicidad y la dosis efectiva de muchos vacunas que están siendo desarrolladas en la actualidad; y (vi) determinar la duración de la inmunidad inducida por una potencial vacuna.
Contribuciones a corto y largo plazo en las áreas descritas anteriormente acelerarán el desarrollo y uso de métodos preventivos y terapéuticos que protejan eficientemente no sólo a la comunidad en general, sino también al personal de salud, a quienes su trabajo en unidades de cuidado de pacientes con COVID-19 los expone frecuentemente a situaciones de riesgo de contagio.
Los ciclos acelerados de multiplicación y destrucción celular de MERS-CoV, SARS-CoV y SARS-CoV-2 provocan respuestas inflamatorias incontrolables inducidas por una intensa y auto-amplificada tormenta de citoquinas. Se ha demostrado que la insuficiencia respiratoria que desarrollan ciertos pacientes con MERS, SARS y COVID-19 se debe a los efectos adversos de citoquinas inflamatorias que circulan en el cuerpo de pacientes causándoles daño. Sin embargo, las manifestaciones clínicas de COVID-19 son bastante complejas y tienen un desenlace fatal más frecuente en pacientes de edad avanzada, con obesidad y/o con comorbilidades de tipo cardiovascular y respiratoria.
Estudios en el área de la inmunopatología deberán determinar el umbral de citoquinas a partir del cual la tormenta de citoquinas alcanza características perjudiciales de auto-perpetuación y auto-amplificación. De este modo, se haría más fácil diseñar tratamientos que bloqueen citoquinas inflamatorias específicas sin afectar otros componentes esenciales de las respuestas inmunes, mejorando así el pronóstico de COVID-19. También se debe construir un cuadro clínico individualizado de pacientes, de todas las edades, con y sin comorbilidades, el cual incluya marcadores moleculares y predictores clínicos con la capacidad de pronosticar si un paciente es propenso a desencadenar estas tormentas de citoquinas.
Estudios clínicos de pacientes con COVID-19 bajo soporte ventilatorio han demostrado que agentes inmunosupresores de tipo esteroide pueden atenuar los efectos adversos de citoquinas inflamatorias, mejorando de esta manera los índices de mortalidad de COVID-19. Una perspectiva interesante que se desprende de estas observaciones es investigar si pacientes con enfermedades genéticas autoinmunes de tipo inflamatorio y bajo tratamiento con este tipo de inmunosupresores son menos susceptibles a padecer complicaciones clínicas severas si contraen COVID-19. Por último, se debe descifrar si algunos individuos asintomáticos o con sintomatología leve desarrollaron inmunidad cruzada a partir de encuentros anteriores con otros coronavirus, tal y como lo sugieren algunos reportes.















