Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Investigaciones Andina
Print version ISSN 0124-8146
Investig. andina vol.14 no.25 Pereira Sept. 2012
DETECCIÓN DE GENES DE TOXINAS PIROGÉNICAS Y TOXINAS EXFOLIATIVAS EN AISLAMIENTOS CLÍNICOS DE STAPHYLOCOCCUS AUREUS EN COLOMBIA
DETECTION OF GENES OF PYROGENIC TOXINS AND EXFOLIATING TOXINS IN CLINICAL ISOLATION FOR STAPHYLOCOCCUS AUREAUS IN COLOMBIA
DETECÇÃO DE GENES DE TOXINAS PIRO GÊNICAS E TOXINAS ESFOLIATIVAS EM ISOLAMENTOS CLÍNICOS DE STAPHYLOCOCCUS AUREUS NA COLÔMBIA
Luisa F Corredor*, José I Moncayo*' Jorge J Santacruz*, Adalucy Álvarez*
* Centro de Biología Molecular y Biotecnología, Laboratorio de Microbiología y Parasitología. Universidad Tecnológica de Pereira. Pereira, Colombia.
Resumen
Introducción: staphylococcus aureus está asociado con graves enfermedades sistémicas causadas por superantígenos (toxinas pirogénicas y exfoliativas).
Métodos: 100 aislamientos clínicos de S. aureus se identificaron por método automatizado y PCR, la prevalencia de genes de superantígenos por PCR múltiple y las correlaciones mediante la prueba exacta de Fischer.
Resultados: en 38 aislamientos se observó que la prevalencia de los genes de enterotoxinas, toxina del síndrome de choque tóxico y toxinas exfoliativas fue 44%, 7% y 4%, respectivamente. La única correlación significativa (p = 0,045) fue entre la presencia de los genes de superantígenos y los aislamientos hospitalarios.
Conclusiones: existe una alta prevalencia de genes de enterotoxinas y una baja de genes de toxinas exfoliativas y del síndrome de choque tóxico en aislamientos de S. aureus en esta población. Esta es la primera investigación que presenta datos de prevalencia de superantígenos en Colombia, y proporciona nueva información para América Latina.
Palabras clave: Staphylococcus Aureus, Superantígenos, Aislamientos Clínicos, Toxinas Pirogénicas, Toxinas Exfoliativas, PCR Múltiple.
Abstract
Introduction: staphylococcus aureus is associated with serious systematic diseases caused by super antigens (pyogenic and exfoliating toxins).
Methods: 100 clinical isolations for S. Aureus were identified by automatic methods and PCR, the prevalence of super antigen genes by multiple PCR and the correlations thru the Fischer exact test.
Results: 38 isolation cases were observed and the prevalence of the enterotoxin genes, toxins of the syndrome of toxic shock and exfoliating toxins was 44%, 7% and 4% respectively. The only significant correlation found (p=0.045) was between the presence of the super antigen genes and the clinical isolations.
Conclussion: there is a high prevalence of enterotoxin genes and a low prevalence of exfoliating genes and the syndrome of toxic shock in isolation of S.Aureus in this population. This is the first investigation that yields data of prevalence of super antigens in Colombia and provides new information for Latin America.
Keywords: Staphylococcus Aureus, Super antigens, Clinical Isolation, Pyrogenic Toxins, Exfoliating Toxins, Multiple PCR
Resumo
Introdução: staphylococcus aureus está associado a graves enfermidades sistêmicas causadas por superantígenos (toxinas pirogênicas e esfoliativas).
Métodos: cem isolamentos clínicos de S. aureus foram identificados por método automatizado e PCR, a prevalência de penas de superantígenos por PCR múltiplos e correlações mediante a prova exata de Fischer.
Resultados: em 38 isolamentos observou-se que a prevalência dos genes de en-terotoxinas, toxina da síndrome de choque tóxico e toxinas esfoliativas foi 44%, 7% e 4%, respectivamente. A única correlação significativa (p = 0,045) foi entre a presença dos genes de superantígenos e os isolamentos hospitalares.
Conclusões: existe uma alta prevalência de genes de enterotoxinas e uma baixa de genes de toxinas esfoliativas e da síndrome do choque tóxico em isolamento de S. aureus nessa população. Esta é a primeira pesquisa que apresenta dados de prevalência de superantígenos na Colômbia e propicia nova informação para América Latina.
Palavras chave: Staphylococcus aureus, superantígenos, isolamentos clínicos, toxinas pirogénicas, toxinas esfoliativas, PCR múltiplo.
Fecha de recibo: Octubre/2011
Fecha aprobación: Diciembre/2011
Introducción
Staphylococcus aureus es una bacteria Gram positiva ampliamente reconocida, debido a su importancia clínica y la expresión de factores de virulencia, como superantígenos (SAg) (1).
Los SAg, junto con otros factores de virulencia, son los responsables de la patogenicidad de este microorganismo. Los SAg de S. aureus comprenden las toxinas exfoliativas (ET), las toxinas pirogénicas (PTSAgs), enterotoxinas (SE) y la toxina del síndrome de choque tóxico 1 (TSST-1) (2, 3,4). Los SAgs del estafilococo causan el síndrome de la piel escaldada (SSSS), el envenenamiento alimentario por estafilococo (SFP) y el síndrome de choque tóxico estafilocócico (SAT) (5,6). Las cepas que sintetizan estos productos son potencialmente más virulentas y peligrosas (7), ya que representan un mayor riesgo para el paciente, porque activan un gran número de linfocitos que funcionan a muy bajas concentraciones y no requieren la especificidad de los antígenos convencionales. Además, evitan la generación de una respuesta inmune efectiva (8, 9). En Colombia, S. aureus es el principal microorganismo aislado en las infecciones nosocomiales (10) y es uno de los más frecuentes en las salas de los hospitales (11).
El objetivo de este estudio fue determinar la prevalencia de los genes que codifican los superantígenos en los aislamientos clínicos de S. aureus y comparar su prevalencia con aislamientos hospitalarios o adquiridos en la comunidad, con la resistencia a los antibióticos y con el tipo de muestra biológica de donde fueron obtenidos los aislamientos.
Materiales y métodos
Las cepas bacterianas. Se estudiaron 100 aislamientos de S. aureus obtenidos de muestras clínicas que fueron enviadas al Laboratorio Clínico del Hospital Universitario San Jorge de Pereira (HUSJ), mediante selección en agar sangre. La identificación por parámetros bioquímicos y el perfil de susceptibilidad a los antibióticos se determinó con el sistema automatizado WalkAway / MicroScan® (Dade Behring). Luego fueron enviadas al Laboratorio de Microbiología y Parasitología de la Universidad Tecnológica de Pereira, para el análisis genotípico. Los aislamientos de S. aureus fueron clasificados de acuerdo con el tipo de muestra biológica en tres categorías: sangre, secreciones (orejas, ojos, músculos, quemaduras, heridas y articulaciones) y otros (catéteres y ventiladores, abscesos, líquido pleural, peritoneal y cefalorraquídeo). Los aislamientos también fueron clasificados como adquiridos en la comunidad (n = 49) o en el hospital (n = 49) en función del origen de la infección. Solo para este último análisis dos cepas fueron descartadas porque la clasificación del origen de la infección no estaba disponible en la historia clínica.
Identificación genotípica de los aislamientos de S. aureus. Se realizaron subcultivos en agar nutritivo de los aislamientos de S. aureus almacenados a -80 °C para la extracción de ADN. Los cultivos fueron sometidos a crecimiento durante 16 horas, a 37°C. El ADN genómico se aisló de células centrifugadas y resuspendidas en 567 μl tampón fosfato salino 1X (8g de NaCl, 0.2 g KCl, 1.44g de Na2HPO4, 0.24g por litro K2PO4, [pH 7.4]) con 10 μl de lisostafina (100 ug/ml) (AMBI BL) y se incubaron a 37°C hasta presentar un estado viscoso (12). Se adicionaron 80 μl de CTAB 1% /NaCl 0,7 M, el ADN fue purificado con fenol-cloroformo y precipitado con etanol (13).
El ADN se resuspendió en 200 ul TE 1X y se cuantificó de acuerdo con los valores A260.
La identificación genotípica de los aislamientos se realizó con una pareja de iniciadores que amplificaron un producto de 108 pb correspondiente al gen Sa442 específico para S. aureus (función no definida) (14). La cepa S. aureus ATCC fue utilizada como control positivo. Los controles negativos fueron aislamientos clínicos de S. epidermidis donado por el HUSJ, Streptococcus pyogenes ATCC 19615 y Pseudomonas aeruginosa ATCC 27853. Finalmente, el agua destilada estéril se utilizó como control negativo de la reacción de amplificación.
PCR Múltiple para los genes de SAg. Se trabajó con dos series de PCR múltiple, la serie A para los genes de enterotoxinas clásicas (sea a see) y la serie B, para los genes de las toxinas exfoliativas (eta y etb) y el gen de la toxina de choque tóxico (tsst-1). Se llevaron a cabo con el método y los iniciadores descritos por otros autores (15). A ambos conjuntos de PCR se les optimizaron condiciones como la disminución del volumen de reacción a 20 μl y de la Taq polimerasa a 1U y con el aumento de la concentración de MgCl2 a 3 mM. Para 35 ciclos de amplificación, el perfil de los ciclos térmicos fue igualmente modificado para la desnaturalización a 94°C durante 1 minuto, hibridación a 61°C durante 30 segundos, y la extensión para la serie A a 74°C durante 1 minuto y para la serie B a 72°C durante 1 minuto.
El gen femA (cuyo producto es parte del peptidoglicano de S. aureus y un factor asociado con la resistencia a la meticilina) se utilizó como control interno en las dos series de PCR, y generó un fragmento de 132 pb (15). Los controles positivos fueron la cepa ATCC 13566, para los genes de enterotoxinas A y B (sea y seb) y la cepa ATCC 700699, para los genes de la enterotoxina C y la toxina de choque tóxico (sec y tsst-1).
Para los genes de las enterotoxinas D y E (sed y see) y para los genes de las toxinas exfoliativas (eta y etb), no estaban disponibles los controles positivos ATCC. Se reemplazaron por una mezcla de ADN que consta de diferentes aislamientos toxigénicos de S. aureus de la colección del proyecto de investigación. Como control negativo se utilizó agua destilada estéril. Los productos de PCR fueron detectados en agarosa al 2,5% (Promega, Madison, WI), teñido con de bromuro de etidio (0,01 mg) (Sigma), en presencia de un marcador de peso molecular de ADN de 100 pb (Invitrogen).
Análisis estadístico. Un análisis de concordancia se llevó a cabo entre los dos métodos de identificación, el automatizado Walkaway/Microscan® y la PCR, implementando la prueba de Kappa. Para establecer diferentes correlaciones clínicas, la prueba exacta de Fisher se utilizó con un valor de probabilidad (p) < 0,05 con un intervalo de confianza del 95%. Las comparaciones estadísticas fueron realizadas entre los aislamientos positivos para SAg, frente a los aislamientos hospitalarios o adquiridos en la comunidad, frente a los aislados con resistencia a antibióticos y en comparación con el tipo de muestra biológica (secreciones, sangre y otros).
Resultados
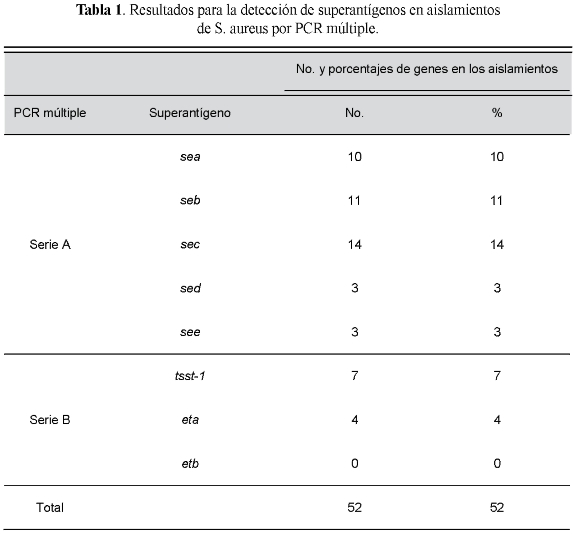
PCR múltiple para genes de superantígenos. Los 100 aislamientos contenían el gen femA y un total de 38% mostraron la presencia de uno o más genes de superantígenos por aislamiento. 26 aislamientos tenían un solo gen, 8 tenían dos genes, 3 tenían tres genes y 1 aislamiento tenía cuatro genes. La prevalencia global de los genes de enterotoxinas en la colección de aislamientos fue de 44% (Tabla 1). Los genes más frecuentes encontrados fueron sec 17% (17/100), seb 11% (11/100) y sea 10% (10/100). Adicionalmente,
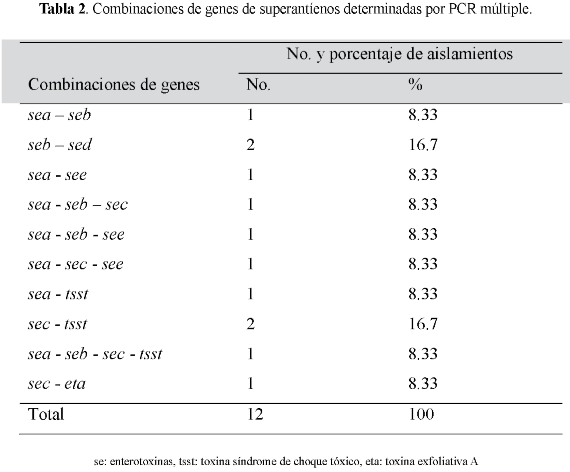
amplificaron tsst-1 7% (7/ 100), eta el 4% (4/100), y sed y see con el 3% (3 / 100), respectivamente. Ningún aislamiento fue positivo para el gen etb y no se observaron amplificaciones en los controles negativos. Ciertas combinaciones de genes se observaron en 12 aislamientos diferentes (Tabla 2).
En 33,3% de los 12 aislamientos, por lo menos uno de los genes de enterotoxinas se encontraban con el gen tsst-1 y un solo aislamiento (8,3%) mostró un gen de enterotoxina (sec) con una toxina exfoliativa (eta). Por último, no se encontraron combinaciones entre los genes de las toxinas exfoliativas.
Identificación de S. aureus por PCR. Todos los aislamientos de S. aureus (100%) amplificaron el gen Sa442, lo que confirma la identificación fenotípica por el sistema automatizado Walkaway/ Microscan®, con el método molecular por PCR.
Análisis estadístico. El análisis de concordancia entre la identificación automatizada Walkaway/Microscan® y la PCR, arrojó un excelente valor Kappa de 1. Con la prueba exacta de Fischer, se observó una correlación significativa (p = 0,045) entre la presencia de los genes de SAg y los aislamientos intrahospitalarios. De los 49 aislamientos intrahospitalarios, 22 tuvieron genes de superantígenos, m ientras que los 49 aislamientos comunitarios, solo amplificaron 13 genes de SAg. No se pudieron establecer correlaciones estadísticas significativas entre la presencia de los genes de SAg y los aislamientos clasificados por tipo de muestra biológica y aquellos que presentaban resistencia a antibióticos (datos no mostrados).
Discusión
De acuerdo con otros investigadores, cuyos resultados mostraron que el 38,3% de los aislamientos tenían genes de enterotoxinas clásicas y de tsst-1 (16) o aproximadamente el 40% (17), este estudio encontró que el 38% de las cepas poseen genes superantígenos de S. aureus. También se ha reportado que el 50% o más de los aislamientos estudiados fueron positivos para los genes de superantígenos (5, 18). Sin embargo, es importante señalar que estas tasas pueden verse afectadas, por una sobrerepresentación de los nuevos genes de enterotoxinas (seg a ser y seu), que al parecer son más frecuentes (19, 20). Dentro del 38% de los aislamientos positivos, este estudio encontró del 44% de los genes de enterotoxinas clásicas, similar a los reportes de 46% (21) y de 42,9% (18). Otros estudios han encontrado tasas de prevalencia menores al 25% (22). También se demostró que la prevalencia del gen de tsst-1 fue del 7%. A diferencia de otros autores que informaron de un 4% (23) y 20% para este gen (21).
Para las toxinas exfoliativas, hay una amplia divergencia de datos. Los estudios que analizaron aislamientos provenientes de infecciones menores, de enfermedades supurativas y poblaciones sanas, mostraron tasas muy bajas (3%) o a veces inexistentes para los genes de eta y etb (21, 24). Sin embargo, la tasa observada es alta (5). Otros estudios han encontrado tasas del 70% para ambas toxinas (25) y de manera individual del 40% para eta y del 25% para etb (26) en la presentación de la enfermedad sistémica causada por toxinas exfoliativas como síndrome de piel escaldada y el impétigo. En esta investigación se utilizaron aislamientos de infecciones menores, infecciones asociadas a los dispositivos, a la sangre o los fluidos, y se encontró un 4% de prevalencia del gen eta y no se observaron resultados positivos para el gen etb.
Los genes con mayor prevalencia detectados en este estudio fueron: sec (17%), seb (11%) y sea (10%). Un reporte anterior mostró que see (15,9%) y sec (11,2%) fueron los más frecuentes (18). Otros investigadores encontraron el gen tsst-1 (24,3%) y sea (19,6%), como los genes más prevalentes (27). De los 38 aislamientos que dieron resultados positivos en esta investigación, 12 amplificaron diferentes combinaciones de genes, de manera similar a lo reportado por otros autores (12, 28). Otros estudios han reportado combinaciones como sec y sed, sea y sed, eta y etb (26, 29), a diferencia de las combinaciones observadas aquí (seb y sed; sea, seb y sec; sea y see; sea, sec y see). Layer et al. 2006, reportaron otras combinaciones diferentes. Además, diferentes estudios mostraron nuevas combinaciones de genes (seb y tsst-1), que normalmente se encuentran localizadas en dos islas patogenicidad diferentes y relacionadas en S. aureus (20). La presencia de algunos genes de enterotoxinas clásicas junto con el gen de la toxina del síndrome del choque tóxico, se estableció aquí y en otros trabajos de investigación (17). Este estudio también demostró que algunos aislamientos contenían un gen de enterotoxinas clásicas en combinación con la toxina exfoliativa A (eta), en contraste con los hallazgos de otras investigaciones que mostraron solo la eta con etb (26,15). Ninguno de los aislamientos mostraron la asociación entre los genes sea y eta, probablemente debido a que ambos genes se encuentran en bacteriófagos atenuados y hasta ahora no se ha reportado ningún aislamiento que tenga más de un fago de cada tipo de familia (28,30).
Al evaluar tres tipos de correlaciones clínicas, solo se observó una correlación significativa entre la presencia de los genes de SAg y los aislamientos intrahospitalarios. Sin embargo, al igual que otros estudios tampoco se ha podido dilucidar claramente estas y otras correlaciones (31,32). En muestras de alimentos, se han establecido perfiles de SAgs en aislamientos resistentes y sensibles a la meticilina (MRSA y MSSA) (3).
En conclusión, la PCR múltiple detectó la presencia de un solo gen o varias combinaciones de genes en los aislamientos clínicos de S. aureus. Se estableció que la prevalencia de los genes de enterotoxinas clásicas en la población estudiada es alta, de acuerdo con las tasas de prevalencia reportada en otros países. En cuanto a los genes de las toxinas exfoliativas y de la tsst-1, la prevalencia es baja, como se reporta en otras investigaciones. Por otra parte, como se estableció una correlación significativa entre la presencia de los genes de superantígenos y los aislamientos adquiridos en el hospital, se requiere profundizar en el análisis de los aislamientos a través de marcadores moleculares, para identificar la clonalidad de los mismos.
Es importante señalar que este estudio es el primer trabajo de investigación que presenta datos de prevalencia de superantígenos en Colombia, y proporciona nueva información para América Latina. Esta técnica solo identifica los genes de toxinas en los aislamientos y no mide la expresión y secreción de las mismas. Esta investigación junto con otros estudios que puedan establecer el comportamiento molecular del organismo (la expresión de genes), ayudarían a diseñar estrategias para el control eficiente de los brotes, además del manejo y tratamiento de las infecciones por estafilococos. Por lo tanto, con base en los ensayos de PCR múltiple, como herramienta para el estudio de los genotipos de los aislamientos de estafilococos, se puede acelerar el proceso de diagnóstico en laboratorios clínicos y de investigación.
Agradecimientos
Este trabajo fue financiado por la Vicerrectoría de Investigaciones, Innovación y Extensión, el Laboratorio de Microbiología y Parasitología y el Centro de Biología Molecular y Biotecnología, de la Universidad Tecnológica de Pereira, Colombia. Agradecemos especialmente a la Directora del Laboratorio Clínico del Hospital Universitario San Jorge de Pereira, Carmen Elisa Llanos Uribe, por la donación de los aislamientos de S. aureus, y la colaboración de las bacteriólogas a su cargo, en la identificación automatizada de las mismas.
REFERENCIAS
1 Götz F. 2002. Staphylococcus and biofilms. Mol Microbiol 43: 1367-1378. [ Links ]
2 Monday ST and Bohach GA. 1999. Use of multiplex PCR to detect classical and newly described pyrogenic toxin genes in staphylococcal isolates. J Clin Microbiol 37: 3411-3414. [ Links ]
3 Ortega E, Abriouel H, Lucas R y Gálvez A. 2010. Multiple Roles of Staphylococcus aureus Enterotoxins: Pathogenicity, Superantigenic Activity, and Correlation to Antibiotic Resistance. Toxins. 2: 117-2131. [ Links ]
4 Macias ES, Pereira FA, Rietkerk W, Safai B. 2011. Superantigens in dermatology. J Am Acad Dermatol. 64: 455-472. [ Links ]
5 Argudín MA, Mendoza MC, Méndez FJ, Martín MC, Guerra B, Rodicio MR. 2009. Clonal Complexes and Diversity of Exotoxin Gene Profiles in Methicillin-Resistant and Methicillin-Susceptible Staphylococcus aureus Isolates from Patients in a Spanish Hospital. J Clin Microbiol 47:2097-2105. [ Links ]
6 Raulin O, Durand G, Gillet Y, Bes M, Lina G, Vandenesch F, et al. 2010. Toxin Profiling of Staphylococcus aureus Strains Involved in Varicella Superinfection. J Clin Microbiol 48:1696-1700. [ Links ]
7 Archer GL. 1998. Staphylococcus aureus: A well-armed pathogen. Clin Infect Dis 26:1179-81. [ Links ]
8 Waclavicek M, Stich N, Rappan I, Bergmeister H, Eibl MM. 2009 Analysis of the early response to TSST-1 reveals VP-unrestricted extravasation, compartmentalization of the response, and unresponsiveness but not anergy to TSST-1. J Leukocyte Biology 85:44-54. [ Links ]
9 Brosnahan AJ and Schlievert PM. 2011. Gram-positive bacterial superantigen outside-in signaling causes toxic shock syndrome. FEBS Journal. 278: 46494667. [ Links ]
10 Tibavizco D, Rodríguez JY, Silva E, Cuervo SI, Cortés JA. 2007. Enfoque terapéutico de la bacteremia por Staphylococcus aureus. Biomédica 27: 294-307. [ Links ]
11 Briceño DF, Correa A, Valencia C, Torres JA, Pacheco R, Montealegre M C, et al. 2010. Actualización de la resistencia a antimicrobianos de bacilos Gram negativos aislados en hospitales de nivel III de Colombia: años 2006, 2007 y 2008. Biomédica. 30:371-81. [ Links ]
12 Johnson WM, Tyler SD, Ewan EP, Ashton FE, Pollard DR, Rozee KR. 1991. Detection of genes for enterotoxins, exfoliative toxins, and toxic shock syndrome toxin 1 in Staphylococcus aureus by the polymerase chain reaction. J Clin Microbiol 29: 426-430. [ Links ]
13 Ausubel FM, Brent R, Kingston RE, Moore DD, Seidman JG, Smith JA, et al. 2003 Preparation of Genomic DNA from Bacteria. Curr Protocols Mol Biol. Vol. 1. John Wiley & Sons, Inc, pp 2.4.1-2.4.5. [ Links ]
14 Martineau F, Picard JR, Roy PR, Ouellette M, Bergeron MG. 1998. Species-specific and ubiquitous-DNA-based assays for rapid identification of Staphylococcus aureus. J Clin Microbiol 36: 618-623. [ Links ]
15 Mehrotra M, Wang G and Johnson WM. 2000. Multiplex PCR for detection of genes for Staphylococcus aureus enterotoxins, exfoliative toxins, toxic shock syndrome toxin 1, and methicillin resistance. J Clin Microbiol 38: 1032-1035. [ Links ]
16 Mempel M, Lina G, Hojka M, Schnopp C, Seidl HP, Schafer T, et al. 2003. High Prevalence of superantigens associated with the egc locus in Saphylococcus aureus isolates from patients with atopic eczema. Eur J Clin Microbiol Infect Dis 22: 306-309. [ Links ]
17 Layer F, Ghebremedhin B, Kõnig W, Kõnig B. Heterogeneity of Methicillin-Susceptible Staphylococcus aureus Strains at a German University Hospital Implicates the Circulating-Strain Pool as a Potential Source of Emerging Methicillin-Resistant S. aureus Clones. J Clin Microbiol 44: 2179-2185. [ Links ]
18 Becker K, Friedrich AW, Lubritz G, Weilert M, Peters G, Von Eiff C. 2003. Prevalence of genes encoding pyrogenic toxin superantigens and exfoliative toxins among strains of Staphylococcus aureus isolated from blood and nasal specimens. J Clin Microbiol 41: 1434-1439. [ Links ]
19 Fueyo JM, Mendoza CM, Alvarez M, Martín C. 2005. Relationships between toxin gene content and genetic background in nasal carried isolates of Staphylococcus aureus from Asturias, Spain. FEMS Microbiol Lett 243: 447-454. [ Links ]
20 Holtfreter S, Grumann D, Schmudde M, Nguyen HT, Eichler P, Strommenger B, et al. 2007. Clonal Distribution of Superantigen Genes in Clinical Staphylococcus aureus Isolates. J Clin Microbiol 45: 2669-2680. [ Links ]
21 Jarraud S, Peyrat MA, Lim A, Tristan A, Bes M, Mougel C, et al. 2001. egc, A highly prevalent operon of enterotoxin gene, forms a putative nursery of superantigens in Staphylococcus aureus. J Immunol 166: 669-677. [ Links ]
22 Banks M, Kamel, NS, Zabriskie JB, Larone DH, Ursea D, Posnett DN. 2003. Staphylococcus aureus Express Unique Superantigens Depending on the Tissue Source. J Infect Dis 187: 77-86. [ Links ]
23 Gubbay JB, Gosbell IB, Barbagiannakos T, Vickery AM, Mercer JL, Watson M. 2008. Clinical features, epidemiology, antimicrobial resistance, and exotoxin genes (including that of Panton-Valentine leukocidin) of gentamicin-susceptible methicillin-resistant Staphylococcus aureus (GS-MRSA) isolated at a paediatric teaching hospital in New South Wales, Australia. Pathology 40: 64 - 71. [ Links ]
24 Ladhani S, Joannou CL, Lochrie DP, Evans RW, Poston SM. 1999. Clinical, microbial, and biochemical aspects of the exfoliative toxins causing staphylococcal scalded-skin syndrome. Clin Microbiol Rev 12: 224-42. [ Links ]
25 Kanzaki H, Ueda M, Morishita Y, Akiyama H, Arata J, Kansaki S. 1997. Producibility of exfoliative toxin and staphylococcal coagulase types of Staphylococcus aureus strains isolated from skin infections and atopic dermatitis. Dermatology 195: 6-9. [ Links ]
26 Koning S, van Belkum A, Snijders S, van Leeuwen W, Verbrugh H, Nouwen J, et al. 2003. Severity of nonbullous Staphylococcus aureus impetigo in children is associated with strains harboring genetic markers for exfoliative toxin B, Panton-Valentine leukocidin, and multidrug resistance plasmid. J Clin Microbiol 41: 3017-3021. [ Links ]
27 Becker K, Roth R and Peters G. 1998. Rapid and specific detection of toxigenic Staphylococcus aureus: use of two multiplex PCR enzyme inmunoassays for amplification and hybridization of staphylococcal enterotoxin genes, exfoliative toxin genes, and toxic shock syndrome toxin 1 gene. J Clin Microbiol 36: 25482553. [ Links ]
28 Kuroda M, Ohta T, Uchiyama I, Baba T, Yuzawa H, Kobayashi I, et al. 2001. Whole genome sequencing of methicillin-resistant Staphylococcus aureus. Lancet 357: 1225-1239. [ Links ]
29 Sharma NK, Rees CED and Dodd ERC. 2000. Development of a Single-Reaction Multiplex PCR Toxin Typin Assay for Staphylococcus aureus Strains. Appl Environ Microbiol 66: 1347-1353. [ Links ]
30 Lindsay JA and Holden MT. 2004. Staphylococcus aureus: superbug, super genome?. Trends Microbiol 12: 379-385. [ Links ]
31 Ferry T, Thomas D, Perpoint T, Lina G, Monneret G, Mohammedi I, et al. 2008. Analysis of superantigenic toxin VP T-cell signatures produced during cases of staphylococcal toxic shock syndrome and septic shock. Clin Microbiol Infect. 14: 546-554. [ Links ]
32 Varshney AK, Mediavilla JR, Robiou N, Guh A, Wang X, Gialanella P, et al. 2009. Diverse Enterotoxin Gene Profiles among Clonal Complexes of Staphylococcus aureus Isolates from the Bronx, New York. Appl Environ Microbiol. 75:6839-6849. [ Links ]